miR-142和miR-144靶向FoxO1基因调节绵羊前体脂肪细胞分化的研究
2018-05-07赵艳艳乔利英景炅婕潘洋洋任端阳王书芳吕宣增刘文忠
赵艳艳,乔利英,景炅婕,潘洋洋,任端阳,王书芳,吕宣增,刘文忠
(山西农业大学动物科技学院,太谷 030801)
动物脂肪组织在体内发挥着重要功能,不仅通过提供能量、维持体温、保护内脏等维持内环境稳态[1],其含量的多少还影响家畜的生产性能,特别是肉品质。脂肪组织的发育和沉积取决于前体脂肪细胞的分化与生长。哺乳动物叉头状蛋白O(Forkhead box O, FoxO)是一个转录因子家族,广泛参与细胞进程,如DNA修复、细胞周期调控、抗逆性、凋亡及代谢等[2]。其中,FoxO1由FoxO1基因编码,其活性受磷酸化和去磷酸化过程的调节[3],P13K/Akt和MAPK/ERK通路使FoxO1磷酸化[4],从而导致FoxO1蛋白的转录活性受到抑制[5]。
miRNA对机体的正常运转发挥极为重要的调节作用,不但参与细胞分化、发育、增殖、凋亡等过程,还对器官的正常发育和肿瘤的形成起着重要作用[6]。miRNA主要是与靶基因mRNA结合,在转录后水平调节基因的表达。研究显示,脂肪细胞分化与miRNA有重要的关系[7]。例如,miR-130可下调PPARγ的表达,从而抑制脂肪形成[8]。
miRNA可直接调节靶基因的表达,也可通过调节转录因子的活性间接影响基因表达。关于miRNA靶向调节FoxO1的机制在人类疾病研究方面报道较多。miR-96通过作用于FoxO1促进人卵巢黄素化颗粒细胞的存活和增加孕激素分泌量[9]。在人工培养的一种非小细胞肝癌(Non-small cell lung cancer)细胞系A549中发现,miR-183通过抑制FoxO1基因表达,促进A549细胞生长[10]。miR-153可下调FoxO1和PTEN的表达促进正常前列腺细胞增殖,进而抑制癌细胞增殖[11]。miR-374可下调FoxO1的表达促进人骨肉瘤细胞的增殖[12]。这些例子说明FoxO1可以通过调节不同的靶基因发挥不同的生物学功能。FoxO1的一个重要靶基因是PPARγ(Peroxisome proliferator-activated receptor γ)。二者结合可抑制脂肪细胞的分化,而PPARγ本身则是促进脂肪细胞分化的,说明FoxO1下调PPARγ的表达。FoxO1在脂肪细胞分化晚期呈高表达趋势[13],应该是FoxO1下调PPARγ的表达而导致的。磷酸化的FoxO1在细胞质中,当其去磷酸化后进入细胞核中靶向作用于PPARγ,使其活性降低[14]。然而,脂肪细胞分化中,FoxO1的miRNA调节机制尚不清楚。
本研究拟首先利用生物信息学方法,预测与FoxO1基因具有靶标关系的miRNAs。然后在体外培养的绵羊前体脂肪细胞中验证FoxO1基因与相关miRNA的靶标关系。由此,揭示FoxO1基因在脂肪细胞分化中的miRNA调节作用,为阐明脂肪细胞分化的网络调控机制提供科学依据。
1 材料与方法
1.1 材料
1.1.1 试验材料 本研究所用试验动物为1只出生1周内的小尾寒羊羔羊。将其屠宰后,迅速采集其皮下脂肪组织。一部分用酒精消毒后放入无菌PBS中带回实验室,用于细胞培养;另一部分装入无RNase的离心管中,置-80 ℃冰箱保存,用于总RNA的提取。
1.1.2 主要试剂 限制性内切酶XhoⅠ和SalⅠ、DNA胶纯化试剂盒、T4连接酶、RNAiso Plus、SYBRPremix Ex TaqTM Ⅱ试剂盒和PrimeScript®RT reagent kit反转录试剂盒均购自TaKaRa公司;氯仿和异丙醇购自天津市风船化学试剂科技有限公司;75%的酒精购自新泰市润生医疗卫生用品科技有限公司;磷酸缓冲盐溶液(PBS)粉末、Tween-20和青链霉素购自索莱宝生物科技有限公司;DMEM/F-12 (HAM)、DMEM/lowGlucose和DMEM/HighGlucose 3种培养基以及胎牛血清购自Biological Industries;Trans5α感受态细胞购自北京全式金生物技术有限公司;Dual-Luciferase Reporter Assay System试剂盒购自Promega公司;发光液购自北京康为世纪生物科技有限公司;Lipofectamine 2000购自Invitrogen公司;II 型胶原酶和胰蛋白酶购自Sigma公司;总蛋白提取试剂盒购自江苏凯基生物公司;FoxO1A和羊抗兔IgG-HRP购自华安生物技术有限公司;Rabbit anti-GAPDH (AB-P-R 001) 购自杭州贤至生物科技有限公司。
1.2 方法
1.2.1 与FoxO1具有靶标关系的miRNA生物信息学预测 利用TargetScan[15]、PITA[16]、StarBase[17]、miRanda[18]4个在线软件,预测与FoxO1基因具有靶标关系的miRNAs。用Venny2.1[19]在线软件对4个靶基因预测软件得出的结果做韦恩图取交集,以聚焦调节FoxO1的miRNAs。各个软件的功能及其网址见表1。
1.2.2 引物设计与合成 根据NCBI核酸数据库(https://www.ncbi.nlm.nih.gov/genbank/)中公布的绵羊FoxO1基因的预测mRNA序列(登 录号:XM_012106873),利用NCBI中Primer BLAST设计绵羊FoxO1-CDS荧光定量PCR引物和FoxO1-3′UTR PCR引物。用18S rRNA基因和β-actin基因为内参基因。miRNA采用茎环法设计引物,以U6为内参基因。引物由北京六合华大基因科技股份有限公司和赛默飞世尔科技(中国)有限公司合成。引物序列见表2。
表1生物信息学在线软件
Table1Onlinebioinformaticssoftwaresusedinthisstudy

软件Software功能Function网址WebsiteTargetScan预测miRNA靶基因http://www.targetscan.org/vert_71/PITA预测miRNA靶基因https://genie.weizmann.ac.il/pubs/mir07/mir07_prediction.htmlStarBase预测miRNA靶基因http://starbase.sysu.edu.cn/miRanda预测miRNA靶基因http://www.microrna.org/microrna/getMrna.do?gene=56458&utr=20147&organism=10090#hdVenny2.1制作韦恩图http://bioinfogp.cnb.csic.es/tools/venny/index.html
表2实时荧光定量PCR和载体构建所用引物序列及内参引物序列
Table2PrimersequencesforqPCR,plasmidconstructionandreferencegenes

引物名称Primername引物序列(5'-3')PrimersequenceFoxO1-3'UTRF:CCGCTCGAGCGGCTTGGCAGCGAGGACACTTTR:ACGCGTCGACGTCGTGAATTATGGGGTGCCGTGAFoxO1-CDSF:GTGACTTGGACGGCATGTTTR:GTCTCCATCCATGAGGTCGTTβ-actinF:CAGCCTTCCTTCTTGGGTATR:TGGCATAGAGGTCTTTACGG18SrRNAF:CAGACAAATCACTCCACCAAR:GAAGGGCACCACCAGGAGTmiR-142F:ACACTCCAGCTGGGTGTAGTGTTTCCTACTTR:CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGTCCATAAAmiR-144F:ACACTCCAGCTGGGTACAGTATAGATGATGR:CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGCTAGTACAU6F:CTCGCTTCGGCAGCACAR:AACGCTTCACGAATTTGCGT统一引物UnifiedreverseprimerTGGTGTCGTGGAGTCG
F. 上游引物;R. 下游引物。标下划线的序列为XhoⅠ、SalⅠ酶切位点,加粗的序列为保护碱基
F. Forward primer; R. Reverse primer. The underlined sequences areXhoⅠ andSalⅠ digestion sites, bold sequences are protection bases
1.2.3 总RNA的提取与反转录 按照RNAiso Plus说明书的步骤,提取绵羊脂肪组织的总RNA,测定总RNA的量和浓度,用2%琼脂糖凝胶电泳检测RNA的完整性。按照PrimeScript®RT reagent kit 反转录试剂盒步骤进行反转录。反转录产物(cDNA)-20 ℃下保存备用。
1.2.4FoxO1-3′UTR 载体构建 根据预测的靶向FoxO1基因的miRNAs,设计包含miR-142、miR-144和FoxO1结合区域的FoxO1 mRNA 3′UTR 序列扩增引物FoxO1-3′UTR。在引物中加入XhoI和SalI两种限制性内切酶的酶切位点及各自的保护碱基(表2中分别标下划线和加粗的序列)。以cDNA为模板,对FoxO1-3′UTR进行扩增。将得到的产物利用琼脂糖凝胶电泳检测分析,将目的条带进行胶回收和纯化。利用XhoI、SalI内切酶对FoxO1-3′UTR的PCR产物和pmir-GLO载体分别进行双酶切。胶回收FoxO1-3′UTR和pmir-GLO载体酶切产物,然后进行连接、转化、菌落的培养和菌液的测序,测序鉴定成功的重组质粒命名为pmir-FoxO1。
1.2.5 前体脂肪细胞培养 将采集的绵羊皮下脂肪组织用PBS反复洗3遍,之后用剪刀剪碎,加入Ⅱ型胶原酶,在37 ℃摇床中200 r·min-1摇晃消化30 min左右。将脂肪组织消化成黏稠状后加入等体积的完全培养基终止消化,700×g 离心10 min。弃上清液,将底部的沉淀用完全培养基重悬,再次离心以洗净组织。分别用200和400目网筛过滤,留下滤液。加入适量的完全培养基,在37 ℃,5% CO2的培养箱中培养。完全培养基为普通培养基DMEM/F-12(HAM)中加入1%双抗和10%胎牛血清(FBS)。
1.2.6 细胞单转染与共转染 单转染中,将绵羊前体脂肪细胞培养于6孔板中,待细胞密度达到80%左右时,按照Lipofectamine 2000说明书对细胞进行转染。试验分为3组,转染miR-142 mimic(或miR-144 mimic)为过表达组,转染miR-142 NC(或miR-144 NC)为阴性对照组,不做任何转染为空白对照组(Blank)。每组设置3个重复。转染后24 h收集细胞,提取总RNA。利用qPCR检测过表达后miR-142、miR-144和FoxO1基因mRNA的表达量。利用Western blotting检测过表达后FoxO1蛋白的表达量。
共转染中,将HEK293T细胞培养于24孔板中,待细胞密度达到80%左右时,按照Lipofectamine 2000说明书对细胞进行转染。试验分为2组,共转染pmir-FoxO1与miR-142 mimic(或miR-144 mimic)为过表达组,共转染miR-142 NC(或miR-144 NC)和pmir-FoxO1为阴性对照组。转染48 h后,利用Dual-Luciferase Reporter Assay System试剂盒进行双荧光素酶检测。
1.2.7 FoxO1 RNA和蛋白表达量检测 按照RNAiso Plus说明书的步骤,从培养的绵羊前体脂肪细胞中提取总RNA,测定RNA的量和浓度,用2%琼脂糖凝胶电泳检测其完整性。根据PrimeScript®RT reagent kit反转录试剂盒说明书对提取的RNA进行反转录获得cDNA。将反转录后的cDNA稀释到50 ng·μL-1,作为荧光定量PCR的模板,用SYBR®Premix Ex TaqTM Ⅱ试剂盒进行实时荧光定量PCR,分别测定miR-142、miR-144和FoxO1 mRNA的表达量。
对于两个miRNAs,反应体系:SYBR®Premix Ex TaqTM (2×) 10 μL,ROX Reference Dye Ⅱ (50×) 0.4 μL,miRNAs Specific primer (10 μmol·L-1) 0.8 μL,统一反向引物(10 μmol·L-1)0.8 μL,cDNA模板2 μL,ddH2O 6 μL,总体积为20 μL。将以上反应体系放入7500荧光定量PCR仪器(ABI 公司)中,按照以下程序运行:(1)预变性:95 ℃ 30 s;(2)PCR反应阶段:95 ℃ 15 s,60 ℃ 34 s(此步骤共进行45个循环);(3)熔解曲线分析阶段:95 ℃ 15 s,60 ℃ 60 s,95 ℃ 15 s。
对于FoxO1 mRNA,反应体系:SYBR®Premix Ex TaqTM (2×) 10 μL,ROX Reference Dye II (50×) 0.4 μL,FoxO1上游引物(10 μmol·L-1) 0.8 μL,FoxO1下游引物 (10 μmol·L-1) 0.8 μL,cDNA产物2 μL,ddH2O 6 μL,总体积为20 μL。反应程序:预变性:95 ℃ 30 s。PCR反应阶段:95 ℃ 5 s,60 ℃ 34 s,共45个循环。熔解曲线分析阶段:95 ℃ 15 s,60 ℃ 60 s,95 ℃ 15 s。
利用Western blotting检测FoxO1蛋白的表达量:收集转染48 h后的绵羊前体脂肪细胞,用全蛋白提取试剂盒按照说明书提取全蛋白。以每孔40 μg上样量进行SDS-PAGE。将目的蛋白从凝胶转移至NC膜上,随后用5%的脱脂奶粉封闭,以1∶1 000比例,孵育一抗(FoxO1A或Rabbit anti-GAPDH (AB-P-R 001)),以1∶4 000比例,孵育二抗(羊抗兔IgG-HRP),用PBST缓冲液(PBST缓冲液是PBS与Tween-20以500∶1比例配置)洗膜后涂布适量发光液,最后拍照观察。最后用Image Lab蛋白灰度值分析软件处理免疫印迹条带图,并进行定量分析。
1.2.8 前体脂肪细胞分化检测 为了直观说明miR-142和miR-144对绵羊前体脂肪细胞分化的影响,设置4组试验,每组3个重复,待细胞密度达到80%左右时,按照Lipofectamine 2000说明书分别转染miR-142 mimic、miR-142 NC、miR-144 mimic和miR-144 NC。但转染miR-142和miR-144的绵羊前体脂肪细胞来自不同批次。将转染后第2天的细胞用诱导分化培养基(DMEM/low Glucose培养基中加10%的胎牛血清、10 μmol·L-1胰岛素、1 μmol·L-1地塞米松、0.5 mmol·L-13-异丁基-1-甲基黄嘌呤和2 mmol·L-1罗格列酮)培养,分化8 d后进行油红O染色,显微镜下拍照观察。
1.2.9 数据分析 双荧光素酶检测结果用萤火虫荧光素酶活性与海肾荧光素酶活性的比值,即荧光值表示。用t-检验分析荧光值的组间差异。用2-ΔΔCt法计算FoxO1 mRNA和miR-142(或miR-144)的相对表达量。用目的蛋白条带灰度值与内参GAPDH条带灰度值的比值计算FoxO1蛋白的相对表达量。对于miR-142、miR-144和FoxO1基因的mRNA和蛋白表达量,配合一般线性模型,以组为因子用SPSS 17.0 (SPSS Inc, Chicago, Ⅲ, USA)进行方差分析。各表达量的柱形图用GraphPad Prism 5.0(GraphPad Software Inc, San Diego, CA, USA)绘制。
2 结 果
2.1 预测的与FoxO1具有靶标关系的miRNAs
用TargetScan、PITA、StarBase和miRanda在线软件,预测出与FoxO1具有靶标关系的miRNAs分别有15、70、48和42个。用Venny2.1.0取交集,结果如图1所示。可见,4个软件共同预测出6个miRNAs,分别为miR-183、miR-96、miR-144、miR-153、miR-374、miR-142。经PubMed数据库检索,发现miR-96、miR-183、miR-153 和miR-374这4个miRNAs与FoxO1的靶标关系已得到验证。于是,本研究以miR-142和miR-144为研究对象。miR-142种子区序列(2~7)碱基与FoxO1-3′UTR第2 524~2 529个碱基完全互补。miR-144种子区序列(2~8)碱基与FoxO1-3′UTR第2 679~2 685个碱基完全互补。在TargetScan中,miR-142的综合排序高于miR-144。这些结果一方面在理论上说明miR-142和miR-144与FoxO1具有靶标关系,另一方面也说明,二者在FoxO1-3′UTR上的结合位点很近。
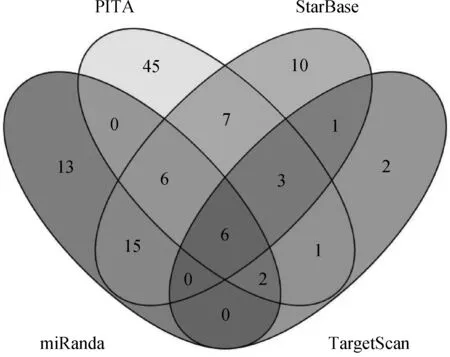
图1 预测的与FoxO1基因具有靶标关系的miRNAsFig.1 Predicted miRNAs potentially targeting to FoxO1 gene
2.2 验证的与FoxO1具有靶标关系的miRNAs
共转染pmir-FoxO1和miR-142 mimic后,过表达组的荧光活性(6.76±0.31)极显著(P<0.01)地低于阴性对照组(14.61±1.09)。与之相似,共转染pmir-FoxO1和miR-144 mimic后,过表达组的荧光活性(9.78±0.46)也显著(P<0.05)低于阴性对照组(15.58±1.23)。说明miR-142和miR-144与FoxO1-3′UTR靶位点的结合抑制了荧光信号的产生。比较而言,miR-142的抑制作用更强一些,这与理论预测得到的miR-142较miR-144的分值高是一致的。
2.3 过表达miR-142对FoxO1 mRNA表达的影响
转染miR-142 mimic的过表达组中,miR-142的表达量(22.97±1.587)极显著(P<0.01)高于阴性对照组(0.64±0.31)和空白对照组(0.43±0.03)(图2A)。由此说明,miR-142 mimic成功转入了绵羊前体脂肪细胞中。相应地,过表达组中FoxO1 的表达量(0.51±0.03)极显著(P<0.01)低于阴性对照组(1.36±0.08)和空白对照组(1.12±0.07)(图2B)。可见,miR-142的过表达导致FoxO1显著低表达,即miR-142负调节FoxO1基因的表达。
2.4 过表达miR-144对FoxO1 mRNA表达的影响
转染miR-144 mimic的过表达组中,miR-144的表达量(506.04±20.00)极显著(P<0.01)高于阴性对照组(0.78±0.11)和空白对照组(0.38±0.06)(图3A)。说明miR-144 mimic成功转入了绵羊前体脂肪细胞。而过表达组中FoxO1 的表达量(0.88±0.05)则极显著(P<0.01)低于阴性(2.55±0.09)和空白对照组(2.91±0.17)(图3B)。同样说明,miR-144的过表达导致FoxO1 显著低表达,即miR-144负调节FoxO1的基因表达。
2.5 过表达miR-142和miR-144对FoxO1蛋白表达的影响
过表达miR-142和miR-144后,FoxO1蛋白的表达情况见图4。图中显示了Western blotting条带(图4A1、4A2)和根据灰度值计算出的蛋白表达量(图4B1、4B2)。过表达组中FoxO1的表达量均显著(P<0.05)低于相应的阴性和空白对照组。由此说明,miR-142和miR-144与FoxO1靶位点结合后,均抑制了FoxO1蛋白的表达。
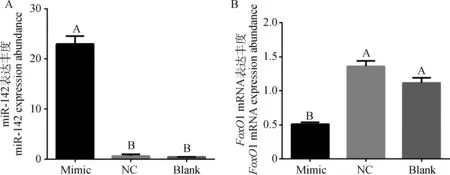
不同大写字母表示差异极显著(P<0.01),图3同The different uppercase letters mean significant difference (P<0.01), the same as Figure3图2 过表达miR-142后miR-142和FoxO1 mRNA在绵羊前体脂肪细胞中的表达Fig.2 The expression of miR-142 and FoxO1 mRNA in ovine preadipocytes after over-expressing miR-142
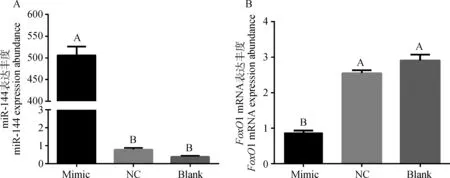
图3 过表达miR-144后miR-144和FoxO1 mRNA在绵羊前体脂肪细胞中的表达Fig.3 The expression of miR-144 and FoxO1 mRNA in ovine preadipocytes after over-expressing miR-144
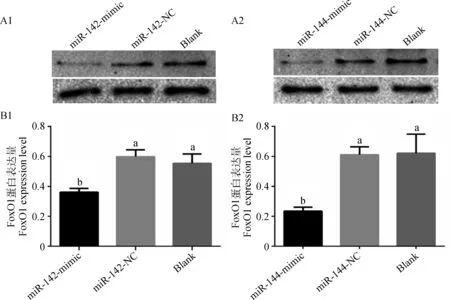
A1、A2.过表达miR-142和miR-144后FoxO1的Western blotting条带图;B1、B2.过表达miR-142和miR-144后FoxO1蛋白表达量。不同小写字母表示组间差异显著(P<0.05)A1, A2. Western blotting diagram of FoxO1 after over-expressing miR-142 and miR-144, respectively; B1, B2.FoxO1 protein expression level after over-expressing miR-142 and miR-144, respectively. The different lower case letters mean significant difference between groups (P<0.05)图4 过表达miR-142和miR-144后FoxO1蛋白在绵羊前体脂肪细胞中的表达Fig.4 The expression of FoxO1 protein in ovine preadipocytes after over-expressing miR-142 and miR-144
2.6 过表达miR-142和miR-144对脂肪细胞分化的影响
在细胞培养的第8天,对培养中的细胞进行油红O染色,发现过表达miR-142和miR-144的两个组的脂滴数分别高于相应的阴性对照组(图5)。说明,过表达miR-142和miR-144均可促进绵羊前体脂肪细胞分化,加快脂滴形成。但因过表达miR-142和miR-144的细胞来自不同批次,二者的过表达组间和阴性对照组间的结果不可比。
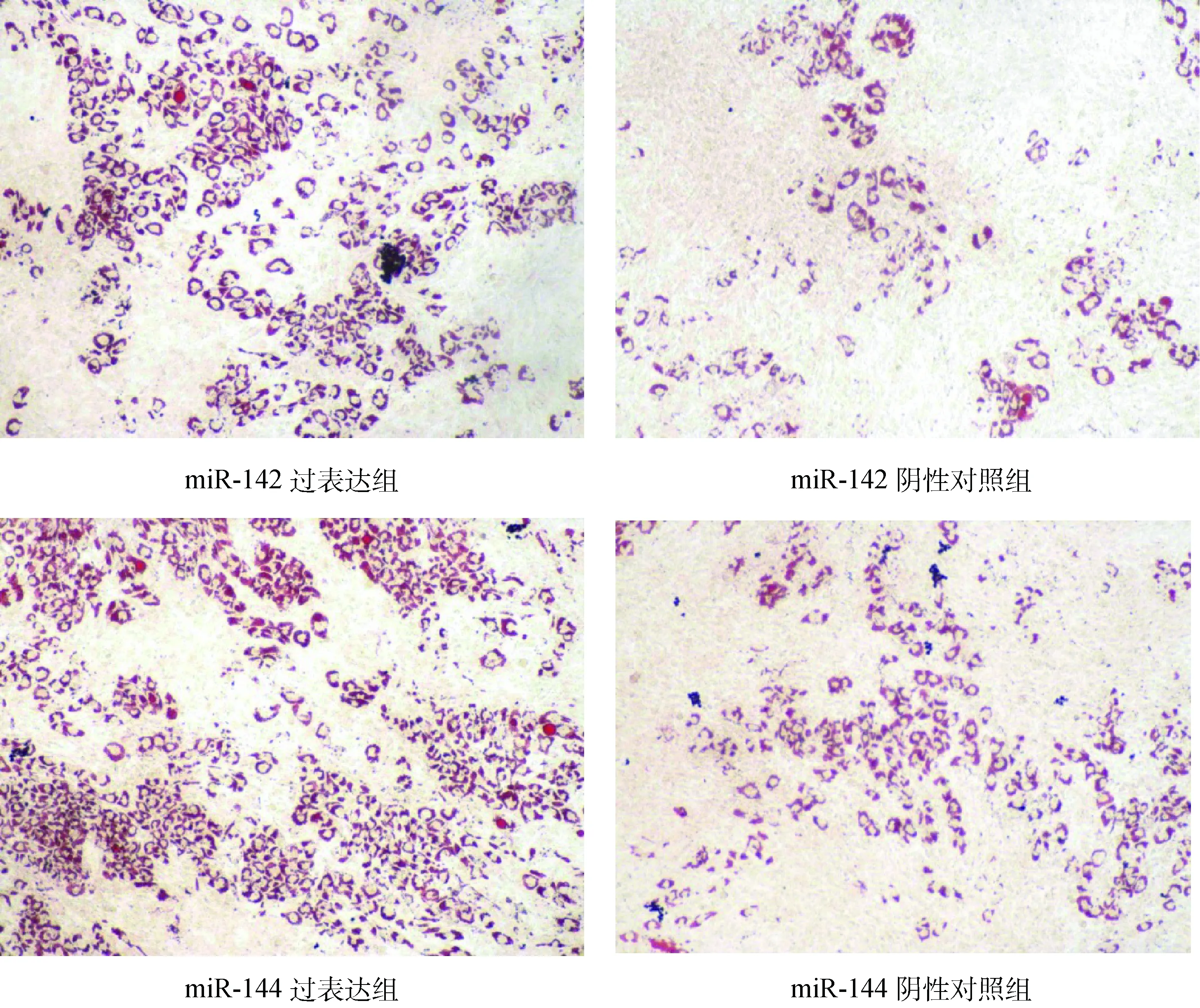
着色区域是被油红O染色后的脂肪细胞The stained areas are the adipocytes stained by Oil red O图5 过表达miR-142和miR-144后绵羊脂肪细胞分化第8天的油红O染色结果(100×)Fig.5 Oil red O staining results of differentiated ovine adipocytes at 8th day by over-expressing miR-142 and miR-144(100×)
3 讨 论
基因和miRNA间的靶标关系通常可以在生物信息学预测的基础上再通过分子生物技术验证确定,这样可以大大提高研究的准确性。本研究先利用4个在线软件对FoxO1基因进行候选miRNAs预测。其中,TargetScan软件最大的特点是强调miRNA的物种间保守性[15],因而预测出的miRNAs最少。本研究的对象是绵羊,在miRBase 21中记录的绵羊miRNA序列只有153个(OARv3.1),远少于常规反刍家畜,更少于人和小鼠。因此,物种保守性对于本研究尤为重要。PITA采用的是自由参数模型计算miRNA-mRNA二聚体的自由能和解除靶标关系所消耗能量差[16],预测范围较大,预测出的miRNAs最多。miRanda和StarBase也有其相应的特性和算法。为此,又结合Venn图,预测得到6个候选miRNAs。在此基础上,结合前人研究结果,确定miR-142和miR-144用于本研究中的后续试验验证。
在人、鼠、猪和牛的研究中发现,FoxO1与能量代谢密不可分。FoxO1通过增加β细胞数量,增强β细胞葡萄糖传感和抗氧化功能,改善了β细胞的补偿,从而调节营养过剩或肥胖[20]。肥胖状态下,胰岛素通过调节FoxO1的磷酸化可改善葡萄糖代谢[21]。FoxO1参与脂肪代谢的调节。α-硫辛酸可以使FoxO1去磷酸化,减少肝中脂肪的积累[22]。减少猪FoxO1基因表达后,甘油-3-磷酸脱氢酶、甘油三酯、脂肪酸结合蛋白、PPARγ显著增加,因而认为FoxO1是前体脂肪细胞分化过程中的负转录调控因子[23]。在牛的脂肪组织中PPARγ表达升高时,FoxO1的含量却降低了[24],这说明FoxO1与PPARγ的关系十分密切。PPARγ在脂肪代谢中的重要性已得到广泛研究,其功能主要是促进脂肪细胞分化[25-26],因而被认为是一个重要的监控脂肪细胞分化的基因[27]。转录因子FoxO1通过P13K/Akt和MAPK/ERK通路去磷酸化后[5],进入细胞核内与PPARγ的启动子区结合,可抑制PPARγ的活性和脂肪细胞分化,从而调节脂肪代谢[14,28]。
本研究利用体外培养的绵羊前体脂肪细胞进行研究,发现过表达miRNA-142和miRNA-144后,FoxO1 的mRNA和蛋白表达量均显著下降,且脂滴明显增多,说明miR-142和miR-144均可下调转录因子FoxO1的表达,促进脂肪细胞分化。相似的研究发现,miR-15a/b也可通过抑制FoxO1的表达,促进猪的前体脂肪细胞分化[29]。PPARγ的表达也受miRNAs的调节。miR-130[8]与PPARγ的3′ UTR 和CDS区结合,miR-301a[30]与PPARγ的3′ UTR区结合,可下调PPARγ的表达,从而抑制脂肪细胞分化。由此,作为转录因子的FoxO1与其靶基因PPARγ及其与二者的调节miRNAs间的关系可以概括如图6所示。
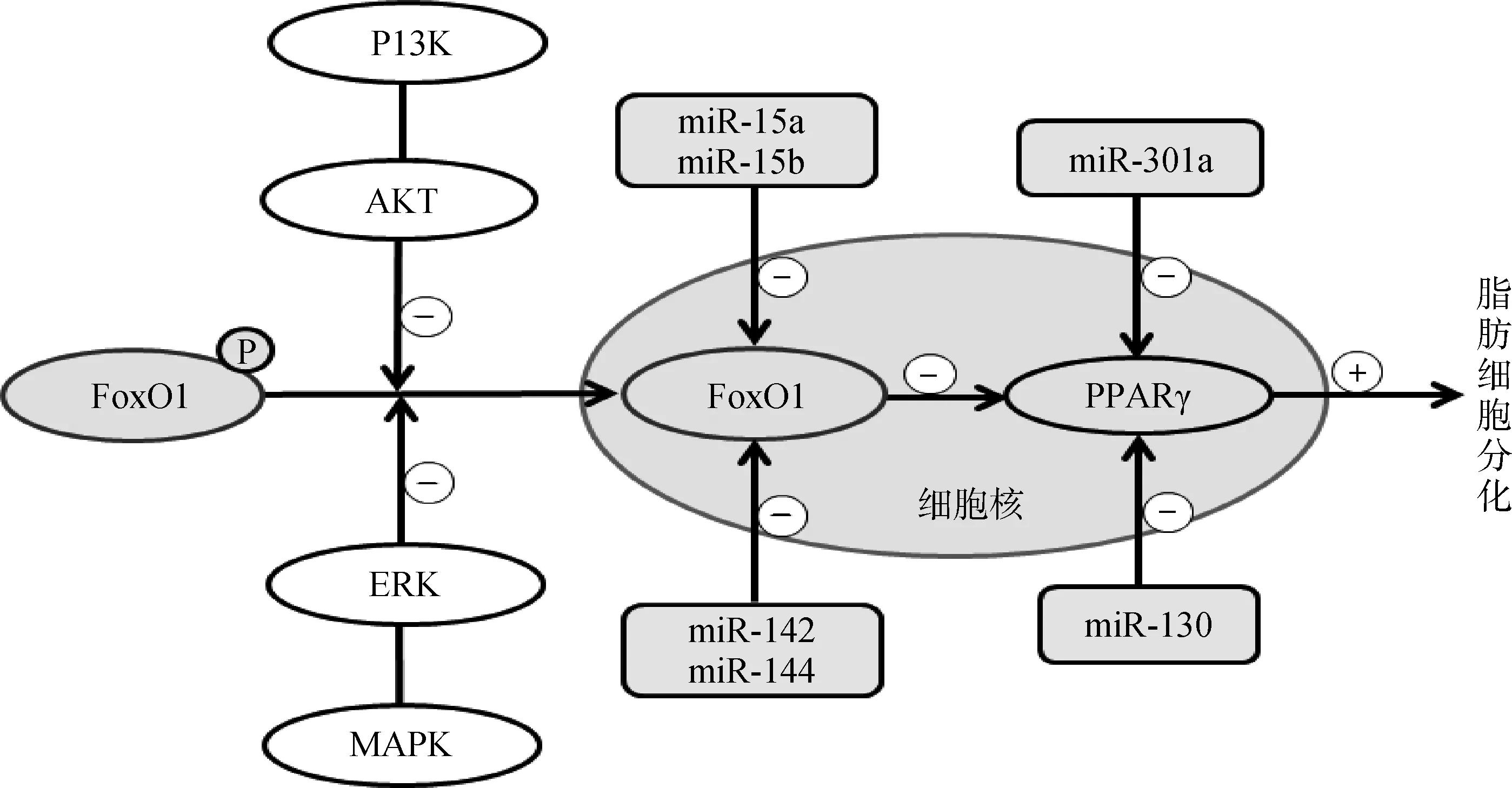
⊕表示正调节;⊖表示负调节⊕means positive regulation;⊖means negative regulation图6 FoxO1作用于PPARγ调节脂肪细胞分化的代谢通路Fig.6 The metabolic pathways regulating adipocyte differentiation by FoxO1 acting on PPARγ
活体试验研究也表明,miR-142和miR-144均可调节脂肪代谢。和牛(高肌内脂肪)背脂中miR-142-3p的表达量显著高于荷斯坦牛(中度肌内脂肪)[31]。高腹脂型北京油鸡中miR-144-3p表达量极显著高于低腹脂型北京油鸡[32]。这说明miR-142-3p和miR-144-3p影响了脂肪沉积,但这些文献并未交待其机制。用图6的通路则可直观地解释其分子机制。
4 结 论
本研究通过理论预测发现,FoxO1与miR-142和miR-144存在靶标关系。用双荧光素酶报告系统发现miR-142和miR-144抑制FoxO1的表达。过表达这两个miRNAs后,FoxO1 mRNA及其编码蛋白的表达量显著下降,证实了这两个miRNAs与FoxO1具有靶标关系,且负调节FoxO1的表达,进而通过FoxO1对PPARγ的负调节促进绵羊前体脂肪细胞的分化。
参考文献(References):
[1] SON Y H, KA S, KIM A Y, et al. Regulation of adipocyte differentiation via microRNAs[J].EndocrinolMetab(Seoul), 2014, 29(2): 122-135.
[2] USHMOROV A, WIRTH T. FOXO in B-cell lymphopoiesis and B cell neoplasia[J].SeminCancerBiol, 2017, doi:10.1016/j.semcancer.2017.07.008.
[3] WANG Q R, REN J. mTOR-Independent autophagy inducer trehaloserescues against insulin resistance-induced myocardial contractile anomalies: role of p38 MAPK and FoxO1[J].PharmacolRes, 2016, 111: 357-373.
[4] ROY S K, SRIVASTAVA R K, SHANKAR S. Inhibition of PI3K/AKT and MAPK/ERK pathways causes activation of FoxO transcription factor, leading to cell cycle arrest and apoptosis in pancreatic cancer[J].JMolSignal, 2010, 5: 10.
[5] PAN C W, JIN X, ZHAO Y, et al. AKT-phosphorylated FOXO1 suppresses ERK activation and chemo-resistance by disrupting IQGAP1-MAPK interaction[J].EMBOJ, 2017, 36(8): 995-1010.
[6] YUAN K, AI W B, WAN L Y, et al. The miR-290-295 cluster as multi-faceted players in mouse embryonic stem cells[J].CellBiosci, 2017, 7: 38.
[7] HILTON C, NEVILLE M J, KARPE F. microRNAs in adipose tissue: their role in adipogenesis and obesity[J].IntJObes(Lond), 2013, 37(3): 325-332.
[8] LEE E K, LEE M J, ABDELMOHSEN K, et al. miR-130 suppresses adipogenesis by inhibiting peroxisome proliferator-activated receptor γ expression[J].MolCellBiol, 2011, 31(4): 626-638.
[9] MOHAMMED B T, SONTAKKE S D, IOANNIDIS J, et al. The adequate corpus luteum: miR-96 promotes luteal cell survival and progesterone production[J].JClinEndocrinolMetab, 2017, 102(7): 2188-2198.
[10] ZHANG L Q, QUAN H Y, WANG S H, et al. miR-183 promotes growth of non-small cell lung cancer cells through FoxO1 inhibition[J].TumorBiol, 2015, 36(10): 8121-8126.
[11] WU Z Q, HE B F, HE J C, et al. Upregulation of miR-153 promotes cell proliferation via downregulation of the PTEN tumor suppressor gene in human prostate cancer[J].Prostate, 2013, 73(6): 596-604.
[12] HE W B, FENG L, XIA D L, et al. miR-374a promotes the proliferation of human osteosarcoma by downregulating FOXO1 expression[J].IntJClinExpMed, 2015, 8(3): 3482-3489.
[13] XIONG Y, PANG W J, WEI N, et al. Knockdown of both FoxO1 and C/EBPβ promotes adipogenesis in porcine preadipocytes through feedback regulation[J].CellBiolInt, 2013, 37(9): 905-916.
[14] BABA S, UENO Y, KIKUCHI T, et al. A limonoid kihadanin B from immatureCitrusunshiupeels suppresses adipogenesis through repression of the Akt-FOXO1-PPARγ axis in adipocytes[J].JAgricFoodChem, 2016, 64(51): 9607-9615.
[15] LEWIS B P, SHIH I H, JONES-RHOADES M W, et al. Prediction of mammalian microRNA targets[J].Cell, 2003, 115(7): 787-798.
[16] KERTESZ M, IOVINO N, UNNERSTALL U, et al. The role of site accessibility in microRNA target recognition[J].NatGenet, 2007, 39(10): 1278-1284.
[17] LI J H, LIU S, ZHOU H, et al. StarBase v2.0: decoding miRNA-ceRNA, miRNA-ncRNA and protein-RNA interaction networks from large-scale CLIP-Seq data[J].NucleicAcidsRes, 2014, 42(D1): D92-D97.
[18] JOHN B, ENRIGHT A J, ARAVIN A, et al. Human microRNA targets[J].PLoSBiol, 2004, 2(11): e363.
[19] LIN G L, CHAI J, YUAN S, et al. VennPainter: a tool for the comparison and identification of candidate genes based on Venn diagrams[J].PLoSOne, 2016, 11(4): e0154315.
[20] ZHANG T, KIM D H, XIAO X W, et al. FoxO1 plays an important role in regulating β-cell compensation for insulin resistance in male mice[J].Endocrinology, 2016, 157(3): 1055-1070.
[21] NERURKAR P V, NISHIOKA A, ECK P O, et al. Regulation of glucose metabolism via hepatic forkhead transcription factor 1 (FoxO1) byMorindacitrifolia(noni) in high-fat diet-induced obese mice[J].BrJNutr, 2012, 108(2): 218-228.
[22] KUO Y T, LIN T H, CHEN W L, et al. Alpha-lipoic acid induces adipose triglyceride lipase expression and decreases intracellular lipid accumulation in HepG2 cells[J].EurJPharmacol, 2012, 692(1-3): 10-18.
[23] PANG W J, YU T Y, BAI L, et al. Tissue expression of porcine FoxO1 and its negative regulation during primary preadipocyte differentiation[J].MolBiolRep, 2009, 36(1): 165-176.
[24] LIU X M, LIU G F, TAN X W, et al. Gene expression profiling of SIRT1, FoxO1, and PPARγ in backfat tissues and subcutaneous adipocytes of Lilu bulls[J].MeatSci, 2014, 96(2): 704-711.
[25] PU Y, VEIGA-LOPEZ A. PPARγ agonist through the terminal differentiation phase is essential for adipogenic differentiation of fetal ovine preadipocytes[J].CellMolBiolLett, 2017, 22: 6.
[26] 林婄婄, 高中元, 袁亚男, 等.PPARα和PPARγ基因在不同脂尾型绵羊脂肪组织中的发育性表达研究[J]. 畜牧兽医学报, 2012, 43(9): 1369-1376.
LIN P P, GAO Z Y, YUAN Y N, et al. Developmental expression ofPPARαandPPARγmRNA in adipose tissues of different fat-tailed sheep[J].ActaVeterinariaetZootechnicaSinica, 2012, 43(9): 1369-1376. (in Chinese)
[27] DE LA ROSA RODRIGUEZ M A, KERSTEN S. Regulation of lipid droplet-associated proteins by peroxisome proliferator-activated receptors[J].BiochimBiophysActa, 2017, 1862(10): 1212-1220.
[28] KIM D H, LEE B, KIM M J, et al. Molecular mechanism of betaine on hepatic lipid metabolism: Inhibition of forkhead Box O1 (FoxO1) binding to peroxisome proliferator-activated receptor gamma (PPARγ)[J].JAgricFoodChem, 2016, 64(36): 6819-6825.
[29] DONG P Y, MAI Y, ZHANG Z Y, et al. miR-15a/b promote adipogenesis in porcine pre-adipocyte via repressing FoxO1[J].ActaBiochimBiophysSin(Shanghai), 2014, 46(7): 565-571.
[30] LI H, XUE M, XU J, et al. miR-301a is involved in adipocyte dysfunction during obesity-related inflammation via suppression of PPARγ[J].Pharmazie, 2016, 71(2): 84-88.
[31] GUO Y T, ZHANG X X, HUANG W L, et al. Identification and characterization of differentially expressed miRNAs in subcutaneous adipose between Wagyu and Holstein cattle[J].SciRep, 2017, 7: 44026.
[32] HUANG H Y, LIU R R, ZHAO G P, et al. Integrated analysis of microRNA and mRNA expression profiles in abdominal adipose tissues in chickens[J].SciRep, 2015, 5: 16132.
