酿酒葡萄品种SSR-PCR体系的优化与建立
2018-04-13马文瑞邹弯魏玉洁严密全莉王雪薇高林龙马静武运薛洁
马文瑞,邹弯,魏玉洁,严密,全莉,王雪薇,高林龙,马静,武运*,薛洁*
1(新疆农业大学 食品科学与药学学院,新疆 乌鲁木齐,830052)2(中国食品发酵工业研究院,北京,100015)
葡萄属于葡萄科(Vitaceae)葡萄属(Vitis L.),为木质藤本植物,是世界最古老的植物之一。优良葡萄品种是生产优质葡萄酒的关键因素之一,因此,酿酒葡萄种质资源的鉴定与分类是葡萄酒产业规范发展的基础。葡萄主要的繁殖方式为无性繁殖,不同产区间互相引种,出现同名异物,同物异名现象频率较高,又因葡萄种间杂交容易产生一些中间型和过渡型杂种,给葡萄的分类鉴定带来困难[1]。随着分子生物学技术在葡萄酒行业的发展,基于DNA指纹图谱技术的品种鉴定方法,应用于葡萄品种鉴别方面,得到了认可与青睐。DNA指纹图谱技术相对于传统的细胞学,孢粉学,细胞学和生物化学鉴定方法,该技术能够快速准确地对亲缘关系很近的品种或个体进行辨别[2]。
简单序列重复 (simple sequence repeats, SSR)技术是基于聚合酶链式反应(polymerase chain reaction, PCR)的DNA指纹图谱技术,SSR技术也称简单重复序列,以少数几个碱基为单位的多次重复序列。该技术具有共显性遗传,可重复性高,多态性强等优点,已经广泛应用于品种鉴定、遗传多样性检测和遗传图谱的构建等方面,如THOMAS等人利用葡萄微卫星位点设计了5对引物,成功了区分了33个葡萄品种[3];郭印山等人筛选出多态性高的49对SSR引物对15份不同原产地的葡萄品种进行遗传多态性分析,结果表明,欧亚种和美洲种葡萄亲缘关系相对较近,而山葡萄与二者亲缘关系相对较远,引物VMC-NG4B9和VMC3G9能将15个葡萄品种区分开[4];张萌等人利用SSR分子标记分析了79份不同原产地的葡萄种质资源的亲缘关系,结果表明说明中国野生葡萄资源和砧木品种与鲜食的栽培种亲缘关系较远[5]。成冰等人利用SSR分析技术对13个酿酒白葡萄品种进行鉴定,结果表明13个品种均有特征性引物且筛选出的引物鉴别效率差异很大, VrZAG62和VVIb66可鉴定所有品种[6]。
本试验采用单因素与正交试验相结合方法,对影响酿酒葡萄 SSR-PCR 扩增体系的主要因素进行优化,以期建立一套适合酿酒葡萄资源的SSR标记反应体系,为葡萄的种质鉴定及亲缘关系提供参考依据。
1 材料与方法
1.1 试验材料
1.1.1试验原料与试剂
酿酒葡萄原料:新疆天山北麓葡萄酒产区的赤霞珠,霞多丽,美乐和玫瑰香酿酒葡萄,置于-80 ℃冰箱贮藏备用。
用于SSR-PCR反应的10×PCR buffer(含 Mg2﹢15 mmol/L)、dNTPs和TaqDNA聚合酶(Taq酶)等购自于大连宝生物有限公司。D15000 DNA marker和D2000 DNA marker购买于北京天根生化科技有限公司。从相关文献中筛选出6对酿酒葡萄SSR引物,见表1,由上海生工生物工程技术有限公司合成。经初步筛选,选用多态性好、条带清晰的引物 VrZAG79作为单因素和正交试验的固定引物。其他试剂均购自北京化工厂。

表1 SSR引物序列及退火温度Table 1 Sequence and annealing temperature of SSR primer
注:F:上游引物;R:下游引物;Tm:退火温度。
1.1.2仪器与设备
JY-CZ-BL垂直电泳槽,北京君意东方电泳设备有限公司; VeritiTM96-Well Thermal Cycler 梯度PCR仪,美国ABI公司;BG-Power600 通用电泳仪电源,北京百晶生物技术有限公司;Tanon 1600凝胶成像系统,上海天能科技有限公司;BioSpec-nano核酸蛋白检测仪,德国Eppendorf公司;微量移液器,德国Eppendorf公司;立式电热压力蒸汽灭菌锅:上海申安医疗器械厂等。
1.2 试验方法
1.2.1基因组DNA提取及质量检测
采用改良的十六烷基三甲基溴化铵法(hexadecyl trimethyl ammonium bromide, CTAB)提取葡萄基因组DNA。参考齐玲倩等人的《水果果肉中总DNA提取方法的比较研究》并根据葡萄的特性加以改进[8]。用0.8%的琼脂糖凝胶电泳检测DNA的片段大小及质量,同时在核酸蛋白仪上测定样品DNA的浓度。赤霞珠葡萄的基因组DNA作为单因素和正交试验的固定DNA模板。
1.2.2PCR基本反应体系
本试验采用25 μL反应体系进行PCR扩增,参照10×PCR Buffer说明书,并根据预实验结果建立初始体系为: 15 mmol/L Mg2﹢2.50 μL, 2.5 mmol/L dNTPs 2.00 μL,5.0 U/μLTaq酶0.30 μL,10 μmol/L引物1.00 μL,50 ng/25 μL DNA模板1.00 μL,剩余用超纯水补足。
1.2.3PCR反应程序及聚丙烯酰胺凝胶电泳检测
95 ℃预变性5 min;94 ℃变性50 s,退火温度60 s,72 ℃延伸1 min,共35循环,最后72 ℃延伸7 min。PCR扩增产物用 8.0%聚丙烯酰胺凝胶电泳,银染检测,在凝胶成像系统下观察,并照相分析。使用 D2000 DNA marker(含100,250,500,750,1 000,2 000 bp 6条带)作为DNA核酸分子量梯度的标准。
1.2.4单因素试验设计
对影响SSR-PCR反应体系的5个因素(Mg2+、dNTPs、Taq酶、引物、模板 DNA)进行单因素试验,见表2。
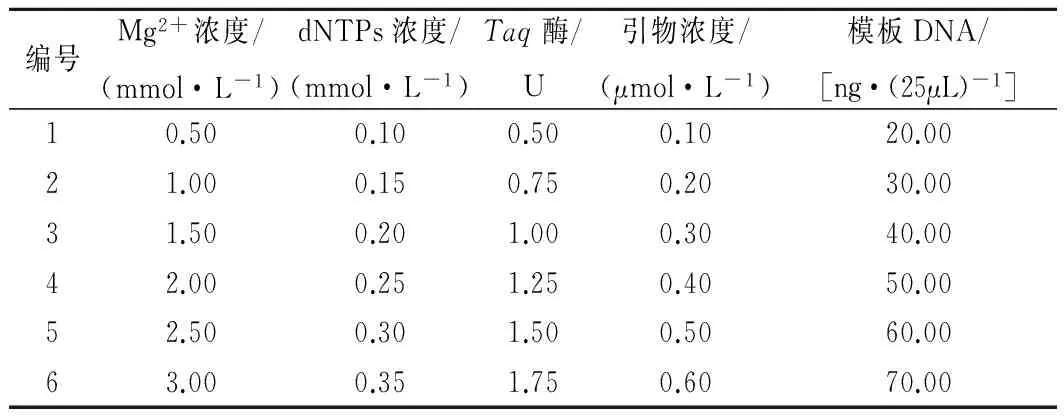
表2 SSR-PCR单因素试验设计Table 2 Single factor experiments design for SSR-PCR
单因素浓度梯度变化范围参照陶巧静等人的《葡萄SSR-PCR体系优化及其诱变单株遗传多样性分析》[9]。当1种因素为单一变量进行试验时,其他因素按照基本反应体系的含量添加,比较不同浓度处理对 SSR-PCR 扩增结果的影响。
1.2.5正交优化试验设计
根据单因素试验确定的各影响因素的浓度范围,设计L16(45)进行5因素4水平正交试验,重复2次,优化条件见表3。PCR反应产物经8%聚丙烯酰胺凝胶电泳检测,以确定适合酿酒葡萄SSR-PCR扩增的最佳反应体系。
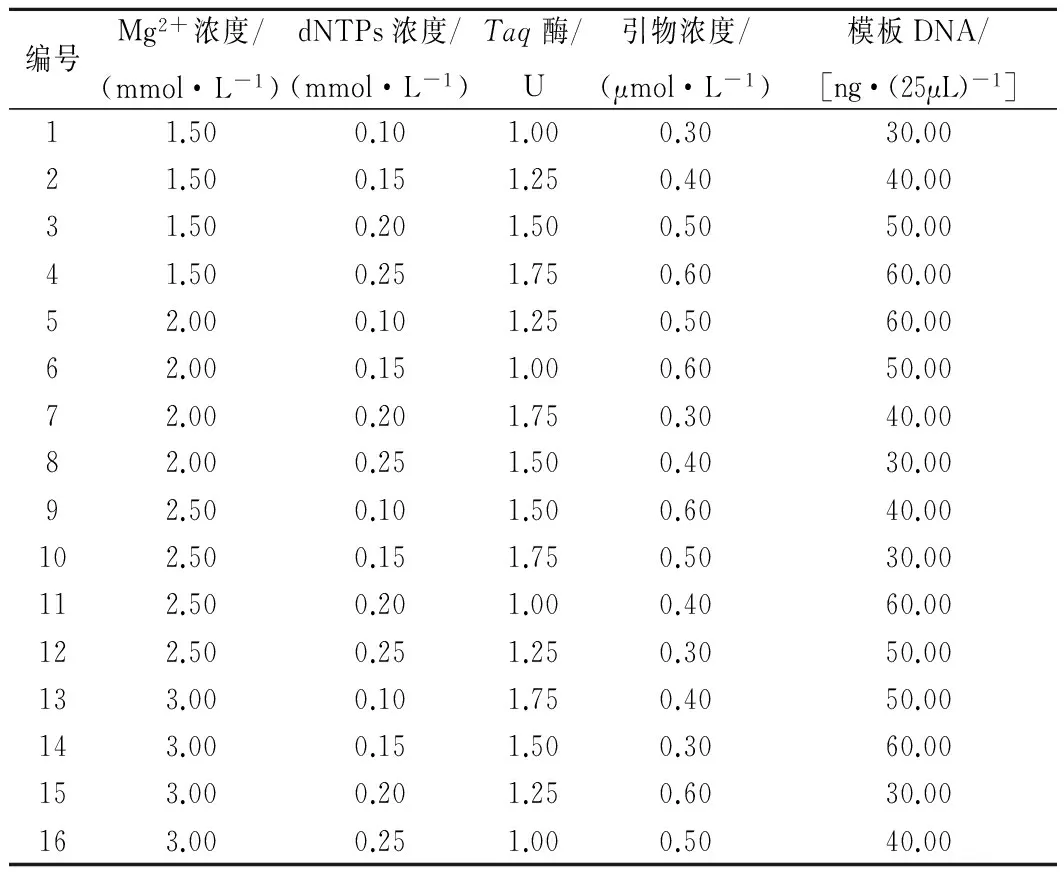
表3 SSR-PCR反应因素水平L16(45)正交设计
1.2.6正交优化试验设计结果分析
结果分析参照段永红等人的《药用植物苦参SSR-PCR体系的优化与验证》[10]。数据使用SPSS 20.0进行分析。
1.2.7最优反应体系检测
将筛选出的引物分别对赤霞珠,霞多丽,美乐和玫瑰香酿酒葡萄的DNA进行扩增,进行最优体系进行稳定性验证。
2 结果与分析
2.1 基因组DNA提取结果检测
由图1可以看出,4个试验样品的基因组DNA提取效果良好,DNA质量较高,条带清晰完整、无杂带、浓度高。
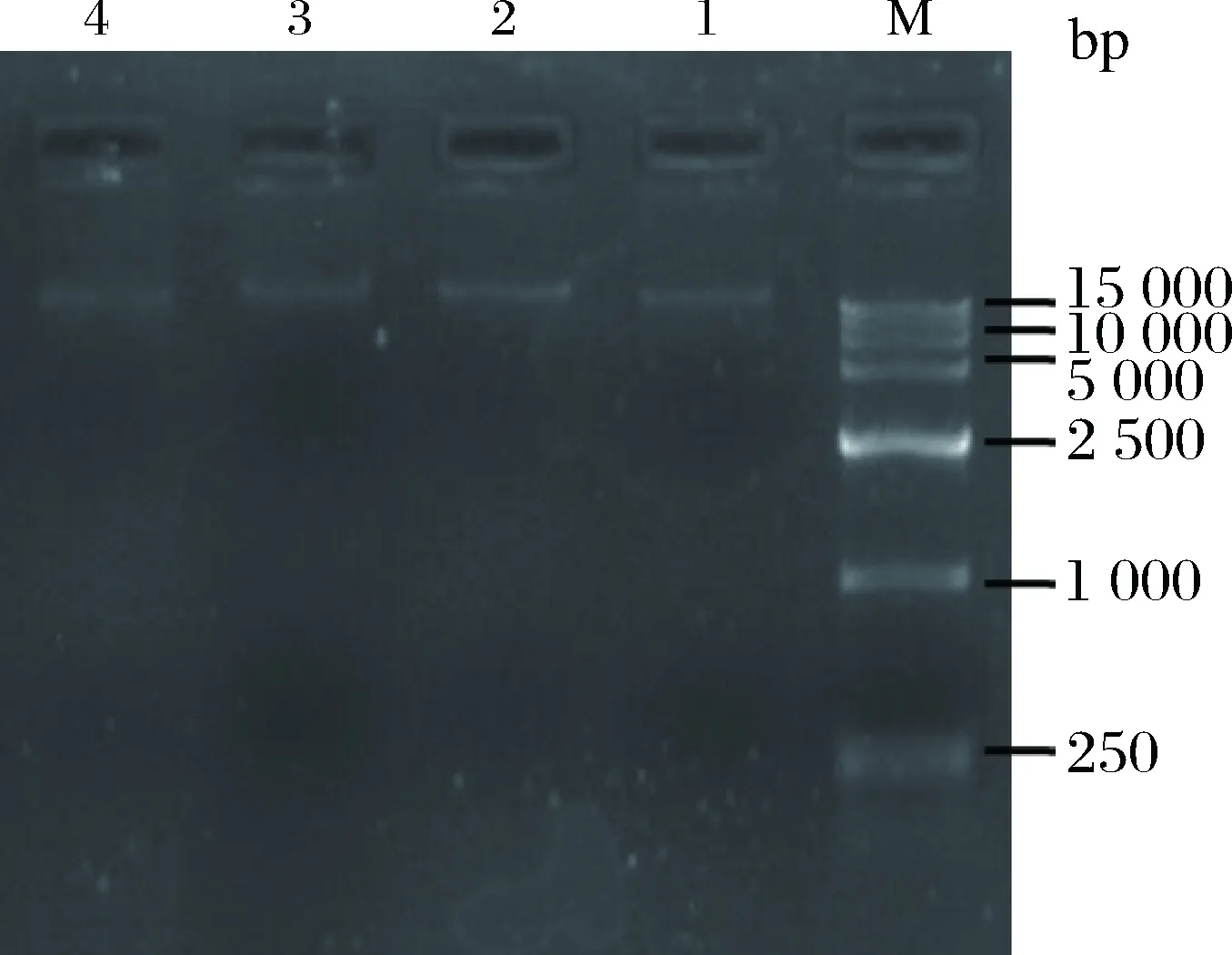
M-D 15000 DNA marker;1-赤霞珠;2-霞多丽;3-美乐;4-玫瑰香图1 酿酒葡萄基因组DNA的琼脂糖凝胶电泳结果Fig.1 Agarose gel electrophoresis of genomic DNA fromVitis vinifera
2.2 单因素试验结果
2.2.1Mg2+浓度对SSR-PCR结果的影响
Mg2+浓度作为PCR体系最重要的变量之一。Mg2+浓度过高,可能会产生非特异性扩增;Mg2+浓度过低,会造成扩增效率降低,因此选择合适的Mg2+浓度,提高PCR产率。图2表明,当Mg2+浓度在0.5和1.0 mmol/L时条带不明显,当Mg2+浓度在1.5 mmol/L以上时出现扩增片段,当Mg2+浓度在2.0和2.5 mmol/L时,条带清晰且多态性良好。综合以上分析,选取Mg2+浓度在1.5~3.0 mmol/L范围进行正交优化试验。
2.2.2dNTPs浓度对SSR-PCR结果的影响
不同浓度的dNTPs均扩增出产物。浓度不同,扩增片段的产率和清晰度不同。图2表明,当dNTPs为0.10和0.15 mmol/L时,条带清晰,扩增产物的多态性最高;在0.20~0.35 mmol/L时,条带清晰,但多态性降低;当dNTPs浓度在0.35 mmol/L时,产物多态性最低。综合经济角度分析,选取dNTPs浓度在0.10~0.25 mmol/L范围进行正交优化试验。

图2 不同浓度Mg2+、dNTPs、Taq酶PCR产物电泳结果Fig.2 Electrophoresis of PCR amplification products withdifferent amounts of Mg2+,dNTPs, Taq DNA polymerase注:M-D2000 DNA marker;Mg2+浓度从右至左依次为0.50、1.00、1.50、2.00、2.50、3.00 mmol/L;dNTPs浓度从右至左依次为0.10、0.15、0.20、0.25、0.30、0.35 mmol/L;Taq酶浓度从右至左依次为0.50、0.75、1.00、1.25、1.50、1.75 U
2.2.3Taq酶浓度对SSR-PCR结果的影响
Taq酶是PCR反应中最重要的因素之一。Taq酶浓度过高,产生非特异性片段的几率增大;浓度过低,PCR扩增效率降低。不同浓度下的Taq酶均扩增出产物且条带清晰,图2表明,Taq酶浓度为1.25~1.75 U的多态性高于0.50~1.00 U。综合以上分析,选取Taq酶浓度在1.00~1.75 U范围进行正交优化试验。
2.2.4引物浓度对SSR-PCR结果的影响
不同浓度的引物均扩增出产物。引物浓度不同,扩增片段的产率不同。图3表明,当引物浓度为0.1~0.2 μmol/L,条带清晰,但多态性较低。当引物浓度为0.3~0.6 μmol/L,条带清晰,并且片段的多态性较高。综合以上分析,选取引物浓度在0.3~0.6 μmol/L范围进行正交优化试验。
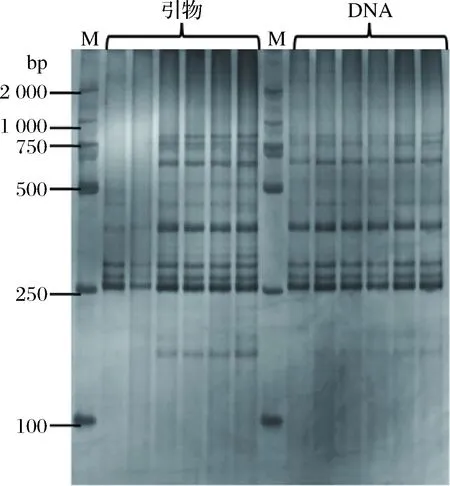
图3 不同浓度引物、DNA PCR产物电泳结果Fig.3 Electrophoresis of PCR amplification products withdifferent amounts of primers and DNAM-D2000 DNA marker;引物浓度从左至右依次为0.10、0.20、0.30、0.40、0.50、0.60 μmol/L;DNA浓度从左至右依次为20.00、30.00、40.00、50.00、60.00、70.00 ng/25 μL
2.2.5DNA浓度对SSR-PCR结果的影响
适当的DNA含量是增加PCR产率和特异性片段合成的首要条件。DNA含量过低,PCR产率下降;DNA含量过高,生成非特异性片段的几率增大。图3表明,不同浓度下的DNA均扩增出产物,但当DNA浓度在20 ng/25μL时背景颜色较淡,条带模糊,当DNA浓度在70 ng/25μL时背景颜色较深。在30~60 ng/25μL范围内,条带清晰且多态性良好。综合以上分析,选取DNA含量在30~60 ng/25μL范围进行正交优化试验。
2.3 正交优化试验设计结果分析
根据正交设计表设计的16个处理组合进行SSR-PCR扩增反应,通过聚丙烯酰胺凝胶电泳检测,得出结果均有扩增产物,见图4,2次重复结果基本一致。由于Mg2+、dNTPs、Taq酶、引物、模板 DNA的浓度组合不同,扩增结果也存在着差异。1和2号组合扩增产物较少,3和4号组合条带模糊,不易观察;而9、10、13和14号组合扩增的条带清晰,多态性较高,便于观察。根据电泳图,结合遗传多样性分析的要求,将扩增产物丰富度高、清晰度高、便于观察分析的最佳产物记为16分,反之,将无扩增条带或条带较少、清晰度差的记为1分。根据电泳结果,分别进行2次独立打分统计。16个处理组合得到的分数分别记为3、4、7、5、7、8、7、6、16、15、5、6、10、11、5、8;3、4、7、5、7、7、7、6、16、15、5、6、10、11、5、8。从两次评分来看,各个组合的结果都有较高的一致性,重复性好。两次评分取平均值,依据平均分结果进行直观分析和方差分析。
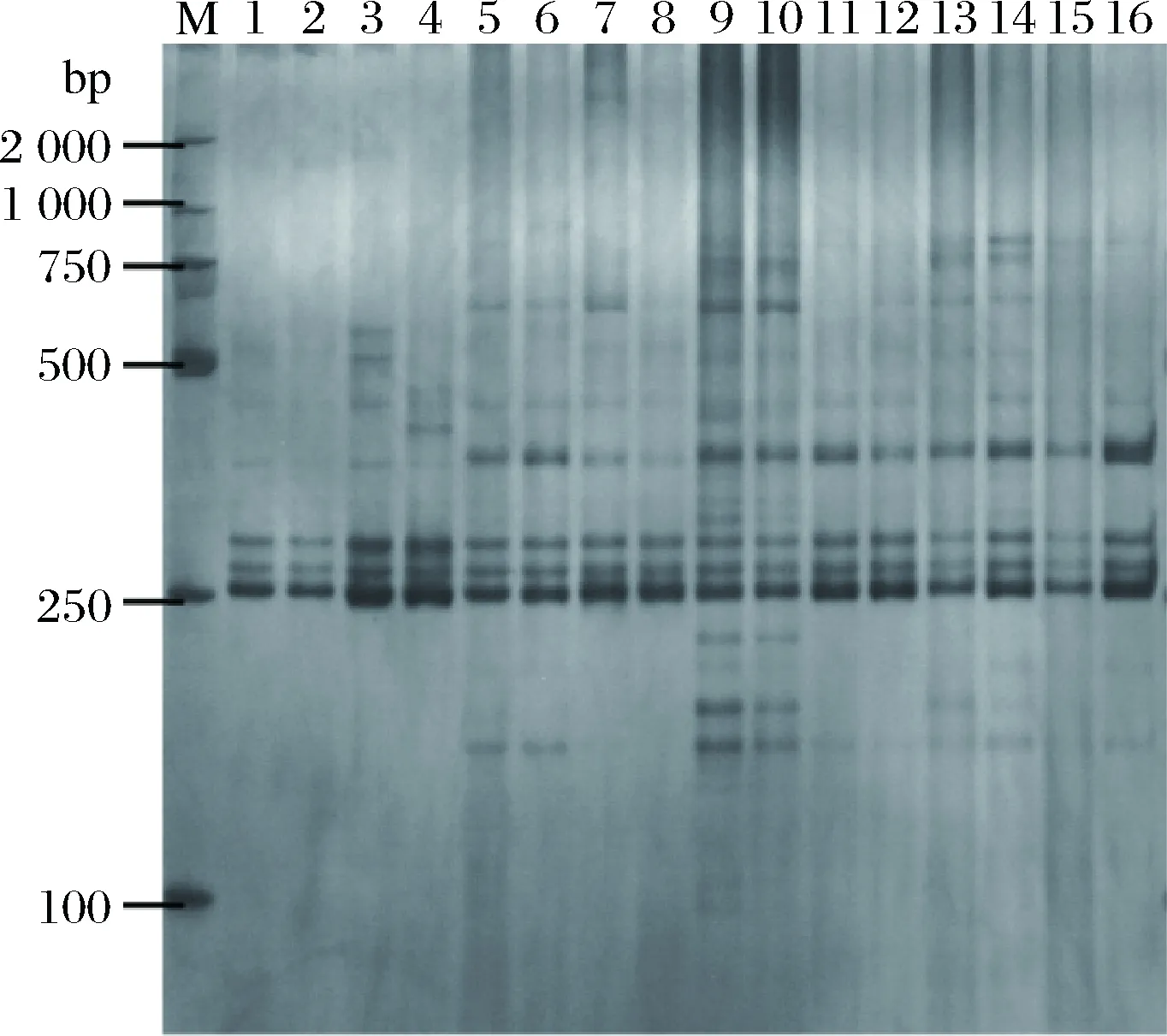
图4 正交设计组合PCR产物电泳结果Fig.4 Electrophoresis of PCR amplification productswith different combinationM-D2000 DNA marker;1~16表示为正交优化设计组合
根据表4,k值为每一因素水平下的数据平均值,反映了各因素不同水平对反应体系的影响情况,均值越大说明该反应水平对扩增效果越好。因此最佳25 μL体系为Mg2+浓度为2.5 mmol/L,dNTPs为0.15 mmol/L,Taq酶为1.5 U,引物为0.5 μmol/L,DNA含量为40 ng/25μL。由直观分析中的相同因素不同水平间的均值极差R值的大小判断各因素对PCR反应效果的影响程度,R值越大,说明该因素对结果影响越大。各因素对结果的影响程度依次为:Mg2+>Taq酶>dNTPs>引物>模板DNA。
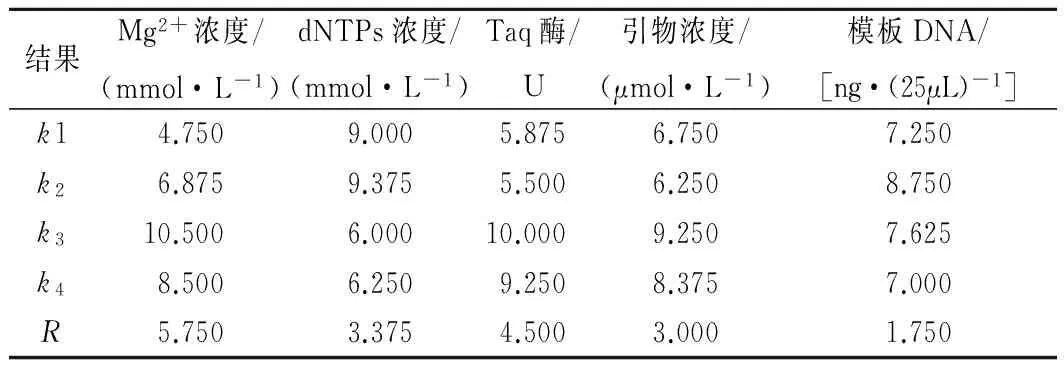
表4 正交设计直观分析Table 4 Intuitive analysis of orthogonal design
对16个处理组合进行方差分析,结果见表5 。由方差分析中的 F值的大小判断各因素对反应效果的影响程度依次为:Mg2+>Taq酶>dNTPs>引物>模板DNA。结果与直观分析一致。
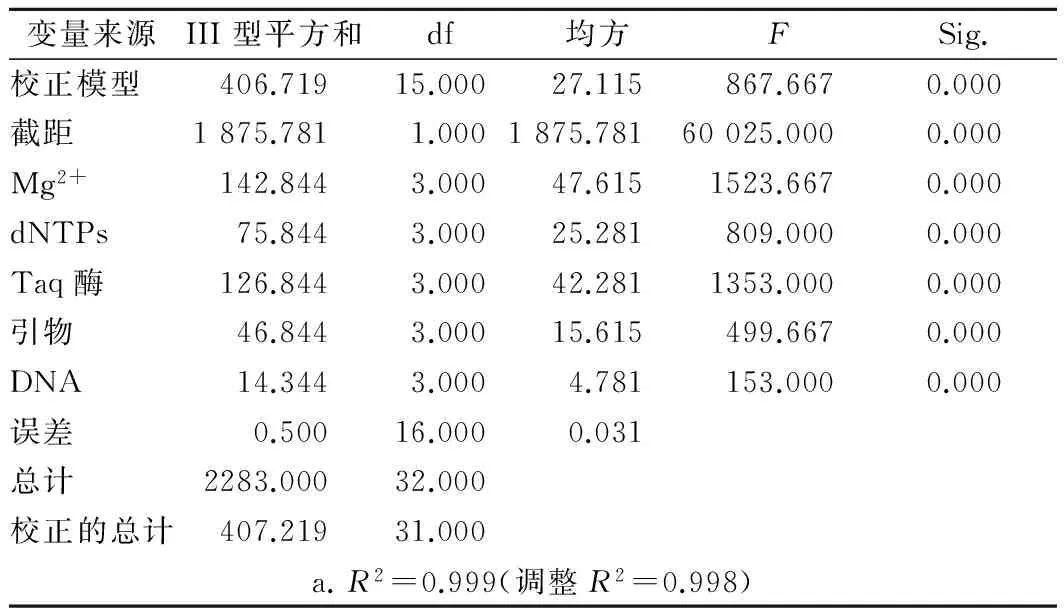
表5 正交设计方差分析Table 5 Variance analysis of orthogonal design
2.4 最优反应体系检测
根据上述试验建立的酿酒葡萄SSR-PCR体系,使用已经筛选出的6对酿酒葡萄SSR引物,对4个酿酒葡萄品种进行聚丙烯酰胺凝胶验证扩增效果。扩增结果如图,均有扩增结果,条带相对清晰,易于观察。条带均集中在100~500 bp左右,不同引物产生的多态性条带为5~13条,见图5,图6。证明单因素和正交试验建立的SSR-PCR体系有较高的稳定性和重复性,该体系适用于酿酒葡萄SSR反应。
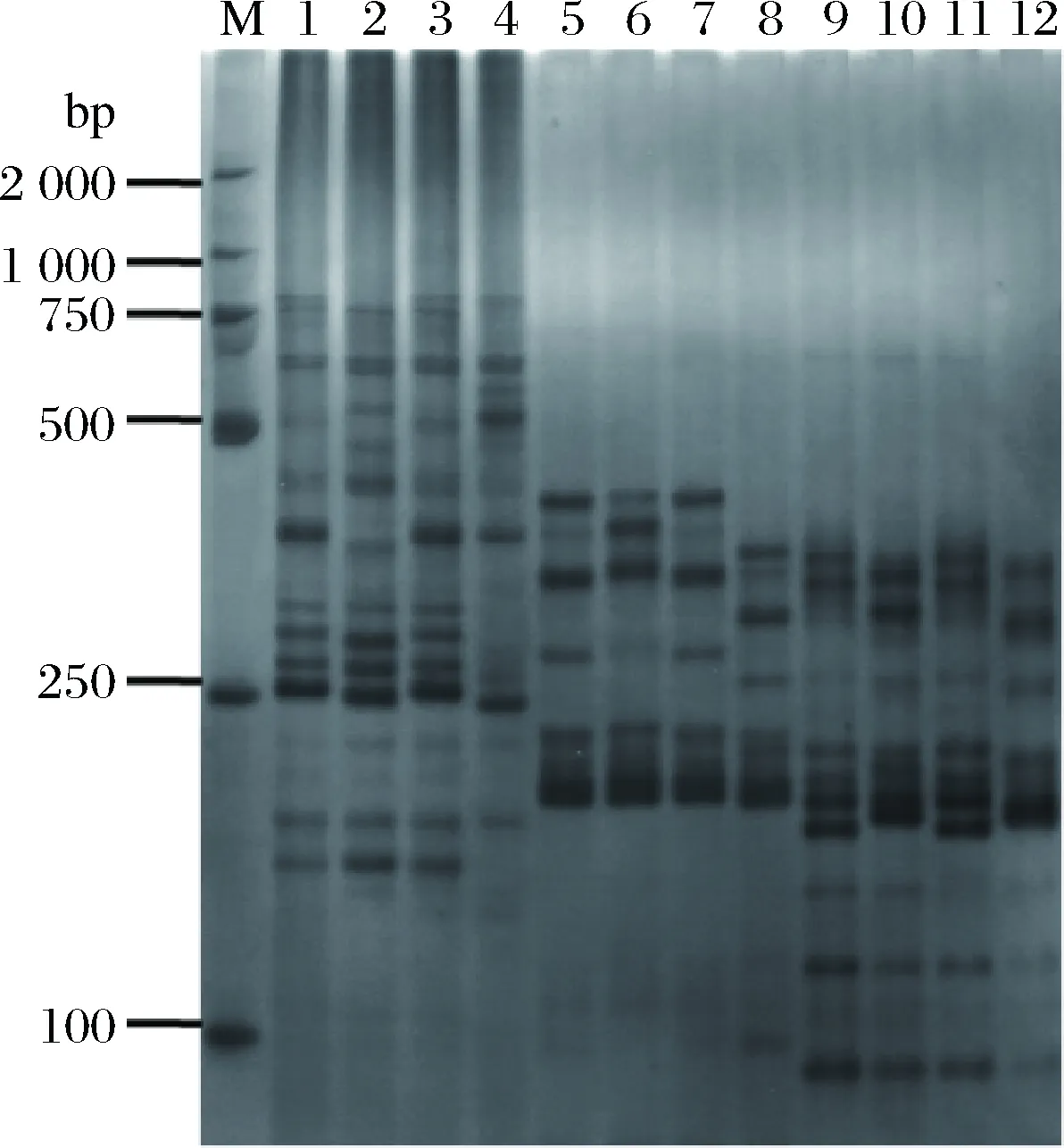
图5 4个酿酒葡萄品种SSR-PCR电泳结果Fig.5 Electrophoresis of PCR amplification products for4 Vitis vinifera varieties注:M-D2000 DNA marker;1~4表示是引物VrZAG79,样品依次为赤霞珠,霞多丽,美乐,玫瑰香;5~8表示是引物VrZAG62,样品依次为赤霞珠,霞多丽,美乐,玫瑰香;9~12表示是引物VVMD27,样品依次为赤霞珠,霞多丽,美乐,玫瑰香
3 讨论与结论
SSR技术是以PCR技术为核心发展起来的DNA分子标记技术,因此SSR 分子标记易受 PCR 反应体系中的主要成分以及成分间的互作影响,Mg2+、Taq酶、引物、dNTPs和模板DNA均有可能影响到扩增的敏感性、特异性以及产量,因此,SSR-PCR 反应体系的建立和优化是基于 SSR 标记研究生物遗传多样性的基础。
Mg2+是Taq酶的激活剂,而且还影响引物与模板的结合效率和产物的特异性,Mg2+浓度过高可降低PCR扩增片段的特异性,同时,非特异性片段产生的几率可能会增大;Mg2+浓度过低会影响PCR扩增产量甚至扩增不出条带。Taq酶浓度直接影响扩增的产量和条带的清晰度,其浓度偏高,易引起非特异性扩增,过低活性降低,降低扩增效率。本试验结果表明,Mg2+和 Taq酶对PCR扩增影响较大,随着浓度增加,PCR扩增效率增加,当Mg2+浓度达到2.0 mmol/L以上时,Taq酶浓度增加,扩增效果变化不明显。这一结果与陶巧静,刘遵春[9,11]等人的研究结果一致。dNTPs作为PCR反应的直接原材料,形成非特异性扩增,浓度过低,则会影响扩增效果;同时dNTPs还会对体系中的Mg2+产生拮抗作用,造成Mg2+浓度下降。因此本试验结果表明dNTPs最佳的添加量为0.1 mmol/L。这一结果与李亚慧,周海兰等人的研究结果一致[12-13]。引物的浓度同样会影响PCR扩增反应,浓度过高会促进非特异性扩增,增加引物二聚体的形成概率,浓度过低则影响扩增效果。DNA的纯度对体系扩增的稳定性有较大的影响,本试验的模板DNA纯度较高,有效提高了电泳效果,DNA浓度对SSR扩增体系影响较低,这一研究结果与潘珍珍,何仁峰等人的研究结果一致[14-15]。
本研究对影响新疆酿酒葡萄SSR-PCR反应的Mg2+、Taq酶、引物、dNTPs和模板DNA等主要成分浓度用量进行了单因素和正交优化试验,确定新疆酿酒葡萄 SSR扩增的25 μL最佳体系:Mg2+浓度2.5 mmol/L,dNTPs为0.15 mmol/L,Taq酶为1.5 U,引物为0.5 μmol/L,DNA含量为40 ng/25μL。
[1]郝宇,张淑静,张世红,等.葡萄品种资源的SSR鉴定及遗传多样性分析[J].河北农业大学学报,2010,33(1):54-59.
[2]HAMED D B,ABOLGHASEM M S,MASSIMO L,et al.Genetic characterization of some wild grape populations (Vitisviniferasubsp. sylvestris) of Zagros mountains (Iran) to indentify a conservation strategy[J].Plant Genetic Resources,2015,13(1):27-35.
[3]THOMAS M R,SCOTT N S.Microsatellite repeats in grapevine reveal DNA polymorphisms when analysed as sequence-tagged sites (STSs) [J].Theoretical & Applied Genetics, 1993, 86(8):985-990.
[4]郭印山,牛早柱,石广丽,等.基于SSR分子标记的葡萄品种遗传多样性分析[J].北方园艺,2016(7):89-92.
[5]张萌,练春兰,松木悠,等.基于SSR分子标记的安徽黄山地区野生刺葡萄的遗传多样性分析[J].江西农业学报,2012,24(6):9-12.
[6]成冰,张京芳,马正强,等.酿酒白葡萄品种的SSR分析与鉴定[J].西北林学院学报,2014,29(2):103-106.
[7]COSTE A,POSTOLACHE D, POPESCU F,et al.Authentication of valuable grapevine varieties from Romania through molecular markers [J].Romanian Biotechnological Letters,2010,15(1):3-10.
[8]齐玲倩,刘秀,丁梦璇,等.水果果肉中总DNA提取方法的比较研究[J].食品研究与开发,2016,37(5):55-58.
[9]陶巧静,曹斌,钱萍仙,等.葡萄SSR-PCR体系优化及其诱变单株遗传多样性分析[J].福建农林大学学报(自然版),2016, 45(2):146-155.
[10]段永红,渠云芳,王长彪,等.药用植物苦参SSR-PCR体系的优化与验证[J].中国农业大学学报,2014,19(5):95-100.
[11]刘遵春,苗卫东,刘大亮,等.新疆野苹果SSR-PCR反应体系的优化[J].广东农业科学,2012,39(4):97-100.
[12]李亚慧,黄丛林,董然.菊花SSR-PCR反应体系的建立和优化[J].北方园艺, 2012(13):127-131.
[13]周海兰,李绍鹏,李卫亮,等.油梨基因组DNA提取、SSR-PCR反应体系优化及引物筛选[J].生物技术通报,2016,32(4):143-150.
[14]潘珍珍,吴才君,刘文睿,等.冬瓜SSR-PCR体系优化及引物筛选[J].分子植物育种,2015,13(4):898-902.
[15]何仁锋,冯尚国,陈喆,等.药用菊花SSR-PCR反应体系优化及引物筛选[J].分子植物育种,2015,13(2):367-378.
