大豆蔗糖合成酶家族成员的全基因组鉴定及表达分析
2018-04-08晁毛妮张自阳王润豪张金宝黄中文
晁毛妮,张自阳,王润豪,张金宝,王 果,黄中文
(河南科技学院,现代生物育种河南省协同创新中心,河南新乡 453003)
蔗糖是植物叶片光合作用的产物,其本身不能被植物利用,需要蔗糖合成酶(Sucrose Synthase, SUS)分解后才能参与植物的各项代谢活动[1-3]。作为调节植物蔗糖代谢的关键酶,蔗糖合成酶的活性与棉花纤维的伸长[4]、水稻颖果的发育[5]、豆科植物氮的固定[6]以及植物对逆境胁迫的响应[7]等生命活动密切相关。鉴定和克隆植物的蔗糖合成酶基因对于提高其产量及抗逆性等具有重要意义。
研究表明,植物的蔗糖合成酶是一个多基因编码的家族,但是不同物种间SUS家族成员数量差别很大。玉米[8]和菜豆[9]的基因组中均鉴定到3个SUS基因;拟南芥[10]、水稻[11]和桃树[12]的基因组中均鉴定到6个SUS基因;陆地棉[13]、大麦[14]、杨树[15]和葡萄[16]的基因组中分别鉴定到15、4、7和5个SUS基因。尽管不同物种间SUS基因数量差别很大,蛋白序列却高度保守,均具有蔗糖合成结构域和糖基转移结构域[10,15]。在表达上,植物SUS家族成员的表达具有组织特异性。例如,在水稻SUS家族中,OsSUS3和OsSUS4在颖果中表达量最高,OsSUS2在所有组织中均有较高的表达,它们具有不同的组织表达模式[17];同样地,豌豆SUS家族成员PsSUS1、PsSUS2和PsSUS3分别在种子、叶片和花中大量表达[9]。除了具有组织特异性,植物SUS家族成员的表达还受发育时期和逆境胁迫的影响。例如,陆地棉[18]和海岛棉[19]纤维发育的不同阶段分别由不同的SUS基因调控;拟南芥SUS家族成员AtSUS1基因的表达受冷和干旱胁迫诱导[10]等。总之,植物SUS家族成员不同的表达调控模式,暗示着它们在植物生长发育过程中可能行驶着不同的功能。
大豆是重要的经济作物,提高大豆产量一直是研究的热点。研究表明,蔗糖合成酶对于光合产物的转运和积累有着重要影响,在作物产量形成的过程中起着重要作用[20-21]。例如,在棉花中过表达马铃薯SUS基因,可显著提高棉花的产量[21],表明SUS基因在提高作物产量方面具有很大潜力。目前,已从陆地棉[13]、玉米[8]和水稻[11]等多种作物中对SUS基因家族进行全基因组鉴定和功能研究,然而,大豆SUS基因家族的系统研究尚很少开展。本研究基于已公布的大豆基因组数据,对大豆SUS基因家族进行全基因组鉴定,并对其序列特征、进化关系及表达模式等进行了全面分析。研究结果为今后深入研究该家族基因的生物学功能及大豆产量的提高奠定理论基础。
1 材料和方法
1.1 大豆蔗糖合成酶基因家族的全基因组鉴定
于Phytozome网站(http://www.phytozome.net/)[22]下载大豆基因组数据,并建立本地Blast数据库。从拟南芥基因组数据库网站(http://www.arabidopsis.org/)下载拟南芥蔗糖合成酶基因家族(AtSUS1~AtSUS6)的蛋白序列,其基因ID号分别为At5g20830、At5g49190、At4g02280、At3g43190、At5g37180和At1g73370。以拟南芥SUS家族成员的蛋白序列作为查询序列,运行Blastp检索程序,筛选阈值设置为1e-10,初步获得大豆SUS基因家族的候选基因。接着将这些候选基因的蛋白序列提交到Pfam数据库[23]进行进一步的蔗糖合成结构域(PF00862)和糖基转移结构域(PF00534)验证,最终鉴定出大豆SUS家族成员。
1.2 蛋白特性和序列分析
利用Expasy在线网站(http://www.expasy.org)预测大豆SUS家族成员的分子量和等电点;蛋白结构域的预测使用InterPro和Pfam数据库在线完成;采用Bioedit7.0软件完成蛋白序列一致性分析;利用ClustalX软件默认设置进行多重序列比对,比对后的序列用GeneDoc进行编辑。
1.3 系统发育分析
在NCBI网站下载用于进化树构建的7个物种的蛋白序列;利用MEGA5.02软件中的邻接法(Neighbor-Joining method, NJ)构建系统进化树[24],其中校验参数bootstrap值设置为1 000次,距离模型设置为p-distance,空位缺失数据的处理设置为pairwise deletion。
1.4 基因结构示意图的绘制
从下载的大豆基因组数据中调取GmSUS基因的编码区(CDS)序列和基因组序列。通过GSDS(http://gsds.cbi.pku.edu.cn/index.php)在线工具绘制大豆SUS家族成员的内含子/外显子结构示意图。
1.5 染色体定位和基因复制分析
根据大豆基因组数据库(http://www.phytozome.net/)提供的每个基因在染色体上的位置信息,通过Maplnspect软件绘制该基因的染色体分布图。根据2个基因序列相互匹配部分的长度大于较长序列长度的80%,且相互匹配部分的相似性大于80%来判定基因复制事件;同时进一步结合基因在染色体上的位置来确定基因复制的类别是串联复制还是片段复制[25-26]。
1.6 基因表达特性分析
大豆SUS基因在根、茎、叶、花、荚、种子和根瘤组织中的表达数据下载自Phyzotome网站(http://www.phy tozome.net/)。利用MeV4.9[27]软件绘制基因表达的热图。
2 结果与分析
2.1 大豆SUS家族成员的鉴定
通过Blastp分析,在大豆基因组中共鉴定到12个蔗糖合成酶基因,根据它们在染色体上的位置,本研究将其命名为GmSUS1~GmSUS12(表1)。12个GmSUS的基因组大小和CDS长度分别在5 184(GmSUS5)~7 720 bp(GmSUS8)和2 409(GmSUS8)~2 766 bp(GmSUS4)之间变化。对其编码蛋白进行分析发现,GmSUS蛋白的氨基酸长度从802 aa(GmSUS8)到921 aa(GmSUS4)不等;分子量从91.58 kD(GmSUS8)到104.16 kD(GmSUS4)不等;等电点从6.03(GmSUS2)到7.46(GmSUS7)不等,其中等电点小于7的蛋白比例达66.67%,表明大豆SUS蛋白主要是以弱酸性蛋白质存在(表1)。
2.2 大豆SUS家族成员的蛋白序列分析
序列一致性分析表明,大豆SUS家族成员间蛋白序列一致性很高(表2),一致性在46.60%~98.80%之间,其中GmSUS11和GmSUS6一致性最高(98.80%),其次是GmSUS7和GmSUS1(97.50%),GmSUS3和GmSUS5一致性最低(46.60%)。通过InterPro和Pfam工具进行蛋白结构域分析发现(表1,图1),大豆GmSUS蛋白均具有植物SUS家族成员特有的蔗糖合成结构域(图1,实线)和糖基转移结构域(图1,虚线),它们分别行驶着蔗糖合成和糖基化合物转移的功能。另外,除GmSUS5外,大豆GmSUS蛋白N端均具有一个保守的丝氨酸(Ser)磷酸化位点(图1,箭头),该位点可被丝/苏蛋白激酶磷酸化[28-29]。
2.3 大豆SUS家族的系统进化分析
先前对植物SUS蛋白进行系统进化分析可知,植物SUS家族成员在进化上可聚为3组(SUS1、SUSA和New Group)[10-11],后续的一些研究将这3组又分别命名为SUSⅠ、SUSⅡ和SUSⅢ,其中SUSI又分为单子叶组(Monocot SUSI)和双子叶组(Dicot SUSI)[12,15,30]。为了了解大豆SUS家族基因在进化上的位置及亲缘关系,本研究构建了大豆和其他7个物种SUS家族成员的系统进化树(图2)。图2表明,大豆SUS家族基因可聚为3组,分别是SUSI的双子叶组(Dicot SUSⅠ)、SUSⅡ和SUSⅢ;其中4个大豆SUS基因(GmSUS9、GmSUS3、GmSUS11和GmSUS6)分布在SUSⅠ组;3个大豆SUS基因(GmSUS2、GmSUS8和GmSUS12)分布在SUSⅡ组,5个大豆SUS基因(GmSUS1、GmSUS4、GmSUS5、GmSUS7和GmSUS10)分布在SUSⅢ组。另外,由图2可知,双子叶和单子叶植物的SUS家族成员在SUSⅠ~SUSⅢ内均有分布,表明SUS家族基因的扩增可能发生在单子叶和双子叶植物分化之前。
2.4 大豆SUS家族成员的基因结构分析
已有研究表明,植物SUS基因家族的内含子数目和基因结构在不同物种间高度保守[31]。本研究对大豆的SUS家族成员的基因结构分析结果(图3)表明,12个GmSUS基因均含有内含子,内含子数目在11~14个之间。在SUSⅠ组中,除GmSUS9含有12个内含子外,其余GmSUS基因均含有11个内含子;SUSⅡ和SUSⅢ组的GmSUS基因均包含14个内含子。另外,位于同一组的GmSUS基因大部分具有相似的内含子/外显子分布模式(图3)。
2.5 大豆SUS家族成员的染色体分布
由图4可以看出,12个GmSUS基因不均匀地分布在大豆的10条染色体上,其中第Chr02、Chr03、Chr11、Chr13、Chr14、Chr16、Chr17和Chr19号染色体各含1个GmSUS基因;第Chr09和Chr15号染色体各含2个GmSUS基因;其余各染色体不含GmSUS基因。进一步基因复制分析发现,该家族成员无串联复制,存在11个片段复制(图4),表明片段复制可能导致了GmSUS基因在大豆基因组中的扩增。


箭头表示丝氨酸(Ser)磷酸化位点;实线和虚线分别表示蔗糖合成结构域和糖基转移结构域图1 大豆蔗糖合成酶基因家族氨基酸序列的多重比对The arrow indicates serine phosphorylation site. The solid and dotted lines indicate a sucrose synthase domain and a glycosyl transferase domain, respectivelyFig.1 Multi alignment of amino acid sequences of sucrose synthase gene family in soybean

大豆SUS蛋白用●标记;分支上的数值表示bootstrap验证中基于1 000次重复节点的可信度;拟南芥AtSUS1~SUS6(At5g20830,At5g49190,At4g02280,At3g43190,At5g37180和At1g73370);豌豆PsSUS1~SUS4(AJ012080,AJ001071,AJ311496和AF079851);蜜桔CuSUS1~SUS2,CuSUSA(AB022092,AB029401和AB022091);杨树PtrSUS1~SUS7(GU559729,GU559730,GU559731,GU559732,GU559733,GU559734和GU559735);水稻OsSUS1~SUS6(Os03g0401300,Os06g0194900,Os07g0616800,Os03g0340500,Os04g0309600和Os02g0831500);玉米ZmSUS1~SUS5(L29418,L22296,BT069288,NM_001111941和EU971052);大麦HvSUS1~SUS2(X69931和Y15802);GmSUS1~GmSUS12,本研究鉴定的12个大豆SUS蛋白图2 大豆SUS蛋白与其他植物SUS蛋白的系统进化分析SUS proteins in soybean was marked by a symbol ●. The numbers on the branches represent the reliability percent of bootstrap values based on 1 000 replication. Arabidopsis thaliana, AtSUS1-SUS6 (At5g20830, At5g49190, At4g02280, At3g43190, At5g37180, At1g73370); Pisum sativum, PsSUS1-SUS4 (AJ012080, AJ001071, AJ311496, AF079851); Citrus unshiu, CuSUS1-SUS2 and CuSUSA (AB022092, AB029401, AB022091); Populus trichocarpa, PtrSUS1-SUS7 (GU559729, GU559730, GU559731, GU559732, GU559733, GU559734, GU559735); Oryza sativa, OsSUS1-SUS6 (Os03g0401300, Os06g0194900, Os07g0616800, Os03g0340500, Os04g0309600, Os02g0831500); Zea mays, ZmSUS1-SUS5 (L29418, L22296, BT069288, NM_001111941, EU971052); Hordeum vulgare, HvSUS1-SUS2(X69931, Y15802); GmSUS1-GmSUS12, 12 soybean SUS proteins identified in this studyFig.2 Phylogenetic relationships between the soybean SUS proteins and other plant SUS proteins
2.6 大豆SUS家族成员的组织表达特性分析
组织表达特性分析表明,大豆SUS家族成员具有不同的组织表达模式(图5)。GmSUS8在大豆种子中表达量最高,其他组织表达量很低,表明其在大豆种子发育过程中可能起着重要作用。GmSUS1、GmSUS7和GmSUS5在大豆根瘤中表达量很高,其他组织表达量很低或者不表达,表明它们可能参与了大豆的根瘤固氮过程。GmSUS3、GmSUS11和GmSUS12在所有被检测的组织均具有较高的表达,表明它们在大豆生长发育的多个过程中都起着重要作用。对不同表达特性GmSUS基因的进化组分布情况进行分析发现,一些特异性表达的基因主要分布在SUSⅢ和SUSⅡ进化组,包括SUSⅢ的全部基因和SUSⅡ的1个基因;另外一些非特异性表达的基因主要分布在SUSⅠ和SUSⅡ进化组,包括SUSⅠ全部基因和SUSⅡ的2个基因(图5)。
3 讨 论
蔗糖是高等植物光合作用的主要产物,而蔗糖合成酶是促使蔗糖进入各种代谢途径的关键酶之一,在植物的生长发育过程中起着重要作用。鉴定和克隆植物的蔗糖合成酶基因是了解其生理功能和代谢机制的第一步。近年来,随着测序技术的发展,利用比较基因组学进行基因家族分析已成为基因功能研究的前提。基于已测序的基因组数据,目前已从玉米[8]、水稻[11]和陆地棉[13]等多种作物中对蔗糖合成酶基因家族进行了全基因组鉴定,并发现其在作物生长和产量形成过程中起着重要的作用[20-21]。然而,目前还未对大豆SUS基因家族进行系统鉴定和研究。本研究在大豆基因组共鉴定到12个SUS基因家族成员,其数量是拟南芥[10]和水稻[11]SUS家族基因数量的2倍。与此相符的是,在大豆漫长的进化过程中,曾经历了2次全基因组复制,分别发生在56.5~60.0 mya (百万年前)和10~13 mya(百万年前)[32],而拟南芥和水稻只经历了1次基因组复制[33-34]。因此,在大豆基因组中,约75%的基因有2个或2个以上的拷贝[35]。本研究中大豆SUS家族基因成员较多,可能与大豆经历的2次全基因组复制事件有关。
许多研究表明,SUS家族基因的蛋白结构及内含子/外显子特征在不同物种间高度保守[16,31,36-38]。在本研究中,大豆的SUS家族成员均具有蔗糖合成结构域和糖基转移结构域。除GmSUS5外,其他GmSUS蛋白N端均有一个保守的丝氨酸(Ser)磷酸化位点,这些蛋白特征与大多数植物SUS家族成员相似。已有研究表明,基因内含子/外显子特征可为基因进化关系的研究提供有价值的信息[39-40]。本研究对大豆内含子/外显子分布特征进行研究发现,大豆SUS基因家族的内含子数目在11~14个之间。同样地,杨树SUS家族的7个PtrSUS基因,海岛棉SUS家族的大部分GaSUS基因(除GaSUS5外),其内含子数目均在11~14个之间[15,31]。以上结果表明,不同物种间SUS基因家族的内含子数目是高度保守的。
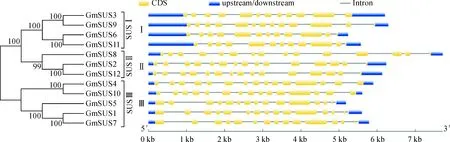
黄色表示外显子,黑线表示内含子,蓝色表示上下游序列;分支上的数值表示bootstrap验证中基于1 000次重复节点的可信度图3 大豆SUS家族基因的系统进化树和基因结构示意图Exons were represented in yellow, introns were represented by black lines, and upstream and downstream were represented in blue. The numbers on the branches represent the reliability percent of bootstrap values based on 1 000 replication.Fig.3 Phylogenetic relationship and gene structures of SUS family genes in soybean
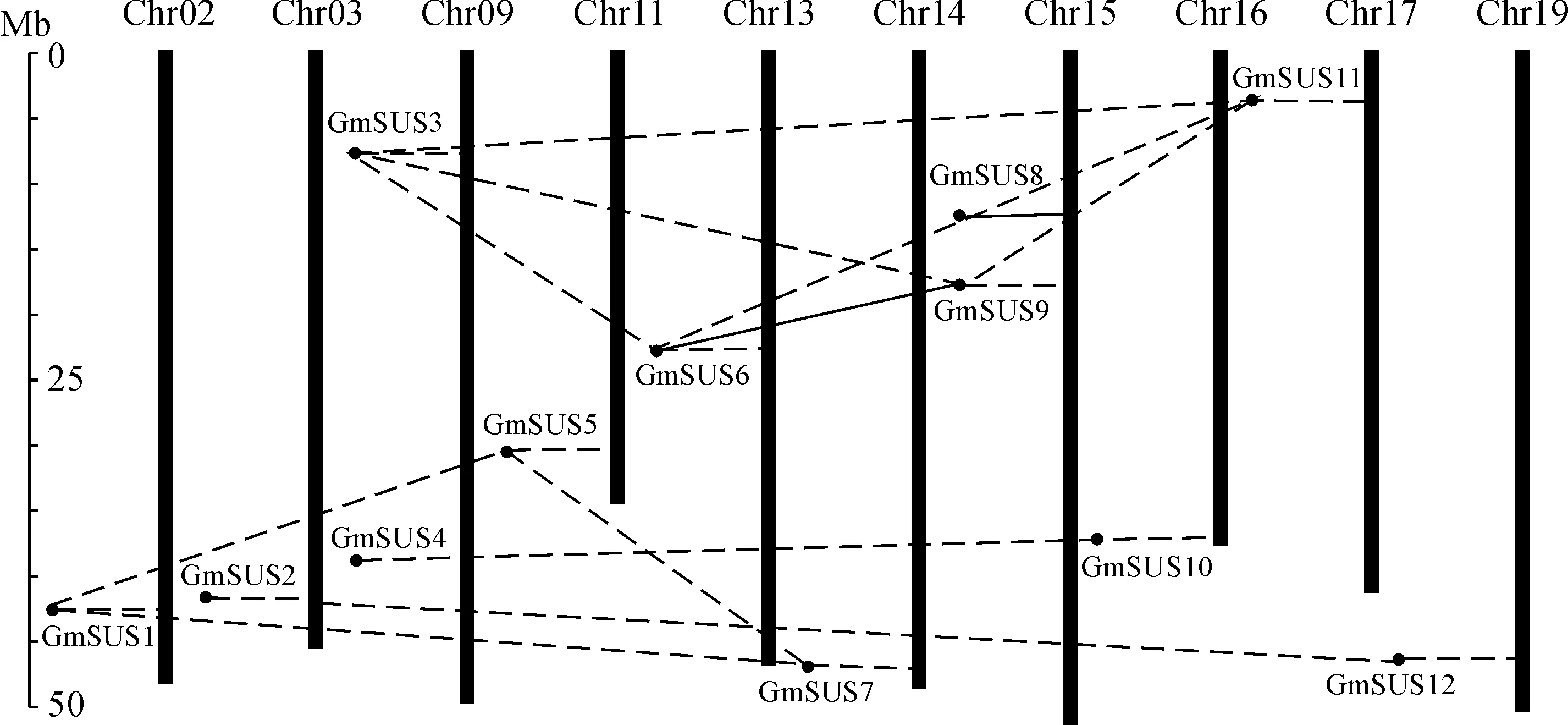
左边标尺的单位是Mb;染色体顶端的数字代表着染色体对应的编号;虚线代表片段复制图4 大豆SUS基因家族在染色体上的分布和基因复制分析The scale on the left is Mb; the chromosome numbers are indicated at the top of each chromosome; the segmental duplicated genes are connected with dotted linesFig.4 Chromosomal distribution and gene duplication analysis of SUS gene family in soybean
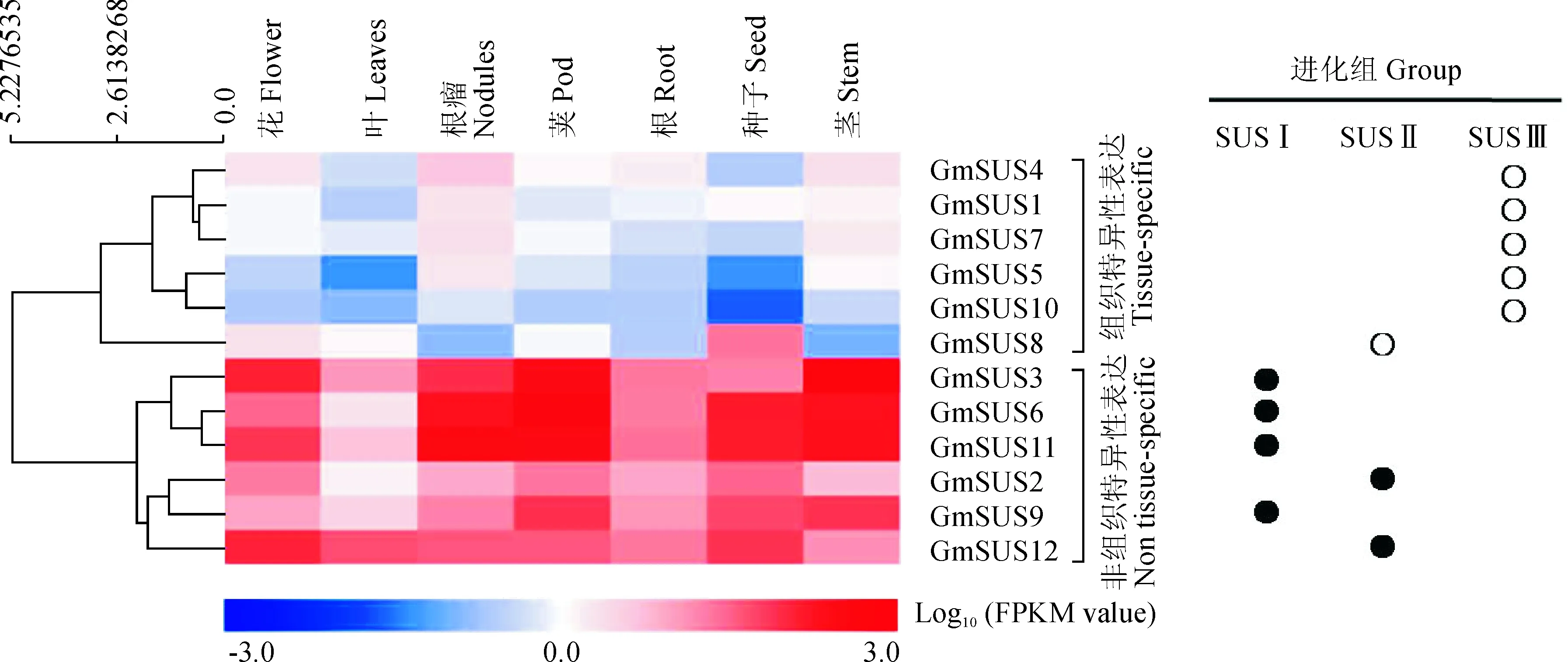
颜色条代表的是GmSUS基因在7个大豆组织中的相对表达水平,其值大小用Log10(FPKM value)表示图5 大豆SUS家族基因的组织表达分析及进化组分布Color bar represents the relative transcript level of GmSUS genes in seven soybean tissues, and its value was showed by Log10(FPKM value)Fig.5 Tissue expression analysis and group distribution of SUS family genes in soybean
对大豆SUS家族成员的组织表达模式进行研究发现,大豆GmSUS基因具有不同的组织表达模式,暗示着它们在大豆生长发育的过程中可能起着不同的功能。GmSUS8基因在种子中表达量最高,其他组织表达量很低,表明该基因在大豆种子发育过程中可能起着重要作用。SUS基因对种子发育的影响,在其他植物中也被观察到。例如,拟南芥的AtSUS2基因特异地在花后12 d的种子中高表达,其可作为种子成熟的标记[10]。在菜豆中,SUS基因表达受到抑制,会推迟胚的发育,使胚和胚柄的发育出现异常,最终导致种子不能正常发育[41];利用RNAi技术抑制棉花SUS基因的表达,可导致棉花种子早期发育受阻[42]。本研究中GmSUS8基因在大豆种子中特异地高表达,但是关于其如何影响大豆的种子发育目前还不清楚,还需通过基因敲除或过表达实验来进一步地研究。先前很多研究表明,豆科植物的根瘤主要依赖蔗糖代谢提供的碳骨架和能量[6],且固氮作用相关的蛋白也依赖于SUS的活性[43],因此,调控SUS的表达或改变SUS的活性可以影响豆科植物的固氮作用。本研究鉴定的GmSUS1、GmSUS7和GmSUS5基因在大豆根瘤中表达量很高,而在其他组织表达量很低或者不表达,表明它们在大豆固氮过程中可能起着重要作用,可作为大豆共生固氮调控分子机制研究的候选基因,进而为通过分子育种提高大豆固氮效率提供理论基础。有趣的是,这些组织特异性表达的基因主要分布在SUSⅢ和SUSⅡ进化组,包括SUSⅢ的全部基因和SUSⅡ的部分基因。另外,SUS家族的一些基因在被检测的各个组织均有较高表达,其表达不具有组织特异性,如大豆GmSUS3、GmSUS11和GmSUS12,这些基因主要分布在SUSⅠ和SUSⅡ进化组,包括SUSⅠ的全部基因和SUSⅡ的部分基因。本研究观察到的这种大豆SUS家族基因不同进化组表达模式或功能的差异,暗示着它们在进化过程中可能受到了选择作用。
大豆在生长发育过程中,会遇到各种逆境胁迫,如低温、干旱和盐胁迫等,它们会严重影响大豆的产量[44-46]。先前很多研究表明,SUS家族基因如拟南芥AtSUS1[10],大麦HvSUS1和HvSUS3[14],橡胶树HbSUS5[7]等在植物应对逆境胁迫过程中起着重要作用,但是关于大豆SUS基因在逆境条件下的表达特性及其对大豆生长发育的影响目前还不清楚。因此,在今后的研究中,有必要在逆境条件下进一步研究大豆SUS基因的表达特性,了解它们响应逆境胁迫的分子机制,以期为将来大豆抗逆育种提供重要的基因信息。
参考文献:
[1]LUTFIYYA L L, XU N, D'ORDINE R L,etal. Phylogenetic and expression analysis of sucrose phosphate synthase isozymes in plants[J].JournalofPlantPhysiology, 2007,164(7): 923-933.
[3]卢合全, 沈法富, 刘凌霄, 等. 植物蔗糖合成酶功能与分子生物学研究进展[J]. 中国农学通报, 2005,21(7): 34-37.
LU H Q, SHEN F F, LIU L X,etal. Recent advances in study on plant sucrose synthase[J].ChineseAgriculturalScienceBulletin, 2005,21(7): 34-37.
[4]BAI W Q, XIAO Y H, ZHAO J,etal. Gibberellin overproduction promotes sucrose synthase expression and secondary cell wall deposition in cotton fibers[J].PlosOne, 2014,9(5): e96537.
[5]YU W P. Complete structures of three rice sucrose synthase isogenes and differential regulation of their expressions[J].Bioscience,Biotechnology,andBiochemistry, 1996,60(2): 233-239.
[6]GORDON A J, MINCHIN F R, JAMES C L,etal. Sucrose synthase in legume nodules is essential for nitrogen fixation[J].PlantPhysiology, 1999,120(3): 867-878.
[7]XIAO X, TANG C, FANG Y,etal. Structure and expression profile of the sucrose synthase gene family in the rubber tree: indicative of roles in stress response and sucrose utilization in the laticifers[J].FebsJournal, 2014,281(1): 291-305.
[8]DUNCAN K A, HARDIN S C, HUBER S C. The three maize sucrose synthase isoforms differ in distribution, localization, and phosphorylation[J].Plant&CellPhysiology, 2006,47(7): 959-971.
[9]BARBER L, SMITH A M, WANG T L,etal. Multiple, distinct isoforms of sucrose synthase in pea[J].PlantPhysiology, 2001,127(2): 655-664.
[10]BAUD S, VAULTIER M N, ROCHAT C. Structure and expression profile of the sucrose synthase multigene family inArabidopsis[J].JournalofExperimentalBotany, 2004,55(396): 397-409.
[11]HIROSE T, SCOFIELD G N, TERAO T. An expression analysis profile for the entire sucrose synthase gene family in rice[J].PlantScience, 2008,174(5): 534-543.
[12]ZHANG C, YU M, MA R,etal. Structure, expression profile, and evolution of the sucrose synthase gene family in peach (Prunuspersica)[J].ActaPhysiologiaePlantarum, 2015,37(4): 1-15.
[13]ZOU C, LU C, SHANG H,etal. Genome-wide analysis of the Sus gene family in cotton[J].JournalofIntegrativePlantBiology, 2013,55(7): 643-653.
[14]BARRERO-SICILIA C, HERNANDO-AMADO S, GONZLEZ-MELENDI P,etal. Structure, expression profile and subcellular localisation of four different sucrose synthase genes from barley[J].Planta, 2011,234(2): 391-403.
[15]ZHANG D, XU B, YANG X,etal. The sucrose synthase gene family inPopulus: structure, expression, and evolution[J].TreeGenetics&Genomes, 2011,7(3): 443-456.
[16]ZHU X, WANG M, LI X,etal. Genome-Wide analysis of the sucrose synthase gene family in grape (Vitisvinifera): structure, evolution, and expression profiles[J].Genes, 2017,8(4),111; doi:10.3390/genes8040111.
[17]ISLAM M Z, HU X M, JIN L F,etal. Genome-Wide identification and expression profile analysis of citrus sucrose synthase genes: investigation of possible roles in the regulation of sugar accumulation[J].PloSOne, 2014,9(11): e113623.
[18]BRILL E, THOURNOUT M V, WHITE R G,etal. A novel isoform of sucrose synthase is targeted to the cell wall during secondary cell wall synthesis in cotton fiber[J].PlantPhysiology, 2011,157(1): 40-54.
[19]黄圣, 何鹏, 田莉莉, 等. 棉花蔗糖合成酶基因家族在纤维发育期时空表达模式分析[J]. 棉花学报, 2015,27(4): 317-328.
HUANG S, HE P, TIAN L L,etal. Expression profiles of the cotton Sus gene family during fiber development[J].CottonScience, 2015,27(4): 317-328.
[21]XU S M, BRILL E, LLEWELLYN D J,etal. Overexpression of a potato sucrose synthase gene in cotton accelerates leaf expansion, reduces seed abortion, and enhances fiber production[J].MolecularPlant, 2012,5(2): 430-441.
[22]GOODSTEIN D M, SHU S, HOWSON R,etal. Phytozome: a comparative platform for green plant genomics[J].NucleicAcidsResearch, 2011,40(D1): D1 178-D1 186.
[23]SONNHAMMER E L, EDDY S R, DURBIN R. Pfam: a comprehensive database of protein domain families based on seed alignments[J].Proteins-structureFunction&Bioinformatics, 1997,28(3): 405-420.
[24]KUMAR S, NEI M, DUDLEY J,etal. MEGA: a biologist-centric software for evolutionary analysis of DNA and protein sequences[J].BriefingsinBioinformatics, 2008,9(4): 299-306.
[25]YANG S, ZHANG X, YUE J X,etal. Recent duplications dominate NBS-encoding gene expansion in two woody species[J].MolecularGeneticsandGenomics, 2008,280(3): 187-198.
[26]JIANG H, WU Q, JIN J,etal. Genome-wide identification and expression profiling of ankyrin-repeat gene family in maize[J]. DevelopmentGenesandEvolution. 2013,223(5): 303-318.
[27]SAEED A I, BHAGABATI N K, BRAISTED J C,etal. TM4 microarray software suite[J].MethodsinEnzymology, 2006,411(2): 134-193.
[28]HUBER S C, HUBER J L, LIAO P C,etal. Phosphorylation of serine-15 of maize leaf sucrose synthase. Occurrence in vivo and possible regulatory significance[J].PlantPhysiology, 1996,112(2): 793-802.
[29]HARDIN S C, HUBER S C. Proteasome activity and the post-translational control of sucrose synthase stability in maize leaves[J].PlantPhysiology&Biochemistry, 2004,42(3): 197-208.
[30]ZHANG J, JIE A, CHEN Y,etal. Haplotype analysis of sucrose synthase gene family in threeSaccharumspecies[J].BMCGenomics, 2013,14: 314.
[31]CHEN A, HE S, LI F,etal. Analyses of the sucrose synthase gene family in cotton: structure, phylogeny and expression patterns[J].BMCPlantBiology, 2012,12: 85.
[32]KIM K D, El B M, Abernathy B,etal. A comparative epigenomic analysis of polyploidy-derived genes in soybean and common bean[J].PlantPhysiology, 2015,168(4): 1433-1447.
[33]BLANC G, BARAKAT A, GUYOT R,etal. Extensive duplication and reshuffling in theArabidopsisgenome[J].PlantCell, 2000,12(7): 1 093-1 101.
[34]WANG X, SHI X, HAO B,etal. Duplication and DNA segmental loss in the rice genome: implications for diploidization[J].NewPhytologist, 2005,165(3): 937-946.
[35]SCHMUTZ J, CANNON S B, SCHLUETER J,etal. Genome sequence of the palaeopolyploid soybean[J].Nature, 2010,463(7278): 178-183.
[36]HAAGENSON D M, KLOTZ K L, MCGRATH J M. Sugarbeet sucrose synthase genes differ in organ-specific and developmental expression[J].JournalofPlantPhysiology, 2006,163(1): 102-106.
[37]TANG H, BOWERS J E, WANG X,etal. Synteny and collinearity in plant genomes[J].Science, 2008,320(5 875): 486-488.
[38]LI F, HAO C, YAN L,etal. Gene structure, phylogeny and expression profile of the sucrose synthase gene family in cacao (TheobromacacaoL.)[J].JournalofGenetics, 2015,94(3): 461-472.
[39]HU L, LIU S. Genome-wide identification and phylogenetic analysis of the ERF gene family in cucumbers[J].Genetics&MolecularBiology, 2011,34(4): 624-633.
[40]LECHARNY A, BOUDET N, GY I,etal. Introns in, introns out in plant gene families: a genomic approach of the dynamics of gene structure[J].JournalofStructural&FunctionalGenomics, 2003,3(1-4): 111-116.
[41]GHASSEN A, YORDAN M, JEAN-MARIE J,etal. Characterization and expression profile analysis of a sucrose synthase gene from common bean (PhaseolusvulgarisL.) during seed development[J].MolecularBiologyReports, 2012,39(2): 1 133-1 143.
[42]YONGLING R, DANNYJ L, LIU Q,etal. Expression of sucrose synthase in the developing endosperm is essential for early seed development in cotton[J].FunctionalPlantBiology, 2008,35(5): 382-393.
[43]CRAIG J, BARRATT P, TATGE H,etal. Mutations at the rug4 locus alter the carbon and nitrogen metabolism of pea plants through an effect on sucrose synthase[J].PlantJournal, 1999,17(4): 353-362.
[44]PHANG T H, SHAO G, LAM H M. Salt tolerance mechanisms in soybean[J].JournalofIntegrativePlantBiology, 2008,50(10): 1 196-1 212.
[45]MANAVALAN L P, GUTTIKONDA S K, PHAN TRAN L S,etal. Physiological and molecular approaches to improve drought resistance in soybean[J].Plant&CellPhysiology, 2009,50(7): 1 260-1 276.
[46]王萍, 宋海星, 马淑英, 等.花期低温对大豆荚和籽粒形成的影响[J]. 中国油料作物学报, 2000,22(2): 31-33.
WANG P, SONG H X, MA S Y,etal. Effects of low temperature at blooming on podding and seed filling in soybean varieties[J].ChineseJournalofOilCropSciences, 2000,22(2): 31-33.
