miRNAs参与养血柔肝法治疗骨关节炎的生物信息学分析
2018-03-20高宁阳熊轶喆石印玉
丁 立,高宁阳,庞 坚,熊轶喆,石印玉
(上海中医药大学附属曙光医院 骨伤科,上海 200000)
骨关节炎(osteoarthritis,OA)是关节炎中最多发的一种,主要累及膝、髋等大关节,以关节软骨的进行性破坏为主要病理特征,是导致中老年人慢性肌肉、骨骼疼痛和功能活动障碍的最主要的原因[1]。据报道,世界上总人口的19%具有影像学膝骨关节炎证据[2]。由于骨关节炎的复杂性和难治性,其已成为现今骨伤科学所面对的重要医学难题和研究热点。微小核糖核酸(microRNAs,miRNAs)具有调节软骨发育及维持软骨代谢平衡的作用,在骨关节炎发病机制中扮演着重要的角色。上海中医药大学附属曙光医院石印玉教授研制的养血柔肝法中成药“养血软坚胶囊”已经被证实了具有提高痛阈、抑制炎性反应、保护软骨等作用,临床研究也发现本药对缓解OA病人疼痛、改善相关症状具有良好效果[3-6]。本研究探求miRNAs生物学功能与软骨代谢的关系,并观察养血柔肝法“养血软坚胶囊”作用后的miRNAs表达水平的变化。
1 材料和方法
1.1 实验材料
1.1.1 实验动物及取材
10周龄健康Sprague-Dawley(SD)大鼠40只,体重200~250 g,雌雄各半。饲养实验室标准为SPF(specific pathogen free,无特定病原体)级实验室。大鼠每4只1笼,保证充分活动空间,自由饮水、进食。
对大鼠进行前交叉切除造模(ACLT)。大鼠造模术后饲养4周,经病理切片、Safranin-O染色、Mankin评分验证造模成功后。随机将40只SD大鼠分为两组:模型组(n=20,造模,不干预)、中药组(n=20,造模,药物干预),中药组按大鼠与人体等效剂量折算的养血软坚胶囊(沪药制字Z04100932)溶液进行灌胃给药,每周6 天(周一至周六),每天1次。灌胃4周后,两组大鼠同时采用 LCM技术捕获术侧膝关节软骨细胞。
1.1.2 主要实验仪器
测序仪(公司:Illumina,型号:Hiseq2500),荧光定量PCR仪(公司:ABI,型号:ABI7500)。
1.1.3 主要实验试剂
养血软坚胶囊(厂家:上海中医药大学附属曙光医院,货号:Z04100932),TruSeqTMSmall RNA Sample Prep Kits(公司:Illumina),RNA提取试剂盒(公司:TAKARA,货号:9109),RNA逆转录试剂盒(公司:TAKARA,货号:RR036A),SYBR GREEN kit(公司:ABI,货号1504490)。
1.2 方 法
1.2.1 RNA提取
捕获后的软骨细胞匀浆,加1 mL Trizol混匀,静置15 min,使其充分裂解,然后转入1.5 mL离心管。每1 mL加入200 μL氯仿,加入氯仿,振荡15 s后,室温放置 15 min。4 ℃ 14000 r/min 离心 15 min。吸取上层透明水相,至另一离心管中。加入0.5 mL异丙醇充分混匀,室温静置15 min。 4 ℃ 14000 r/min离心10 min,弃去上清液,RNA 沉于管底。加入75%乙醇洗涤RNA,温和振荡离心管,悬浮沉淀。4 ℃ 9500 r/min 离心 5 min,小心吸弃上清。室温晾干。用 20 mL 1‰DEPC水溶解RNA样品,室温静置30 min。
1.2.2 制备文库及测序实验
提取总RNA后,选取模型组和中药组每组各3个样品,利用miRNAs 5’端为磷酸基团,3’端为羟基基团的特殊属性。首先利用T4 RNA Ligase连接酶2(Truncated)将1个腺苷化单链DNA的 3’接头和5’接头分别连接到small RNA之上,其中3’端接头的5’端为rAPP,同时3’端有氨基起保护作用,故而能够在很大程度上减少miRNAs的自连。5’端接头可以特异性地捕获5’端为磷酸基团的small RNA序列。通过与其3’端互补的RT引物反转录带有3’端与5’端链接接头的small RNA 序列,然后使用PCR扩增所得的cDNA序列。通过6%polyacrylamide Tris-borate-EDTA 胶回收长度140 bp 碱基的 PCR 产物。
参照说明书,利用Illumina Hiseq2500测序仪对已构建好的miRNAs测序文库进行测序,测序读长设为单端 1×50 bp。
1.2.3 数据处理
使用FASTX工具包(http://hannonlab.cshl.edu/fastx_toolkit)对数据进行预处理,去掉3’端接头序列,再去掉两端测序分值<20的碱基序列,过滤掉已经去掉30%或以上的测序序列。过滤的测序序列用Bowtie工具(Bowtie2 version 2.1.0 http://sourceforge.net/projects/bowtie-bio/files/bowtie2) 比对到参考基因组上生成sam文件,只允许每个reads有1~8个碱基的错配。过滤sam文件只保留比对分值>60的序列,去掉比对分值低的序列。
从miRbase下载最新的人类microRNA序列和位置信息。然后通过perl程序统计每个miRNA比对的序列个数。再统计每个样本总的个数计算每个miRNA的RPKM的值,得到miRNA表达值矩阵。通过bioconducter 里的Deseq软件包(Version 1.24.0 http://www.bioconductor.org/packages/release/bioc/html/DESeq.html )对6个样本的RPKM值进行标准化和利用鉴定差异表达的miRNA。选择阈值为logFC>1,且P<0.01。miRanda[7]鉴定差异表达的miRNA的靶基因,只挑选align_score> 150。利用DAVID工具对差异表达的靶基因进行GO的富集分析和pathway富集分析,选择P<0.05作为显著性富集的阈值。利用string v10[8]数据鉴定蛋白的相互作用,选择combine score值>0.9。在获得这些作用网络后,使用Cytoscape(v 3.1.0)[9]对他们进行可视化和生成图片,构建网络图。
1.2.4 miRNA定量验证
在冰上配置反转录体系的反应液(2×miRNA RT Reaction Buffer 10 μL,miRNA RT Enzyme Mix 2 μL,RNase-Free ddH2O 补至20 μL)。移液器轻轻混匀上述配制的反应液进行逆转录反应(42 ℃ for 60 min,95 ℃ for 3 min)。利用相应引物进行荧光定量PCR实验(SYBR Premix Ex Taq (2×) 10 μL,For ward primer 1 μL,Reverse primer 1 μL,cDNA 8 μL)。反应条件为:变形:95 ℃ 3 min,退火:95 ℃ 10 s,延伸:60 ℃ 30 s,共40个循环。以U6为内参,miRNA名称及序列见表1。
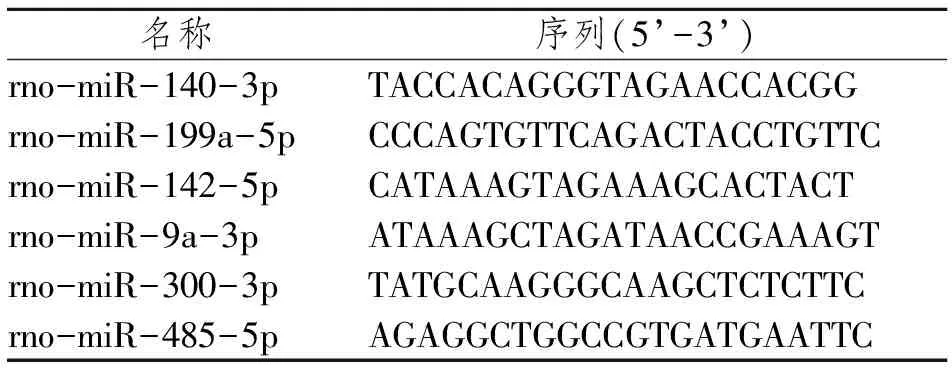
表1 miRNA名称及序列
1.3 统计学方法
所有结果以均值±SEM值的形式呈现,并制成表格。数据所用的统计分析方法为t检验,数据分析使用的软件为Graphpad prism (Graphpad Software, San Diego,CA),P<0.05为显著性差异的筛选标准。
2 结 果
2.1 差异表达的miRNA的鉴定
差异表达miRNA分析结果:7个上调,28个下调,对所有的差异表达miRNA做聚类热图。见图1。
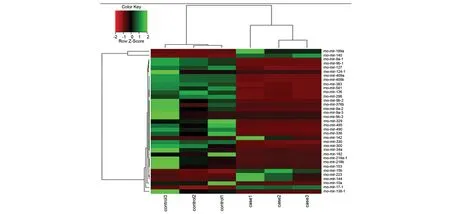
control-模型组,case-中药组,列代表不同的样品,行代表不同的miRNA,通过颜色变化展示miRNA在不同实验条件下的表达变化,颜色由红色-黑色-绿色变化,其中红色代表低表达,绿色代表高表达,差异表达miRNA在不同实验条件下表达变化相同或相似的miRNA聚集成类图1 差异表达miRNA的聚类热图Fig 1 Clustering image of the miRNA different expression
2.2 差异基因的靶基因鉴定
通过miRanda工具计算差异表达miRNA的靶基因。其中7个上调miRNA共调控了1174个靶基因,28个下调miRNA共调控了2216个靶基因。上调的靶基因多在离子、蛋白转运,维持内稳态,细胞增殖等方面起作用。见表2。富集比较显著的通路包括长期衰减、趋化因子信号通路等。见图2。下调的miRNA靶基因多在离子结合、细胞运输等方面起作用。见表3。下调的miRNA靶基因最显著的信号通路包括血管平滑肌收缩,过氧化物酶体增生物激活受体信号通路,神经活性配体受体相互作用等。见图3。

表2 上调差异miRNA调控的靶基因的GO BP富集分析(TOP15)
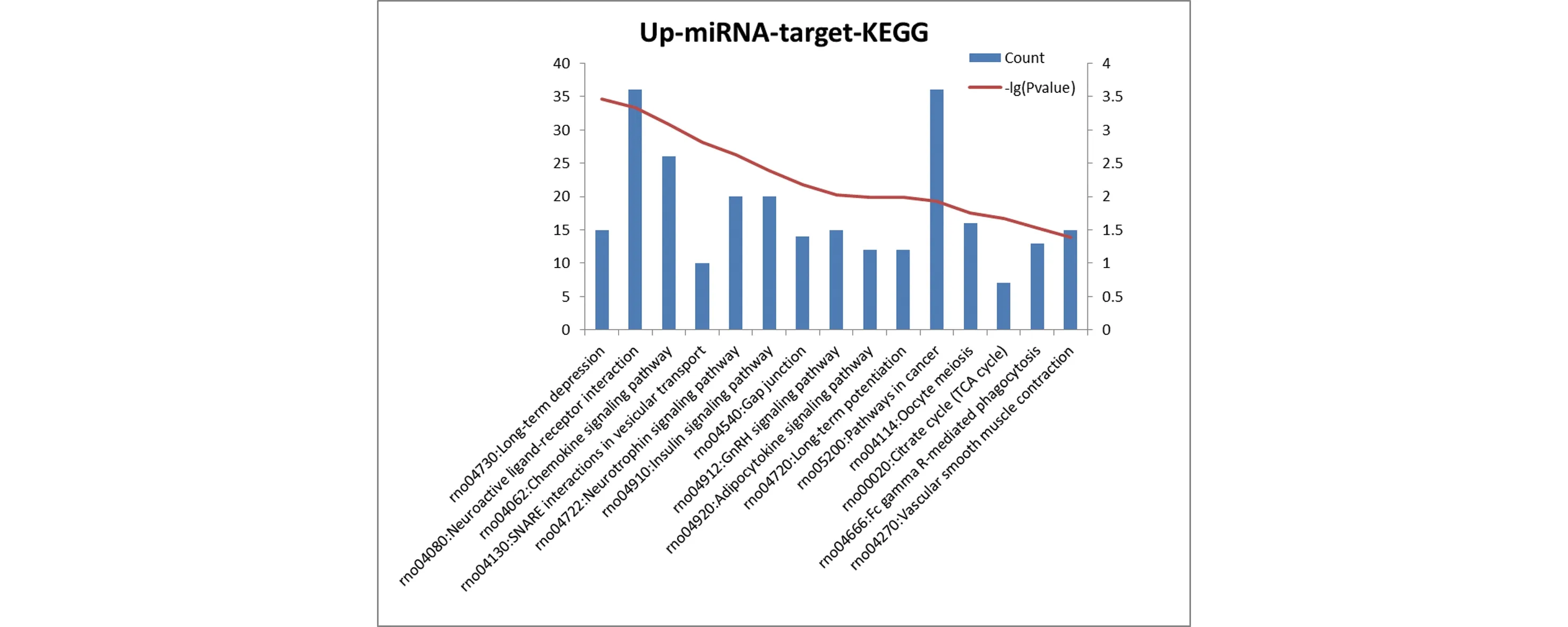
横坐标为KEGG数据库中KEGG通路的ID及描述,左侧纵坐标为富集到该KEGG条目的靶基因的数目,右侧纵坐标为-lg(Pvalue),P表示该通路中的富集水平的显著程度,P的取值范围为[0,1],越接近于0,表示富集越显著图2 上调miRNA的靶基因KEGG pathway 富集分析结果图Fig 2 KEGG pathway enrichment analysis of the differentially regulated miRNA with up regulation

GO号注释下调靶基因数量P0006811ionbinding4925.15E-110046872metalionbinding4771.32E-100043169cationbinding4821.67E-100015293symporteractivity422.16E-100031420alkalimetalionbinding571.47E-090042802identicalproteinbinding1252.35E-080015294solute:cationsymporteractivity312.99E-080046983proteindimerizationactivity1138.45E-080005275aminetransmembranetransporteractivity262.96E-060019899enzymebinding1005.76E-060046982proteinheterodimerizationactivity556.88E-060019001guanylnucleotidebinding727.85E-060032561guanylribonucleotidebinding727.85E-060008509aniontransmembranetransporteractivity358.74E-060015179L-aminoacidtransmembranetransporteractivity162.00E-05
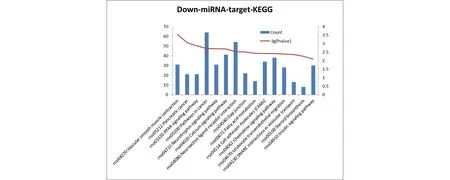
横坐标为KEGG数据库中KEGG通路的ID及描述,左侧纵坐标为富集到该KEGG条目的靶基因的数目,右侧纵坐标为-lg(Pvalue),P表示该通路中的富集水平的显著程度,P的取值范围为[0,1],越接近于0,表示富集越显著图3 下调miRNA的靶基因KEGG pathway富集分析结果Fig 3 KEGG pathway enrichment analysis of the differentially regulated miRNA with down regulation
2.3 PPI网络分析
上调miRNA调控靶基因有Adcy5、Gnai3、Lpar1等。见图4,表4。下调miRNA调控靶基因有Gnb1、Adcy3、Adcy5等。见图5,表4。这些靶基因多为趋化因子信号通路、黑素生成等方面起作用。见表5。

每个圆形节点代表一个靶基因,节点之间连接线表示两靶基因之间存在相互作用关系,靶基因节点上连接新越多,表示该靶基因节点连通度越高,越可能是hub基因图4 上调miRNA调控靶基因的PPI网络图Fig 4 PPI network map of the target genes of the miRNA with up regulation expression

上调miRNA靶基因连接度下调miRNA靶基因连接度Adcy536Gnb169Gnai329Adcy356Lpar155Adcy555Cxcr323Adcy655Cxcl1223Pomc47Ccr123Lpar343Ccr722Lpar143Agtr222Ccl943Cxcl1121Ccl643Ccr921Casr43

每个圆形节点代表一个靶基因,节点之间连接线表示两靶基因之间存在相互作用关系,靶基因节点上连接新越多,表示该靶基因节点连通度越高,越可能是hub基因图5 下调miRNA的靶基因PPI网络图Fig 5 PPI network map of the target genes of the miRNA with down regulation expression
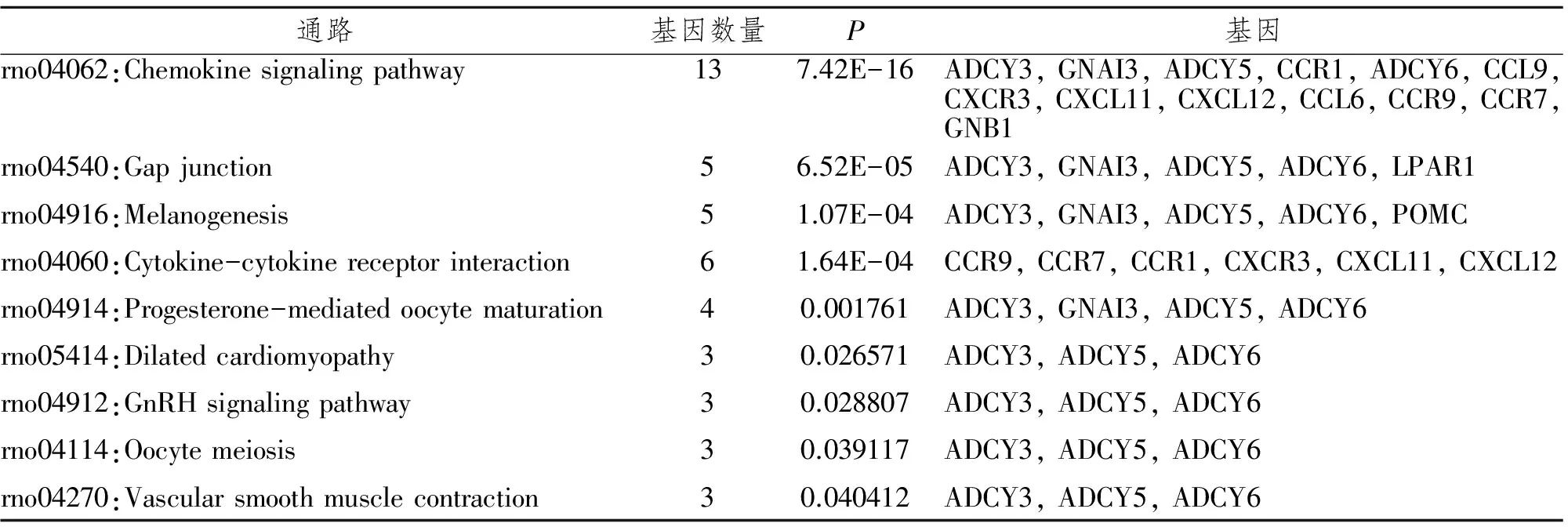
通路基因数量P基因rno04062:Chemokinesignalingpathway137.42E-16ADCY3,GNAI3,ADCY5,CCR1,ADCY6,CCL9,CXCR3,CXCL11,CXCL12,CCL6,CCR9,CCR7,GNB1rno04540:Gapjunction56.52E-05ADCY3,GNAI3,ADCY5,ADCY6,LPAR1rno04916:Melanogenesis51.07E-04ADCY3,GNAI3,ADCY5,ADCY6,POMCrno04060:Cytokine-cytokinereceptorinteraction61.64E-04CCR9,CCR7,CCR1,CXCR3,CXCL11,CXCL12rno04914:Progesterone-mediatedoocytematuration40.001761ADCY3,GNAI3,ADCY5,ADCY6rno05414:Dilatedcardiomyopathy30.026571ADCY3,ADCY5,ADCY6rno04912:GnRHsignalingpathway30.028807ADCY3,ADCY5,ADCY6rno04114:Oocytemeiosis30.039117ADCY3,ADCY5,ADCY6rno04270:Vascularsmoothmusclecontraction30.040412ADCY3,ADCY5,ADCY6
2.4 重要的miRNA调控靶基因网络图
一些重要的miRNA调控了比较多的hub 基因,例如miR-140 ,miR-9, miR-15b, miR-223等。见图6。

橙色圆形节点为靶基因,蓝色菱形节点为下调miRNA,红色菱形为上调miRNA,菱形节点和圆形节点之间的连接线表示两节点之间存在调控关系图6 重要miRNA调控靶基因网络图Fig 6 Important miRNA regulatory target gene network map
2.5 RT-PCR验证关节软骨中miRNAs的表达
关节软骨中,中药组rno-miR-199a-5p、rno-miR-140-3p的表达高于模型组,中药组rno-miR-300-3p、rno-miR-9a-3p的表达低于模型组,差异具有统计学意义(P<0.05)。而中药组rno-miR-142-5p、rno-miR-485-5p表达与模型组比较,差异无统计学意义。见图7。

Model为模型组,Treatment为中药组,*与模型组相比较, P<0.05图7 关节软骨中miRNAs的表达差异Fig 7 Expression levels of miRNA
3 讨 论
3.1 养血柔肝法
上海中医药大学附属曙光医院骨伤科石印玉教授根据骨关节炎经典理论,结合自身多年临床经验,创立了养血柔肝法治疗骨关节炎,疗效甚佳。已研制了中成药——“养血软坚胶囊”。养血软坚胶囊主要由白芍、秦艽、牡蛎等中药组成,前期试验已经证实了养血软坚胶囊可以提高C57黑鼠的痛阈,并具有较强的抑炎作用,其抑炎率分别为66.7%和75%[3];具有上调软骨蛋白多糖核心蛋白基因表达的作用,但不会引起软骨细胞异常分泌MMP-3[4-6]。临床研究也发现本药对缓解OA病人疼痛、改善相关症状具有良好效果。
3.2 相关miRNAs与骨关节炎和养血柔肝法的关系
近期,随着对关节软骨代谢分子机制的不断深入了解,已有较多研究认为:miRNAs在骨关节炎病理过程中对调节软骨发育及维持软骨代谢平衡具有重要作用,是治疗骨关节炎的新靶点[10]。本课题通过观察养血柔肝法治疗的骨关节炎大鼠模型,发现rno-miR-199a-5p、rno-miR-140-3p、rno-miR-300-3p、rno-miR-9a-3p等的表达受到调控。
3.2.1 miR-140
本研究通过测序技术以及qRT-PCR发现,在养血柔肝法治疗的大鼠骨关节炎中药组中,软骨里的rno-miR-140-3p呈高表达状态,且与模型组有明显差异。并上调了Ccr1、Ccr7、Agtr2等因子。
作为第一个被报道在斑马鱼、鼠类等动物的关节软骨中特异性高表达的miRNA[11-12],miR-140在软骨生长和稳定过程中,起到一系列的作用。同时,人体研究表明,在骨关节炎患者关节软骨中的miR-140表达较正常人关节软骨中的表达明显降低,其过度表达能够抑制小鼠骨关节炎的进展。这与本课题所做的大鼠动物实验结果相符。Miyaki等[13]研究发现,miR-140基因敲除的小鼠会出现软骨内成骨生长缺陷,小鼠四肢短小伴有颅面骨骼畸形,随着年龄的增长,关节软骨也会出现骨关节炎样改变,说明miR-140参与到了软骨生长发育的过程当中。Nakamura等[14]在miR-140基因敲除小鼠中发现,小鼠软骨内成骨生长发育速度加快,并判断这是由于miR-140的缺失致使小鼠的软骨细胞加速向肥大软骨细胞分化而导致的。以上实验均说明了miR-140的表达与软骨细胞分化相关,且IL-1β可以下调miR-140的表达以致在骨关节炎软骨中促进骨关节炎基因的异常表达。Karlsen等[15]通过抑制miR-140的表达,发现会导致软骨生成调控因子Sox9和aggrecan蛋白表达水平的下调,且这种下调不会引起Sox9和ACAN mRNA的改变。这些实验均证实了miR-140可以和Sox9等软骨生成调控因子互相影响,但是具体的调控机制需进一步研究。miR-140被Sox9激活后可以调节Sp1并与Wwp2-c共表达,进而在软骨退变和骨关节炎的发病机制上具有重要作用。金属基质蛋白酶13(matrix metalloproteinase,MMP13)、含血小板结合蛋白的基序的聚蛋白样金属蛋白酶5(a disintegrin and metalloproteinase with thrombospondin motifs,ADAMTS5)、胰岛素样生长因子结合蛋白 5(IGFBP5)都是参与关节软骨代谢的重要因子,miR-140可以靶向地对它们调控,从而维持关节软骨的稳定[16]。目前研究已经表明,miR-140可以通过调控TGF-β信号通路、BMP信号通路、Wnt/β-catenin信号通路等途径对软骨细胞的生长进行调控[17]。
3.2.2 miR-199a
从本研究结果可以看到,与模型组比较在大鼠骨关节炎模型中药组关节软骨样本里,rno-miR-199a-5p表达上调,且有统计学差异。
研究发现,在兔骨关节炎模型的软骨下骨中,miR-199-5p的表达量明显处于低水平,而软骨代谢相关因子MMP-3和IL-1β的表达量均明显升高[18]。在骨关节炎软骨细胞中作为COX-2的调节因子,miR-199a与p38-MAPK的激活呈负相关,并在软骨细胞的代谢中具有重要的调节作用,可能是骨关节炎新的治疗靶点。同时,miR-199a 是骨形态发生蛋白(BMP2)的响应microRNA,其可通过直接靶点SMAD1蛋白负向调节早期软骨细胞分化的过程[19]。以往的研究也表明,miR-193b和miR-199a-5p与软骨细胞的衰老相关。
3.2.3 miR-300
本研究中,与模型组比较养血柔肝法治疗的大鼠骨关节炎中药组中关节软骨的rno-miR-300-3p表达下调,差异有统计学意义。
根据本课题测序实验,miR-300-3p可以靶向调节趋化因子配体6[Chemokine (C-C motif) ligand 6,Ccl6]。作为CC趋化因子,Ccl6会在炎症和重构疾病中大量产生。这就解释了大鼠骨关节炎模型组的rno-miR-300-3p表达上调的原因,而养血柔肝法治疗的大鼠骨关节炎中药组中,rno-miR-300-3p在关节软骨中表达低于模型组,这可能也体现了养血软坚胶囊具有抑制炎症的功效。
3.2.4 miR-9a
通过本次课题软骨中miRNAs表达结果可以看出,养血柔肝法治疗的大鼠骨关节炎中药组在关节软骨中的rno-miR-9a-3p的表达低于大鼠骨关节炎模型组,差异具有统计学意义。
在IL-1β和IL-6诱导的骨关节炎软骨细胞中发现,miR-9可以抑制单核细胞趋化蛋白诱导蛋白-1(MCPIP1)在软骨细胞中的表达。MCPIP1是一类具有免疫调节作用的CCCH型锌指家族分子,可被多种炎性因子激活,可通过下调IL-6等炎症因子表达,从而抑制炎症过程。同时,MCPIP1也可负向调控NF-κB信号通路的活化[20]。NF-κB信号通路的激活会导致进展性的细胞外基质损伤和关节软骨降解。故而中药组rno-miR-9a-3p相对于模型组的低表达,是养血软坚胶囊保护软骨和抑制炎症的重要佐证。
3.2.5 miR-142
本课题中,尽管结果并无统计学意义,但中药组的rno-miR-142-5p在关节软骨中低表达明确,且以往的研究已经证实miR-142在软骨代谢、炎症水平方面有一定的调控作用。
NF-κB信号通路在关节软骨的生长发育及骨关节炎的发生发展中起到明显作用[21],可以调节关节软骨细胞的增殖凋亡、炎性因子的表达。研究表明,miR-142可以通过HMGB1调控NF-κB信号通路以达到抑制炎症和骨关节炎软骨细胞凋亡的作用。而白芍中的白芍总苷能够通过下调TLR-4 / NF-κB的表达水平来减轻脓毒症大鼠的炎性反应[22],同时能够下调NF-κB信号通路的蛋白表达,进而抑制骨关节炎大鼠模型的炎症反应[23]。据此可以推测白芍总苷是通过抑制骨关节炎软骨细胞中NF-κB信号通路的表达,达到保护软骨细胞的作用,从而对骨关节炎起到一定的治疗作用。通过以后进一步实验或可解释养血柔肝法中药“养血软坚胶囊”的作用机理。
[1] Buckwalter JA, Martin JA. Osteoarthritis[J]. Adv Drug Deliv Rev, 2006, 58(2):150-167.
[2] Carmona-Terés V,Lumillo-GutiérrezéI,Jodar-Fernández L, et al. Effectiveness and cost-effectiveness of a health coaching intervention to improve the lifestyle of patients with knee osteoarthritis: cluster randomized clinical trial[J].BMC Musculoskelet Disord,2015,16(1): 38.
[3] 石印玉,汪青春.分期诊治膝骨关节炎的对比研究(第一部分抗炎镇痛实验)[J].中国学术期刊文摘,1997, 3(7):890-892.
[4] 冯伟.中药对软骨细胞生物学功能的影响[D].上海中医药大学,1997.
[5] 冯伟,石印玉,沈培芝,等.MTT法分析中药含药血清对体外软骨细胞增殖影响的研究[J].上海中医药大学学报, 2000, 14(1):43-45.
[6] 高宁阳, 曹月龙, 庞坚, 等. C57黑鼠骨关节炎模型的关节病理积分与血清及滑膜中MMP-3、IL-1水平的相关性研究[J].中华风湿病学杂志, 2010, 14(3):195-198.
[7] John B, Enright AJ,Aravin A, et al. Human MicroRNA targets[J].PLoS Biol, 2004,2(11):e363.
[8] Szklarczyk D, Franceschini A,Wyder S,et al.STRING v10:protein-protein interaction networks, integrated over the tree of life[J].Nucleic Acids Res, 2015,43(Database issue):D447-D452.
[9] Kohl M, Wiese S, Warscheid B. Cytoscape: software for visualization and analysis of biological networks[J]. Methods Mol Biol,2011, 696(696):291-303.
[10] Alcaraz MJ, Megías J, García-Arnandis I, et al. New molecular targets for the treatment of osteoarthritis[J]. Biochem Pharmacol, 2010, 80(1):13-21.
[11] Ason B, Darnell DK, Wittbrodt B, et al. Differences in vertebrate microRNA expression[J].Proc Natl Acad SciUSA,2006,103(39):14385-14389.
[12] Wienholds E,Kloosterman WP,Miska E,et al. MicroRNA expression in zebrafish embryonic development[J].Science,2005,309(5732):310-311.
[13] Miyaki S,Sato T,Inane A,et al.MicroRNA-140 plays dual roles in both cartilage development and homeostasis[J].Gene Dev,2010,24(11):1173-1185.
[14] Nakamura Y,Inloes JB,Kataqiri T,et al.Chondrocyte specific microRNA 140 regulates endochondral bone development and targets dnpep to modulate bone morphogenetic protein signaling[J].Mol Cell Biol,2011,31(14):3019-3028.
[15] Karlsen TA,Jakobsen RB,Mikkelsen TS,et al. microRNA-140 targets RALA and regulates chondrogenic differentiation of human mesenchymal stem cells by translational enhancement of SOX9 and ACAN[J].Stem Cells Dev,2014,23(3):290-304.
[16] 殷春明,潘晓华.MicroRNA-140在骨关节炎中的分子机制研究进展[J].中国骨质疏松杂志,2015,21(10):1277-1286.
[17] Swingler TE,Wheeler G,Carmont V,et al. The expression and function of microRNAs in chondrogenesis and osteoarthritis[J]. Arthritis Rheum,2012,64(6): 1909-1919.
[18] Lin EA, Kong L, Bai XH, et al. miR-199a, a bone morphogenicprotein 2 -responsive MicroRNA, regulates chondrogenesis via direct targeting to Smad1[J]. J Biol Chem, 2009,28(4):11326-11335.
[19] 张静,王秀丽,彭方,等. miR-199-3p在兔膝骨关节炎软骨下骨中的表达及其意义[J].解放军医学院学报,2016,37(12):1289-1292.
[20] 李敏,郭婧,汪冽.单核细胞趋化蛋白诱导蛋白-1的研究进展[J]. 生命科学,2013,25(10):1015-1021.
[21] Brager MA,Patterson MJ,Connolly JF,et al. Osteogenic growth peptide normally stimulated by blood loss and marrow ablation has local and systemic effectson fracture healing in rats[J]. J Orthop Res, 2000,18(1):133-139.
[22] Chen C,Li H,Kong X,et al. Immobilizing osteogenic growth peptide with and without fibronectin on a titanium surface: effects of loading methods on mesenchymal stem cell differentiation[J]. Int J Nanomed, 2014,10: 283-295.
[23] 刘俊丽,周学慧,张军,等.成骨生长肽(OGP10-14 )在不同微重力条件下生物学活性研究[J].中国骨质疏松杂志,2012,18 (7):596-599.
