水稻肽链释放因子OseRF1-3基因克隆、表达及启动子分析
2018-03-15唐跃辉包欣欣张慧聪赵君苇娄慧敏李成伟
唐跃辉,包欣欣,刘 坤,张慧聪,王 双,赵君苇,娄慧敏,王 箐,梁 静,乔 蓉,李成伟
(1.周口师范学院 植物遗传与分子育种重点实验室,河南 周口 466000;2.河南省作物分子育种与生物反应器重点实验室,河南 周口 466000)
蛋白质的合成是细胞将遗传信息由核酸水平传递到蛋白质水平的重要步骤,在维持细胞结构的稳定中起重要的作用。蛋白质的合成分为起始、延伸和终止3个过程,然而,真核生物蛋白质的终止需要2类肽链释放因子eRF1(第1类肽链释放因子)和eRF3(第2类肽链释放因子)的参与[1-5],eRF1在核糖体的A位点专一识别终止密码子UAA、UAG、UGA,在P位点催化新生肽链与tRNA之间酯键水解,促使新生肽链的释放,进而引起翻译过程终止[1]。eRF3是鸟嘌呤核苷酸结合蛋白,具有GTPase活性,与eRF1相互作用形成复合体,协助eRF1完成核糖体循环和新生肽链从核糖体的释放[6]。
eRF1蛋白由N、M、C这3个保守的结构域组成,其中,N结构域包含3个保守的NIKS、GTS、YxCxxxF模块基序(motif),该结构域主要负责识别终止密码子[7-9];M结构域包含1个高度保守的GGQ基序,负责肽酰tRNA酯键的水解[8];C结构域是eRF1和eRF3相互作用的区域,其中第410-415位置的氨基酸(GGXLRY基序)在eRF1和eRF3相互作用中起重要的作用,第416-437位的氨基酸富含酸性氨基酸,也是eRF1和eRF3相互作用所必需的[8-11]。
eRF1在不同物种中都具有很高的保守性,在多个物种中都有所研究,但植物中研究较少,仅在拟南芥方面有些报道。此外,eRF1除了终止翻译的功能外还发现了其他功能,例如,抑制酵母eRF1影响酵母有丝分裂[12-13];共抑制拟南芥eRF1-1影响细胞伸长与径向细胞的分离,导致韧皮部异位木质化[14],拟南芥中过表达eRF1-2表现出对葡萄糖超敏感,并且发现,eRF1-2在GA信号转导途径中可能起负调控的作用[15]。在水稻中关于eRF1基因功能还没有相关的报道。为此,本研究克隆了1个水稻eRF1基因,命名为OseRF1-3,首先对OseRF1-3进行了组织特异性表达和生物信息学分析,并构建了OseRF1-3基因启动子融合GUS植物表达载体,并转化到水稻愈伤组织,通过GUS组织化学染色进一步分析了OseRF1-3基因的表达模式,旨在为进一步研究OseRF1-3基因在水稻生长与发育中的功能奠定基础。
1 材料和方法
1.1 试验材料
本试验所需的水稻材料为粳稻中花11(ZH11),种植于周口师范学院试验田,取生长28 d的水稻幼苗根、茎、幼叶及分蘖芽,5~9 cm的水稻穗和授粉后10 d的种子用于组织特异性表达分析,锡箔纸包裹并立即放液氮速冻,然后放于-80 ℃保存待用。
本试验所用的pMD18-T载体,LATAG酶,Hind Ⅲ和BamH Ⅰ,T4DNA连接酶均购买于大连宝生物有限公司,pCAMBIA1391Z载体为河南省作物分子育种与生物反应器重点实验室保存,其他试剂均为国产分析纯。
1.2 水稻DNA和RNA提取
水稻DNA的提取采用上海捷锐生物工程有限公司新型快速植物基因组DNA提取试剂盒(离心柱型)提取方法,具体的操作参考试剂盒说明书进行。待水稻生长到14 d后,取地上部分叶片液氮速冻,根据试剂盒说明进行水稻DNA提取。
水稻各组织RNA提取使用Magen公司植物小量RNA提取方法,具体操作参照试剂盒说明书进行,取2 μg提取RNA作为模板,参照Magen公司逆转录试剂盒说明书进行第1链cDNA合成。
1.3 系统发育和氨基酸序列分析
拟南芥AteRF1蛋白序列来自于TAIR拟南芥基因组数据库,水稻和别的物种的eRF1蛋白序列从GenBank(http://www.ncbi.nlm.nih.gov/)下载,通过ClustalW软件进行eRF1蛋白质全长序列比对,通过Mega software version 5软件进行系统发育树构建(NJ法,Bootstraps:1 000)。
1.4 基因表达量分析
以F:5′-ACTACGGTAAAGCACCTCAACAA-3′;R:5′-ATAGCGGAGAATCCCCCCT-3′为定量PCR引物,采用定量PCR仪(LightCycler 480)对OseRF1-3基因表达进行检测,条件如下:95 ℃,30 s;95 ℃,5 s,60 ℃,20 s,72 ℃,20 s,40 个循环。选用水稻的Ubiquitin1 (Os06g0681400)F:5′-TTCCATGCTGCTC
TACCACAG-3′;R:5′-AGGGTTCACAAGTCTGCCTA
TT-3′作为定量 PCR 内参引物。设3次重复。
1.5 基因及启动子克隆
按以下引物F:5′-ATGGCTGACAGCCATGAAA
C-3′; R:5′-GCCAGCACAGGCTCCTCC-3′进行OseRF1-3基因开放阅读框克隆,条件如下:94 ℃,5 min;94 ℃,30 s,54 ℃,30 s,72 ℃,90 s,32 个循环;72 ℃ 7 min;16 ℃保存。按以下引物F:5′-ATCGAATTCCA
AACCCGTTAGCCAAACACT-3′; R:5′-AAAGGATCCTG
TCGTGCTCGCAGAGGTGT-3′进行OseRF1-3基因启动子克隆,条件如下:94 ℃,5 min;94 ℃,30 s,54 ℃,30 s,72 ℃,2 min,31个循环;72 ℃,10 min;16 ℃保存。将克隆的片段连接到pMD18-T载体并送南京金斯瑞生物公司测序。
1.6 启动子生物信息学分析
使用tssp和plantCARE软件对启动子进行生物信息学分析。
1.7 水稻稳定遗传转化及转基因株系的获得
以粳稻中花11的愈伤组织作为受体,参考Tang等[16]水稻转化方法进行水稻的稳定遗传转化。通过潮霉素对转基因株系进行初次筛选,然后通过GUS染色对初次筛选的结果进行进一步确认。
1.8 GUS组织化学染色分析
取生长30 d的水稻幼苗根、叶、叶夹角、根茎接触处以及60 d的茎,授粉后8 d的水稻种子、颖壳和授粉前的花进行GUS染色,首先将这些组织放入GUS染液中,然后放入37 ℃,2 h后取出,取出后倒掉GUS染液,并向EP管里加入1 mL 75%的酒精,放入70 ℃温箱脱色,脱色后拍照保存。
2 结果与分析
2.1 OseRF1-3基因系统发育和氨基酸序列分析
水稻基因组中存在4个eRF1基因,分别命名为OseRF1-1、OseRF1-2、OseRF1-3、OseRF1-4,系统发育分析表明,OseRF1-1和OseRF1-3在禾本科中形成独立的分支(图 1-A),说明OseRF1-1和OseRF1-3在植物进化过程中有可能分化出了新的功能,因此,OseRF1-3被用来做进一步的功能分析。
设计特异引物,以水稻穗和种子混合cDNA为模板,通过RT-PCR技术克隆出了OseRF1-3的CDS序列,然后进一步将该序列在NCBI进行比对,结果表明,该序列与NP_001045349 (OseRF1-3)在Genbank登录序列完全一致。对OseRF1-3的序列分析发现,该基因位于水稻1号染色体,mRNA全长2 059 bp,CDS序列长1 308 bp,编码436个氨基酸,包含N、M、C这3个保守结构域,属于eRF1家族。氨基酸序列比对分析表明,OseRF1-3蛋白与拟南芥AteRF1和水稻OseRF1家族别的成员相似性为90.6%,且OseRF1-3包含N结构域中保守的NIKS、GTS、YxCxxxF基序以及M结构域中GGQ基序和C结构域中GGXLRY基序(图1-B)。进一步表明,eRF1基因在单子叶植物和双子叶植物中高度保守。

A.OseRF1-3同源性蛋白系统发育树分枝;Cr.莱茵衣藻;Vv.葡萄;Rc.蓖麻;Pt.毛果杨;Mt.苜蓿;Gm.大豆;Ps.北美云杉;At.拟南芥;St.马铃薯;Bo.花椰菜;Sb.高粱;Zm.玉米;Os.水稻;Ta.小麦;Hv.大麦;B.OseRF1-3蛋白NIKS、GTS、YxCxxxF、GGQ和GGXLRY基序通过下划线显示。
A. Phylogenetic tree analysis of OseRF1-3 and homologous proteins from other species:Cr.Chlamydomonasreinhardtii;Vv.Vitisvinifera;Rc.Ricinuscommunis;Pt.Populustrichocarpa;Mt.Medicagotruncatula;Gm.Glycinemax;Ps.Piceasitchensis;At.Arabidopsisthaliana;St.Solanumtuberosum;Bo.Brassicaoleraceavar.botrytis;Sb.Sorghumbicolor;Zm.Zeamays;Os.OryzasativaJaponica;Ta.Triticumaestivum;Hv.Hordeumvulgare;B. The conserved NIKS,GTS,YxCxxxF,GGQ and GGXLRY motifs of OseRF1-3 protein are indicated as the underlined segment.
图1OseRF1-3与其他物种同源蛋白的系统发育树和多重序列比对分析
Fig.1PhylogenetictreeandmultiplesequencealignmentanalysisofOseRF1-3andhomologousproteinsfromotherspecies
2.2 水稻OseRF1-3组织特异性表达分析
通过qRT-PCR技术检测了OseRF1-3基因在水稻不同发育时期的表达模式,结果表明,OseRF1-3是组成型表达,在根中表达最低,在水稻穗中表达最高(图2)。
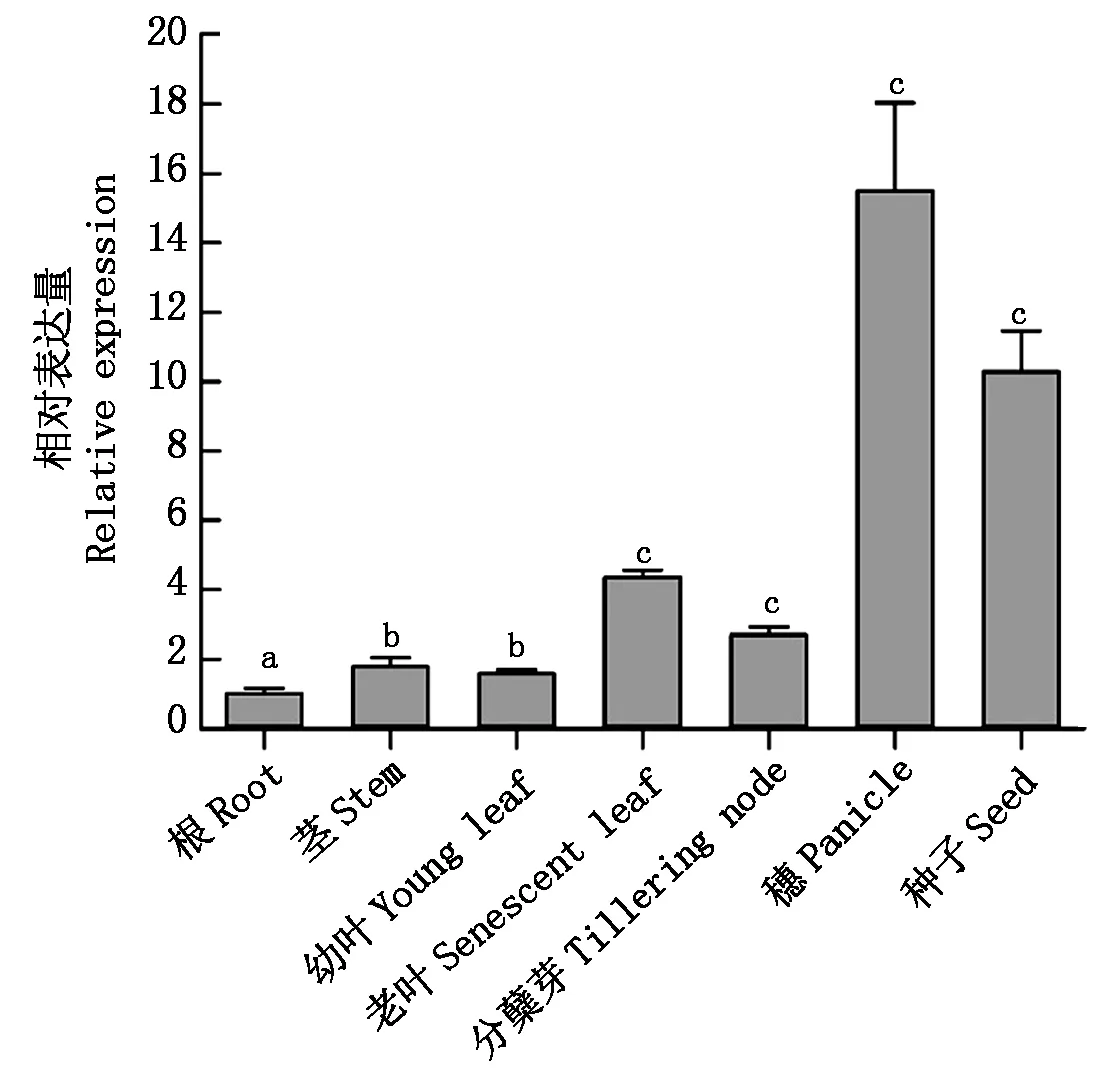
试验设 3 个生物学重复,每个重复设 2 个技术性重复。字母b和c表示相对于根差异显著(P<0.05)。
The experiment included three biological replicates, each with two technical replicates. Letters b and c indicate significant differences from the corresponding root(P<0.05).
图2OseRF1-3表达模式分析
Fig.2ExpressionlevelsofOseRF1-3
2.3 OseRF1-3基因启动子克隆
根据OseRF1-3基因组序列设计特异引物,通过PCR技术扩增出OseRF1-3基因启动子序列,然后送公司测序,结果表明,该序列长2 120 bp(图 3)。将该序列与扩增出来的OseRF1-3基因CDS序列进行比对,结果表明,该序列与OseRF1-3基因CDS序列A开始连续49 bp碱基重合,说明该序列为OseRF1-3基因启动子序列。去掉与CDS重合序列,扩增出来的OseRF1-3基因启动子序列全长2 071 bp。

1~3.启动子序列扩增产物;M.Marker。
2.4 OseRF1-3启动子生物信息学分析
利用在线软件tssp和plantCARE (http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对水稻OseRF1-3启动子序列进行分析,结果表明,OseRF1-3基因启动子区共包含12个增强转录元件CAAT-box,11个RNA聚合酶结合位点TATA-box,这些元件在启动子发挥作用中起重要的调控作用,预测的-796位置的A可能为转录起始位点(图4)。此外,OseRF1-3基因启动子还包含1个光应答元件TTTCAAA;3个WRKY转录因子结合元件WRKY71OS和3个细胞分裂素调节相关元件NGATT(图4)。

图4 OseRF1-3基因启动子序列分析Fig.4 Sequence analysis of OseRF1-3 gene promoter
2.5 构建OseRF1-3启动子植物表达载体
将测序正确的OseRF1-3启动子序列通过T4DNA连接酶连接到植物表达载体pCAMBIA1391Z上Hind Ⅲ和BamH Ⅰ位点之间,获得POseRF1-3∷GUS载体,并送公司进行再次测序,进一步通过Hind Ⅲ和BamH Ⅰ对构建完成的POseRF1-3∷GUS载体进行酶切验证(图 5-A),测序和酶切结果表明,该载体为OseRF1-3启动子驱动GUS植物表达载体(图 5-B)。

A .OseRF1-3启动子植物表达载体酶切结果;B.OseRF1-3启动子植物表达载体图谱。NOS.胭脂氨酸合成酶基因终止转录区;Hph.潮霉素磷酸转移酶基因;GUS.β-葡萄糖醛酸报告基因;MCS.多克隆微点。A.The enzyme result of plant expression vector from OseRF1-3 promoter;B.Plant expression vector spectrum of OseRF1-3 promoter.NOS. Nopaline synthase transcription termination region;Hph.Hygromycin phosphotransferase gene;GUS.β-glucuronidase reporter gene; MCS. Multiple clone site.
2.6 POseRF1-3∷GUS转基因株系获得和组织GUS染色
以水稻愈伤组织为受体,通过农杆菌介导法将构建好的OseRF1-3启动子驱动GUS植物表达载体转入水稻愈伤组织,通过潮霉素抗性筛选和PCR技术鉴定阳性转基因株系,通过T2分离比,挑选单拷贝插入转基因株系(分离比为3∶1)进行进一步分析。取T3转基因水稻不同组织部位进行GUS染色,37 ℃,2 h后观察GUS染色结果,进而确定OseRF1-3基因启动子的表达特性。GUS染色结果表明,OseRF1-3基因是组成型表达,在根中表达较弱,然而,在水稻花、穗中有高水平的表达(图 6)。GUS检测OseRF1-3基因启动子的表达特性的结果与qRT-PCR得出的结果相一致。

从上到下、从左到右依次为:根、茎、叶、叶夹角、茎结合部位、花、种子、根茎结合部和穗。From top to bottom and from Left to right in turn:root,stem,leaf,leaf angle,stem-stem junction,flower,seed,root-stem junction and spike.
3 结论与讨论
基因的功能往往与该基因在植物组织中的表达模式密切相关。为了研究水稻肽链释放因子OseRF1-3在水稻发育中的功能,通过qRT-PCR技术检测了该基因的组织表达模式,其在穗中有强的表达,在其他组织都有较强表达。此外,克隆了OseRF1-3基因起始密码子ATG上游2 071 bp序列,并构建了启动子融合GUS基因植物表达载体,该基因启动子融合GUS基因组织化学染色结果与qRT-PCR结果一致,该结果表明,OseRF1-3为组成型表达。该结果与拟南芥AteRF1表达模式相似[8-9],表明肽链释放因子eRF1表达模式具有一定的保守性,推测该基因的功能也存在一定的保守性。
在过去的研究中,对作物抗逆、抗虫害等优良品种的分子育种中,选用的是外源花椰菜花叶病毒CaMV35S组成型启动子。近几年,一些植物内源组成型启动子被相继克隆和功能分析。Wu等[17]研究表明,浒苔(Olavprolifera)Actin1启动子是组成型启动子,驱动基因表达的强度高于CaMV 35S启动子。拟南芥AtTCTP启动子融合GUS报告基因在所有的组织中都表达,能够替代35S启动子作为驱动目的基因表达的组成型启动子[18]。Masura等[19]证明棕榈油Type2启动子为组成型启动子。Huang等[20]证明田旋花EPSPS启动子为组成型启动子。在本研究中,OseRF1-3启动子融合GUS基因在转基因水稻中为组成型表达。
在水稻高产、稳产和优质品种的分子模块设计育种中,往往采用的是花椰菜花叶病毒35S或玉米泛素基因启动子作为常用的植物表达载体激活靶基因表达的启动子,然而,选用水稻内源组成型启动子仍然很少。因此,从植物中挖掘更多的组成型启动子对优良基因在作物生产上的应用和基础研究具有重要的意义。在本研究中,GUS活性分析结果表明,水稻肽链释放因子OseRF1-3基因的启动子具有明显的组成型特性,因此,该启动子可以作为内源启动子应用于水稻植物过表达载体的构建,进而在水稻高产、抗逆、耐虫害等品种的培育中具有很好的应用前景。
[1] 闫 静, 石文鑫, 王 美, 等. 第一类肽链释放因子C端结构域参与调控终止密码子的识别过程[J]. 中国生物化学与分子生物学报, 2017, 33(2): 184-189.
[2] 黄红英. 纤毛虫肽链释放因子识别终止密码子特异性的功能位点分析[D]. 太原: 山西大学, 2016.
[3] Vaishya S, Kumar V, Gupta A, et al. Polypeptide release factors and stop codon recognition in the apicoplast and mitochondrion ofPlasmodiumfalciparum[J]. Molecular Microbiology, 2016, 100(6): 1080-1095.
[4] Trappl K, Joseph S. Ribosome induces a closed to open conformational change in release factor 1[J]. Journal of Molecular Biology, 2016, 428(6): 1333-1344.
[5] Polshakov V I, Eliseev B D, Birdsall B, et al. Structure and dynamics in solution of the stop codon decoding N-terminal domain of the human polypeptide chain release factor eRF1[J]. Protein Science:a Publication of the Protein Society, 2012, 21(6): 896-903.
[6] Urakov V N, Mitkevich O V, Safenkova I V, et al. Ribosome-bound Pub1 modulates stop codon decoding during translation termination in yeast[J]. The FEBS Journal, 2017, 284(12): 1914-1930.
[7] Frolova L, Seit-Nebi A, Kisselev L. Highly conserved NIKS tetrapeptide is functionally essential in eukaryotic translation termination factor eRF1[J]. RNA, 2002, 8(2): 129-136.
[8] Inagaki Y, Blouin C, Doolittle W F, et al. Convergence and constraint in eukaryotic release factor 1 (eRF1) domain 1: the evolution of stop codon specificity[J]. Nucleic Acids Research, 2002, 30(2): 532-544.
[9] Song H, Mugnier P, Das A K, et al. The crystal structure of human eukaryotic release factor eRF1-mechanism of stop codon recognition and peptidyl-tRNA hydrolysis[J]. Cell, 2000, 100(3): 311-321.
[10] Ito K, Ebihara K, Nakamura Y. The stretch of C-terminal acidic amino acids of translational release factor eRF1 is a primary binding site for eRF3 of fission yeast[J]. RNA, 1998, 4(8): 958-972.
[11] Pillay S, Li Y, Wong L E, et al. Structural characterization of eRF1 mutants indicate a complex mechanism of stop codon recognition[J]. Scientific Reports, 2016, 6: 18644.
[12] Valouev I A, Kushnirov V, Teravanesyan M D. Yeast polypeptide chain release factors eRF1 and eRF3 are involved in cytoskeleton organization and cell cycle regulation[J]. Cytoskeleton, 2002, 52(3): 161-173.
[13] Kochneva-Pervukhova N V, Alexandrov A I. Amyloid-Mediated sequestration of essential proteins contributes to mutant huntingtin toxicity in yeast[J]. PLoS One, 2012, 7(1): e29832.
[14] Petsch K A, Mylne J, Botella J R. Cosuppression of eukaryotic release factor 1-1 inArabidopsisaffects cell elongation and radial cell division[J]. Plant Physiology, 2005, 139(1): 115-126.
[15] Zhou X, Peter C, Li L.Eukaryotic release factor 1-2 affectsArabidopsisresponses to glucose and phytohormones during germination and early seedling development[J]. Journal of Experimental Botany, 2010, 61(2): 357-367.
[16] Tang Y E, Liu K, Zhang J, et al.JcDREB2, a physic nut AP2/ERF gene, alters plant growth and salinity stress responses in transgenic rice[J]. Frontiers in Plant Science, 2017, 8(e0131599): 306-313.
[17] Wu C, Jiang P, Guo Y, et al. Isolation and characterization of Ulva proliferaactin1 gene and function verification of the 5' flanking region as a strong promoter[J]. Bioengineered, 2017, 21(2): 1-10.
[18] Han Y J, Kim Y M, Hwang O J, et al. Characterization of a small constitutive promoter fromArabidopsistranslationally controlled tumor protein (AtTCTP) gene for plant transformation[J]. Plant Cell Reports, 2015, 34(2): 265-275.
[19] Masura S S, Parveez G K, Ti L L. Isolation and characterization of an oil palm constitutive promoter derived from a translationally control tumor protein (TCTP) gene[J]. Plant Physiology and Biochemistry, 2011, 49(7): 701-708.
[20] Huang Z F, Wang G R, Huang H J, et al. Isolation and functional analysis of convolvulus arvensis EPSPS promoter[J]. Plant Molecular Biology Reporter, 2015, 33(6): 1650-1658.
