GPⅢa PLA2、PEAR1、PTGS1基因多态性与阿司匹林临床抗血栓疗效关联性研究
2018-03-13石秀锦胡志旭彭文星徐晓宇续茜桥首都医科大学附属北京安贞医院药事部北京
石秀锦,胡志旭,彭文星,李 骁,徐晓宇,续茜桥,林 阳(.首都医科大学附属北京安贞医院药事部,北京
100029;2.首都医科大学化学生物学与药学院临床药学系,北京 100069;3.首都医科大学附属北京世纪坛医院药剂科,北京 100038)
阿司匹林和氯吡格雷双抗治疗成为介入术后预防血栓、脑中风等缺血性心血管事件的常规方案。阿司匹林抗血小板作用机制为直接并不可逆地抑制环氧化酶1和2(COX-1和COX-2),减少前列腺素的合成,抑制血小板合成血栓素A2(TXA2),从而抑制血小板聚集。同时,阿司匹林也可抑制低浓度胶原、凝血酶、抗原-抗体复合物等所致的血小板聚集和释放反应及自发性聚集,由此发挥预防血栓的作用。但阿司匹林抵抗(aspirin resistance,AR)现象十分常见,发生率在5.5%~60%之间[1]。研究[2]表明,有AR的患者发生急性冠脉综合征和心血管缺血事件的风险会增加。AR的确切机制还不明确,但研究发现基因多态性与AR有一定关系,目前研究的基因多态性包括:血小板糖蛋白(GP)Ⅱb/Ⅲa基因多态性、环氧合酶(COX-1)基因的多态性、血小板糖蛋白Ⅰa/Ⅱa受体基因多态性、ADP受体P2Y1基因的变化等[3-6]。若这些位点突变为失功能等位基因可使阿司匹林的疗效降低,并增加患者心血管事件的发生率[7]。本研究分析GPⅢa PLA2、PEAR1、PTGS1三个基因位点的突变情况及其与阿司匹林临床疗效的相关性,进一步探讨影响阿司匹林疗效的遗传因素,为临床制定合理的抗血小板方案提供理论依据。
1 资料与方法
1.1 研究人群
回顾性收集2015年1月–2016年5月入院并进行阿司匹林基因检测的747例患者信息资料,最终筛选出138例患者。纳入标准:服用标准剂量的阿司匹林100 mg·d-1和氯吡格雷75 mg·d-1抗血小板治疗,持续至少3 d,用药3 d后进行血小板聚集率检测,已完成GPⅢa PLA2、PTGS1、PEAR1的基因检测。排除标准:慢性肝病病史,严重肾功能不全(Cr < 30 mL·min-1),合并出血性疾病,有阿司匹林或氯吡格雷的禁忌症,只使用单抗治疗,血小板计数小于100×109·L-1,合并使用华法林,同时使用其他非类固醇类抗炎药物。
1.2 血小板聚集率
枸橼酸抗凝管采血3~5 mL,比浊法进行血小板功能检测,先将试管在4000~5000 r·min-1离心5 min,得到富含血小板的血浆(PRP),再于22 500 r·min-1高速离心15 min获得含少量血小板的血浆(PPP),将PPP加入PRP中,将血小板浓度稀释至2×105·µL-1,加入6 µmol·L-1的花生四烯酸诱导血小板聚集,之后采用仪器Chrono-Log Lumi-aggregometer(model 700,美国Chrono-Log公司)测定血小板聚集率。
1.3 基因位点测定
用乙二胺四乙酸(EDTA)抗凝管采血2~3 mL,采用荧光染色原位杂交检测法。取150 µL血加入1 mL红细胞裂解液,静置5 min后,在3000 r·min-1离心5 min,弃上清,再加入核酸纯化试剂(北京华夏时代基因科技发展有限公司),混匀后加入相应的测序反应通用试剂(北京华夏时代基因科技发展有限公司),采用双通道实时荧光定量PCR仪(TL988A,西安天隆科技有限公司)进行检测。
1.4 分组定义
本次研究中,由于样本特性,以PEAR1基因型进行实验分组,分为野生组和突变组。野生组定义为GPⅢa PLA2、PTGS1基因型为TT、AA,PEAR1基因型为GG;而突变组定义为GPⅢa PLA2、PTGS1基因型为TT、AA,PEAR1基因型为AG或AA。
1.5 统计学分析
采用SPSS 20.0对数据进行统计学分析。正态性连续性变量采用(均值±标准差)表示,非正态性资料采用中位数和四分位数表示,计数资料采用百分比表示。正态性连续性变量采用双侧t检验比较两组之间的差异,计量资料采用卡方检验或者Fisher精确检验。基因型的比较采用单因素ANOVA分析,两组之间基因型或者位点的比较采用卡方检验,以P< 0.05为差异有统计学意义。
2 结果
2.1 三个相关基因位点多态性
根据纳入标准,统计纳入的138例患者基因型,其中GPⅢa PLA2、PEAR1、PTGS1位点的野生基因型分别为TT、GG、AA。结果显示GPⅢa PLA2基因在亚洲人群中突变频率较低,突变率不足1%,C等位基因频率为0.004,T为0.996。PTGS1基因突变频率亦较低,突变率约为2%,G等位基因频率为0.01,A为0.99。PEAR1基因突变率较高,为63.8%, A等位基因频率为0.376,G为0.624,详见表1。
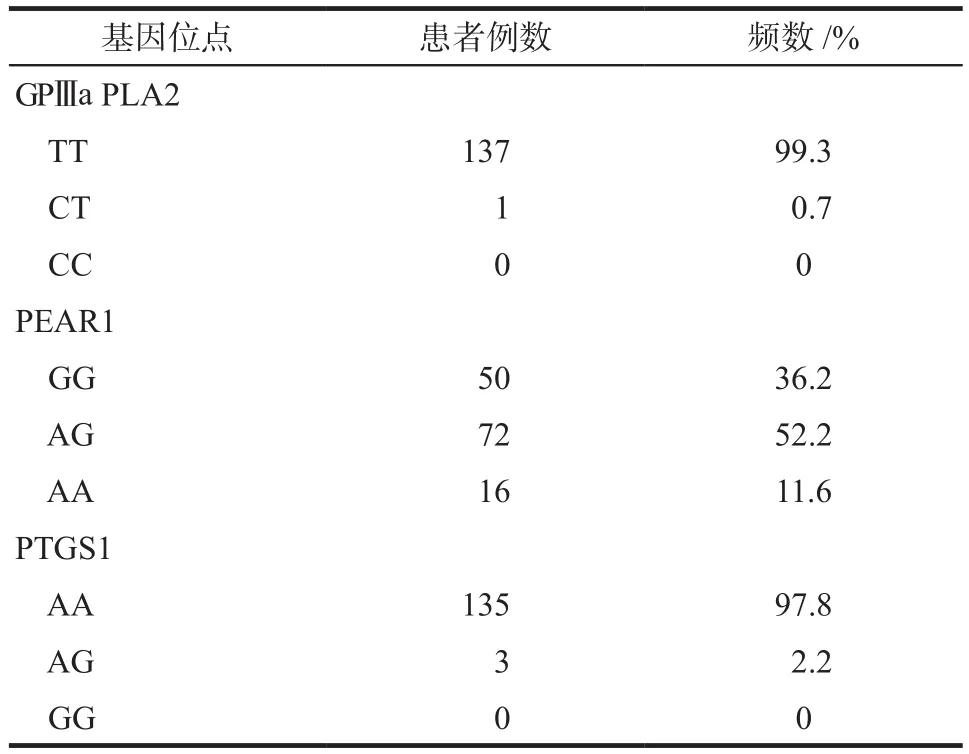
表1 三个基因位点的突变情况Tab 1 Mutation of three geneloci
2.2 研究人群的基线水平
根据纳入排除标准,最终纳入138例目标患者,在进行数据处理中排除1例GPⅢa PLA2位点突变杂合型(该患者同时PTGS1位点为杂合型)和3例PTGS1位点突变杂合型,基于GPⅢa PLA2、PTGS1均为野生基因型,即基因型分别为TT、AA,依据PEAR1基因型分组,PEAR1基因为野生型(GG)组共49例(36.3%),突变组(AG和AA)共86例(63.7%)。135例患者的平均年龄为(62.05±11.24)岁。结果显示两组人群一般情况均无显著性差异,表明患者的年龄、性别、BMI、合并症(高血压、糖尿病、高脂血症)和合并用药(他汀类、ACEI、ARB、β受体阻滞剂、质子泵抑制剂、肝素类、前列地尔类)等均不是引起阿司匹林疗效差异的危险因素,详见表2。
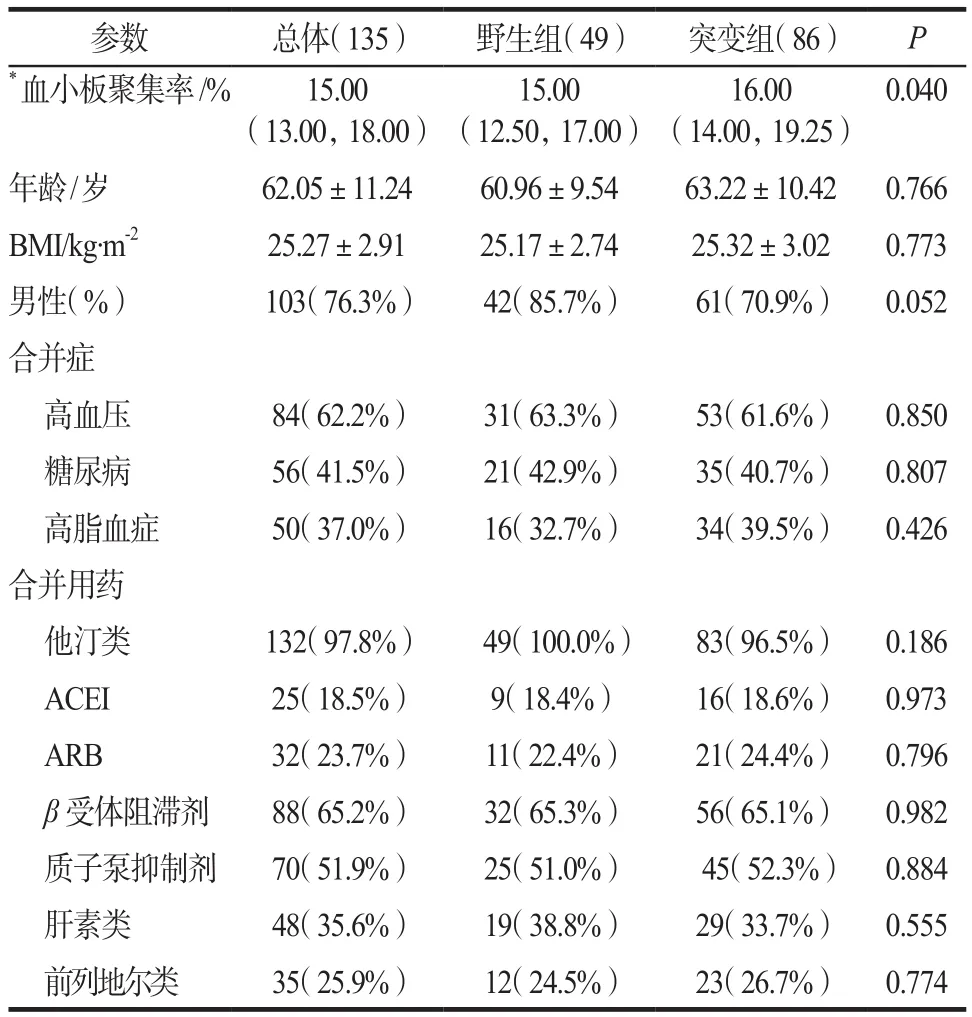
表2 研究人群的基线信息Tab 2 Baseline characteristics of the study population
2.3 PEAR1基因多态性与血小板聚集率的关联性
不同基因型的PEAR1基因位点花生四烯酸诱导的血小板聚集率的差异见表3。结果显示,PEAR1突变纯合型的血小板聚集率显著高于野生型(P=0.007),而突变杂合型与野生型相比未表现出显著性差异(P=0.135)。PEAR1基因突变型(AG和AA)的血小板聚集率显著高于野生型GG(P=0.040),从总体来看,突变位点数目越多,对应的血小板聚集率就越高。
3 讨论
3.1 结果讨论与相关研究
阿司匹林抵抗及其临床疗效问题是由多种因素造成的,目前已知的因素有基因多态性(GPⅢa PLA2、PEAR1、PTGS1等)、患者的依从性、生活习惯方式(有无吸烟、酗酒等)以及多种药物同时服用的相互作用等,而基因多态性是其中非常重要且研究较多的危险因素。本研究拟对GPⅢa PLA2、PTGS1及PEAR1基因与阿司匹林抗血栓疗效的关联性进行研究。
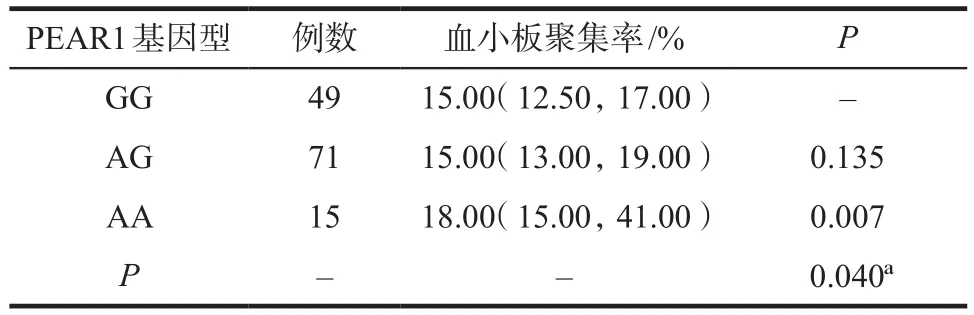
表3 PEAR1基因多态性与血小板聚集率的关联性Tab 3 The correlation between PEAR1 gene polymorphisms and platelet aggregation rate
Verschuren等[8]纳入1327例ST段抬高型心肌梗死患者的队列研究发现,PTGS1基因位点的突变显著增加了阿司匹林抵抗的风险(P=0.001),故建议对心血管事件复发风险高的ST段抬高型心肌梗死患者进行基因型检测。Lepäntalo等[9]以血小板聚集率为标准定义阿司匹林抵抗,发现60%的阿司匹林抵抗患者均携带突变位点G,而无阿司匹林抵抗患者只有17%携带突变基因(OR=7.3,P=0.017)。Dropinski等[10]的研究表明,GPⅢa基因PLA1/A2多态性位点中野生型PLA1在使用阿司匹林后血栓形成被抑制和出血时间延长比突变型PLA2明显。Goodman等[11]综合多项研究结果认为,PLA1/A2 多态性位点是影响阿司匹林疗效的一个重要因素,与阿司匹林抵抗的发生相关。但本研究由于PTGS1及GPⅢa PLA2基因位点多态性罕见,无法进行关联性分析,PTGS1和GPⅢa PLA2基因与阿司匹林抗血栓疗效的相关性还有待于大样本的进一步研究。
既往研究[12]显示中国人群中PEAR1的野生G基因的基因频率为0.61,突变A基因的频率为0.39,与本研究PEAR1突变频率非常一致。本研究根据PEAR1基因有无突变情况分为野生组和突变组,两组患者的基线水平没有显著性差异,结果显示PEAR1基因的突变型显著增加了阿司匹林抵抗的风险,纯合突变基因型尤为显著(P=0.007)。Lewis等[13]对1000例患者的研究发现,PEAR1基因与患者对抗血小板治疗的反应具有强烈的相关性(P=7.66×10-9)。高加索人与美国黑人携带突变基因患者PCI术后比携带野生型基因患者有更高的心血管事件发生率和死亡率[风险比(HR)=2.62,P=0.059;HR=3.97,P=0.035]。在服用阿司匹林和氯吡格雷双抗治疗的患者中,携带突变基因患者比野生型基因患者心肌梗死的发生率更高[比值比(OR)=2.03,P=0.048]。Würtz等[6]在一项985例稳定性冠心病患者服用阿司匹林单药治疗的研究中发现,PEAR1基因突变与血小板高活性具有相关性,突变基因型AA或AG的患者使用阿司匹林(或联合氯吡格雷)时的心肌梗死发生率和死亡率会增高。本研究也得出了阿司匹林抵抗与PEAR1基因型密切相关的结论,但本研究PEAR1突变杂合型者较野生型者的血小板聚集率增加程度尚不具有显著性差异(P=0.135),原因可能是本研究样本量小,也可能是突变杂合型并未使血小板内皮聚合受体有大量表达,因而血小板聚集率并未显著提高,但仍需大样本研究加以验证。
另外,有研究统计临床资料发现,年龄大、有糖尿病史、HDL水平低的患者阿司匹林抵抗发生率高,而本研究对于糖尿病、高血脂等危险因素并未得出阳性结论,可能与本次研究样本量偏小有关,因而需要更大样本量、严谨的实验加以验证。
3.2 实验方法
本研究采用比浊法进行血小板功能检测,与先前的研究采用了相似的检测方法[14]。血小板聚集试验是一种体外的血小板功能检测,虽然该方法有一定的局限性,如重复性较差和繁琐的制作样本流程,但仍是最常用的血小板功能检测的方法之一。有研究显示,根据血小板功能检测调整给药方案不能改善患者的预后,但这些研究主要研究人群并不是有缺血性疾病患者,同时这些患者本身发生心血管不良事件风险就比较低。
3.3 局限性
本研究并未获得患者的预后信息,仅根据血小板聚集率指标反映药物疗效,未获得药物的临床反应性。其次,采用比浊法进行血小板功能检测,不能获得良好的重现性,导致结果可能有所偏差。本研究为回顾性分析,每个患者进行血小板功能检测的时间不完全相同,可能造成实验室检测结果的差异。此外,由于本研究样本量小,且GPⅢa PLA2和PTGS1位点的突变率低,因而本研究无法分析GPⅢa PLA2、PTGS1基因位点与阿司匹林临床疗效的关联性情况,需大样本研究加以分析比较。
4 小结
本研究揭示了基因检测是预测患者对血小板反应性的方法之一,在患者进行治疗之前进行基因型检测,可根据患者的基因型选择合适的治疗方案,避免因药物未起效而发生急性缺血性事件。且基因检测较其他检测方法更方便快捷,能及时为临床提供准确的药物基因信息,并可初步预测患者的阿司匹林抵抗风险,为患者选择合适的抗血小板药物提供参考依据。
[1] 彭文星,冯频频,石秀锦,等.阿司匹林抵抗的基因多态性及个体化治疗[J].中国药房,2016,27(23):3172-3174.
[2] Pamukcu B, O fl az H, Oncul A,et al. The role of aspirin resistance on outcome in patients with acute coronary syndrome and the effect of clopidogrel therapy in the prevention of major cardiovascular events[J]. J Thromb Thrombolysis, 2006, 22(2):103-110.
[3] 韩雅玲.冠心病阿司匹林抵抗的研究与对策[J].中国实用内科杂志,2015,35(1):3-7.
[4] Li Y, Chen F, Zhang X,et al. Angiotensin type 1 receptor A1166C gene polymorphism is associated with endothelial dysfunction and in-stent restenosis after percutaneous coronary intervention[J].Int J Clin Exp Pathol, 2015, 8(6): 7350-7357.
[5] Yao Y, Tang XF, Zhang JH,et al. Association of PEAR1 genetic variants with platelet reactivity in response to dual antiplatelet therapy with aspirin and clopidogrel in the Chinese patient population after percutaneous coronary intervention.[J]. Thromb Res, 2016, 141: 28-34.
[6] Würtz M, Nissen PH, Grove EL,et al. Genetic determinants of on-aspirin platelet reactivity: focus on the in fl uence of PEAR1[J].PLoS One, 2014, 9(10): e111816.
[7] Oh MS, Yu KH, Lee JH,et al. Aspirin resistance is associated with increased stroke severity and infarct volume[J]. Neurology,2016, 86(19): 1808-1817.
[8] Verschuren JJ, Boden H, Wessels JA,et al. Value of platelet pharmacogenetics in common clinical practice of patients with ST-segment elevation myocardial infarction[J]. Int J Cardiol,2013, 167(6): 2882-2888.
[9] Lepäntalo A, Mikkelsson J, Reséndiz JC,et al. Polymorphisms of COX-1 and GPVI associate with the antiplatelet effect of aspirin in coronary artery disease patients[J]. Thromb Haemost, 2006,95(2): 253-259.
[10] Dropinski J, Musial J, Sanak M,et al. Antithrombotic effects of aspirin based on PLA1/A2 glycoproteinⅢa polymorphism in patients with coronary artery disease[J]. Thromb Res, 2007,119(3): 301-303.
[11] Goodman T, Ferro A, Sharma P. Pharmacogenetics of aspirin resistance: a comprehensive systematic review[J]. Br J Clin Pharmacol, 2008, 66(2): 222-232.
[12] Allele Frequencies. rs12041331 at chr1:156869714(hg38)in PEAR1[EB/OL]. (2015-05-04)[2017-09-01]. https://www.pharmgkb.org/rsid/rs12041331.
[13] Lewis JP, Ryan K, O'Connell JR,et al. Genetic variation in PEAR1 is associated with platelet aggregation and cardiovascular outcomes[J]. Circ Cardiovasc Genet, 2013, 6(2): 184-192.
[14] 彭文星,徐晓宇,石秀锦,等.PCI术后患者CYP2C19基因多态性与氯吡格雷抵抗关联性研究[J].中国药物应用与监测,2015,12(6):334-338.
