43份葡萄种质遗传多样性的SSR分析
2018-01-31杨航宇杨哲何非陈为凯王军
杨航宇,杨哲,何非,陈为凯,王军*
(中国农业大学食品科学与营养工程学院/农业部葡萄酒加工重点实验室,北京 100083)
葡萄是葡萄科(Vitaceae)葡萄属(Vitis)落叶藤本植物,是世界第二大水果[1],可用于酿酒、鲜食、制汁和制干,具有极高的经济价值。葡萄种质资源繁多,且容易发生芽变,所以在引种和栽培过程中常发生同名异物或同物异名现象,给葡萄的选育和推广带来很多不便。传统的利用形态学特征或利用花色苷等酚类物质组成和含量作为化学指纹的鉴别方法[2-4],由于容易受环境、气候影响,不能有效区分葡萄的无性系等,在品种鉴别时会受到限制。近年来,随着DNA分子标记技术的发展,SSR(Simple Sequence Repeat,简单序列重复)标记技术以其多态性高、符合孟德尔共显性遗传、实验重复性好等优点[5],被广泛应用于葡萄的品种鉴定、遗传多样性研究和亲缘关系分析。比如,Díaz Losada等[6]利用SSR标记对生长在西班牙的葡萄品种进行鉴别,发现了某些品种的同物异名现象;尹玲等[7]利用SSR标记建立了近年来我国新育成的24个葡萄品种的指纹图谱,并明确了它们的亲缘关系;DNA分子标记还可以作为鉴定品种的附加描述符,与形态学标记相结合来对葡萄种质资源进行描述[8-9],如Bosco等[8]在叶片形态和SSR标记两个方面对西班牙西北部的9个葡萄品种进行了描述和区分。
本研究选取了国内外常见的和近年来我国选育的43个用于酿酒或鲜食的葡萄品种,利用6对SSR引物进行扩增,对上述葡萄品种的遗传多样性进行分析,通过建立DNA指纹图谱进行品种鉴别,并在分子水平上探究了各葡萄种质的亲缘关系,以期为葡萄种质资源的鉴别、管理和推广提供理论依据和参考价值。

表1 43份供试葡萄种质资源Table 1 43 grape germplasms resource in this research
1 材料和方法
1.1 材料
供试材料名称及起源地见表1。43个栽培葡萄品种,其中琼瑶浆采自新西兰种质资源圃,引进冬季休眠枝条,回国解除休眠,扦插成活;其余42份葡萄种质采自中国农业大学上庄实验站(北京,40.14°N,116.19°E)。采样时每个品种选择2~3片生长良好的幼叶装入冰盒中带回实验室,液氮速冻后放-80 ℃冰箱保存,用于基因组DNA的提取。
1.2 DNA提取
采用王军等[10]改良的CTA B法提取葡萄幼叶中的基因组DNA,以λDNA(48 kb,50 ng/μL,天根生化科技有限公司)为标准,利用1%琼脂糖凝胶电泳检测DNA溶液的质量,并以λDNA亮度为标准,选择适当比例将提取的基因组DNA稀释至50 ng/μL,于-20 ℃冰箱保存备用。
1.3 DNA扩增和电泳
选用6对VIVC葡萄种质资源数据库提供的引物用于葡萄品种的SSR扩增,这6对引物被This等[11]推荐为用于葡萄品种的核心标记,引物由上海生工公司合成,序列及退火温度见表2。采用如下比例的20 μL PCR扩增反应体系:2 μL 10×PCR buffer,1.5 mmol/L MgCl2,0.2 mmol/L dNTPs,1U TaqDNA聚合酶,50 ng模板DNA,0.4 μmol/L引物(每条)。体系中反应物均购自宝生物工程(大连)有限公司。

表2 6对SSR引物信息[11]Table 2 Information of six pairs of SSR primers
实验在Prime G02型PCR仪(英国Bibby Scientific公司)中进行。PCR反应程序:94 ℃预变性5 min;94 ℃变性30 s,50~60 ℃退火30 s,72 ℃延伸1 min,循环35次;最后72 ℃延伸10 min。
扩增产物经8%聚丙烯酰胺变性凝胶电泳分离检验。对正向引物5'端加FAM荧光标记,重新合成荧光引物,重新进行PCR扩增,扩增产物送苏州金唯智生物科技有限公司进行毛细管电泳,ABI3730xI全自动分析仪进行片段收集,利用GenemapperV3.2进行条带分析,确定每个等位基因的片段大小。
1.4 数据分析
利用popgene 32软件对6对引物扩增的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、Shannon多样性指数(I)及多态性位点百分比等进行计算;利用NTSYSpc Version 2.10e软件计算成对种质间Jaccord(J)遗传相似系数,并根据遗传相似系数,利用MEGA 5.1软件,采用非加权平均法(UPGMA)对供试材料进行聚类分析。
2 结果与分析
2.1 引物扩增结果及多态性分析
6对SSR引物对43份葡萄种质的扩增结果见表3。由表3可以看出,在43份葡萄品种中共扩增出64个等位基因,片段大小为123~269 bp,其中每个位点扩增出的等位基因数为8~13个,平均等位基因数为10.67,平均有效等位基因6.20个。
各引物的观测杂合度(Ho)为0.74~0.84,平均Ho为0.80;期望杂合度(He)为0.82~0.88,平均He为0.84。Shannon多样性指数(I)为1.86~2.22,平均I为2.01。其中,Ho(观测到杂和个体数/样本个体数总数)能够反应实际群体中某一位点杂合子所占比例,杂合度越高,该位点处的基因型越多,遗传变异越丰富[12]。在6对引物中,VVMD7,VRZAG62和VRZAG79的Ho在0.8以上,扩增出丰富的基因型,显示出了良好的多态性。
所有引物的多态性条带比例均为100%,表明所选引物遗传多态性高,进而表明了不同葡萄品种之间的遗传差异。此外,6对引物均扩增出了特征性条带,这将对葡萄品种鉴定具有贡献。
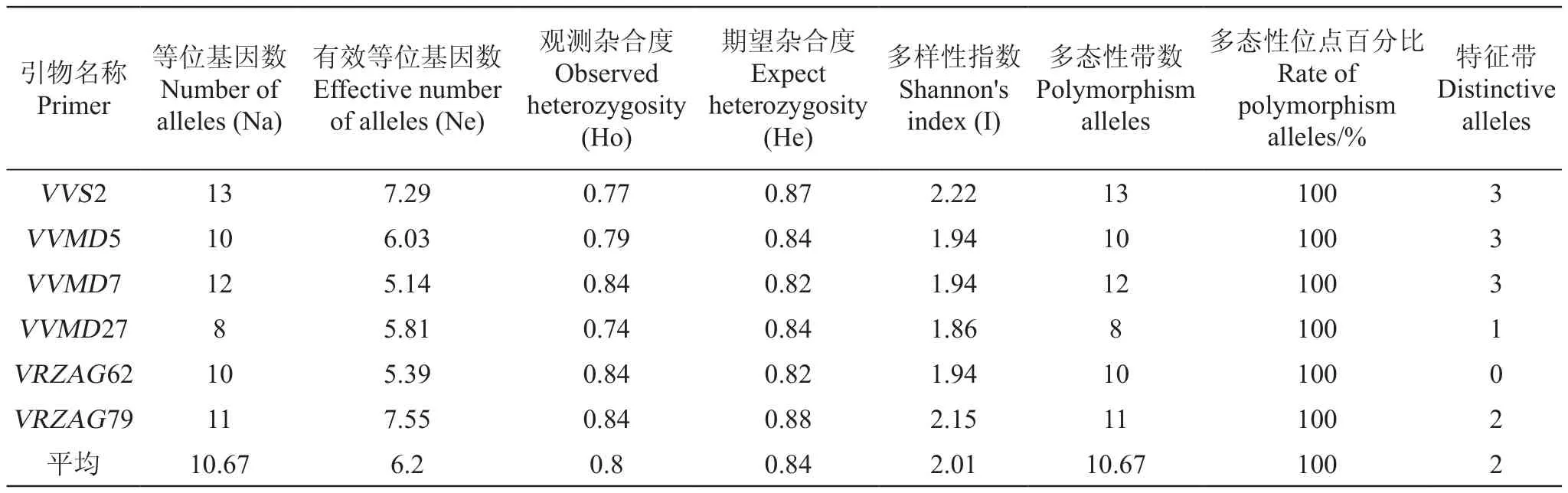
表3 6对SSR引物的遗传多态性指标Table 3 Genetic parameters of 6 SSR loci
2.2 DNA指纹数据及图谱的建立
PCR扩增得到的产物经毛细管电泳分离并进行条带分析,可得到扩增片段的大小,整理得到6对SSR引物对43份葡萄种质扩增的DNA指纹数据(表4)。
根据各个品种在不同位点的指纹数据,并与VIVC标准数据库(http://www.vivc.de)比对,可以进行品种的鉴别。由表4可以看出,除了21个品种的指纹信息VIVC标准数据库中未收录之外,其余22个品种的指纹信息均与标准数据库一致,可以实现品种的鉴定。
对扩增出的等位基因按顺序排列,按照各品种中每个等位基因的有无赋予“1, 0”两个数值,由此可以得到二元数据矩阵,根据二元数据矩阵构建43份供试葡萄种质的分子指纹图谱,结果如图1所示。综合6个位点的SSR数据可知,43个品种的指纹图谱各不相同,这表明可以利用SSR扩增得到的指纹数据进行品种鉴别,同时也说明了供试的样品中不存在同物异名的现象。
2.3 亲缘关系分析
由6对SSR引物的扩增结果,利用NTSYSpc Version 2.10e软件,根据各品种之间的相似系数,采用非加权平均法(UPGMA)对所有品种进行聚类分析,结果如图2所示。6对SSR引物标记可以将43个葡萄品种完全分开,它们的遗传相似系数在0.13~0.75,在0.13处,全部供试品种可以被划分为3大类。
第一大类(A组)包含17个品种,其中在遗传相似系数0.19处又可被细分为两个组:第一组(A1)包括‘赤霞珠’‘长相思’‘小芒森’‘琼瑶浆’‘美乐’‘贵人香’‘西拉’‘维欧尼’‘胡桑’‘威代尔’‘小味儿多’‘丹娜’‘马尔贝克’和‘宝石无核’,其中‘长相思’是‘赤霞珠’的亲本之一,二者被聚到了一起;第二组(A2):包括‘雷司令’‘霞多丽’和‘烟73’,3个品种均为酿酒品种。
第二大类(B组)包含22个品种,在遗传相似系数0.21处可以被进一步的分为3个组:第一组(B1)包括‘马瑟兰’‘阿拉奈尔’‘Kolor’‘北冰红’‘丽红宝’‘无核白鸡心’‘无核翠宝’‘Fresno’和‘克瑞森无核’等9个品种,其中‘马瑟兰’和‘阿拉奈尔’起源于法国,‘丽红宝’和‘无核翠宝’与其共同亲本‘无核白鸡心’聚为一类;第二组(B2)包括‘玫瑰香’‘魏可’‘奥古斯特’‘维多利亚’‘爱神玫瑰’‘碧香无核’‘夏至红’‘瑞都无核怡’‘金田皇家无核’‘瑞都香玉’‘瑞都翠霞’‘沈农金皇后’12个品种,这12个品种均为鲜食品种,其中8个品种为我国选育的品种;第三组(B3)是‘秋红宝’。
第三大类(C组):包括‘希姆劳特’‘白香蕉’‘Neptune’和‘Jupiter’4个品种,全部为欧美杂种,且起源地均为美国。
3 讨论
3.1 DNA指纹数据建立和品种鉴别
根据每个引物扩增片段的大小,整理可以得到基于
6对SSR引物对43份葡萄种质扩增的DNA指纹数据。在葡萄引种和种质资源推广时,常会出现同物异名或同名异物等现象,相较于传统的形态学鉴定,SSR标记可以提高鉴别的准确性,如Vilanova等[17]利用SSR对25种生长在西班牙的葡萄品种进行鉴别,并对其中同名异物的品种进行了区分;利用不同引物扩增出来的片段大小的不同,可以实现品种区分,如杜晶晶[13]和王慧玲[14]等通过对各引物扩增的等位基因数进行赋值,构建葡萄种质的分子身份证对品种进行区分;除了分子身份证之外,还可以将DNA指纹数据按照各条带的有无转化成二元数据矩阵,但相关内容研究结果呈现主要是以“0, 1”或“+,-”的形式[15-16],本研究在二元数据的基础上进一步构建了基于SSR标记的指纹图谱,更加简单明了的实现了品种的区分。本研究结果显示,43个品种的指纹图谱各不相同,进一步表明了SSR指纹数据可以应用于对引种时错误命名情况的鉴别和纠正。
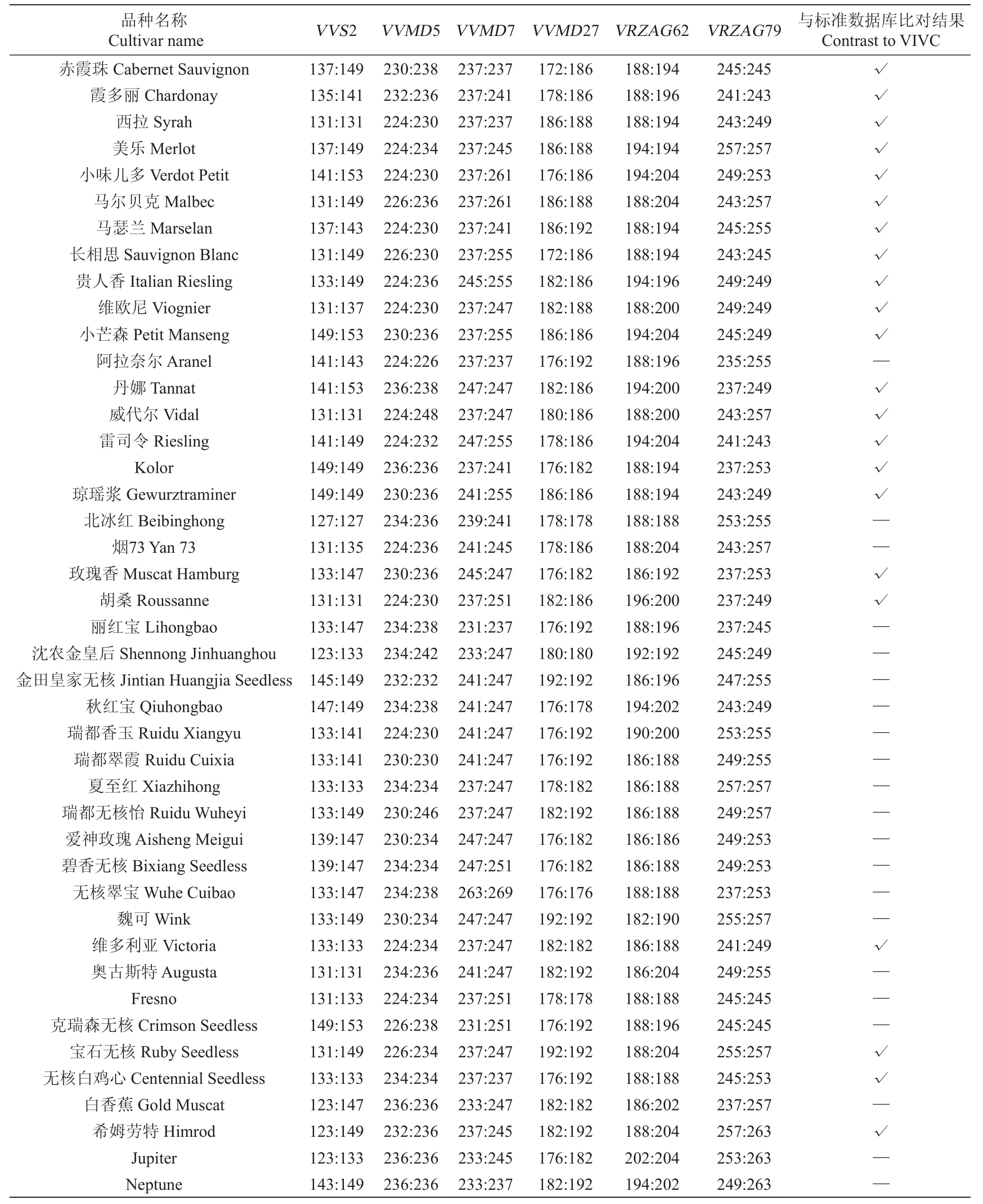
表4 43个葡萄品种在6个SSR位点的DNA指纹数据Table 4 DNA proɦle data of 43 cultivars at six SSR loci

图1 基于SSR标记构建的43份葡萄种质指纹图谱Figure 1 Molecular ɦngerprint of 43 grape cultivars based on SSR markers
除了对品种进行区分外,整理得到的DNA指纹数据,可以与VIVC标准数据库进行比对,本试验所研究的43个葡萄品种中,除未收录的品种外,22个品种的指纹数据均与标准数据库一致,表明利用SSR扩增得到的指纹数据可以对品种进行鉴定。
3.2 亲缘关系分析
本研究利用6对SSR引物扩增的数据,根据各品种之间的相似系数,采用非加权平均法对所有品种进行聚类,并对这43份鲜食与酿酒葡萄种质进行了亲缘关系分析。
从用途方面来看,通过聚类基本上可以将酿酒葡萄与鲜食葡萄区分开,如A组中除‘胡桑’(两用)和‘宝石无核’(鲜食)外,其余均为酿酒品种,而B、C两组中除‘玫瑰香’(两用)和‘北冰红’(酿酒)外,其余均为鲜食品种,这可能与性状连锁有关,即本文研究的6个SSR位点可能与某些决定葡萄果实品质的基因连锁,酿酒葡萄与鲜食葡萄由于果实大小、糖酸含量等性状的不同,可以被区分开来,如Leocirj等[18]利用SSR分子标记对欧亚种葡萄中与叶片形态有关的位点进行了定位;牛早柱[19]分析了葡萄果实中的糖、酸和香气物质,并把这些表型性状与SSR分子标记进行关联分析研究,找出了与上述物质合成显著相关联的位点。本试验中可能也是由于调控某些性状的基因与SSR位点连锁,从而将酿酒品种与鲜食品种区分开,这有待于进一步的研究。
从地理起源方面来看,A组的各个品种大多数起源于法国和德国,B组中大多是近年来我国育成的品种,其中也有少部分起源于欧洲国家的品种,由于亲缘关系相近而与我国选育的品种聚在一起,C组中则是起源于美国的品种,且均为欧美杂种。葡萄种群的形成与其起源地的地理及气候特征有密切的联系,前人[20-21]常利用花色苷或酚类物质的组成和含量作为化学指纹,结合多元统计分析对葡萄或葡萄酒的产地进行区分;Martínez等[22]利用SSR标记对葡萄品种进行聚类时发现聚类结果与形态学形状有关,但未与产地形成关联,因而未能区分秘鲁和阿根廷的品种。本试验研究结果表明,利用SSR标记可以较好的实现对葡萄起源地的区分,这启示我们可以将DNA指纹和化学指纹综合起来运用于葡萄起源地和产地的分析。此外,本研究中供试的酿酒葡萄大多起源于欧洲(法国、德国等),鲜食葡萄大多起源于亚洲东部(中国、日本等),所以本文中所论述的鲜食葡萄与酿酒葡萄被区分开,可能是起源地的差异和性状连锁综合作用的结果。
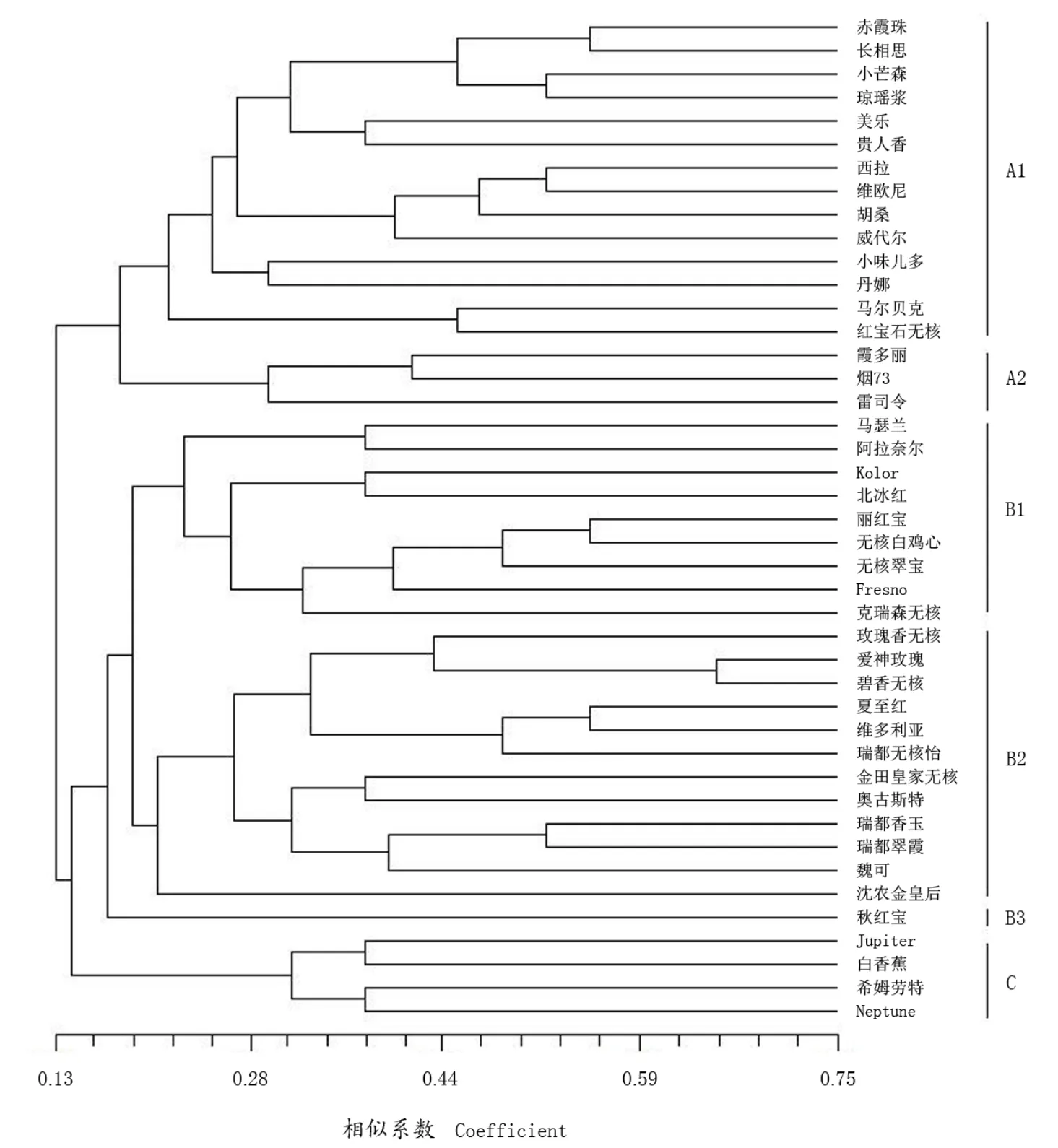
图2 基于SSR标记的43份葡萄种质聚类分析图Figure 2 Dendrogram of 43 grape cultivars based on SSR markers
从亲本关系方面来看,如前文所述,本研究发现多对子代与其亲本或拥有共同亲本的品种聚为一类的现象,这表明了SSR标记在亲缘关系分析中的作用。有些葡萄品种由于栽培历史悠久或栽培品种之间进行杂交和自交等,导致其亲缘关系不明确,SSR标记是一种研究亲缘关系的好方法,对SSR扩增结果进行分析,可以研究品种之间的亲缘关系以及寻找子代的亲本[23-24],如尹玲等[7]利用6对SSR引物的扩增结果,结合聚类分析确定了我国育成的品种昌黎21号的亲本为‘巨峰’ב早黑宝’,而不是此前认为的‘巨峰’ב玫瑰香’。此外,本研究中4个欧美杂种(C组)与欧亚种葡萄(A、B组)明显区分开,与李雪雁等[25]研究结果一致,说明我国葡萄种质资源中的欧亚种和欧美杂种存在明显区别。
4 结论
SSR标记是葡萄种质资源鉴定的有效方法,本文以43个酿酒和鲜食品种为研究对象,利用6对SSR引物对上述品种进行扩增并进行多态性分析,通过建立DNA指纹图谱对品种进行区分和鉴别,之后对所有品种进行聚类分析。结果表明:根据建立的DNA指纹数据,可以区分供试的葡萄品种,并可以通过与VIVC标准数据库比对实现品种鉴定;通过聚类分析基本上可以从用途和起源地两方面实现对各种质的区分,并可以有效的分析各种质之间的亲缘关系。本研究从分子水平上为葡萄种质资源的鉴别、遗传多样性和亲缘关系分析提供了参考价值。
[1]KELLER M. 葡萄学: 解剖学与生理学[M]. 王军, 段长青, 何非,等译. 北京: 科学出版社, 2016: 1-3.
[2]刘崇怀, 帖锋, 潘兴, 等. 无核葡萄品种资源性状的聚类分析[J].果树学报, 2006, 23(4): 531-533.
[3]FRAIGE K, PEREIRA-FILHO E R, CARRILHO E.Fingerprinting of anthocyanins from grapes produced in Brazil using HPLC-DAD-MS and exploratory analysis by principal component analysis[J]. Food Chemistry, 2014, 145(4): 395-403.
[4]GUSTAVO G, JORGE F, LAURA B, et al. Varietal differentiation of Tannat, Cabernet-Sauvignon and Merlot grapes and wines according to their anthocyanic composition[J]. European Food Research and Technology, 2007, 225(1): 111-117.
[5]刘闯萍, 王军. SSR标记及其在葡萄上的应用[J]. 果树学报,2008, 25(1): 93-101.
[6]DIAZ LOSADA E, TATO SALGADO A, RAMOS-CABRER A M, et al. Twenty microsatellites (SSRs) reveal two main origins of variability in grapevine cultivars from northwestern Spain[J].Vitis, 2010, 49(2): 55-62.
[7]尹玲, 张晨, 向江, 等. 我国新育成葡萄品种SSR指纹图谱的建立[J]. 果树学报, 2015, 32(3): 366-373.
[8]BOSCO S, MARCIDE J M O, BLANCO J L S, et al.Characterisation and identification of grapevine cultivars (Vitis vinifera L.) from northwestern Spain using microsatellite markers and ampelometric methods[J]. Vitis, 2005, 44(2): 67-72.
[9]ZULINI L, PETERLUNGER E, FABRO E. Characterisation of the grapevine cultivar Picolit by means of morphological descriptors and molecular markers[J]. Vitis, 2005, 44(1): 35-38.
[10]王军, 杨传平, 刘桂丰. 木本植物基因组DNA提取及鉴定[J].植物研究, 2006, 26(5): 589-594.
[11]THIS P, JUNG A, BOCCACCI P, et al. Development of a standard set of microsatellite reference alleles for identification of grape cultivars[J]. Theoretical and Applied Genetics, 2004,109(7): 1448-1458.
[12]NEI M. Analysis of gene diversity in subdivided populations[J].Proceedings of the National Academy of Sciences, 1973, 70(12):3321-3323.
[13]杜晶晶, 刘国银, 魏军亚, 等. 基于SSR标记构建葡萄种质资源分子身份证[J]. 植物研究, 2013, 33(2): 232-237.
[14]王慧玲, 闫爱玲, 孙磊,等. 13个中国葡萄优新品种的分子身份证构建[J]. 生物技术通报, 2016, 32(4): 137-142.
[15]SCIENZA A, VILLA P, TEDESCO G, et al. A chemotaxonomic investigation on Vitis vinifera L. II.Comparison among ssp.sativa traditional cultivars and wild biotypes of ssp. silvestris from various Italian regions[J]. Vitis, 1994, 33(4): 217-224.
[16]RGENER F, WIEDECK E, STADLBAUER A. Differentiation and identification of White Riesling clones by genetic markers[J]. Vitis, 2000, 39(3): 103-107.
[17]VILANOVA M, FUENTE M D L, FERNÁNDEZGONZÁLEZ M, et al. Identification of new synonymies in minority grapevine cultivars from Galicia (Spain) using microsatellite analysis[J].American Journal of Enology and Viticulture, 2009, 60(2): 236-240.
[18]LEOCIRJ W, NILGÜN G, MURAT A, et al. Genetic mapping and localization of quantitative trait loci affecting fungal disease resistance and leaf morphology in grapevine (Vitis vinifera L.)[J]. Molecular Breeding, 2007, 20(4): 359-374.
[19]牛早柱. 葡萄果实香气及糖酸物质的SSR标记关联分析[D].沈阳农业大学, 2016.
[20]VESNA R, GORAN S, MARICA-MEDIC S. Polyphenolic composition of Croatian wines with different geographical origins[J]. Food Chemistry, 2009, 115(1): 54-60.
[21]JAITZ L, SIEGL K, EDER R, et al. LC-MS/MS analysis of phenols for classification of red wine according to geographic origin, grape variety and vintage[J]. Food Chemistry, 2010,122(1): 366-372.
[22]MARTÍNEZ L E, CZVAGNARO P F, MASUELLI R W, et al.SSR-based assessment of genetic diversity in South American Vitis vinifera varieties[J]. Plant Science, 2006, 170(6): 1036-1044.
[23]VECCHI STARAZ M D, BANDINELLI R, BOSELLI M, et al. Genetic structuring and parentage analysis for evolutionary studies in grapevine: kin group and origin of the cultivar Sangiovese revealed[J]. Journal of the American Society for Horticultural Science, 2007, 132(132): 514-524.
[24]VOUILLAMOZ J, MAIGRE D, MEREDITH C P. Microsatellite analysis of ancient alpine grape cultivars: pedigree reconstruction of Vitis vinifera L. 'Cornalin du Valais'[J].Theoretical and Applied Genetics, 2003, 107(3): 448-454.
[25]李雪雁, 梁海永, 宪立杰, 等. 基于SSR技术对七十五份栽培葡萄品种的鉴定研究[J]. 北方园艺, 2015(13): 115-119.
