基于DNA双链取代策略免标记检测铅离子的研究
2018-01-25吕菊波张亚会孟凡斌
吕菊波,张亚会,孟凡斌,刘 刚,徐 慧
(鲁东大学 化学与材料科学学院, 山东 烟台 264025)
铅离子是一种常见的重金属离子,主要用于铅酸蓄电池、电缆和化工行业。铅离子可以通过生物富集作用被人体吸收,极少量的铅离子就会对人体健康和生物环境产生极大的危害[1-4]。所以寻求新的技术有效检测铅离子对于人类的健康和环境的保护显得十分重要。
目前常见的铅离子的检测方法主要有物理化学方法和生物传感器法等,物理化学方法主要有原子吸收光谱(AAS)[5-7]、电感耦合等离子体发射光谱(ICP-AES)[8-10]、阳极溶出伏安法(ASV)[11-13]、X射线荧光光谱法[14]等,这些方法操作要求高、仪器昂贵、对铅离子的检测不够敏感,不能满足较低浓度铅离子的检测需求。生物传感器法是通过生物分子与金属离子之间的特异性结合,定性、定量检测金属离子的方法,具有特异性强、灵敏度高、检测快速等优点。
功能核酸是一类具有特定生物功能的核酸分子,包括核酸适配体(Aptamer)[15-17]、脱氧核酶DNA酶[18-20]和蛋白酶[21]等,其功能远超出传统核酸的遗传作用。近年来功能核酸已被广泛应用于生物传感等研究领域,并对蛋白质、金属离子以及小分子等进行检测[22-25]。
1990年,核酸适配体首次被报道[26-27]之后,即被广泛应用在分析检测、分离、医疗诊断和生物成像等领域。Taghdisi等[28]基于荧光标记的铅离子核酸适配体和单壁碳纳米管(SWCNT)的超猝灭效应发展了一种超灵敏检测铅离子的方法。无铅离子存在时,荧光标记的核酸适配体吸附到SWCNT表面,荧光被猝灭。加入铅离子后,核酸适配体与铅离子结合,形成G-四面体/Pb2+复合物,不被SWCNT吸附,荧光增强。该传感器对Pb2+有很高的选择性,检出限达0.42 nmol/L。Lu等[29]发展了一种利用共轭高分子和核酸适配体高灵敏度检测铅离子的方法,当铅离子存在时,将诱导其特异性DNA形成四面体结构,共轭高分子缠绕在四面体上,溶液颜色由红色变成黄色,并且其最大吸收峰和荧光峰发生蓝移。用此方法肉眼可辨别出2 μmol/L的Pb2+。
此外,依赖Pb2+的DNA酶已广泛应用于Pb2+的检测,Li和Lu[30]设计了一种基于铅离子的8-17 DNA酶的荧光传感器检测铅离子。该传感策略在10 nmol/L~4 μmol/L内具有较好的动力学范围,在4 ℃下具有良好的选择性,但室温下背景荧光较高。为克服这个缺点,在基底DNA链的3'端引入分子内猝灭剂,可在室温下抑制背景荧光[31],除此之外,在8-17 DNA酶中引入错配防止温度引起的变化[32],使用GR-5 DNA酶代替8-17 DNA酶[33],可以获得更好的选择性。而将基于铅离子的8-17 DNA酶的基底DNA链设计成分子信标,可以有效猝灭荧光强度[34],当存在铅离子时,分子信标的环部分可被8-17 DNA酶裂解,荧光基团与猝灭剂分离,荧光增强,检出限达到0.6 nmol/L。Guo等[35]设计了一种能同时检测汞离子、铅离子和铜离子的生物传感方法,将这3种金属离子分别加入到木瓜酶包裹的纳米金溶液中,三者与木瓜酶结合,使纳米金聚集,溶液的颜色由酒红色转变为蓝色。且汞离子与木瓜酶结合能力较强,溶液颜色变化更加明显,具有较好的选择性,检测限为200 μmol/L。
上述方法大多需荧光标记、合成高分子、纳米材料辅助检测或操作繁琐,因此亟需发展简单、快速、免标记且高灵敏度的检测铅离子的传感方法。本文基于DNA双链取代的策略,首先,适配体与其互补链结合形成稳定的双链DNA结构,无Pb2+时,向溶液中加入SYBR Green I(SG),SG能够插入双链DNA,使荧光强度明显增强;当溶液中加入铅离子后,铅离子与适配体特异性结合,并形成稳定的G-四面体结构,双链结构被打开。铅离子的加入使溶液中双链DNA的数量减少,体系的荧光强度降低,因而可以定量检测铅离子。
1 实验部分
1.1 试剂与仪器
铅离子特异性DNA(Pb aptamer)、其部分互补链DNA(com)、另一对完全互补的单链DNA1和DNA2,所有DNA均由Takara公司合成并经高效液相色谱(HPLC)纯化,所用DNA序列有:Pb aptamer:(5’-GGGTGGGTGGGTGGGT-3’);com:(5’-CACCCTCCCAC-3’);DNA1:(5’-CCG TCA GAA GTT CCT TTG CCG(A)9-3’);DNA2:(5’-CGG CAA AGG AAC TTC TGA CGG TTCC-3’)。Sybr Green Ⅰ购于Inviotrogen公司,母液浓度为10 000×(19.6 mmol/L)[36],使用时用二甲亚砜(DMSO)稀释至所需浓度。Pb2+、K+、Na+、Ca2+、Mg2+、Cu2+、Zn2+、Cd2+、Hg2+的水溶液由其硝酸盐配制,所有溶液均用18.25 MΩ·cm的超纯水配制而成。二甲亚砜(国药化学试剂有限公司),所有试剂均为分析纯。杂交缓冲液(杂交Buffer):10 mmol/L Tris-HAc(0.2 mol/L NaAc),pH 7.5。
LS-55荧光分光光度计(美国Perkins Elmer仪器有限公司)测试条件为:脉冲式氙灯激发,激发波长为490 nm,荧光光谱的扫描范围为495~600 nm,激发和发射狭缝宽度均为8 nm,用宽度10 mm石英比色皿进行测量,样品体积2 mL。
1.2 实验方法
1.2.1SG浓度的优化通过改变染料碱基对比例(即染料与碱基对的比值dbpr),进行染料用量优化。首先固定DNA的浓度为20 nmol/L,改变染料的浓度,其中dbpr分别为0.1、0.3、0.57、0.9、1.2,通过检测加入终浓度为500 nmol/L铅离子前后荧光强度的变化,确定最佳染料浓度。
1.2.2反应时间的优化在2 mL杂交Buffer体系中加入20 μL 2 μmol/L Pb aptamer和com,两DNA终浓度为20 nmol/L,混合均匀,32 ℃反应30 min。然后加入10 μL 20 μmol/L的铅离子,终浓度为100 nmol/L,32 ℃反应不同时间。按照dbpr为0.57加入23.26 μL 的9.8 μmol/L SG,反应5 min,测定荧光强度。
1.2.3最佳pH值与离子强度的优化在2 mL不同pH值的杂交Buffer(pH 7.3、7.5、7.8、8.0)或10 mmol/L Tris-HAc(含不同NaAc)的杂交Buffer中,加入20 μL 2 μmol/L Pb aptamer和com,两DNA终浓度为20 nmol/L,混合均匀,32 ℃反应30 min。然后加入10 μL 100 μmol/L的铅离子,终浓度为500 nmol/L,32 ℃反应不同时间。按照dbpr为0.57加入23.26 μL 的9.8 μmol/L SG,反应5 min,测定荧光强度。
1.2.4灵敏度分析在2 mL 杂交Buffer体系中加入20 μL 2 μmol/L Pb aptamer和com,两DNA终浓度为20 nmol/L,混合均匀,在32 ℃下反应30 min。加入不同浓度的铅离子,32 ℃下反应2 h。按照dbpr为0.57加入23.26 μL 的9.8 μmol/L SG,反应5 min,测定荧光强度。
1.2.5铅离子对SG荧光强度的影响在2 mL 杂交Buffer体系中加入10 μL 4 μmol/L DNA1和DNA2,两DNA终浓度为20 nmol/L,混合均匀,32 ℃反应30 min。加入不同浓度的铅离子,32 ℃反应2 h,按照dbpr为0.57加入9.77 μL 的49.0 μmol/L SG,反应5 min,测定荧光强度。
1.2.6选择性实验在2 mL 杂交Buffer体系中加入20 μL 2 μmol/L Pb aptamer和com,两DNA终浓度为20 nmol/L,混合均匀,32 ℃反应30 min。加入10 μL 100 μmol/L终浓度为500 nmol/L的不同金属离子,32 ℃反应2 h。按照dbpr为0.57加入23.26 μL 9.8 μmol/L SG,反应5 min,测定荧光强度。
以上所有实验平行3组,作误差棒分析。
2 结果与讨论
2.1 实验原理

图1 铅离子检测原理Fig.1 Principle for detection of lead ion
本实验基于DNA双链取代的策略,核酸适配体可以与靶向分子特异性结合,改变适配体的构象,而荧光染料SG能够插入双链DNA使其荧光强度明显增强,基于此设计了一种检测铅离子的生物传感策略,检测原理如图1所示。铅离子适配体首先与其互补链杂交形成稳定的双链DNA结构,此时溶液中无Pb2+,向溶液中加入SG,SG能够插入双链DNA使SG的荧光强度明显增强,此时体系的荧光强度较高。当溶液中加入铅离子时,铅离子与其适配体特异性结合,形成稳定的G-四面体DNA结构,双链结构被打开,双链数量减少,体系的荧光强度降低,荧光强度的降低量与铅离子的加入量相关,因此,这种方法可用以定性、定量检测铅离子,且方法简单快速,无需荧光标记DNA。
2.2 SG浓度的优化
SG能够插入双链DNA使其荧光强度明显增强,若SG用量太少则不能完全插入双链DNA,而用量太多又会使多余的SG游离在溶液中,增强检测的背景荧光,因此选择合适的SG用量对生物传感器的研究相当重要。实验通过不同的染料碱基对比例(dbpr)与加入铅离子前后荧光强度比值(F0/F)的关系对SG浓度进行优化。dbpr分别为0.1、0.3、0.57、0.9、1.2,F0表示未加铅离子时的荧光强度,F表示加入铅离子时的荧光强度。结果显示,随着dbpr的增加,F0/F呈先增大后减小的趋势,当dbpr为0.57时F0/F最大,说明此时SG的用量最佳,因此选择dbpr为0.57,即加入23.26 μL 的9.8 μmol/L SG。
2.3 反应时间的优化
固定铅离子的终浓度为100 nmol/L,考察了不同的反应时间(0、1、3、5、10 min)条件下体系的荧光强度。结果表明,随着反应时间的增加,体系的荧光强度逐渐降低,并在反应0~1 min时降幅最大,当反应时间为5 min时体系的荧光强度趋于平稳。因此,铅离子的最佳反应时间为5 min。
2.4 pH值与离子强度的优化
溶液的pH值和离子强度会影响DNA的杂交、反应动力学和SG的荧光强度,因此实验考察了pH值和离子强度的影响。F0表示未加Pb2+时的荧光强度,F表示加入500 nmol/L Pb2+时的荧光强度。F0/F在生理pH值(7.3~8.0)附近改变很小,因此选择pH 7.5为反应的最佳酸度。此外,F0/F随NaAc浓度的增加而增加,当NaAc浓度大于0.2 mol/L后F0/F保持不变。因此,最佳离子强度为0.2 mol/L NaAc。
2.5 灵敏度分析
在最优实验条件下,通过检测体系中荧光强度随铅离子浓度的变化,得到荧光强度与铅离子浓度变化的光谱图和关系图(图2)。由图2可见,随着铅离子浓度的不断增加,体系的荧光强度不断减小。这是由于随着铅离子的不断加入,铅离子与其适配体特异性结合,双链DNA数量减少,而SG从双链DNA上释放出来,体系的荧光强度减弱。
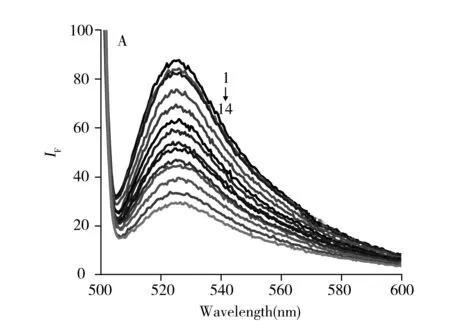

图2 不同浓度铅离子存在时的荧光光谱图(A)以及铅离子浓度与荧光强度的关系(B)Fig.2 The fluorescence spectra of different concentrations of Pb2+(A) and the relationship between Pb2+ concentration and fluorescence intensity(B)1-14:0,2,5,20,50,70,100,200,300,400,500,1 000,2 000,5 000 nmol/L
分析发现体系的荧光强度(Y)与铅离子浓度(X,nmoI/L)之间呈良好的线性关系。其中,当铅离子浓度为2~100 nmol/L时,线性方程为Y=83.22-0.26X,r2=0.991 4;当铅离子浓度为100~500 nmol/L时,线性方程为Y=60.80-0.03X,r2=0.969 2。结果显示,该方法可以用于铅离子的定量检测,检出限(S/N=3)为1.6 nmol/L。
2.6 铅离子对SG荧光的影响
实验设计了1对与铅离子不能结合的两条互补链DNA分别取代Pb aptamer和com,通过加入不同量的铅离子和SG检测体系的荧光强度。结果显示,随着铅离子的不断增加,体系的荧光强度基本保持不变。其中DNA1和DNA2是完全互补的两条单链DNA,铅离子不能与任何一条单链DNA作用打开双链DNA,说明铅离子对SG的荧光强度不会产生猝灭作用,而本实验荧光强度的降低是由于铅离子与其适配体结合打开双链DNA,使体系荧光强度降低,铅离子对SG的荧光强度没有影响。
2.7 选择性分析

图3 选择性实验Fig.3 Experiments for selection
实验分别向杂交Buffer中加入500 nmol/L的不同金属离子,通过检测体系的荧光强度,考察铅离子的选择性(图3)。由图3可知,只有当铅离子存在时,荧光强度才有非常明显的降低,说明此实验只对铅离子具有良好选择性,对其他金属离子影响不大,适合用于铅离子检测。
2.8 实际样品检测
为验证该传感器在实际样品检测中的可靠性,选取本实验室的自来水进行了加标回收实验。实际水样用反应的缓冲溶液进行稀释,然后加入不同浓度的铅离子,采用本生物传感器进行检测。如表1所示,加入10、100、200 nmol/L铅离子时,实际样品的回收率为92.5%~108%,相对标准偏差(RSD)为2.5%~8.6%,方法显示了在实际样品检测中的良好性能。

表1 实际样品检测结果Table 1 The results of real sample
3 结 论
本文设计了一种基于DNA双链取代的策略和SG作为荧光染料插入剂区别单双链进行铅离子检测的荧光传感方法。研究发现体系的荧光强度变化由铅离子加入引起。在最佳条件下,铅离子浓度与荧光强度分段呈现良好的线性关系,最低检测浓度为2 nmol/L,检出限(S/N=3)为1.6 nmol/L。本方法具有灵敏度高、特异性强、简便快速等优点,而且采用无标记的方法,降低了检测成本,适用于实际样品的检测。
[1] Needleman H.Annu.Rev.Med.,2004,55(55):209-222.
[2] Peng C F,Xie Z J,Wang Y Y,Song S S.J.Instrum.Anal.(彭池方,谢正军,汪雅云,宋姗姗.分析测试学报),2014,33(10):1194-1198.
[3] Ji T Y,Xu L R,Zhou P.J.Instrum.Anal.(戢太云,徐鲁荣,周培.分析测试学报),2010,29(1):51-54.
[4] Lee Y F,Huang C C.ACSAppl.Mater.Interfaces,2011,3(7):2747-2754.
[5] Rivaro P,Ianni C,Soggia F,Frache R.Microchim.Acta,2007,158(3/4):345-352.

[7] Ceccarelli C,Picón A R,Mariangel P P,Greaves E D.Pet.Sci.Technol.,2000,18(9/10):1055-1075.
[8] Zougagh M,Garcia de Torres A,Vereda Alonso E,Cano Pavon J M.Talanta,2004,62(3):503-510.
[9] AlRashdan A,Heitkemper D,Caruso J A.J.Chromatogr.Sci.,1991,29(3):98-102.
[10] Abdi M M,Abdullah L C,Sadrolhosseini A R,Wan M M Y,Moksin M M,Tahir P M.PlosOne,2011,6(9):e24578.
[11] Keebaugh S,Kalkan A K,Nam W J,Fonash S J.Electrochem.Solid-StateLett.,2006,9(9):H88-H91.
[12] Zejli H,Hidalgo-Hidalgo de Cisneros J L,Naranjo-Rodriguez I,Elbouhouti H,Choukairi M,Bouchta D,Temsamani K R.Anal.Lett.,2007,40(14):2788-2798.
[13] Zhu Z Q,Su Y Y,Li J,Li D,Zhang J,Song S P,Zhao Y,Li G X,Fan C H.Anal.Chem.,2009,81(18):7660-7666.
[14] Nie D D,Wu H Y,Zheng Q S,Guo L Q,Ye P R,Hao Y L,Li Y N,Fu F F,Guo Y H.Chem.Commun.,2012,48(8):1150-1152.
[15] Qian Z S,Shan X Y,Chai L J,Chen J R,Feng H.Biosens.Bioelectron.,2015,68:225-231.
[16] Zhang D P,Yin L,Meng Z H,Yu A C,Guo L H,Wang H L.Anal.Chim.Acta,2014,812:161-167.
[17] Guo L Q,Nie D D,Qiu C Y,Zheng Q S,Wu H Y,Ye P R,Hao Y L,Fu F F,Chen G.Biosens.Bioelectron.,2012,35(1):123-127.
[18] Wang F L,Wu Z,Lu Y X,Wang J,Jiang J H,Yu R Q.Anal.Biochem.,2010,405(2):168-173.
[19] Liang G,Man Y,Li A,Jin X X,Liu X H,Pan L G.Microchem.J.,2017,131:145-153.
[20] Zhang H X,Jiang B Y,Xiang Y,Su J,Chai Y Q,Yuan R.Biosens.Bioelectron.,2011,28(1):135-138.
[21] Guo Y M,Wang Z,Qu W S,Shao H W,Jiang X Y.Biosens.Bioelectron.,2011,26(10):4064-4069.
[22] Xu H,Gao S L,Wang L H.Chem.Sens.(徐慧,高淑丽,王丽华.化学传感器),2010,30(1):13-24.
[23] Eissa S,Zourob M.Biosens.Bioelectron.,2017,91:169-174.
[24] Taghdisi S M,Danesh N M,Lavaee P,Ramezani M,Abnous K.Sens.Actuators.B,2016,234:462-469.
[25] Dolati S,Ramezani M,Abnous K,Taghdisi S M.Sens.Actuators.B,2017,246:864-878.
[26] Tuerk C,Gold L.Science,1990,249(4968):505-510.
[27] Ellington A D,Szostak J W.Nature,1990,346(6287):818-822.
[28] Taghdisi S M,Emrani S S,Tabrizian K,Ramezani M,Abnous K,Emrani A S.Environ.Toxicol.Pharmacol.,2014,37(3):1236-1242.
[29] Lu Y,Li X,Wang G K,Tang W.Biosens.Bioelectron.,2013,39(1):231-235.
[30] Li J,Lu Y.J.Am.Chem.Soc.,2000,122(42):10466-10467.
[31] Liu J W,Lu Y.Anal.Chem.,2003,75(23):6666-6672.
[32] Nagraj N,Liu J,Sterling S,Wu J,Lu Y.Chem.Commun.,2009,27(27):4103-4105.
[33] Lan T,Furuya K,Lu Y.Chem.Commun.,2010,46(22):3896-3898.
[34] Zhang X B,Wang Z,Xing H,Xiang Y,Lu Y.Anal.Chem.,2010,82(12):5005-5011.
[35] Guo Y M,Wang Z,Qu W S,Shao H W,Jiang X Y.Biosens.Bioelectron.,2011,26(10):4064-4069.
[36] Zipper H,Brunner H,Vitzthum F.NucleicAcidsRes.,2004,32(12):e103.
