浙江近海鰓属鱼类形态描述及中国鰓属鱼类分子系统发育分析
2018-01-24肖家光张少秋高天翔韩志强
肖家光 张少秋 高天翔, 韩志强
(1. 中国海洋大学水产学院, 青岛 266003; 2. 浙江海洋大学水产学院, 舟山 316000; 3. 浙江省海洋渔业资源可持续利用技术研究重点实验室, 舟山 316021)
物种的准确鉴定是开展渔业资源调查和管理以及渔业生物多样性研究的基础, 也是海洋生物多样性保护的重要内容。由于传统形态学鉴定的局限性和形态分类学家队伍的持续缩减, 海洋生物分类学的发展举步维艰。近年来, 随着分子生物学技术的进步和发展, DNA条形码技术(DNA barcoding)凭借其简单、快速、准确的优点, 逐渐成为生物分类研究中的热点[1]。与传统形态学相比, DNA条形码在揭示隐存种、鉴定缺少形态数据或形态不易区分的物种方面优势明显[2—4]。线粒体DNA的COⅠ基因长度适宜、进化速率慢且富含系统发育的信号, 大多数动物的COⅠ基因都能被通用引物所扩增, 因此常被选作物种鉴定的条形码[5—8]。
鲵属(Sillago)鱼类隶属硬骨鱼纲(Osteichthyes)、鲈形目(Perciformes)、鲈亚目(Percoidei)、鲵科(Sillaginidae), 广泛分布于印度洋-西太平洋近岸水域,具有较高的经济价值[9,10]。目前, 全世界发布并描述鲵属鱼类33种, 中国沿海分布有12种, 其中较常见的有多鳞鲵Sillago sihama、少鳞鲵Sillago japonica、斑鲵Sillago aeolus等[4,9,11—13]。由于鲵属鱼类形态和体色相近, 使得该属鱼类的分类一直存在诸多问题。澳大利亚鱼类学家McKay和日本的学者Kaga曾根据鱼鳔及骨骼等形态特征对鲵属鱼类的分类及系统进化进行了较为详细的研究[14,15], 但近缘类群以及部分种属界限仍难以确定, 且以外部形态特征建立的分类系统迄今仍存有争议。近年来随着分子生物技术的成熟, 越来越多的鲵属新种及隐存种被发掘(Sillago caudiculaKaga, Imamura& Nakaya, 2010;Sillago sinicaGao & Xue, 2011;Sillago suezensisGolani, Fricke & Tikochinski, 2013;Sillago shaoiGao & Xiao, 2016)[4,11—13]。这些新种分布较广, 并都曾被误认为多鳞鲵。有关浙江近海的鲵属鱼类一直缺乏系统整理, 诸多鱼类志和文献中记载有多鳞鲵和少鳞鲵两种[16—18]。本研究采集了浙江近海的411尾鲵属鱼类样品, 结合传统形态学和DNA条形码对其物种组成、序列差异和系统发育关系进行了探究, 以期为浙江近海鲵属鱼类分类及资源保护提供更多的基础资料。
1 材料与方法
1.1 实验材料
研究所用鲵鱼样品共计411尾, 其中229尾于2016年5月8日采自温州近海, 舟山近海182尾鲵鱼于2016年6月14日采集(图1)。所有样品经形态学分类鉴定后取肌肉组织于95%酒精中固定, 样品现保存于浙江海洋大学渔业生态与生物多样性实验室。

图1 鲵鱼采样地点示意图Fig. 1 Sampling sites of Sillago species
1.2 实验方法
形态学研究对所有样品的主要可数与可量性状进行测定并观察其形态特征。其中可数性状包括: 背鳍鳍条、臀鳍鳍条、胸鳍鳍条、腹鳍鳍条、尾鳍鳍条、第一鳃弓鳃耙、侧线鳞、侧线上鳞和脊椎骨; 可量性状包括: 体长、头长、眼后头长、吻长、眼径、眼间距、尾柄长和尾柄高。所有可量性状均由游标卡尺测得并精确至0.1 mm。电子天平称取样品体重(精确至0.1 g)后解剖观察鱼鳔特征。
遗传学研究经形态鉴定后各种取适量肌肉组织, 采用酚-氯仿方法[19]提取基因组DNA, 将乙醇沉淀后的基因组DNA溶解于100 μL蒸馏水中,4℃保存备用。用于扩增线粒体DNACOⅠ片段的引物为L5956-COⅠ: 5′-CACAAAGACATTGGC ACCCT-3′; H6558-COⅠ: 5′-CCTCCTGCAGGGTCA AAGAA-3′[20]。PCR反应体系为25 μL, 包括1.25 UTaqDNA聚合酶, 各种反应组分的终浓度为200 nmol/L的正反向引物; 200 μmol/L的dNTP, 10 mmol/L Tris (pH 8.3), 50 mmol/L KCl和1.5 mmol/L MgCl2;反应条件为94℃预变性5min; 94℃变性45s, 50℃退火45s, 72℃延伸45s, 35个循环; 72℃延伸10min。以上反应均设阴性对照以排除DNA污染的情况。取2 μL PCR扩增产物进行1%琼脂糖凝胶电泳检测(U=5 V/cm), 送上海桑尼生物科技有限公司进行双向测序。
1.3 数据处理
将测得的COⅠ基因片段序列用DNAStar(DNASTAR, Inc)软件包中的Seqman软件进行比对,并辅以人工校正。使用MEGA 5.05计算所得序列的碱基组成、序列间的碱基变异频率、转换颠换频率及其比率; 并基于Kimura-2-parameter (K2P)双参数模型计算种间及各种内的遗传距离, 采用邻接法(Neighbour-Joining, NJ)构建分子系统发育树, 经1000次重复抽样(Bootstraps)检测其置信度。选取中国近海常见鲵属鱼种作为比较并扩增其COⅠ基因序列, 与本研究所检测的2种鲵属鱼类共计7种45个个体的COⅠ基因同源序列进行系统发育分析,另选取同科斑似鲵属斑似鲵(Sillaginodes punctatus)的同源序列作为外群。本研究所分析物种的COⅠ基因及其相关信息见表 1。
2 结果
2.1 形态描述
实验测得舟山近海182个个体都为少鳞鲵。其主要形态特征如图2A: 体呈长圆柱形, 稍侧扁。吻部尖细, 背部稍隆起, 腹侧稍平直; 尾部细长, 尾柄较短。头中大, 颊部宽大; 眼睛较小, 位于头部两侧中上部, 眼上缘与头部背缘平齐。头背侧眼前部两侧各具一鼻孔, 分离。口小, 前位, 上下颌前端近平齐。体背部青灰色, 头部背侧深色, 腹部近于白色。体侧中部具一模糊锯齿状银色条带; 第一及第二背鳍近似透明, 背鳍前几个棘之间具有黑色小点,第二背鳍边缘具黑色小点; 胸鳍透明, 胸鳍基部银白色不具有黑斑; 腹鳍和臀鳍浅白色或近似透明;尾叉较浅, 尾鳍深灰色, 边缘偏黑。第一背鳍具X—XII硬棘; 第二背鳍具I硬棘及21—23软条, 臀鳍具II硬棘及21—24软条, 胸鳍具14—15软条, 腹鳍具I硬棘及5软条; 第一鳃弓外鳃耙3—5/7—10; 侧线上鳞3—4, 侧线下鳞8—10, 具68—73侧线鳞; 脊椎骨数34—36。
实验测得温州近海229个个体都为中国鲵。其主要形态特征如图2B: 体细长, 略侧扁, 吻尖细, 背部稍隆起, 腹部相对平直。中国鲵整体呈黄褐色,背侧颜色深褐色, 腹部渐银白色。背鳍2个, 第一背鳍起点在胸鳍基部后上方, 具X—XII硬棘, 鳍膜上具有排列不规则的黑色小斑点; 第二背鳍起始于体中央, 由I根硬棘和20—22根软鳍条组成, 沿鳍条有3—4行规则排列的黑色小斑点; 臀鳍浅黄色, 与第二背鳍相对, 具有II硬棘和20—24根软条, 鳍膜上具细小黑点; 胸鳍细长, 向斜后方延伸, 腹鳍略呈三角形; 尾叉较浅, 尾鳍上下缘密布黑点。侧线上鳞5—6, 侧线鳞75—80; 脊椎骨37—39。
本研究对浙江近海少鳞鲵和中国鲵的主要可数与可量性状做了比较(表 2), 并与参考文献中的描述相对比。少鳞鲵头长为体长的26.38%—28.6%,吻长为头长的36.1%—42.9%, 眼径为头长的16.4%—21.3%, 眼间距为头长的14.3%—19.1%, 尾柄高为体长的5.6%—6.8%, 尾柄长为体长的5.5%—9.5%, 胸鳍长为体长的12.4%—18.6%, 腹鳍长为体长的11.9%—18.5%。中国鲵头长为体长的23.5%—27.6%, 吻长为头长的40.4%—46.1%, 眼径为头长的14.8%—22.7%, 眼间距为头长的20.0%—27.3%, 尾柄高为体长的6.2%—7.4%, 尾柄长为体长的7.9%—12.5%, 胸鳍长为体长的12.0%—22.1%,腹鳍长为体长的11.4%—15.7%。
少鳞鲵鱼鳔与中国鲵鱼鳔形态存在明显差异。少鳞鲵鱼鳔(图3A)具一个前部延伸, 位于鳔体前端中间, 与枕骨相连; 左右各具一前外侧延伸, 皆伸向前部且一般比前部延伸略短; 具一个后部延伸, 细长并向尾部延伸; 具一小管与肛门相连。中国鲵鱼鳔(图3B)具两个前部延伸, 伸向前部且与枕骨相连; 两侧各具一前外侧延伸, 前外侧延伸分前后两部分, 前部向体前部延伸, 纤细, 与前部延伸等长或略长, 后部沿鳔体向后延伸, 长度因个体而异,达鳔体长的1/3—2/3, 且延伸上具向外侧的小刺状结构, 数目不定; 具两个后部延伸, 紧密并排向尾部延伸, 一般一个略长于另一个, 且两后部延伸基部连接紧密; 具一小管与肛门相连。

表 1 本研究中鰓属7种鱼类的COⅠ基因信息Tab. 1 Information of COⅠgenes of 7 Sillago species in this study

图2 少鳞鲵(A)和中国鲵(B)Fig. 2 Sillago japonica (A) and Sillago sinica (B)
2.2 COⅠ基因序列特征及其变异
通过PCR扩增获得鲵属7种45个个体的COⅠ基因序列, 经测序比对得到同源片段序列600 bp, 编码氨基酸200个, 没有碱基插入和缺失, 序列长度上不存在变异。全部位点中变异位点有193个, 占32.17%; 简约信息位点有192个, 占32%; 不变位点有407个, 占67.83%。平均碱基组成为A: 23.2%、T: 28.4%、C: 28.5%、G: 19.9%, A+T含量(51.6%)高于G+C含量(48.4%)。符合线粒体编码基因核苷酸碱基构成的基本特征[7]。其中, 密码子第一位点G+C含量最高55.8%, 第二位点G+C含量为43.9%,第三位点G+C含量为45.6%。

表 2 少鳞鰓与中国鰓主要可数与可量性状比较Tab. 2 The countable and measurable characters of Sillago japonica and S. sinica
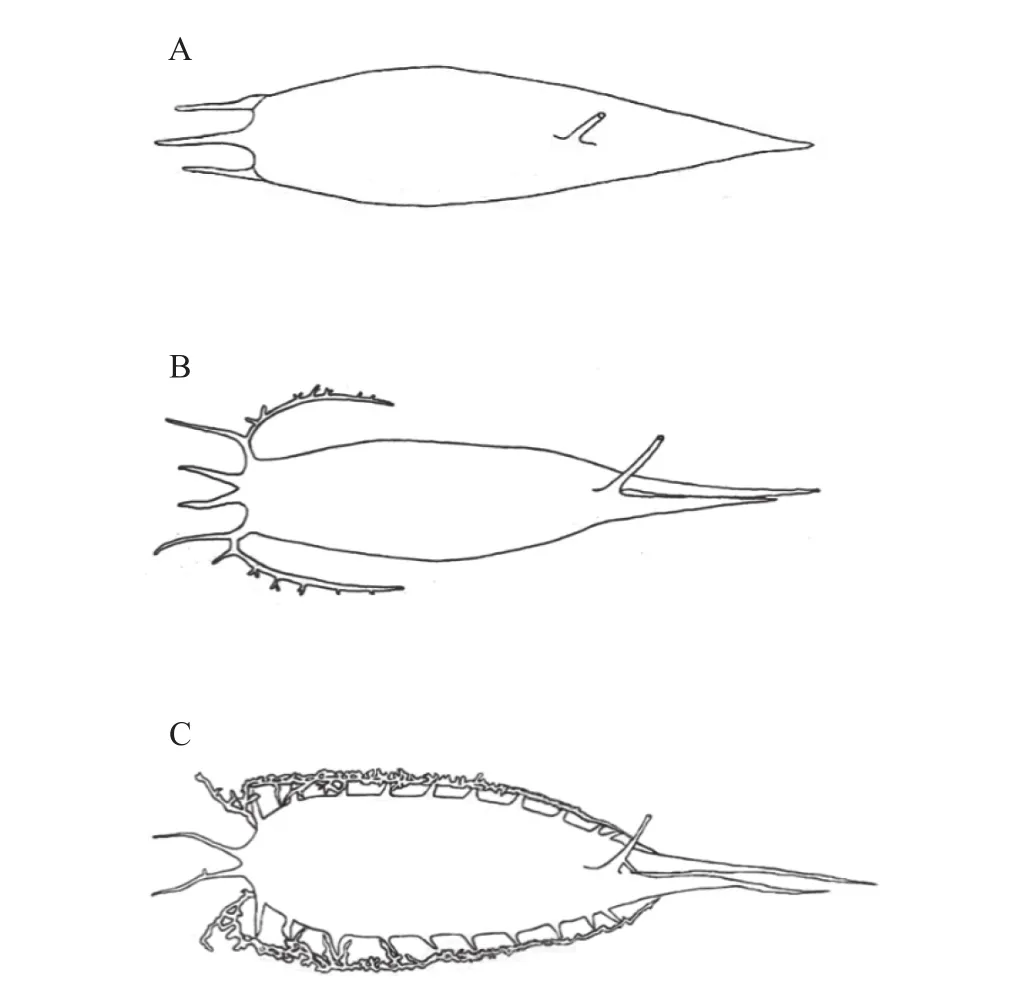
图3 少鳞鲵鱼鳔(A)、中国鲵鱼鳔(B)和多鳞鲵鱼鳔(C)Fig. 3 Swim bladder of Sillago japonica (A), S. sinica (B) and S.sihama (C)
COⅠ序列核苷酸变异情况: 其中保守位点512个, 转换位点56个, 颠换位点32个。其中第二位点最为保守, 第三位点变异最大。鲵属7种鱼类COⅠ基因片段GC含量差异情况: 其中邵氏鲵G+C含量最高(51.1%), 湾鲵G+C含量最低(45.1%)。各种均为密码子第一位点G+C含量最高。
2.3 种内、种间遗传距离及系统发育
采用MEGA 5.05, 基于Kimura-2-parameter计算7种鲵属鱼类的种内和种间遗传距离(表 3)。结果表明, 7种鲵属鱼类种内遗传距离为0.000—0.003,种间遗传距离为0.119—0.223, 种间遗传距离远远大于种内遗传距离。其中最大遗传距离出现在少鳞鲵和斑鲵之间, 最小遗传距离出现在中国鲵和邵氏鲵之间。根据Hebert等[1]提出的物种鉴定最小种间遗传距离标准(0.020), 使用COⅠ条形码所识别出的浙江近海少鳞鲵和中国鲵鉴定准确, 且两者之间的遗传距离为0.211。
基于COⅠ基因片段构建的7种鲵属鱼类的NJ系统发育树(图4)显示, 所有物种均聚为独立分支, 且都具有较高的节点支持率。舟山近海少鳞鲵与青岛胶南的少鳞鲵聚在一起, 且有共享单倍型;温州近海中国鲵与东营近海的中国鲵聚为一支, 共享一个单倍型。此外, 中国鲵先跟邵氏鲵聚类, 然后再和少鳞鲵聚在一起, 而与形态相近的多鳞鲵关系较远。

表 3 鰓属7种鱼类的种间遗传距离(对角线以下)和种内遗传距离(对角线)Tab. 3 Net genetic distance (K2P) between (blow the diagonal) and within (on the diagonal) in 7 Sillago species

图4 基于COⅠ基因构建的鲵属鱼类NJ系统发育树Fig. 4 Phylogenetic tree estimated by the Neighbor-Joining method based on COⅠ sequence data of 7 Sillago species
3 讨论
本研究所用温州和舟山近海411尾鲵鱼样品中,仅出现了少鳞鲵和中国鲵两种, 并未发现文献中记载的多鳞鲵[16]及其他鲵属鱼种。事实上, 多鳞鲵作为鲵属中分布最为广泛的种类, 其分布范围遍及印度洋及西太平洋沿岸[9,14], 国内许多文献记载多鳞鲵在我国沿海均有分布[21—24]。然而, 有关多鳞鲵的原始描述模糊不清, 形态指标与同属其他种类互有交叉, 缺乏详细的鱼鳔描述, 这使得鉴定该种时会出现很大误差。近年来新发现的鲵属鱼种S. caudicula、S. sinica、S. suezensis和S. shaoi无一不是和多鳞鲵混在一起[4,11—13]。此外, 近年来我们一直在中国沿海广泛采集鲵属鱼类样品, 结果发现在我国福建晋江以北海域未发现多鳞鲵分布[25]。所以,广泛分布的多鳞鲵极有可能包含多个隐存种; 在我国, 真正的多鳞鲵仅分布于福建晋江以南海域。少鳞鲵作为一种分布范围较广的鲵属鱼类, 在我国黄渤海、东海及南海均有分布[9], 其形态特征与多鳞鲵有较明显不同: 少鳞鲵侧线上鳞3—4(不会达到5), 而多鳞鲵侧线上鳞5—6。中国鲵作为2011年发布的新种, 可以根据侧线鳞和脊椎骨数与多鳞鲵区分开来: 中国鲵侧线鳞75—80, 脊椎骨37—39; 多鳞鲵侧线鳞68—72, 脊椎骨34—35; 且三种鲵鱼的鱼鳔差异极明显(图3)。
DNA条形码技术自提出至今已被广泛应用于鸟类、蜘蛛、鱼类等众多生物类群中[7,26,27]。该技术很好地克服了传统的形态学分类存在的缺陷, 已成为生物分类学研究中引人注目的新方向和研究热点。本研究中7种鲵属鱼类均可以形成单系群,且各物种种间遗传距离明显大于种内的遗传距离,这符合DNA条形码技术鉴定物种的标准和前提[24]。最小遗传距离出现在中国鲵和邵氏鲵之间(0.119),远远大于Hebert等[1]2003年通过研究DNA条形码确定的基于COⅠ基因进行物种鉴定的遗传距离阀值(0.020)。由此证明COⅠ基因序列作为条形码, 能够用于鲵属鱼类的有效物种鉴定。分子系统发育树显示, 中国鲵先跟邵氏鲵聚类, 然后和少鳞鲵聚在一起, 而和形态相似的多鳞鲵亲缘关系较远, 这和传统形态学分类的结果并不一致。事实上, 依据传统形态学得到的系统发育关系一直备受争议。Kaga[15]基于解剖学进行了详细的鲵科鱼类系统进化关系研究, 其结果与基于分子标记的分子系统发育树也并不一致。因此, 在进行鲵属鱼类系统发育关系研究时, 不同的方法可能得到不一致的结果,基于序列构建的基因树可能会与物种树有所不同,将传统形态分类方法与DNA条形码技术相结合可以准确鉴别鱼种, 为后续分类地位确定和系统发育关系研究的开展奠定基础。
[1]Hebert P D N, Cywinska A, Ball S L,et al. Biological identifications through DNA barcodes [J].Proceedings of the Royal Society of London. Series B:Biological Sciences, 2003, 270(1512): 313—321
[2]Tavares E S, Baker A J. Single mitochondrial gene barcodes reliably identify sister species in diverse clades of birds [J].BMC Evolutionary Biology, 2008, 8(1): 1—14
[3]Smith M A, Fisher B L. Invasions, DNA barcodes and rapid biodiversity assessment using ants of Mauritius [J].Frontiers in Zoology, 2009, 6(1): 1—12
[4]Xiao J G, Song N, Han Z Q,et al. Description and DNA barcoding of a newSillagospecies,Sillago shaoi(Perciformes: Sillaginidae), in the Taiwan Strait [J].Zoological Studies, 2016, 55: 47
[5]Hebert P D N, Ratnasingham S, deWaard J R. Barcoding animal life: cytochrome c oxidase subunit I divergences among closely related species [J].Proceedings of the Royal Society of London. Series B, 2003, 270(suppl.1): 96—99
[6]Vences M, Thomas M, Bonett R M,et al. Deciphering amphibian diversity through DNA barcoding: chances and challenges [J].Philosophical Transactions of the Royal Society of London, 2005, 360(1462): 1859—1868
[7]Ward R D, Zemlak T S, Innes B H,et al. DNA barcoding Australia’s fish species [J].Philosophical Transactions of the Royal Society of London, 2005, 360(1462):1847—1857
[8]Witt J D S, Therlof D L, Hebert P D N. DNA barcoding reveals extraordinary cryptic diversity in an amphipod genus: implications for desert spring conservation [J].Molecular Ecology, 2006, 15(10): 3073—3082
[9]McKay R J. FAO Species Catalogue. volume14. Sillaginid Fishes of the World (Family Sillaginidae). An Annotated and Illustrated Catalogue of the Sillago, Smelt or Indo-Pacific Whiting Species Known to Date [M]. FAO,Rome. 1992, 1—83
[10]Nelson J S. Fishes of the World [M]. John Wiley & Sons,New Jersey. 2016, 503
[11]Kaga T, Imamura H, Nakaya K. A new sand whiting,Sillago(Sillago)caudicula, from Oman, the Indian Ocean(Perciformes: Sillaginidae) [J].Ichthyological Research,2010, 57(4): 367—372
[12]Gao T X, Ji D P, Xiao Y S,et al. Description and DNA barcoding of a newSillagospecies,Sillago sinica(Perciformes: Sillaginidae), from coastal waters of China [J].Zoological Studies, 2011, 50(2): 254—263
[13]Golani D, Fricke R, Tikochinski Y.Sillago suezensis, a new whiting from the northern Red Sea, and status ofSillago erythraeaCuvier (Teleostei: Sillaginidae) [J].Journal of Natural History, 2014, 48(7-8): 413—428
[14]McKay R J. A Revision of the Fishes of the Family Sillaginidae [M]. Memoirs of the Queensland Museum. 1985,42—43
[15]Kaga T. Phylogenetic systematics of the family Sillaginidae (Percomorpha: order Perciformes) [J].Zootaxa,2013, 3642(1): 1—105
[16]Zhu Y D, Zhang C L, Cheng Q T. Fishes of the East China Sea [M]. Beijing: Science Press. 1963, 238—240[朱元鼎, 张春霖, 成庆泰. 东海鱼类志. 北京: 科学出版社. 1963, 238—240]
[17]Cheng Q T, Zheng B S. Systeatic Synopsis of Fishes in China [M]. Beijing: Science Press. 1987, 303—304 [成庆泰, 郑葆珊. 中国鱼类系统检索. 北京: 科学出版社.1987, 303—304]
[18]Zhao S L, Xu H X, Zhong J S,et al. Marine Fishes of Zhejiang [M]. Hangzhou: Zhejiang Science and Technology Press. 2016, 601—602 [赵盛龙, 徐汉祥, 钟俊生,等, 浙江海洋鱼类志. 杭州: 浙江科学技术出版社. 2016,601—602]
[19]Sambrook J, Fritsch E F, Maniatis T. Molecular cloning:a laboratory manual (2nd ed.) [J].Immunology, 1989,49(1): 895—909
[20]Inoue J G, Miya M, Tsukamoto K,et al. A mitogenomic perspective on the basal teleostean phylogeny: resolving higher-lever relationships with longer DNA sequences [J].Molecular Phylogenetics and Evolution, 2001, 20(2):275—285
[21]Chen D G, Zhang M Z. Marine Fishes of China [M].Qingdao: China Ocean University Press. 2015,1060—1064 [陈大刚, 张美昭. 中国海洋鱼类. 青岛: 中国海洋大学出版社. 2015, 1060—1064]
[22]Su Y Q, Wang J, Dai T Y,et al. Atlas of Common Sea Fishes in Taiwan Strait [M]. Xiamen: Xiamen University Press. 2011, 98—100 [苏永全, 王军, 戴天元, 等. 台湾海峡常见鱼类图谱. 厦门: 厦门大学出版社. 2011,98—100]
[23]Sun D Y, Chen Z. Synopsis of Fishes in the South China Sea [M]. Beijing: Ocean Press. 2013, 468—470 [孙典荣,陈铮. 南海鱼类检索. 北京: 海洋出版社. 2013,468—470]
[24]Zhao S L, Zhong J S. Photographic Guide to Fishes of Zhoushan [M]. Hangzhou: Zhejiang Science and Technology Press. 2006, 104 [赵盛龙, 钟俊生. 舟山海域鱼类原色图鉴. 杭州:浙江科学技术出版社. 2006, 104]
[25]Yang Y F, Song N, Xiao J G,et al. The description of morphological charactersSillago japonicafrom Laizhou Bay [J].Shandong Fisheries, 2016, 33(10): 8—10 [杨亚峰, 宋娜, 肖家光, 等. 莱州湾少鳞鱚的形态特征描述.齐鲁渔业, 2016, 33(10): 8—10]
[26]Hebert P D N, Stoeckle M Y, Zemlak T S,et al. Identification of birds through DNA barcodes [J].PloS Biology,2004, 2(10): e312
[27]Barrett R D H, Hebert P D N. Identifying spiders through DNA barcodes [J].Canadian Journal of Zoology, 2005,83(3): 481—491
