水稻OsENO2-2基因过表达对水稻抽穗期的影响
2018-01-22刘小云李晓李腾飞苏鲁方
刘小云 李晓 李腾飞 苏鲁方
水稻基因过表达对水稻抽穗期的影响
刘小云 李晓 李腾飞 苏鲁方
(江汉大学 交叉学科研究院, 武汉 430056; E-mail: lxytcw@webmail.hzau.edu.cn)
是通过可变剪切产生的短转录本,其cDNA序列从粳稻中花11中分离获得。本研究的主要目的是初步分析在水稻抽穗期调控中的作用。构建了的超量表达载体,获得转基因植株,通过对表型的观察和统计,分析目的基因在过量表达条件下的功能,并利用反向遗传学方法验证该基因的功能。过量表达导致水稻在长日照条件下的抽穗期推迟,而短日照条件下抽穗期无明显变化。通过qRT-PCR方法对水稻开花关键基因的检测发现,在长日照条件下,的表达量在超表达材料中显著下调,其他开花重要基因表达量在野生型和超表达材料中没有显著变化;而在短日照条件下,所有检测基因的表达量在野生型和超表达材料中均没有显著变化。在长日照条件下,主要通过调控基因的表达来调控水稻抽穗期。
水稻; 过量表达;;烯醇化酶基因;抽穗期
水稻抽穗期与产量密切相关,抽穗期调控基因直接或间接地影响水稻株高、穗数、一次枝梗数及二次枝梗数等农艺性状。因为较长的生育期往往伴随着更高的株高和更多的分蘖,所以在作物产量的研究中,经常把长生育期和高产联系在一起[1]。
光周期途径是水稻抽穗期调控的主要途径。水稻是短日照植物即长夜植物。目前已知水稻光周期调控主要由两条途径调控,一条是途径,另一条是不依赖的光诱导途径即途径。其中途径与拟南芥途径类似,受光照和节律钟调控[2-5],是水稻中与拟南芥()同源的基因,它在调控水稻抽穗的过程中有着双重作用,在短日照条件下促进抽穗而在长日照条件下抑制水稻抽穗[6]。()是水稻中与拟南芥()同源的成花素基因,促进成花转变[7]。()在短日照条件下促进的表达,但是在长日照条件下,由光敏色素B转换为的阻抑物抑制的表达[8]。因此,在短日照条件下有较高的表达量而在长日照条件下表达量较低。是的同源基因,主要调控长日照条件下的成花转变[9]。同时的表达受到水稻抽穗节律钟中的关键基因()的正向调控[10]。另一条途径是不依赖的光诱导途径即途径,该途径是水稻中特有的。()是受短日照促开花因子调控的B型反应调节子,它可以促进、和的表达,是短日照抽穗调控的正调控因子[11]。在短日照条件下,和同时被正调控。而在长日照条件下则被正调控,被()负调控[1],是一个具有CCT结构域的转录因子,是长日照条件下的抽穗抑制子[12]。在长日照和短日照条件下,均可被//正调控[13-14],促进和的表达从而促进和的表达,最终促进水稻抽穗[15],是位于光周期途径较下游的促开花因子[16,17]。
烯醇化酶(enolase, ENO)是糖酵解中的关键酶之一,催化2-磷酸甘油酸形成高能化合物磷酸式丙酮酸,在细胞能量代谢中起重要作用。烯醇化酶基因在原核生物中研究较多,除去催化作用外,还在原核生物入侵动植物过程中有重要作用,在动物中也参与转录、凋亡调控以及细胞分化过程[18, 19]。在拟南芥中编码该酶的基因为,其可变剪切体基因在超表达后对开花期有影响,并且在逆境响应中也有重要作用[20,21]。
本研究通过构建转基因材料研究对抽穗期的调控作用,揭示该基因在水稻高产育种中可能存在的应用价值。
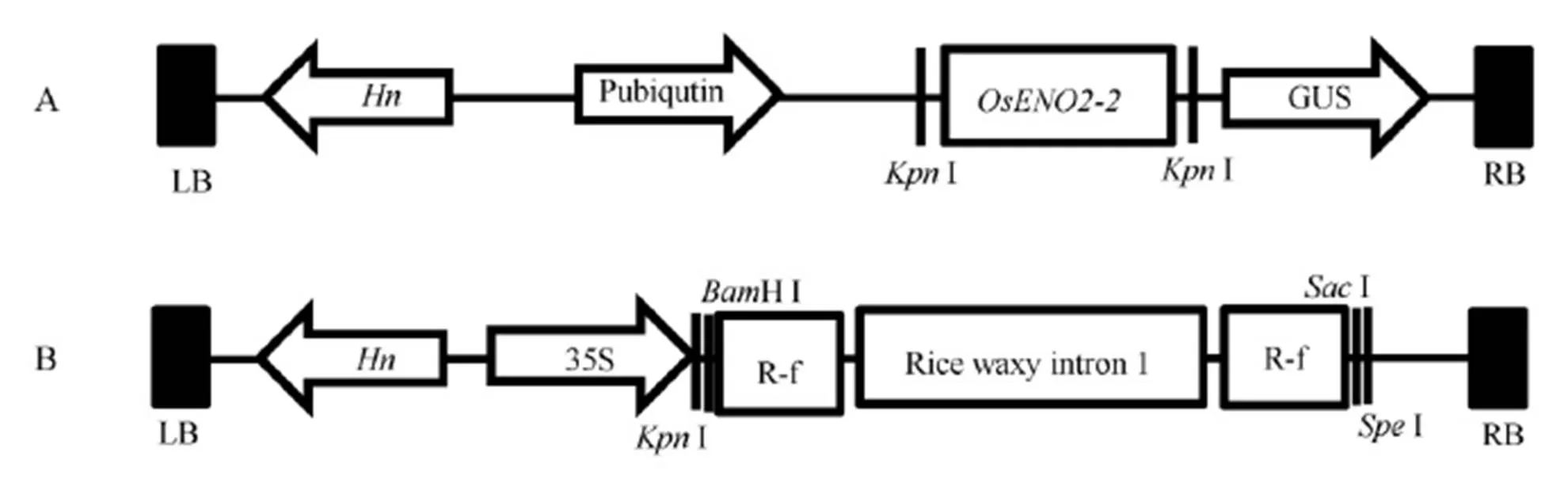
A–超表达载体结构。LB为T-DNA左边界;Hn–潮霉素抗性筛选标记;Pubiqutin为玉米ubiqutin基因启动子;GUS为β-葡萄糖苷酸酶基因;RB为T-DNA右边界。B–RNAi载体结构。35S–花椰菜花叶病毒启动子;R-f为OsENO2-2 RNAi片段。
Fig. 1. Construction of overexpression vector and RNAi vector.
1 材料与方法
1.1 植物材料与菌株
粳稻中花11(野生型)、超表达转基因株系的T1和T2代植株、RNAi抑制转基因株系的T1和T2植株均种植于华中农业大学作物遗传改良国家重点实验室试验地。田间常规管理。
所用菌株为大肠杆菌()菌株DH5α和根癌农杆菌()菌株EHA105,均由本实验室保存。
1.2 OsENO2-2基因全长和RNAi片段的扩增
利用Trizol抽提试剂盒(Invitrogen公司)抽提中花11幼苗叶片的RNA,逆转录成cDNA(逆转录试剂盒, Bio-Rad公司)。根据RGAP数据库(http://rice. plantbiology.msu.edu/)里(Os06g04510.2)的cDNA序列,设计基因全长引物(-F和-R,两端加上Ⅰ酶切位点,图1-A)和RNAi片段引物(R--F和R--R,两端分别加Ⅰ、Ⅰ和Ⅰ、HⅠ酶切位点,图1-B,表1)。以上述cDNA为模板进行PCR扩增,PCR条件如下:94℃下4 min; 94℃下30 s,60℃下30 s,72℃下60 s,循环35次; 72℃下延伸7 min,4℃下保存。将扩增出的全长连接到T/A克隆载体(pGEMT-vector,Promega公司)上,用T7和SP6引物测序验证。

表1 本研究所用引物
1.3 OsENO2-2过表达载体和RNAi载体的构建
将带有片段的T/A克隆用Ⅰ单酶切,回收目标条带,与Ⅰ单酶切的表达载体质粒pU1301连接,pU1301是在国际上常用的植物遗传转化载体pCAMBIA1301[22]基础上改造的,是携带具有组成型和超量表达特征的玉米泛素启动子的农杆菌介导的遗传转化载体。pCAMBIA1301载体由澳大利亚CAMBIA实验室惠赠。内切酶Ⅰ和连接所使用的T4连接酶均购自宝生物工程大连有限公司。将连接产物导入到DH5α细胞中,菌液经PCR检测呈阳性后送至测序。将测序正确的pU1301-OsENO2-2质粒电击到EHA105,菌液检测呈阳性者作为遗传转化菌株。
用Ⅰ和HⅠ酶切带有RNAi片段的T/A克隆,回收目标条带,与Ⅰ和HⅠ酶切的表达载体质粒pDS1301连接获得pDS1301-1载体(所使用的内切酶均购自宝生物工程大连有限公司,连接酶为上海英俊生物技术公司产品);再将带有RNAi片段的T/A克隆载体+pDS1301-1载体用Ⅰ和Ⅰ酶切,回收目标条带,与Ⅰ和Ⅰ酶切的质粒pDS1301-1连接,得到RNAi抑制载体pDS1301-2。
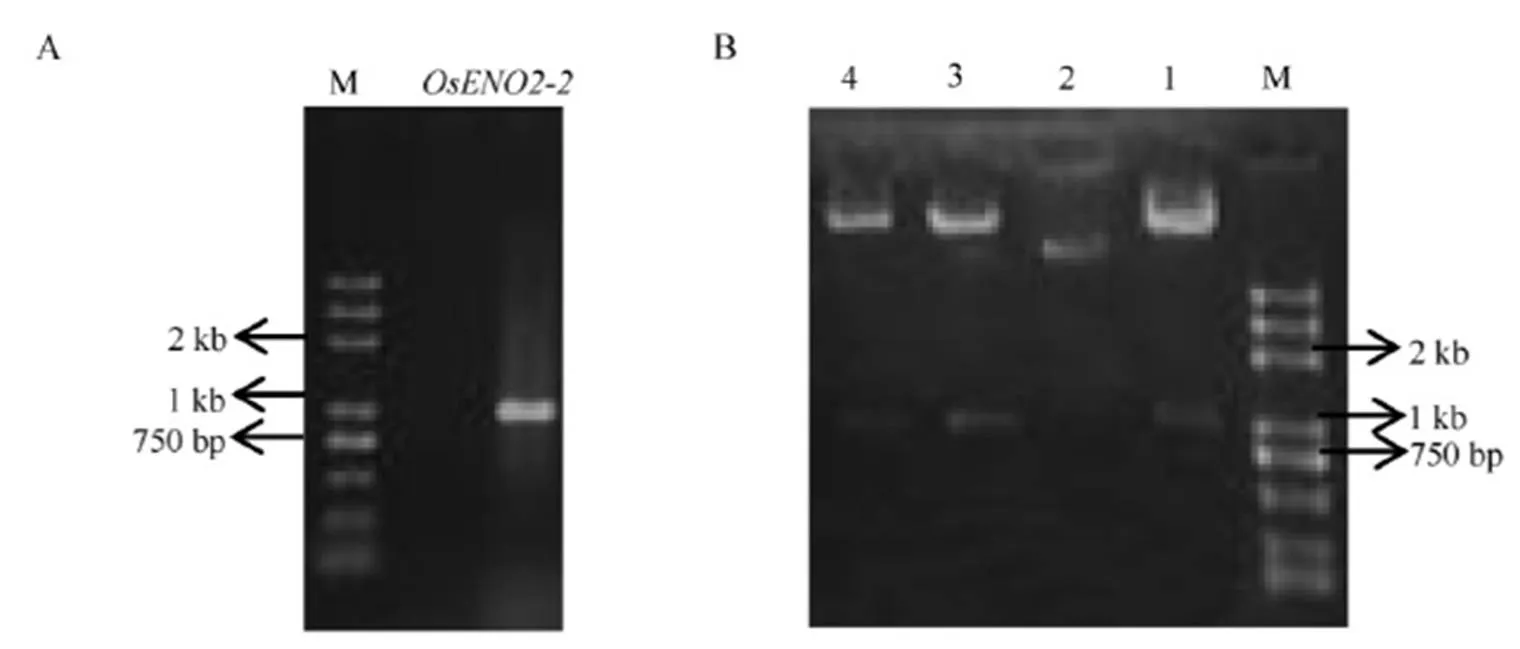
A–OsENO2-2全长开放阅读框的PCR扩增。B–过表达载体的酶切鉴定。1~4为4个不同单克隆,其中1、3、4为阳性克隆;2–阴性对照;M为DNA标记。
Fig. 2. PCR amplification ofand identification of recombinant plasmid.
1.4 农杆菌介导的水稻遗传转化
利用农杆菌介导的遗传转化方法将全长以及RNAi片段转入到水稻基因组中,培养基以及使用的试剂主要参照华中农业大学授权专利(专利号ZL200710053552.9)。并用空载体产生转基因植株作对照。
1.5 转基因植株的DNA检测
用CTAB法[23]从叶片中提取基因组DNA,用载体中的潮霉素抗性基因()对获得的过表达转基因植株进行PCR检测。所用引物为-F和-R,引物序列如表1所示。反应条件如下: 94℃下4min;94℃下30 s,57℃下30 s,72℃下30 s,30次循环;之后72℃下延伸5 min。通过PCR筛选出阳性株系。
1.6 OsENO2-2在野生型材料不同组织的特异性表达分析以及转基因植株中的表达分析
为了检测目的基因在野生型材料不同组织的特异性表达量以及在超表达植株中目标基因的表达量,采用实时PCR的方法进行了表达分析。实验所用的总RNA分别来自来中花11的幼苗,抽穗期的倒2叶、剑叶、叶鞘、茎秆、根、幼穗和胚乳以及转基因材料分蘖期的水稻叶片,采用Trizol抽提试剂盒(Invitrogen公司)抽提RNA,逆转录成cDNA(Bio-Rad公司逆转录试剂盒),用实时荧光定量PCR检测表达量(试剂购自宝生物工程大连有限公司,仪器为美国ABI公司7500 PCR仪),PCR参数为95℃下预变性10 s;95℃下变性5 s,60℃下退火延伸40 s,45个循环。以内源为参照基因,对目的基因在不同组织中特异性表达量进行相对定量分析;转基因受体材料中花11为参照样本,对在转基因植株叶片中的表达进行相对定量分析。用2法[24]计算基因表达的相对变化,实验设置3次技术重复。实时PCR所用引物序列为:-QF和-QR,-QF和-QR(序列见表1)。
1.7 光周期材料的处理及目的基因的表达量分析
将转基因植株和野生型植株分别在长日照条件(14 h光照30℃/10 h黑暗28℃)下培养6周,在短日照条件(10 h光照30℃/14 h黑暗28℃)下培养8周后,每4 h取一次样,取样部位为植株顶端3叶,抽提叶片RNA,反转录产物检测抽穗相关基因的表达量。取叶片总RNA,逆转录成cDNA,用实时荧光定量PCR的方法检测抽穗相关基因的表达量(PCR同1.6,所用引物列于表1,引物名称含QF或QR)。以内源为参照基因,用2法[24]计算基因表达的相对变化,设置3次重复。
2 结果与分析
2.1 OsENO2-2克隆与超量表达载体和RNAi载体的构建
用RT-PCR方法从水稻叶片中获得基因的全长ORF,目的片段长度与RGAP数据库公布的片段长度1062 bp吻合,符合预期设计(图2-A)。将该片段连接到T/A载体上,通过测序分析目的片段序列,并与网站中序列进行比对验证。随后,用Ⅰ将连接有外源的T/A载体和pU1301载体分别进行单酶切后连接,转化大肠杆菌后,经过PCR检测以及酶切检测,得到预期大小片段(图2-B),表明目的片段已成功连入空载体中,获得最终的过表达载体。同样,用RT-PCR从叶片中获得基因的RNAi片段,利用酶切方法构建RNAi载体。将构建成功的重组质粒进行测序验证后,转化到农杆菌EHA105中,用于后续的转基因实验。
2.2 OsENO2-2基因的组织特异性表达分析
为了确定在不同组织材料中的表达量,分别提取中花11的幼苗,抽穗期的倒2叶、剑叶、叶鞘、茎秆、根、幼穗和胚乳的总RNA,反转录成cDNA,通过实时PCR方法进行表达量检测。检测结果显示,在水稻各个部位均有表达,但不同组织部位表达差异显著,在叶片中表达量显著高于其他部位,尤其是剑叶中表达量最高,而幼穗、胚乳中表达量最低(图3)。
2.3 转基因株系的DNA检测和OsENO2-2在转基因植株中的表达分析
通过农杆菌介导的遗传转化[26]将pU1301载体和pU1301空载体以及pDS1301-2载体导入野生型中花11中,抽提DNA通过PCR检测载体上抗性基因后,最终共获得50个T0转基因阳性株系,其中pU1301载体转化后阳性株系(超表达阳性株系)有18个,pU1301空载体转化后阳性株系(阴性对照株系)有16个,pDS1301-2载体转化后阳性株系15个(图4-A)。共选取15个转基因阳性株系包括7个超表达阳性株系(OX-1,OX-2,OX-3,OX-4,OX-5,OX-6,OX-8)、1个超表达阴性对照株系(OX-7)和7个RNAi株系(R-1,R-2,R-3,R-4,R-5,R-6,R-7)种植到大田里,获得了T1代转基因株系,通过实时PCR方法检测转基因中目的基因的表达,发现7个超表达阳性株系(OX-1,OX-2,OX-3,OX-4,OX-5,OX-6,OX-8)中的表达量明显增强,与野生型相比,的表达量上升3~15倍。阴性对照株系OX-7中基因的表达量与野生型相比没有明显差异(图4-B)。7个RNAi株系(R-1,R-2,R-3,R-4,R-5,R-6,R-7)中除了R-5表达量与野生型无显著差异外,其他株系目的基因表达量均显著降低,其中R-1和R-7中的表达量下降约4倍,R-6中下降约3倍,R-2、R-3和R-4中目的基因下降约2倍(图4-B)。上述结果表明构建的过表达载体和RNAi载体能够正常工作,超表达以及抑制效果均良好。

S–幼苗;FL–剑叶;L–抽穗期倒2叶;LS–叶鞘;ST–茎秆;R–根;PA–幼穗;EN–胚乳。
Fig. 3. Tissue-specific expression of
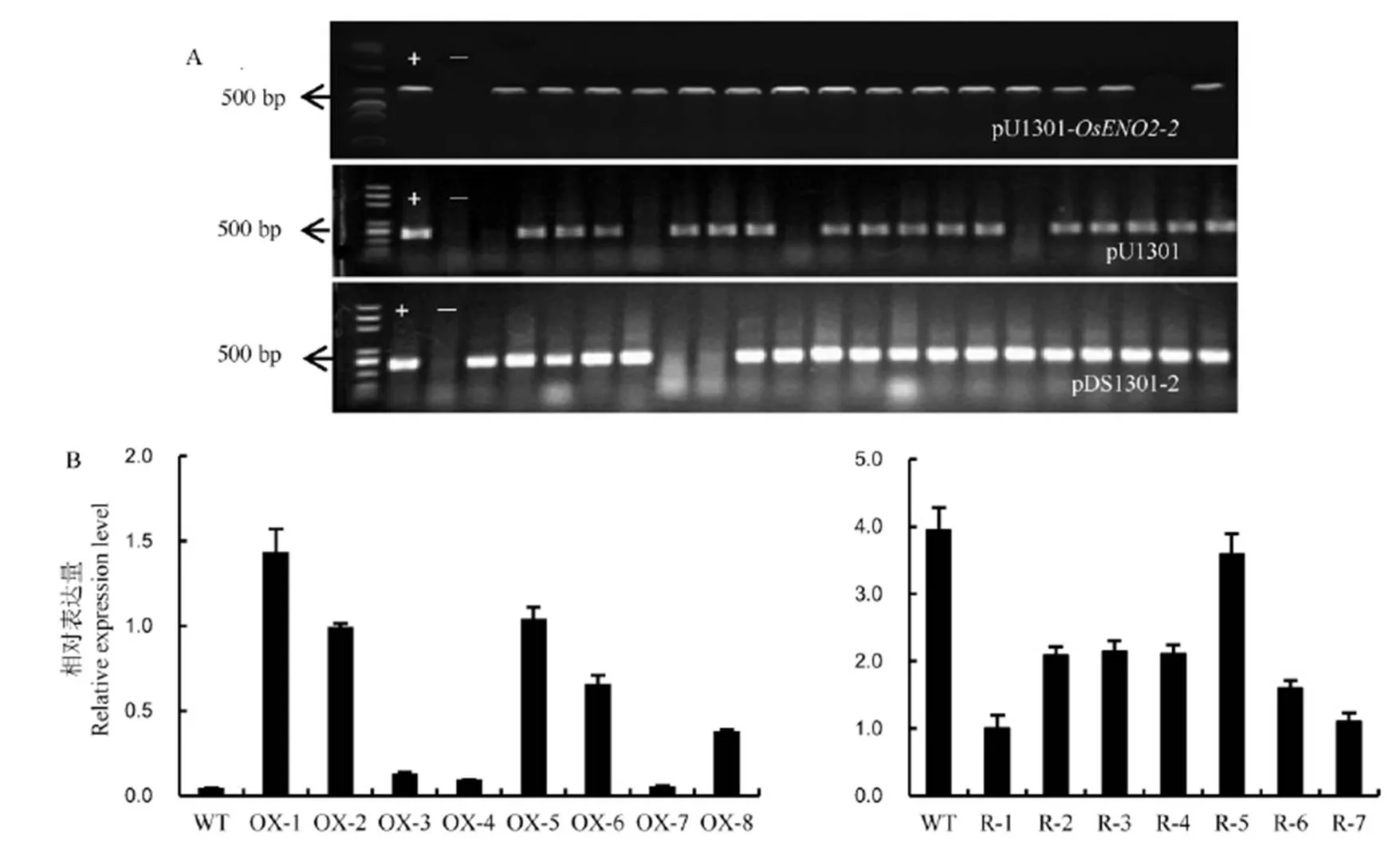
A–转基因阳性植株Hn基因PCR鉴定,自上到下依次为超表达载体、空载体对照组、RNAi载体的转基因植株。B–转基因植株OsENO2-2的表达量(平均值±标准差)。WT–野生型;OX-1~OX-8,8个超表达转基因植株;R-1~R-7,7个RNAi转基因植株。
Fig. 4. PCR analysis ofgene in T0transgenic positive plants and expression analysis ofin transgenic lines.

A–野生型和超表达植株的抽穗期表型。WT–野生型植株,OX–超表达植株; B–野生型和超表达植株在长日照大田的抽穗期(n=30)。WT–野生型植株,OX-1, OX-2, OX-3为超表达阳性株系,OX-7为转基因对照株系。LD–长日照条件。C–在长日照条件下(光照14 h/黑暗10 h)超表达,RNAi株系以及野生型株系的抽穗期(n=10)。R-1,R-2,R-5为RNAi抑制阳性株系。D–在短日照条件下(光照10 h/黑暗14 h)超表达,RNAi株系以及野生型株系的抽穗期(n=10)。SD–短日照条件。**表示与野生型植株相比差异达极显著水平(P<0.01,t检验)。
Fig. 5. Heading date of the transgenic plants(Mean±).
2.4 转基因植株的表型分析
将上述8个超表达转基因株系的T2代植株种植到大田(武汉6月中旬-8月中旬属于长日照时间),观察统计表型时发现,7个转基因株系的植株抽穗期较野生型显著推迟(图5-A),1个阴性株系植株与野生型相比没有差异。选取4个株系,分别取30株植株进行统计分析,其中3个超量表达株系的抽穗期显著推迟,1个阴性对照株系与野生型无明显变化(图5-B)。根据3个株系的表达量以及相应抽穗期分析得知,转基因植株的抽穗期推迟是由于的过量表达所致。随后,我们选取大田种植的这3个株系在培养箱中进行严格日照条件控制,分别取10株植株进行统计分析,结果显示在长日照条件下2个超量表达的株系抽穗期较野生型显著推迟,1个阴性对照株系与野生型相比无显著变化(图5-C);短日照条件下,3个株系与野生型相比均无显著差异(图5-D)。
将7个RNAi抑制转基因株系的T2植株种植到大田(武汉6月中旬-8月中旬属于长日照时间),并未发现抽穗期与野生型有差异。在严格控制长短日照条件的培养箱中种植R-1、R-2和R-7株系,不管是长日照还是短日照条件下,抽穗期与野生型相比均无显著变化(图5-C、D)。
2.5 OsENO2-2基因在不同光周期条件下昼夜节律性表达
由于超表达植株抽穗期推迟,推测表达可能与光周期有关。在长、短日照条件下检测野生型和超表达植株中的相对表达量,结果表明野生型植株的基因在长、短日照条件下均有节律性表达,并且在长日照条件下表达量显著高于短日照条件下(图6)。不管是在长日照条件还是短日照条件下,在光照条件下表达量较高,进入黑暗条件后表达量降到最低(图6)。在超表达OX-2株系纯合植株中,在长、短日照条件下的任何时间点均较野生型超量表达。
2.6 转基因植株中抽穗相关基因的表达量检测
根据表型统计分析的结果,在长日照条件下过量表达导致抽穗推迟。为了研究基因在水稻抽穗过程中的功能,将转基因植株(OX-2株系纯合植株)和野生型植株分别在长日照条件下和短日照条件下处理后,反转录产物检测抽穗相关的关键基因的表达量。qPCR检测结果显示,在长日照条件下,的表达量在超表达材料中显著下调,下游基因和也相应显著下调表达,而其他抽穗相关基因、、、、表达量在野生型和超表达材料中基本没有变化(图7);而水稻在短日照条件下,、、、、、以及的表达量在野生型和超表达材料中均没有显著变化(图8)。
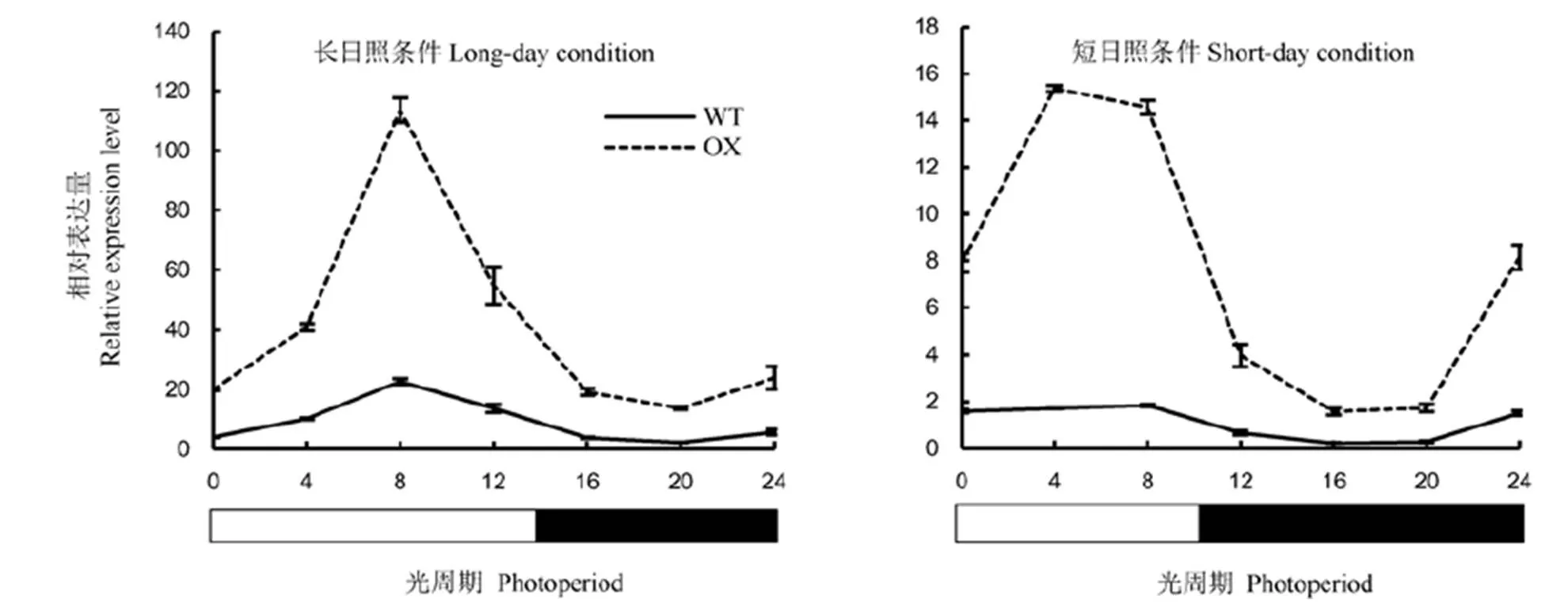
LD–长日照条件(光照14 h/黑暗10 h); SD–短日照条件(光照10 h/黑暗14 h); WT–野生型植株; OX–OsENO2-2超表达植株。
Fig. 6. Circadian rhythm expression ofunder different photoperiods(Mean±).
3 讨论
近年来,调控水稻抽穗期的光周期途径中的关键基因被克隆和鉴定,这些基因在短日照促进水稻抽穗途径、长日照抑制水稻抽穗途径和长日照诱导水稻成花途径中发挥着重要的作用。研究报道表明,和分别在长日照和短日照下对水稻成花转换起到主导作用,而在短日照条件下表达量高,长日照条件下表达量低,表明在短日照条件下,主要由调控来调控水稻抽穗期,是在不表达的情况下在短日照下起作用;长日照条件下主要由正调控水稻抽穗期[9],而目前研究比较清楚的是作为的上游调控因子来调节水稻抽穗期,而在长日照条件下表达量低,那么在长日照条件下作为的上游调控因子可能作用有限,也就是说还有可能通过其他上游调控因子来调控其表达来影响水稻长日照条件下抽穗期。而我们的研究发现基因正是在长日照条件下作为RFT1的上游正调控因子来调节水稻抽穗(图9)。因此,我们的研究结果是对于水稻长日照条件下抽穗调控机理的补充。至于基因是否是直接影响的表达以及调控的表达的机制目前尚不清楚还有待研究。
另一方面,我们在研究中发现基因仅在超量表达的情况下对水稻长日照条件下正常抽穗有显著影响,基因被抑制后并未观察到水稻抽穗表型的变化。这一结论表明在水稻中可能存在基因的同源基因,并在部分功能上与存在冗余的作用,当的表达量显著降低时,有其他基因来互补它的功能,使植物的生理状态不受影响进而保证植株能够正常生长发育。
植物的代谢调节是植物适应不同环境保证植物正常生长的机制之一,例如植物糖酵解是由不同的生物和非生物胁迫引起的,如病原体攻击、氧化应激和缺氧等。在受到胁迫条件的植物中,增加糖酵解对于抵抗逆境提供额外的能量和活性氧清除剂(如丙酮酸和NADH)是非常重要的。烯醇酶是糖酵解途径中的关键酶之一,烯醇酶活性直接影响糖酵解的进程,因此编码烯醇酶的基因在植物中的功能也是非常重要的。是拟南芥中烯醇酶基因的同源基因,它有可能影响水稻中烯醇酶的活性从而与水稻的代谢过程息息相关。那么,水稻的代谢与抽穗过程中间是否存在一定的联系也有待进一步研究。
此外,我们的研究还发现,基因在超表达后,不仅水稻抽穗时间显著推迟,而且转基因植株的种子的形态也发生了显著改变,种子的胚乳显著增大,千粒重显著增多。这些性状的变化也表明,水稻的抽穗时间与水稻产量之间是存在着一定的关系的,水稻抽穗时间推迟,营养生长时期延长,营养物质积累多,导致种子的营养物质也积累多从而使种子增大,但其中的分子机制目前也并不清楚,还在研究中。

Fig. 7. Expression levels of key heading regulatory genes in leaves ofoverexpression plants(OX) compared to wild type(WT) under long-day condition(Mean±).

Fig. 8. Expression levels of key heading regulatory genes in leaves ofoverexpression plants(OX) compared to wild type(WT) under short-day condition(Mean±).

图9 OsENO2-2基因在水稻长日照条件下调控开花途径中的作用
Fig. 9. Summary offunction in LD pathways of rice flowering control.
[1] 徐铨, 奥本裕, 王晓雪. 水稻开花期调控分子机理研究进展. 植物遗传资源学报, 2014, 15(1): 129-136.
Xu Q, Okumoto Y, Wang X E. Research progress on regulatory molecular mechanisms of flowering time in rice, 2014, 15(1): 129-136. (in Chinese with English abstract)
[2] Imaizumi T, Kay S. Photoperiodic control of flowering: not only by coincidence., 2006, 11(11): 550-558.
[3] Itoh H, Nonoue Y, Yano M, Izawa T. A pair of floral regulators sets critical day length forflorigen expression in rice., 2010, 42(7): 635-638.
[4] Tsuji H, Taoka K I, Shimamoto K. Regulation of flowering in rice: Two florigen genes, a complex gene network, and natural variation., 2011, 14(1): 45-52.
[5] Yano M, Katayose Y, Ashikari M, Yamanouchi U, Monna L, Fuse T, Baba T, Yamamoto K, Umehara Y, Nagamura Y, Sasaki T., a major photoperiod sensitivity quantitative trait locus in rice, is closely related to theflowering time gene., 2000, 12(12): 2473-2484.
[6] Tamaki S, Matsuo S, Wong H L, Yokoi S, Shimamoto K. Hd3a protein is a mobile flowering signal in rice., 2007, 316(5827): 1033-1036.
[7] Hayama R, Coupland G. The molecular basis of diversity in the photoperiodic flowering responses ofand rice., 2004, 135(2): 677-684.
[8] Komiya R, Yokoi S, Shimamoto K. A gene network for long-day flowering activates RFT1 encoding a mobile flowering signal in rice., 2009, 136(20): 3443-3450.
[9] Hayama R, Yokoi S, Tamaki S, Yano M, Shimamoto K. Adaptation of photoperiodic control pathways produces short-day flowering in rice., 2003, 422: 719-722.
[10] Kim S L, Lee S, Kim H J, Nam H G, An G. OsMADS51 is a short-day flowering promoter that functions upstream of,, and., 2007, 145(4): 1484-1494.
[11] Xue W, Xing Y, Weng X, Zhao Y, Tang W, Wang L, Zhou H, Yu S, Xu C, Li X, Zhang Q. Natural variation inis an important regulator of heading date and yield potential in rice., 2008, 40(6): 761-767.
[12] Wu C, You C, Li C, Long T, Chen G, Byrne M E, Zhang Q. RID1, encoding a Cys2/His2-type zinc finger transcription factor, acts as a master switch from vegetative to floral development in rice., 2008, 105(35): 12915-12920.
[13] Matsubara K, Yamanouchi U, Wang Z X, Minobe Y, Izawa T, Yano M., a rice ortholog of the maizegene, promotes flowering by up-regulating., 2008, 148(3): 1425-1435.
[14] Park S J, Kim S L, Lee S, Je B I, Piao H L, Park S H, Kim C M, Ryu C H, Xuan Y H, Colasanti J, An G, Han C D. Rice() is necessary for the expression of() regardless of photoperiod., 2008, 56(6): 1018-1029.
[15] Lu S J, Wei H, Wang Y, Wang H M, Yang R F, Zhang X B, Tu J M. Overexpression of a transcription factormodifies plant architecture and flowering time in rice (L.)., 2012, 30(6): 1461-1469.
[16] Arora R, Agarwal P, Ray S, Singh A, Singh V, Tyagi A K, Kapoor S. MADS-box gene family in rice: Genome-wide identification, organization and expression profiling during reproductive development and stress., 2007, 8(1): 242.
[17] Pancholi V. Multifunctional α-enolase: Its role in diseases., 2001, 58: 902-920.
[18] Àngels D R, Anna R B, Ana G M, Roser L A. α-enolase, a multifunctional protein: Its role on pathophysiological situations., 2012(7): 1-12.
[19] Kang M, Abdelmageed H, Lee S, Reichert A, Mysore K S, Allen R D. AtMBP-1, an alternative translation product of LOS2, affects abscisic acid responses and is modulated by the E3 ubiquitin ligase AtSAP5., 2013, 76(3): 481-493.
[20] Eremina M, Rozhon W, Yang S, Poppenberger B. ENO2 activity is required for the development and reproductive success of plants, and is feedback-repressed by AtMBP-1., 2015, 81(6): 895-906.
[21] Sun X, Cao Y, Yang Z, Xu C, Li X, Wang S., a gene conferring resistance topv.in rice, encodes an LRR receptor kinase-like protein., 2004, 37: 517-527.
[22] 卢扬江, 郑康乐. 提取水稻DNA 的一种简易方法. 中国水稻科学, 1992, 6(1): 47-48.
Lu Y J, Zheng K L. A simple method for isolation of rice DNA., 1992, 6(1): 47-48. (in Chinese with English abstract)
[23] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2[-Delta Delta C(T)] method., 2001, 25(4): 402-408.
[24] Hiei Y, Ohta S, Komari T, Kumashiro T. Efficient transformation of rice (L.) mediated byand sequence analysis of the boundaries of the T-DNA., 1994, 6(2): 271-282.
Overexpression ofAffects Heading Date in Rice
LIU Xiaoyun, LI Xiao, LI Tengfei, SU Lufang
(Interdisciplinary Research Institute, Jianghan University, Wuhan 430056, China; E-mail: lxytcw@webmail.hzau.edu.cn)
was isolated fromcultivar Zhonghua 11as a short transcript produced by alternative splicing ofwhich is homologous to. To reveal the function of,we constructed an overexpression vector ofand obtained the transgenetic plants. Based on the observation and statistics of phenotype in overexpressed plant and wild-type, we validated the function ofusing reverse genetics method.Overexpression oftransgenetic plants delayed heading date only under long-day conditions but not short-day conditions. qRT-PCRexpressionanalysis of the critical flowering time genes showedthat in long-day conditions, the expression ofin overexpressed plants was downregulated, and the expression levels of the other tested genes did not change significantly in wild-type and overexpressed plants; but in short-day conditions, the expression of all the tested genes in wild-type and over-expression plants did not change significantly.mightbeinvolvedinthephotoperiodicpathwaystoregulatetheheading dateinriceby regulating the expression ofgene.
rice; overexpression;; enolase gene; heading date
10.16819/j.1001-7216.2018.7055
Q786; S511.032
A
1001-7216(2018)01-0012-11
2017-05-18;
2017-09-15。
国家自然科学基金资助项目(31600981);江汉大学科研启动项目(1019-06210002)。
