CRISPR/Cas9系统编辑水稻Wx基因
2018-01-22汪秉琨张慧洪汝科张锦文杨睿罗琼曾千春
汪秉琨 张慧,# 洪汝科 张锦文 杨睿 罗琼,* 曾千春,*
CRISPR/Cas9系统编辑水稻基因
汪秉琨1,2,#张慧1,#洪汝科1张锦文3杨睿2罗琼2,*曾千春1,*
(1云南农业大学 农学与生物技术学院, 昆明 650201;2云南农业大学 云南生物资源保护与利用国家重点实验室, 昆明 650201;3云南省农业科学院粮食作物研究所, 昆明 650205;#共同第一作者;*通讯联系人, E-mail: qiongbf@aliyun.com, zengqianchun@qq.com)
直链淀粉含量与稻米品质密切相关。基因是控制水稻直链淀粉合成的主效基因,通过对基因定点编辑以获得稳定遗传、直链淀粉含量适宜的突变体。构建CRISPR/Cas9表达载体pGK03-Wx-gRNA (靶点1和2分别在基因第1和第2外显子),利用工程菌EHA105遗传转化超级稻楚粳27,潮霉素筛选获得转化株系,对转化株系及其后代进行分子检测、测序、基因表达和遗传稳定性分析以及直链淀粉含量测定。【获得9个独立的T0代转化株系,靶点1(L1~L5) 5个株系,突变频率100%,靶点2(L6~L9) 4个株系,突变频率75%。由T0代突变体衍生出T1和T2代株系,测序发现T0、T1和T2代株系出现缺失(单、双、多碱基缺失)和单碱基插入两种突变类型;T0至T1代部分株系(L1、L2、L3和L6)发生再编辑,T1至T2代遗传稳定。与野生型相比,突变株系RNA水平基因表达量显著下降(<0.01),稻米直链淀粉含量显著降低(<0.01),从17.5%降到1.93%。利用CRISPR/Cas9系统成功编辑水稻基因,获得了稳定遗传、低直链淀粉含量的突变体,为稻米品质改良提供了材料。
水稻; CRISPR/Cas9系统;; 基因编辑; 直链淀粉含量
水稻是世界上最重要粮食作物之一,养活了全世界半数以上人口。近30年来,中国水稻生产经过高产育种、超高产育种和超级稻育种,单产不断提高,超级稻百亩连片平均单产达到13.5 t/hm2,但存在高产不优质问题。随着人们生活水平的提高,优质稻米需求愈显迫切[1]。淀粉是评价稻米品质重要指标之一,稻米胚乳90%为淀粉[2]。因此,淀粉品质改良是水稻育种的重要方向。
稻米淀粉包括直链淀粉和支链淀粉,水稻蜡质基因编码的颗粒淀粉合成酶Ⅰ(GBSSⅠ),是控制直链淀粉合成的主效基因。在非糯品种中,基因分化为等位基因Wx和Wx,籼稻以Wx为主,直链淀粉含量较高,粳稻以Wx为主,直链淀粉含量较低[3,4]。的外显子或内含子的结构改变都将影响基因的表达量和蛋白质功能[5]。目前在水稻中已经报道了7个基因的等位基因和7个功能位点,包括Wx等位基因的第1内含子5′端剪切处发生GT→TT的突变[6]、Wx和Wx等位基因的第4外显子A→G突变[7-8]、Wx等位基因的第4外显子G→A突变,第5外显子T→C突变[9]、Wx等位基因的第6外显子A→C突变[10]和第10外显子C→T的突变[11]、等位基因的第2外显子23 bp碱基重复插入[12]。这7个功能位点都被证实与稻米直链淀粉含量显著相关。
CRISPR/Cas9系统是近几年发展迅速的高效、准确、便捷的基因组编辑技术。该技术利用向导RNA(gRNA)与基因靶序列结合,在Cas9内切酶作用下对靶DNA进行剪切,断裂的DNA会启动修复机制进行修复,修复过程中可能会出现碱基插入、缺失和替换突变[13-14]。2013年两个实验室分别报道了CRISPR/Cas9系统定点编辑水稻基因的成功事例:高彩霞实验室[15]定点突变、、和Os02g23823等4个基因,并且在T0代获得了基因功能缺失的纯合突变体;朱健康实验室[16]利用CRISPR/Cas9系统定点突变水稻基因,T0代10%的水稻叶片有白化现象。超级稻楚粳27,高产、高抗稻瘟病,但是直链淀粉含量偏高(17.5%)[17]。本研究利用CRISPR/Cas9系统对楚粳27基因进行定点编辑,以期获得直链淀粉含量适宜的突变体,为稻米品质育种提供材料。
1 材料与方法
1.1 水稻材料以及载体试剂盒
楚粳27为云南省楚雄州农业科学研究所选育的常规超级稻,米质达国标优质米一级标准;CRISPR/Cas载体试剂盒购自百格生物科技有限公司,Oligo序列和检测引物由北京擎科新业生物技术有限公司合成。
1.2 gRNA靶点选择及寡核苷酸链的合成
根据水稻基因组注释网站(http://rice. plantbiology.msu.edu/)提供的(LOC_Os06g04200)序列,在第1和第2外显子区域分别设计一个gRNA靶点序列。靶点长20 bp并且其后面是PAM序列(protospacer adjacent motif) NGG。在NCBI (https:// blast.ncbi.nlm.nih.gov/Blast.cgi)中比对设计好的gRNA序列,排除非特异性酶切位点,并在线(http:// www.biogle.com)生成寡核苷酸(Oligo)序列(表1)。

表1 寡核苷酸序列及检测引物序列
1.3 CRISPR/Cas9载体构建
按试剂盒说明,分别构建靶点1和靶点2的pGK03-Wx-gRNA载体。寡核苷酸(Oligo)二聚体制备:将Oligo序列加水溶解至10 µmol/L,按照总体系20 µL(18 µL退火缓冲溶液+1 µL Oligo-F+1 µL Oligo-R)混匀后,95℃下3 min,然后以约0.2℃/s降至20℃。Oligo二聚体构建至CRISPR/Cas载体:总体系10 µL,于冰上依次加入6 µL ddH2O, 2 µL CRISPR/Cas载体,1 µL Oligo二聚体和1 µL 酶混合液,混匀,室温静置1 h,形成pGK03-Wx-gRNA载体。取包含pGK03-Wx-gRNA载体的反应液5 µL与50 µL DH5α感受态细胞混匀,冰浴静置30 min;轻轻取出,42℃热激60 s,置冰上2 min;加500 µL LB,37℃、200 r/min下培养1 h;取适量菌液涂布于含有100 mg/L卡那霉素的LB平板上,37℃下倒置培养过夜。挑取单菌落在1 mL LB培养基中摇菌过夜(含卡那霉素,37℃,200 r/min)。菌液检测:选取阳性菌液提取质粒,电激转化获得农杆菌工程菌EHA105。
1.4 农杆菌介导的遗传转化
参照曾千春等[18],利用EHA105,遗传转化楚粳27胚性愈伤组织,潮霉素筛选获得转化植株。
1.5 水稻转化植株分子检测
上述转化植株顶盖时,炼苗,移栽至温室,常规水肥管理。CTAB法[19]提取转化植株叶片基因组DNA进行PCR检测,检测引物序列见表1。PCR扩增体系20 µL:15 µL金牌Mix(北京擎科新业生物技术有限公司)、1 µL模版DNA、正反引物各0.5 µL、3 µL ddH2O。扩增条件:98℃下2 min,98℃下10 s,55℃下15 s,72℃下15 s,72℃下5 min,32个循环。扩增产物用1%琼脂糖凝胶电泳检测,并送北京擎科新业生物技术有限公司测序。
1.6 T1植株qPCR检测
取野生型和突变株系灌浆7 d后的籽粒,Trizol法提取RNA,用反转录试剂盒(TransGen)合成第1链cDNA。qPCR仪为Bio-Rad CFX96,内参基因引物:Act-Q-F ACTTCATGCTTTCGTCCT ACGCTCCA,Act-Q-R CTGGGAGGCTGGTAG TTGATTC;引物:Wx-Q-F TGCGTTTCAGCCT TCTTTGCC,Wx-Q-R CGATTGCCGTGGTTGCG TCGT。反应体系20 µL:2 µL cDNA,正反引物各0.4 µL,10 µL Top Green qPCR Mix,7.2 µL ddH2O。扩增程序:94℃下30 s,94℃下5 s,60℃下30 s,45个循环。每样品3个生物学重复,基因相对表达量按照2法计算[20]。
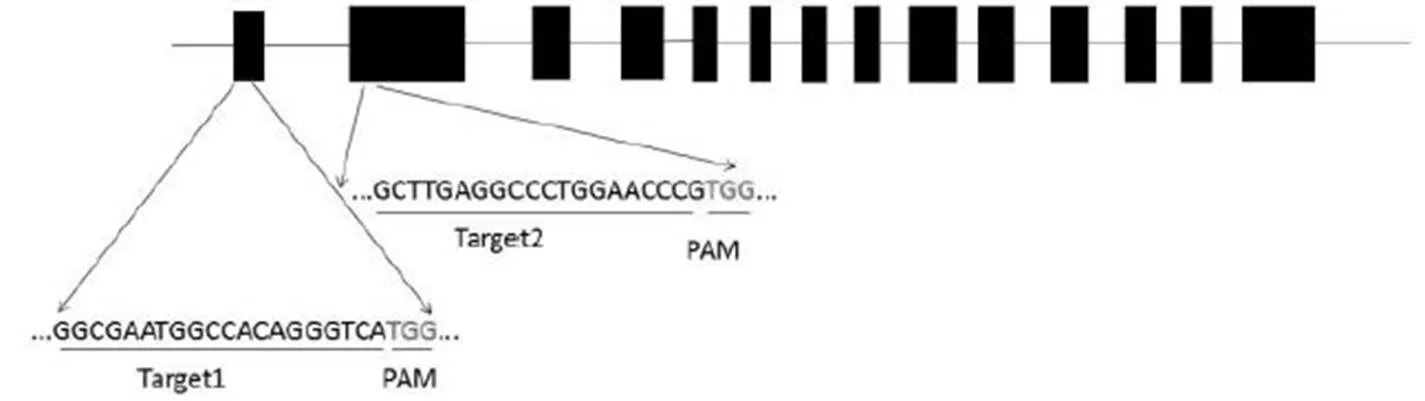
图1 2个gRNA靶点在Wx基因的位置
Fig. 1. Positions of the two targets ingene.

LB-左边界;U6-水稻U6启动子;SG-向导RNA;UBI-UBI启动子;NOS Ter-NOS终止子;35S-35S启动子;Hpt-潮霉素基因;PolyA Ter-PolyA终止子;RB-右边界。
Fig. 2. Vector map of pGK-Wx-gRNA.
1.7 直链淀粉含量测定
阳性植株T0种子和野生型,正季种植于云南宜良,收获T1代和野生型种子,烘干并存放3个月以上,待理化性质稳定后,参照齐万清等[21]方法,测定稻米直链淀粉含量。

图3 转化株系L1~L9的PCR扩增结果
Fig. 3. PCR results of transformants L1-L9.
2 结果与分析
2.1 gRNA靶位点选择及表达载体构建
如图1所示,靶点1在基因第1外显子,序列为GGCGAATGGCCACAGGGTCATGG,靶点2在第2外显子,序列为GCTTGAGGCCCTGGAAC CCGTGG。表达载体pGK-Wx-gRNA如图2所示,能同时表达密码子优化过的Cas9蛋白和gRNA序列,可以高效编辑水稻基因。
2.2 转化植株的获得及T0代检测
每个靶点各获得了8个独立的转基因株系,T0代测序结果显示:靶点1,3个为双峰(杂合)株系,5个为纯合株系;靶点2,7个株系为成功敲除株系,4个为双峰(杂合)株系,3个为纯合株系。由于部分材料T0未收获到种子且植株死亡,仅详细分析9个代表性株系(靶点1的L1~L5和靶点2的L6~L9,其中L9为未成功敲除株系)。用相应的检测引物1和2(表1),PCR扩增L1~L5和L6~L9,包含靶序列在内的扩增片段约390 bp(图3)。扩增产物测序结果显示,靶点1的L1~L4单碱基T缺失,L5单碱基A插入,突变频率为100%;靶点2的L6~L8单碱基G缺失,L9无突变,突变频率为75%(图4)。氨基酸比对发现,L1~L8翻译提前终止(图5)。
2.3 T1和T2代突变植株分子检测
T1代测序结果显示,L1、L2和L3发生了再编辑,缺失相同的14个碱基;L4和L5突变情况与T0代相同;L6发生了再编辑,缺失双碱基GT和单碱基G;L7和L8突变情况同T0代。T2代测序结果显示,突变情况与T1代一致,表明T2代已经稳定(图6)。这些结果与前人报道一致,即T1代转基因植株靶序列的编辑位点还可能存在gRNA靶点识别的序列,导致发生再次编辑,从而使得T1突变具有不确定性,但是到了T2代以后该突变能够稳定遗传[22]。

下划线表示gRNA靶序列;阴影表示PAM序列;“-”示碱基缺失;a示碱基插入。-1表示单碱基缺失;+1表示单碱基插入。
Fig. 4. DNA sequence alignment for wild type and the nine transformants in T0generation.
Fig. 5. Amino acid sequence alignment for wild type and nine transformants in T0generation.

下划线表示gRNA靶序列;阴影表示PAM序列;“-”示碱基缺失;a示碱基插入。
Fig. 6. DNA sequence alignment for the wild type and the L1-L8 in T1and T2generations.
2.4 T1植株Wx基因qPCR检测
qPCR检测T1植株籽粒基因RNA相对表达量,结果发现8个突变株系都显著降低(<0.01,图7)。说明突变株系中基因转录极不正常,导致基因的表达量显著下调。
2.5 T1代稻米直链淀粉含量
L1~L8突变株系T1代稻米直链淀粉含量测定结果如表2所示,所有8个株系的直链淀粉含量均显著下降(<0.01),从17.5%下降到1.93%。与野生型糙米相比,突变株系米粒蜡质状,呈糯性,从表型上佐证了基因的成功编辑(图8)。

表2 野生型与转化株系L1~L8的直链淀粉含量
**表示野生型与转化株系在<0.01下差异显著(检验)。
**indicates significant difference between the wild type and transformants at<0.01(-test).
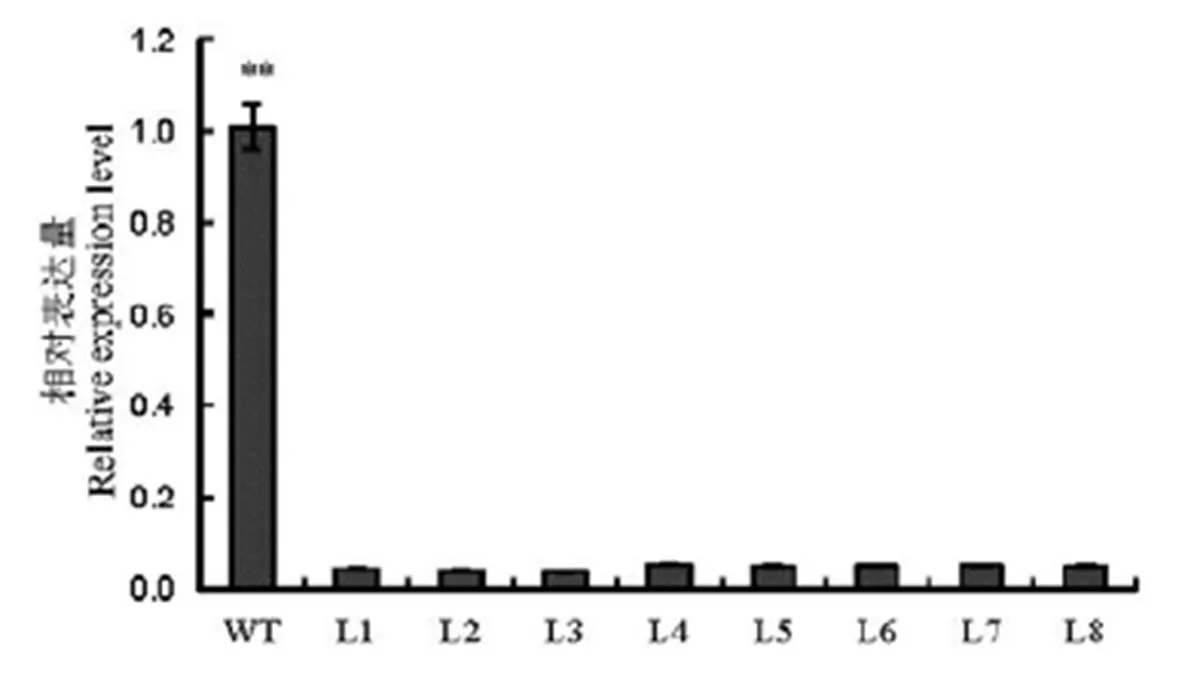
**表示野生型与T1突变体在P<0.01下差异显著(t检验)。
Fig. 7. Relative expression level ofgene in wild type and transformants in T1generation(Mean±).
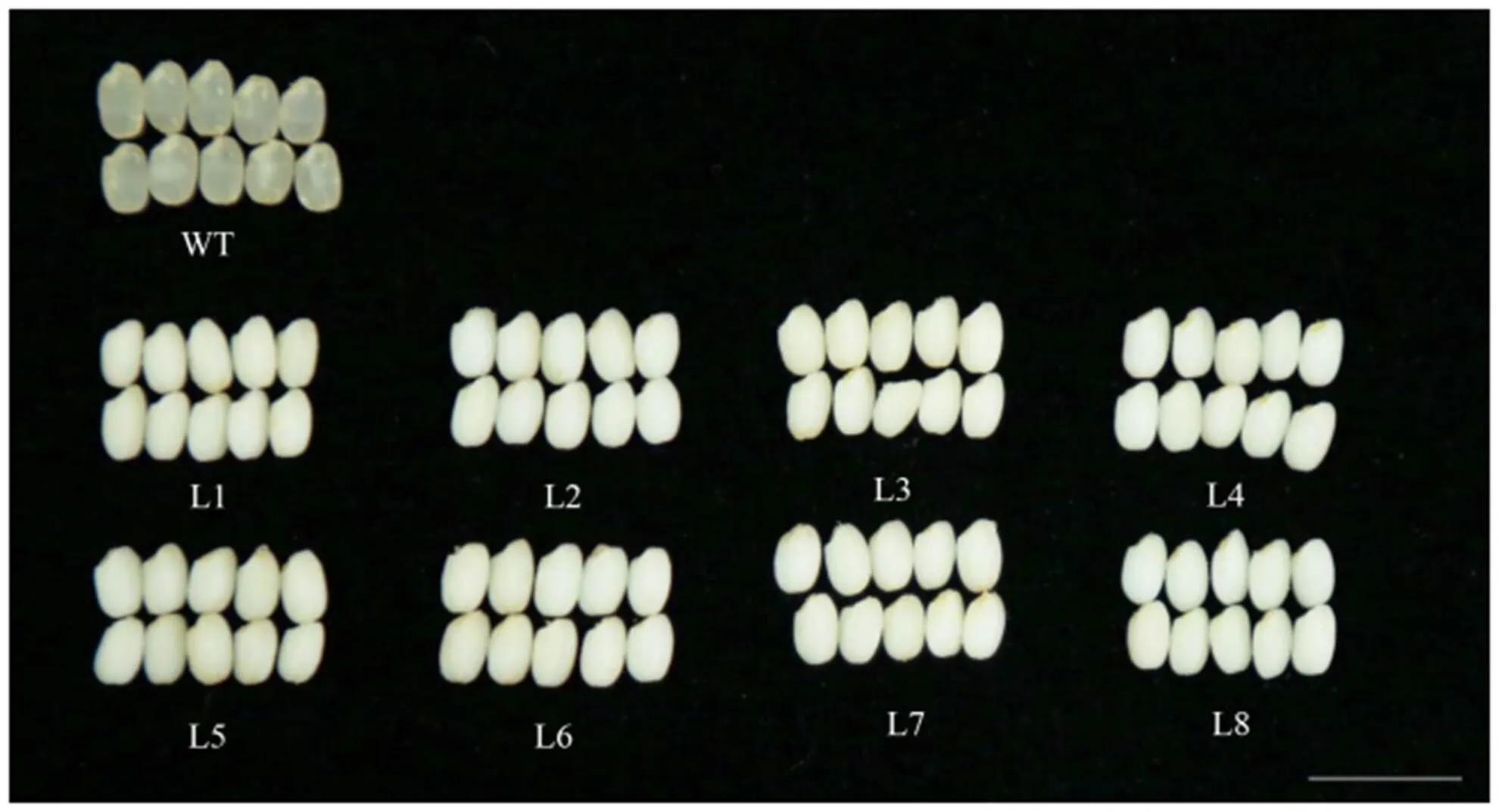
WT为楚粳27的糙米表型;L1~L8为突变株系T1代的糙米表型;比例尺,1 cm
Fig. 8. Brown rice of wild type and mutants.
3 讨论
CRISPR/Cas9是近年发展的基因组编辑技术,只需一个gRNA和一个Cas9蛋白就能对靶基因进行特异性修饰[23],该技术已经成功应用于水稻多个基因的编辑。编辑分蘖基因,突变体分蘖夹角显著增大[24];定点突变叶绿素合成基因,突变体叶片呈淡绿色[25];敲除基因,突变体幼苗表现出白化和矮化[15];编辑中花11香味基因,突变体籽粒中香味物质2-乙酰-1吡咯啉显著增加[26]。
有关水稻基因的定点编辑,Ma等[27]利用CRISPR/Cas9系统,在第1外显子近C端、第4内含子与第5外显子之间和第6内含子与第7外显子之间共设计了3个靶点,相应的启动子为U6a、U3和U6b,农杆菌介导转化台中65,获得3个独立的突变植株(Os-1~Os-3)。T1代检测发现Os-1在靶点1和靶点2均发生突变;Os-2在靶点2发生突变;Os-3在靶点1和靶点3均发生突变。稻米直链淀粉含量从14.6%降到2.6%,与航香糯相似。
与Ma等[27]突变位点不同,本研究设计的gRNA靶点位于基因第1外显子和第2外显子近N端,U6启动子驱动,超级稻楚粳27为受体,获得8个独立的突变体,靶点1(GC含量65%)突变频率100%,靶点2(GC含量70%)突变频率75%,与Ma等[27]认为靶点适宜的GC含量为50%~70%时突变频率高吻合。L1、L2、L3和L6在T1代发生再编辑,T2代稳定,与Xu等[22]研究结果一致。T1代突变体籽粒基因表达量显著降低,稻米直链淀粉含量从17.5%降到1.93%(表2),表现为糯性(图8)。
基因组编辑技术之前,稻米淀粉品质改良主要利用反义RNA或RNAi技术降低或抑制基因表达。Terada等[28]将反义基因分别导入粳稻和籼稻,得到稻米直链淀粉含量均显著降低的转基因植株;刘巧泉等[29]将转反义基因导入到武云粳7号,获得转反义基因植株,其稻米直链淀粉含量下降,粒重降低,实际产量变化不大;郭健[30]应用RNAi调控中花11的基因,转基因植株稻米直链淀粉含量从15.32%降到6.28%。本研究采用CRISPR/Cas9系统,定点编辑楚粳27的基因,能够实现基因的精准高效修饰,突变体的检测十分简便,基因是否被成功编辑可以从序列上直观地体现出来,这是传统转基因技术无法实现的。本研究的目标是获得直链淀粉含量适宜的楚粳27突变体,结果仅获得糯性的楚粳27突变体。稻米淀粉合成是由18个相关基因组成的复杂遗传调控网络[31],想要获得直链淀粉含量适宜和优质的水稻新材料,可能需要同时对多个淀粉品质形成相关基因进行编辑,这有待进一步研究。
[1] Hsu Y C, Tseng M C, Wu Y P, Lin M Y, Wei F J, Hwu K K, Hsing Y I, Lin Y R. Genetic factors responsible for eating and cooking qualities of rice grains in a recombinant inbred population of an inter-subspecific cross., 2014, 34(2): 655-673.
[2] Wani A A, Singh P, Shah M A, Schweiggert W U, Gul K, Idrees A. Rice starch diversity: Effects on structural, morphological, thermal, and physicochemical properties., 2012, 11(5): 417-436.
[3] Sano Y. Differential regulation of waxy gene expression in rice endosperm., 1984, 68(5): 467-473.
[4] Wang Z Y, Zheng F Q, Shen G Z, Gao J P, Snustad D P, Li M G, Zhang J L, Hong M M. The amylose content in rice endosperm is related to the post-transcriptional regulation of the waxy gene., 1995, 7(4): 613-622.
[5] 陈雅玲, 包劲松. 水稻胚乳淀粉合成相关酶的结构、功能及其互作研究进展. 中国水稻科学, 2017, 31(1): 1-12.
Chen Y L, Bao J S. Progress in structures, functions and interactions of starch synthesis related enzymes in rice endosperm., 2017, 31(1): 1-12. (in Chinese with English abstract)
[6] Frances H, Bligh J, Larkin P D, Roach P S, Jones C A, Fu H, Park W D. Use of alternate splice sites in granule-bound starch synthase mRNA from low-amylose rice varieties., 1998, 38(3): 407-415.
[7] Mikami I, Aikawa M, Hirano H Y, Sano Y. Altered tissue-specific expression at thegene of the opaque mutants in rice., 1999, 105(2): 91-97.
[8] Liu L, Ma X, Liu S, Zhu C, Ling J, Yi H W, Yi S, Yu L R, Hui D, Liang C, Xi L, Zhi Z, Hu Z, Jian W. Identification and characterization of a novelallele from a Yunnan rice landrace., 2009, 71(6): 609-626.
[9] Sato H, Suzuli Y, Sakai M, Lmbe T. Molecular characterization of Wx-mq, a novel mutant gene for low-amylose content in endosperm of rice (L.)., 2002, 52(2): 131-135.
[10] Larkin P D, Park W D. Association of waxy gene single nucleotide polymorphisms with starch characteristics in rice (L.)., 2003, 12(4): 335-339.
[11] Bergman C J, Delgado J T, McClung A M, Fjellstrom R G. An improved method for using a microsatellite in the rice Waxy gene to determine amylose class., 2001, 78(3): 257-260.
[12] Inukai T, Sako A, Hirano H Y, Sano Y. Analysis of intragenic recombination atin rice: Correlation between the molecular and genetic maps within the locus., 2000, 43(4): 589-596.
[13] Shen B, Zhang J, Wu H, Ma K, Li Z, Zhang X, Zhang P, Huang X. Generation of gene-modified mice via Cas9/ RNA-mediated gene targeting., 2013, 23(5): 720-723.
[14] Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna J A, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity., 2012, 337(6096): 816-821.
[15] Shan Q W, Wang Y P, Li J, Zhang Y, Chen K L, Liang Z, Zhang K, Liu J X, Xi J J, Qiu J L, Gao C X. Targeted genome modification of crop plants using a CRISPR-Cas system., 2013, 31(8): 686-688.
[16] Feng Z, Zhang B, Ding W, Liu X, Yang D L, Wei P, Cao F, Zhu S, Zhang F, Mao Y, Zhu J K. Efficient genome editing in plants using a CRISPR/Cas system., 2013, 23(10): 1229-1232.
[17] 李开斌, 张天春, 阮文忠, 徐加平, 王正伟, 黄文兴. 超级粳稻楚粳27号的选育及应用. 中国稻米, 2012, 18(5): 71-72.
Li K B, Zhang T C, Ruan W Z, Xu J P, Wang Z W, Huang W X. Breeding and application of super japonica rice variety Chujing 27., 2012, 18(5): 71-72. (in Chinese with English abstract).
[18] 曾千春, 李旭刚, 马炳田, 陈松彪, 徐鸿林, 孟昆, 魏晓丽, 朱祯. 有效去除农杆菌和籼稻转化系统优化. 分子植物育种, 2003, 1(5): 783-790.
Zeng Q C, Li X G, Ma B T, Chen S B, Xu H L, Meng K, Wei X L, Zhu Z. Efficient elimination ofand optimization of-mediated transfor- mation ofrice., 2003, 1(5): 783-790. (in Chinese with English abstract)
[19] Zhang G H, Gao M G, Zhang G Z, Sun J J, Jin X M, Wang C Y, Zhao Y, Li S S. A high through-put protocol of plant genomic DNA preparation for PCR., 2013, 39(7): 1200-1205.
[20] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2[-Delta Delta C(T)] method., 2001, 25(4): 402-408.
[21] 齐万清, 蒋刚生, 谢建华, 秦宣才, 李铮友, 曾千春. 杂交早粳稻米品质在四个生态点的变化. 西南农业学报, 2008, 21(6): 1500-1504.
Qi W Q, Jiang G S, Xie J H, Qin X C, Li Z Y, Zeng Q C. Quality variation of earlyhybrid rice from four ecological sites., 2008, 21(6): 1500-1504. (in Chinese with English abstract)
[22] Xu R F, Li H, Qin R Y, Li J, Qiu C H, Yang Y C, Ma H, Li L, Wei P C, Yang J B. Generation of inheritable and “transgene clean” targeted genome-modified rice in later generations using the CRISPR/Cas9 system., 2015, 5: 11491.
[23] Mussolino C, Cathomen T. RNA guides genome engineering., 2013, 31(3): 208-209.
[24] Miao J, Guo D, Zhang J, Huang Q, Qin G, Zhang X, Gu H, Qu L J. Targeted mutagenesis in rice using CRISPR- Cas system., 2013, 23(10): 1233-1236.
[25] Shan Q W, Wang Y, Li J, Gao C X. Genome editing in rice and wheat using the CRISPR/Cas system., 2014, 9(10): 2395-2410.
[26] 邵高能, 谢黎虹, 焦桂爱, 魏祥进, 圣忠华, 唐绍清, 胡培松. 利用CRISPR/CAS9技术编辑水稻香味基因. 中国水稻科学, 2017, 31(2): 216-222.
Shao G N, Xie L H, Jiao G A, Wei X J, Sheng Z H, Tang S Q, Hu P S. CRISPR/CAS9-mediated editing of the fragrant genein rice., 2017, 31(2): 216-222. (in Chinese with English abstract)
[27] Ma X L, Zhang Q Y, Zhu Q L, Liu W, Chen Y, Qiu R, Wang B, Yang Z F, Li H Y, Lin Y R, Xie Y Y, Shen R X, Chen S F, Wang Z, Chen Y L, Guo J X, Chen L T, Zhao X C, Dong Z C, Liu Y G. A Robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in Monocot and Dicot plants., 2015, 8(8): 1274-1284.
[28] Terada R, Nakajima M, Isshiki M, Okagaki R J, Wessler S R, Shimamoto K. Antisense Waxy genes with highly active promoters effectively suppressgene expression in transgenic rice., 2000, 41(7): 881-888.
[29] 刘巧泉, 王兴稳, 陈秀花, 王宗阳, 汤述翥, 洪孟明, 顾铭洪. 转反义基因糯稻的显性遗传及对稻米粒重的效应分析. 中国农业科学, 2002, 35(2): 117-122.
Liu Q Q, Wang X W, Chen X H, Wang Z Y, Tang S Z, Hong M M, Gu M H. Effect of dominant Waxy character on kernel weight of transgenic rice with antisensegene., 2002, 35(2): 117-122. (in Chinese with English abstract)
[30] 郭健. 应用RNA干涉技术改变水稻淀粉组成的研究. 广州: 华南农业大学, 2008.
Guo J. Improving the composition of rice with RNA interference technology. Guangzhou: South China Agricultural University, 2008. (in Chinese with English abstract)
[31] Tian Z X, Qian Q, Liu Q Q, Yan M X, Liu X F, Yan C J, Liu G F, Gao Z Y, Tang S Z, Zeng D L, Wang Y H, Yu J M, Gu M H, Li J Y. Allelic diversities in rice starch biosynthesis lead to a diverse array of rice eating and cooking qualities., 2009, 106(51): 21760-21765.
Gene Editing via CRISPR/Cas9 System in Rice
WANG Bingkun1,2,#, ZHANG Hui1,#, HONG Ruke1,ZHANG Jinwen3,YANG Rui2, LUO Qiong2,*, ZENG Qianchun1,*
(1College of Agronomy, Yunnan Agricultural University, Kunming 650201, China;2State Key Laboratory for Conservation and Utilization of Bio-Resources in Yunnan, Yunnan Agricultural University, Kunming 650201, China;3Institute of Food Crops, Yunnan Academy of Agricultural Sciences, Kunming 650205, China;#These authors contributed equally to this work;*Corresponding author, E-mail:qiongbf@aliyun.com,zengqianchun@qq.com)
Amylose content is closely related to rice quality. The objective of this research is to knockoutgene for obtaining rice mutants with suitable amylose content.Vector pGK03-Wx-gRNA, a CRISPR/Cas9 system, targeted either the first (target 1) or the second (target 2) exon ofgene was constructed and transformed into super-rice Chujing 27to edit thegene. Independent transformants were obtained via-mediated transformation with hygromycin selection. The transgenic lines of T0, T1and T2generationswereidentified byDNA sequencing, in addition to qPCR and amylose content assay of T1.Nine T0independent transgenic lines were generated, the L1-L5 from target 1 and L6-L9 from target 2. Eight lines (L1-L8) were mutants and L9 was not confirmed as mutant by sequencing. Therefore, the mutation rate was 100% for target 1, and 75% for target 2. T1and T2generations of the eight lines (L1-L8) were further sequenced, which revealed two types of mutation: base deletion (single base, double and up to fourteen bases), and base insertion. Some lines (L1, L2, L3 and L6) were reedited at T1generation when compared to its T0generation, while the rest lines were stable at the mutation sites. All the eight mutants were stable when comparing their target gene sequence at T1and T2generations. qPCR results indicated thatgene expression at RNA level was considerably reduced in the mutant. The seed amylose content was reduced from 17.5%to 1.93%(<0.01).Thegene was successfully edited by CRISPR/Cas9 system in rice, the inheritable mutants with average amylose content at 1.93% were obtained, which would provide valuable germplasm for rice quality improvement.
rice; CRISPR/Cas9 system;gene; gene editing; amylose content
10.16819/j.1001-7216.2018.7061
Q755; S511.033
A
1001-7216(2018)01-0035-08
2017-05-21;
2017-08-24。
国家转基因生物新品种培育重大专项(2014ZX0800939B); 国家现代农业产业技术体系资助项目(CARS-11-YNZQC); 云南农业大学自然科学青年科研基金资助项目(2016ZR11)。
