基于小尺度高原鼢鼠种群遗传结构研究
2018-01-19刘丽王贵珍周延山楚彬马素洁姬程鹏田永亮花立民
刘丽,王贵珍,周延山,楚彬,马素洁,姬程鹏,田永亮,花立民
(甘肃农业大学草业学院,教育部草业生态系统重点实验室,甘肃农业大学-新西兰梅西大学草地多样性研究中心,甘肃 兰州 730070)
青藏高原是我国以及东亚气候系统稳定的重要屏障,有丰富多样、独具特色的特殊生态系统类型和珍稀动植物种类,是全球生物多样性保护的重点区域[1]。广布于该区的高寒草甸在畜牧业生产和水源涵养等生态保育功能中发挥着极其重要的作用[2]。然而随着气候变化、人类干扰等因素,高寒草甸生态系统退化严重,其中草原鼠害是加剧退化的重要因素[3]。高原鼢鼠(Eospalaxbaileyi)是青藏高原高寒草甸生态系统优势地下害鼠,当其种群密度超过环境容量时,显著影响到草地生产力和植物群落结构[4]。但是,高原鼢鼠是土壤疏松、生物多样性维持的关键物种之一,有着 “生态系统工程师”的美誉[5-6]。
种群遗传分化研究是遗传资源利用和物种保护的基础[7]。遗传多样性是决定一个物种进化潜力和抵御不良环境能力的主要驱动力[8-9]。高原鼢鼠依靠挖掘地下洞道系统完成取食、扩散等行为[10]。稳定的地下生活环境和有限的迁移能力使不同地理种群间存在严重的限制性基因流[11]和独特的地域种群遗传结构[12]。蔡振媛等[12]通过测定线粒体控制区序列变异发现地理屏障与高原鼢鼠种群间遗传距离没有显著相关性,种群间遗传分化中大约79.6%的变异可以由地理隔离解释。但这些研究多集中在较大尺度的地理区域和历史事件对其种群结构的影响[11-12],小尺度特殊地理环境等对鼢鼠种群遗传结构影响的研究较少。研究尺度的差异会对遗传结构结果解释各异,而小尺度上的种群遗传结构则受到栖息地景观模式、个体永久性迁移频率及由于交配事件导致的基因扩散情况等多重因素的影响[13]。为进一步研究小尺度生境中环境因素对种群遗传的影响,本试验采用微卫星标记的方法,在祁连山东段高寒草甸区小尺度下,研究不同地理位置的高原鼢鼠种群遗传结构特点、基因交流及其影响因素,为该物种生物多样性保护和草原鼠害防治提供相关参考依据。
1 材料与方法
1.1 试验区概况
试验地选在甘肃省天祝县抓喜秀龙乡高寒草甸区。该区位于东祁连山的天祝金强河河谷,地理坐标为北纬37°11′,东经102°29′,海拔 2710~3080 m。境内地形受马牙雪山和雷公山隆起的影响,形成东西向的峡谷地带,西高东低。气候寒冷潮湿,太阳辐射强。年均温-0.1 ℃,>0 ℃年积温1380 ℃;年降水量416 mm,多为地形雨,集中于 7、8、9 三个月。无绝对无霜期,仅分冷热两季。天然草地主要为高寒草甸。主要植物有垂穗披碱草(Elymusdahuricus)、矮嵩草(Kobresiahumilis)、线叶嵩草(K.capillifolia)、二裂委陵菜(Potentillabifurca)、秦艽(Gentianamacrophylla)、扁蓿豆(Ruthenianmedic)、早熟禾(Poaannua)、狗娃花(Heteropappushispidus)、黄芪(Astragalusmembranaceus)、棘豆(Oxytropisbella)、蕨麻(Potentillaanserina)、苔草(Carexhumilis)等。
1.2 试验材料
试验材料总共包括163个高原鼢鼠个体,试验采样点分别在甘肃祁连山东段高寒草甸区的马营滩(MYT)、下南泥沟(XNG)、马营河东(MYR)和马营河西(MYL)共4个样地,如图1所示。MYT、XNG、MYL和MYR 的样地面积分别为7.50,1.60,1.25,1.25 hm2。其中,MYT样地中高原鼢鼠的个体采样点的最远直线距离为600 m,采样点范围约3 hm2;XNG样地中采样点的最远直线距离为500 m,采样点范围约1 hm2;MYL样地中采样点的最远直线距离为200 m,采样点范围约1 hm2;MYR样地采样点的最远直线距离为300 m,采样点范围约1.2 hm2。4个样地共采集鼢鼠的雌性个体数量为87;雄性个体数量为76。采取典型种群随机抽样法,分别在2014、2015年的5、6、8月用弓箭捕获鼢鼠,记录鼢鼠的性别、年龄后解剖获取部分肝脏组织,置于1.5 mL离心管中,先保存于液氮罐中,在实验室内转入-80 ℃超低温冰箱中保存。
1.3 DNA提取及微卫星扩增
鼢鼠肝脏组织的总DNA用上海生工生物技术有限公司提供的试剂盒(Ezup柱式基因组DNA抽提试剂盒,批号:12111677ZS)提取。操作过程中,将蛋白酶K的用量由原来的20 μL增加为40 μL,其他步骤不变。将提取好的DNA保存于-20 ℃的冰箱用于后续试验。
试验中SSR扩增用10个核苷酸微卫星位点的引物(ECTI5、ECTI6、ECTI8、ECTI10、ECTI21、ECTI22、ECTI23、ECTI33、 ECTI48、ECTI49)[14],这些位点在高原鼢鼠种群中具有多态性,可用于种群位点扩增。PCR扩增用荧光标记的引物,分别用HEX(绿光)、FAM(蓝光)和TAM(黄色)荧光基团在5′端标记合成荧光引物。
扩增体系为25 μL:其中包括1 μL的模板,上下游引物各0.5 μL,dNTP 10 mmol/L 0.5 μL,Taq Buffer 2.5 μL,25 mmol/L MgCl22.0 μL,Taq 酶0.2 μL,ddH2O 17.8 μL。用GeneAmp的PCR扩增系统9700,反应循环包括95 ℃下3 min的预变性(聚合酶激活);接着是10个循环95 ℃ 下30 s的变性;在60 ℃下退火30 s,在72 ℃延伸30 s;20个循环在95 ℃ 下变性30 s, 在55 ℃退火30 s,在72 ℃延伸30 s;最终72 ℃温度下再修复延伸6 min。PCR银光标记产物用毛细管电泳装置ABIPRISM3130XL的Gene Scan 500 LIZ分析计算扩增片段的大小。试验中引物扩增以及微卫星扩增反应委托上海生工生物工程有限公司完成。

图1 高原鼢鼠采样点地理分布Fig.1 Geographic distribution of plateau zokor sampling sites along the study area MYT,MYL, MYR, XNG, 分别代马营滩、马营河西边、马营河东边、下南泥沟种群。下同。MYT, MYL, MYR, XNG, replace Mayingtan, the west and east side of Maying river, Xianannigou population. The same below.
1.4 统计分析
微卫星属共显性遗传,通过STR分型实验得到每一扩增片段的具体大小。使用Popgene(Version 1.3)软件GenAlex 6.4统计微卫星基因座的等位基因数(observed number of alleles,Na)、有效等位基因数(effective number of alleles,Ne)、观测杂合度(observed heterozygosity,Ho)、期望杂合度(expected heterozygosity,He)、遗传相似系数(genetic similarity index,I)、种群间遗传距离(genetic distance,Ds)、种群间近交系数(FST)、基因流(Nm)等遗传分化系数及卡方检验种群哈迪温伯格平衡[15-17]。参照Botstein等[18]的方法计算多态信息含量(polymorphism information content,PIC)。利用 Structure和Distruct软件对等位基因数据进行遗传结构推导分析,确定种群类聚数K值[13]等。用Excel整理制作相关图、表。
2 结果与分析
2.1 遗传多样性
来自祁连山东段高寒草甸区4个采样点的163个高原鼢鼠DNA样品,在10个微卫星位点上进行基因分型,4个种群共获得164个等位基因(Na),有效等位基因(Ne)为83.6。MYT、MYL、MYR、XNG种群的平均有效等位基因分别为1.79,1.66,2.17,2.75,平均观测杂合度(Ho)分别为0.32,0.15,0.23,0.24,平均期望杂合度(He)分别为0.42,0.31,0.42,0.50,平均多态信息含量(PIC)分别为0.35,0.34,0.47,0.43。处于中等多态性水平(表1)。

表1 高原鼢鼠4个地理种群在10个微卫星基因座的多样性指数Table 1 The genetic diversity indices of 10 microsatellite loci for four plateau zokor populations
Ne:有效等位基因数;He:期望杂合度;Ho:表观杂合度;PIC:多态信息含量; -:无效数值。
Ne: Number of effective alleles;He: Expected heterozygosity;Ho: Observed heterozygosity;PIC: Polymorphism information content; -: Behalf invalid value.
2.2 种群哈迪温伯格平衡检验(卡方检验 )
基于最小Pearsonχ2估计的哈迪温伯格平衡(Hardy-Weinberg equilibrium)检验精确P值的无偏估测对各种群的多基因座检测(multi-locus test)发现,4个种群在10个位点上大多处于极显著不平衡状态(P<0.001)。MYT种群的位点ECTI16、ECTI48和ECTI49、MYL和MYR种群的位点ECTI48、XNG种群的位点ECTI8和ECTI21处于遗传平衡状态(P>0.05)。种群MYL的ECTI8 和ECTI22位点处的基因型单一(表2)。种群偏离哈迪温伯格平衡,表明高原鼢鼠种群间自由交流具有一定的局限性,或近亲交配现象严重。

表2 种群哈迪温伯格平衡检验(卡方检验 )Table 2 Hardy-Weinberg equilibrium testing (chi-square test) of plateau zokor populations
ns,无差异显著性(P>0.05), **差异极显著(P<0.01),***差异极显著(P<0.001), -,单型。
ns mean no significant difference,P>0.05, ** mean very significant difference,P<0.01, *** mean extremely significant differences,P<0.001, -, monomorphic.
2.3 遗传结构分析

图2 用Structure推导的4个高原鼢鼠种群遗传结构(K=2, 3, 4, 5)Fig.2 Structure analysis of 4 plateau zokor populations (K=2, 3, 4, 5)
依据贝叶斯群聚方法[19],利用Structure和Distruct软件[20]对研究的4个鼢鼠种群10个微卫星座位等位基因数据进行遗传结构推导分析。结果如图2所示,分别为当遗传聚类种群数K=2, 3, 4, 5时的遗传结构图。根据K值对后验概率自然对数lnP(D)[21]所做的曲线图(图3)表明,在K=2时,lnP(D)的值最大,随后数值都较小。分析各种群的遗传结构可知,本研究中 4个高原鼢鼠种群被分为 2个分类簇,分别用红、绿两种颜色表示,如图2所示。即当K=2时,4个种群被分成2组:MYT种群为一组,MYL、MYR、XNG种群聚在一起为一组。
同时参照 Evanno 等[22]的方法利用 Structure 软件对来自 4个不同高原鼢鼠种群进行贝叶斯聚类分析,聚类分类从K=1 到K=5,每个K重复运行 10 次,每次进行了 1000000 次马尔科夫链蒙特卡罗重复搜索(MCMC),舍弃最初的 50000 次。根据 ΔK方法(基于K的似然函数变动率)计算最大可能性的K值,结果为K=2(图4)。这一结果与根据K值对后验概率自然对数lnP(D)做曲线所得结果一致。

图3 后验概率自然对数[ln P(D)]与遗传聚类种群数(K)的关系Fig.3 Relationship of ln probability data [ln P(D)] and population clusters (K)

图4 利用 Evanno K 方法绘制不同种群分类簇K值变动Fig.4 The number of genetic clusters (K) by Delta K for different populations
用GenAlEx软件进行高原鼢鼠居群4个不同区域163个个体基于10对SSR引物的遗传距离的主坐标分析(PCoA),前2个极轴坐标(Coordinate)分别解释了高原鼢鼠种群整体遗传变异的47.58%和8.20%。从图 5 中可看到位于MYT样地的个体聚为一组,而XNG、MYL、MYR三个样地的个体倾向于聚为一组。得到类似的遗传结构。

图5 基于遗传距离的主坐标分析(PCoA)Fig.5 Principal coordinate plot of genetic distance(PCoA)
2.4 种群遗传分化的因素分析
以不同种群间的遗传结构分析结果为基础,结合近交系数(FST)与基因流(Nm)分析这4个不同地理种群间的遗传分化情况。种群间无遗传分化的标准值为FST=0~0.05[23],阻止种群间遗传分化的基因流值标准是Nm>1。如表3所示,4个不同地理种群两两之间的FST值都大于0.05,种群间的基因流值都小于1。种群XNG和MYR之间的基因流最大(0.616),MYT和MYL之间的基因流最小(0.040)。XNG样地与MYR、MYL样地间的地理距离相当,但MYR和MYL样地间存在天然河流,XNG与MYR间的遗传分化程度(0.061)小于XNG与 MYL间的(0.120);并且河流两侧样地MYR与MYL、MYT与MYL间遗传分化程度分别为0.130、0.470。XNG与MYT样地间存在公路,XNG与MYT 间的遗传分化程度(0.339)大于XNG与 MYR间的(0.061)。综上所述,天然河流和公路可能对种群扩散有一定的阻碍作用,特殊栖息地环境影响种群与其他种群间的信息交流。
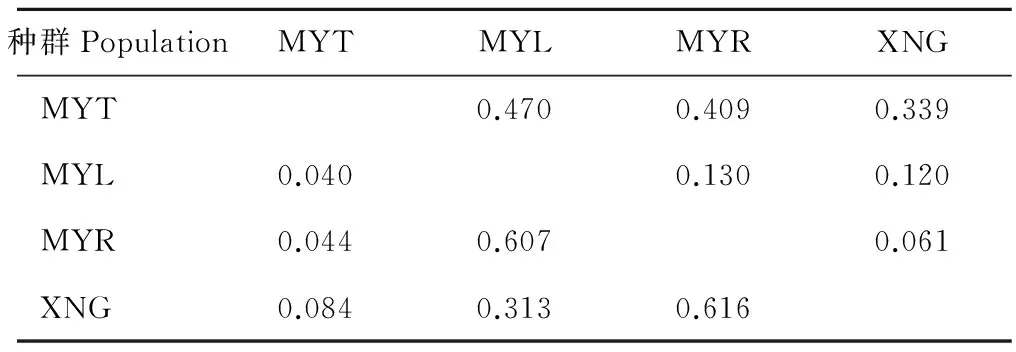
表3 4个高原鼢鼠种群的遗传分化程度(FST,右上角)和基因流(Nm,左下角)Table 3 Fixation index resulting from comparing subpopulations to the total population (FST, above the diagonal) and gene flow (Nm, below the diagonal) populations
3 讨论与结论
遗传多样性是生物固有的特性,是生物长期适应与进化的产物。每一生物的遗传多样性越丰富,对环境变化的适应能力就越强,就越容易扩展其分布范围和开拓新的环境[24]。等位基因数是种群遗传多样性分析的重要参数之一[25],本研究中高原鼢鼠种群共获得164个等位基因,有效等位基因为83.6。种群平均多态信息含量(PIC)为0.40,遗传多态性处于中等水平,与苏军虎[26]的研究结果一致。栖息地破碎化及严重的近亲繁殖,阻碍种群间基因流动,是物种遗传多样性水平降低主要原因。高原鼢鼠有独特的生活方式,主要依靠挖掘地下洞道系统完成活动、取食、扩散等行为。高耗能的挖掘行为严重制约其长距离的迁移扩散活动,使扩散率低于地上活动的动物[27]。低的迁移能力使种群间的基因流受到限制,增加种群内部近亲繁殖的可能,最终使种群遗传多样性降低,产生明显的种群遗传结构模式[28-30]。
对种群遗传研究发现种群的遗传隔离会有潜在的种的分化,严重的限制性基因流会导致种群分化[31]。高原鼢鼠种群进行遗传结构推导分析中,在K=2时,lnP(D)和ΔK的值都最大,4个种群被分成2组:MYT种群为一组,MYL、MYR和XNG种群聚在一起为一组。4个种群两两之间的FST值都大于0.05。种群MYT和MYL之间的遗传分化程度较高(0.770),并且种群之间较小的基因流值(小于1)不能阻止其分化。种群遗传学认为空间距离、地理障碍等通常是阻碍基因交流、导致分化的重要因素[32]。XNG与MYR间的遗传分化程度(0.061)小于XNG与MYL间的(0.120),并且种群XNG和MYR之间的基因流(0.616)大于XNG和MYL之间的(0.313)。MYL和MYR之间的距离不到200 m,中间有河流隔离。XNG与MYT 间的遗传分化程度(0.339)大于XNG与MYR间的(0.061),XNG与MYT样地间存在公路。表明在试验范围内特殊岛状栖息地限制了高原鼢鼠种群基因流,河流和公路对种群内和种群间个体交流可能有阻碍作用。蔡振媛等[12]研究发现种群间遗传分化中大约79.6%的变异可以由地理隔离解释。唐利洲等[11]研究结果表明高原鼢鼠不同地理种群间存在严重限制性基因流。国外其他地下啮齿动物的研究结果表明地理距离是造成种群遗传分化的主要原因[33],并且在整个种群扩散过程中河流、裂谷、火山对其有阻碍作用[34-36]。除了MYL种群,MYT种群的多态信息含量、等位基因数、期望杂合度都比其他2个种群的要低。岛状地理环境限制该种群与外界的交流,增加种群内部的个体交流的机会,长久积累不利于增加种群遗传多样性,进而减弱对环境变化的适应能力[37]。
从遗传学的角度分析高原鼢鼠遗传多样性、遗传结构特征及影响因素,种群遗传多样性处于中等水平,河流和公路对种群基因交流可能有阻碍作用。对高原鼢鼠来说,长期的特殊生存环境对整个种群的遗传特征有较大影响,不利于适应新的栖息环境。高原鼢鼠作为草原害鼠,严重破坏草地植被,控制其种群数量是草原保护工作的主要内容之一。由于高原鼢鼠对新环境的适应能力较差,可通过改变其栖息地环境达到减少种群数量的目的,有利于草地生态系统健康持续发展。
References:
[1] Sun H L, Zheng D, Yao T D,etal. Protection and construction of the national ecological security shelter zone on Tibetan Plateau. Journal of Geographical Sciences, 2012, 67(1): 3-12.
孙鸿烈, 郑度, 姚檀栋, 等. 青藏高原国家生态安全屏障保护与建设. 地理学报, 2012, 67(1): 3-12.
[2] Zhou X R, Guo Z G, Guo X H. The role of plateau pika and plateau zokor in alpine meadow. Pratacultural Science, 2010, 27(5): 38-44.
周雪荣, 郭正刚, 郭兴华. 高原鼠兔和高原鼢鼠在高寒草甸中的作用. 草业科学, 2010, 27(5): 38-44.
[3] Fan N C, Wang Q Y, Zhou W Y,etal. The relationship between plateau zokor populations and vegetation damage. Essays of Alpine Meadow Ecosystem International Academic Discussion. Beijing: Science Press, 1988: 109-115.
樊乃昌, 王权业, 周文扬, 等. 高原鼢鼠种群数量与植被破坏程度的关系. 高寒草甸生态系统国际学术讨论文集. 北京: 科学出版社, 1988: 109-115.
[4] Smith A T, Foggin J M. The plateau pika (Ochotonacurzoniae) is a keystone species for biodiversity on the Tibetan plateau. Animal Conservation, 1999, (2): 235-240.
[5] Golley F B, Petrusewicz K, Ryszkowski L. Smallmam-mals: their productivity and population dynamics. International Biological Programme. England: Cambridge University Press, 1975: 1-23.
[6] Jones C G, Lawton J H, Shachak M. Organisms as ecosystem engineers. Oikos, 1994, 69: 373-386.
[7] Zhang F M, Ge S. Data analysis in population genetics I. analysis of RAPD data with AMOVA. Biodiversity Science, 2002, 10(4): 438-444.
张富民, 葛颂. 种群遗传学研究中的数据处理方法Ⅰ.RAPD数据的AMOVA分析. 生物多样性, 2002, 10(4): 438-444.
[8] Chen L Z. Biodiversity Situation and It’s Conservation Measure in China. Beijing: Science Press, 1993: 99-113.
陈灵芝. 中国的生物多样性现状及其保护对策. 北京: 科学出版社, 1993: 99-113.
[9] Hedrick P W. Genetics of Populations. 2nd ed. Massachusetts: Jones and Bartlett Publishers, 2000: 1-61.
[10] Zhang Y M, Liu J K. Effects of plateau zokor (Myospalaxfontanierii) on plant community and soil in an alpine meadow. Journal of Mammalogy, 2003, 84: 644-651.
[11] Tang L Z, Yu L, Wang J J,etal. Gene flows of eospalax baileyi geographical populations. Journal of Anhui Agricultural Sciences, 2010, (10): 5123-5124.
唐利洲, 于龙, 王俊杰, 等. 高原鼢鼠种群间基因流研究. 安徽农业科学, 2010, (10): 5123-5124.
[12] Cai Z Y, Zhang T Z, Ci H X,etal. Mitochondrial phylogeography and genetic diversity of plateau zokor (Myospalaxbaileyi). Acta Theriologica Sinica, 2007, 27(2): 130-137.
蔡振媛, 张同作, 慈海鑫, 等. 高原鼢鼠线粒体谱系地理学和遗传多样性. 兽类学报, 2007, 27(2): 130-137.
[13] Mora M S, Mapelli F J, Gaggiotti O E,etal. Dispersal and population structure at different spatial scales in the subterranean rodent (Ctenomysaustralis). BMC Genetics, 2010, 11(9): 1-14.
[14] Su J H, Ji W H, Howitt R,etal. Novel microsatellite markers obtained from Gansu zokor (Eospalaxcansus) and cross-species amplification in Plateau zokor (Eospalaxbaileyi). Biochemical Systematics and Ecology, 2014, 57: 128-132.
[15] Yeh F C, Boyle T. POPGENE version 1.3-Microsoft Windows-based freeware for population genetic analysis. [2016-06-24]. https://sites.ualberta.ca/~fyeh/popgene.pdf. html.
[16] Piggott M P, Banks S C, Taylor A C. Population structure of brush-tailed rock-wallaby (Petrogalepenicillata) colonies inferred from analysis of faecal DNA. Molecular Ecology, 2006, 15(1): 93-105.
[17] Favre L, Balloux F, Goudet J,etal. Female-biased dispersal in the monogamous mammalCrocidurarussula: evidence from field data and microsatellite patterns. Biological Sciences, 1997, 264(1): 127-132.
[18] Botstein D, White R L, Skolnick M. Construction of a genetic linkage map in man using restriction fragment length polymorphism. American Journal of Human Genetics, 1980, 32(3): 314-331.
[19] Falush D, Stephens M, Pritchard J K. Inference of population structure using multilocus genotype data: linked loci and correlated allele frequencies. Genetics, 2003, 164(4): 1567-1587.
[20] Rosenberg N A. Distruct: a program for the graphical display of population structure. Molecular Ecology Notes, 2004, 4: 137-138.
[21] Rossiter S J, Benda P, Dietz C,etal. Rangewide phylogeography in the greater horseshoe bat inferred from microsatellites: implications for population history, taxonomy and conservation. Molecular Ecology, 2007, 16(22): 4699-4714.
[22] Evanno G, Regnaut S, Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study. Molecular Ecology, 2005, 14(8): 2611-2620.
[23] Wright. Evolution and the Genetics of Populations. America: University of Chicago Press, 1978.
[24] Jie X M, Yun J F. Genetic diversity and detective methods of plant. Grassland of China, 2000, (6): 51-59.
解新明, 云锦凤. 植物遗传多样性及其检测方法. 中国草地, 2000, (6): 51-59.
[25] Masatoshi N, Sudhir K. Molecular Evolution and Phylogeny. Lv B Z, Zhong Y, Gao L P, translate. Beijing: Higher Education Press, 2002: 204-207.
Masatoshi N, Sudhir K. 分子进化与系统发育. 吕宝忠, 钟扬, 高莉萍, 译. 北京: 高等教育出版社, 2002: 204-207.
[26] Su J H. Study on Population Genetic Structure of Two Kinds of Typical Native Animals in the Eastern Qinghai-Tibetan Plateau. Lanzhou: Agricultural University, 2014.
苏军虎. 青藏高原东缘两类典型土著动物种群遗传结构分析. 兰州: 甘肃农业大学, 2014.
[27] Wei W H, Wang Q Y, Zhou W Y,etal. The population dynamics and dispersal of plateau zokor after removing. Acta Theriologica Sinica, 1997, 17(1): 53-61.
魏万红, 王权业, 周文扬, 等. 灭鼠干扰后高原鼢鼠的种群动态与扩散. 兽类学报, 1997, 17(1): 53-61.
[28] Duffy J E. Genetic population structure in two tropical sponge-dwelling shrimps that differ in dispersal potential. Marine Biology, 1993, 116(3): 459-470.
[29] Duran S, Palacín C, Becerro M A,etal. Genetic diversity and population structure of the commercially harvested sea urchinParacentrotuslividus(Echinodermata,Echinoidea). Molecular Ecology, 2004, 13(11): 3317-3328.
[30] Duran S, Pascual M, Estoup A,etal. Strong population structure in the marine sponge Crambe crambe (Poecilosclerida) as revealed by microsatellite markers. Molecular Ecology, 2004, 13(3): 511-522.
[31] Quan Y C, Li D Y, Cao D C,etal. Population genetic variation and structure analysis on five populations of mirror carpCyprinuscarpioL. using microsatellites. Hereditas, 2006, 28(12): 1541-1548.
全迎春, 李大宇, 曹鼎辰, 等. 微卫星DNA标记探讨镜鲤的种群结构与遗传变异. 遗传, 2006, 28(12): 1541-1548.
[32] Hewitt G M. Genetic consequences of climatic oscillations in the Quaternary. Philosophical Transactions of the Royal Society of London Series B: Biological Sciences, 2004, 359: 183-195.
[33] Mora M S, Cutrera A P, Lessa E P,etal. Phylogeography and population genetic structure of the Talas tuco-tuco (Ctenomystalarum): integrating demographic and habitat histories. Journal of Mammalogy, 2013, 94(2): 459-476.
[34] Van Daele P, Verheyen E, Corkery I,etal. Trends in skull morphology in relation to differential molecular evolution in African mole-rats of the chromosomally hyperdivers genusFukomys(Bathyergidae, Rodentia) from the Zambezian region. Italian Journal of Mammalogy, 2006, (Suppl 1): 143.
[35] Brown G G, Gadaleta G, Pepe G,etal. Structural conservation and variation in the D-loop-containing region of vertebrate mitochondrial DNA. Journal of Molecular Biology, 1986, 192(3): 503-511.
[36] Jianping S U. Energy cost of foraging and optimal foraging in the fossorial rodent (MyospalaxbaileyI). Acta Theriologica Sinica, 1992, 2: 4.
[37] Huenneke L F. Ecological implications of genetic variation in plant populations//Falk D A, Holsinger K E. Genetics and Conservation of Rare Plants. New York: Oxford University Press, 1991: 31-44.
