干扰MHR1对嗜热毁丝霉纤维素酶活性的影响
2018-01-17龚艳芬来亚鹏邓婷婷
王 娟,龚艳芬,2,来亚鹏,邓婷婷,谢 宁,刘 刚
1)深圳大学生命与海洋科学学院,深圳市微生物基因工程重点实验室,广东深圳 518060; 2)深圳市海洋生物资源与生态环境重点实验室,广东深圳 518060
自然界中,木质纤维素的年产量超过1.5×103亿t,是储存量巨大的可再生碳源,具有极高的开发潜力和应用前景[1].纤维素酶是一类可催化水解纤维素和半纤维素,生成寡糖或单糖的酶类[2].
嗜热真菌是一类适宜在40~50 ℃生长的真菌,其最高生长温度可达60 ℃[3-4].嗜热真菌耐高温,所产纤维素酶在高温条件下更稳定,活性更高,具有更强的降解纤维素的能力[5-6].嗜热毁丝霉(Myceliophthorathermophile)ATCC42464是一种丝状子囊真菌,是潜在的高效中高温酶库[7-8].嗜热毁丝霉由于耐高温,纤维素降解酶含量丰富引起了人们极大的兴趣[9-12].近年来,WANG等[13-14]分别对该菌转录调控因子Xyr1、Cre1和Ace1进行了研究,发现同源过表达xyr1基因,可提高该菌木聚糖酶活性;沉默阻遏因子Cre1或Ace1的基因表达,均可提高纤维素酶活性[15-16].相关研究也表明,其他丝状真菌中与纤维素酶调控相关的转录因子Cre1、Ace1和Xyr1在嗜热毁丝霉中也发挥着相似功能[17-18].
本研究应用RNA-Seq技术[19-21],对嗜热毁丝霉分别在葡萄糖和微晶纤维素培养条件下的基因表达进行差异分析[22-23],筛选到可能的负调控因子MHR1(GenBank 序列号为AEO57591.1).该因子由733个氨基酸组成,含有锌离子和DNA结合区,其中,135~558个氨基酸区段为真菌转录因子同源区域.该序列在NCBI蛋白数据库中BLAST的结果显示,MHR1与少数几个已知蛋白具有60%作用的同源性,其中,与嗜热毛壳菌(Chaetomiumthermophilum)的一种假定转录因子的同源度为63%.与其同源的转录因子功能均未知.本通过设计mhr1基因的干扰片段,构建RNA干扰表达载体,研究了沉默mhr1基因表达对嗜热毁丝霉纤维素酶活性、木聚糖酶活性及相关基因表达的影响.
1 材料与方法
1.1 实验材料
1.1.1 质粒与菌种
pAN7-1质粒是带有真菌筛选标记潮霉素抗性基因hph及大肠杆菌筛选标记氨苄青霉素抗性基因Ampr; pUC19-M质粒是本实验室构建并保存的pUC19-MtPpdc-MtTpdc通用型表达载体[13].
大肠杆菌JM107由深圳大学苟德明教授惠赠;嗜热毁丝霉(MyceliophthorathermophilaATCC42464)购于美国菌种保藏中心(American Type Culture Collection,ATCC).
1.1.2 培养基
1)LB培养基.用于大肠杆菌的培养,含10.0 g/L 胰蛋白胨、5.0 g/L酵母提取物、10.0 g/L的NaCl、15.0 g/L的琼脂(固体培养基),pH=7.0.筛选重组质粒时,加入终质量浓度为100.0 μg/mL氨卞青霉素.
2)PDA培养基.含200.0 g/L土豆浸出液,10.0 g/L葡萄糖、20.0 g/L琼脂.200.0 g/L土豆浸出液作法如下,将土豆去皮切碎,每200.0 g土豆加水1 L,煮沸20 min,用3层纱布过滤,加入葡萄糖、琼脂后定容.筛选转化子时加入终质量浓度为50 μg/mL的潮霉素B.
3)孢子洗涤液.体积分数为0.1%的吐温-80溶液,用于制备嗜热毁丝霉孢子悬液.
4)Mandels营养盐浓缩液为(NH4)2SO414.0 g/L,尿素 3.0 g/L、KH2PO420.0 g/L、CaCl2·2H2O 4.0 g/L(或CaCl23.0 g/L)、MgSO4·7H2O 3.0 g/L,加水至1 L.
5)Mandels 微量元素浓缩液为FeSO4·7H2O 5.0 g/L、ZnSO4·7H2O 1.7 g/L(或ZnCl20.7 g/L)、CoCl2·6H2O 3.7 g/L(或CoCl22.0 g/L)、MnSO4·H2O 1.6 g/L(或 MnCl21.7 g/L或 MnSO4·7H2O 2.6 g/L),加水至1 L.
6)非诱导培养基.Mandels营养液浓缩液100 mL/L、Mandels微量元素浓缩液1.0 mL/L、蛋白胨1.0 g/L、葡萄糖20.0 g/L、1 mol/L的柠檬酸缓冲液(pH=4.5)50 mL/L、吐温80为1.0 mL/L.
7)诱导培养基.Mandels营养盐浓缩液100 mL/L、Mandels微量元素浓缩液1.0 mL/L、蛋白胨1.0 g/L、1 mol/L的柠檬酸缓冲液(pH=4.5)50 mL/L、吐温80为1.0 mL/L、麦秸秆粉30.0 g/L.
1.1.3 试 剂
DNA限制性内切酶购于美国ThermoFisher公司;DNA marker、 DNA连接酶、 RNA提取试剂(RNAiso Plus)、反转录试剂盒(PrimeScript reagent kit)、实时荧光定量PCR试剂盒SYBR®Premix ex taq Ⅱ(Tli RNaseH Plus)购于美国Takara公司;质粒提取试剂盒、PCR产物纯化试剂盒,DNA切胶回收试剂盒购于美国Omega公司;超保真PCR试剂盒(Phusion®High-Fidelity PCR kit)购于NEB公司;木聚糖、溶壁酶购于美国Sigma公司,改良型Bradford法蛋白浓度测定试剂盒及其它生化试剂和试剂盒均购于中国上海生工生物工程股份有限公司.
1.2 实验方法
1.2.1 mhr1基因的RNAi设计
在NCBI数据库中获取mhr1基因的全序列,运用RNAi设计软件Invitrogen Block-iT RNAi Designer设计选筛选合适的干扰序列,正义和反义序列分别为(5′-3′)mhr1senseGGCCGCAAGGCTTCTTGCGAAAT TTCAAGAGAATTTCGCAAGAAGCCTTGC;mhr1antiCTAGGCAAGGCTTCTTGCGAAATTCTCTTGAAATTTC GCAAGAAGCCTTGC.正义链和反义链的5′末端序列分别含有NotI和XbaI的限制性酶切位点,两处单下划线是互补的寡核苷酸序列,中间未划线部分为茎环结构.
1.2.2 嗜热毁丝霉基因组DNA、RNA的提取和cDNA的合成
接种嗜热毁丝霉孢子于PDA平板上,于45 ℃恒温培养7 d后,制备适量浓度为1×107mL-1的孢子悬液接种于液体基本培养基中,于45 ℃、250 r/min 条件下培养2 d后.取30 mL菌液进行抽滤,刮取滤纸表面的菌体,用于液氮研磨.使用基因组DNA提取试剂盒进行基因组DNA的提取.使用RNAiso Plus试剂提取RNA后反转录得到相应的cDNA模板,冻存于-20 ℃用于后续实验.
1.2.3 嗜热毁丝霉原生质体的制备与转化
嗜热毁丝霉原生质体的制备转化方法参照里氏木霉原生质体转化方法并进行了优化[13,24].将转化子涂布于潮霉素抗性平板和普通PDA平板上纯化分离3次,直至得到单一的纯菌.将纯菌接种于PDA平板上,45 ℃倒置培养7 d.阳性转化子利用基因组PCR测序进行验证.
1.2.4 实时荧光定量PCR
以cDNA为模板,使用SYBR Green I嵌合实时荧光定量聚合酶链式反应(quantitative real-time polymerase chain reaction,RT-qPCR)方法进行实验并分析目的基因的相对表达,以beta-tubulin基因(GenBank编号为AEO58945.1)为内参对照,所需引物序列见表2.反应程序为 ① 预变性95 ℃ 30 s;② 变性95 ℃ 5 s,退火、延伸60 ℃ 31 s,共40个循环;③ 融解曲线95 ℃ 30 s,60 ℃ 30 s,95 ℃ 15 s.利用2-ΔΔCt方法计算样品中目的基因相对表达量[25].
1.2.5 酶活与胞外蛋白浓度测定
取少许保种孢子液接种于PDA平板中,45 ℃倒置培养7 d,刮取孢子,制备孢子悬液.取1 mL浓度为1×107mL-1的孢子于非诱导培养基或诱导培养基中,45 ℃,250 r/min,摇床培养.从第3天至第8天取发酵上清液,1.2×104r/min离心10 min后进行酶活和胞外蛋白浓度的测定.滤纸酶活(filter paper activity, FPA)和内切葡聚糖酶活(EGase activity)的测定参考标准QB 2583—2003进行修改测定[26-27],使用96孔板反应体系为200 μL.木聚糖酶酶活(xylanase activity)的测定参照BAILEY等[28-29]的方法并进行优化.酶活单位定义为1 mL酶液在50 ℃、指定pH值(酸性纤维素酶pH=4.8),1 h水解底物,产生出相当于1 mg葡萄糖的还原糖量,为1个酶活力单位,以U/g(或U/mL)表示.以上酶活测定实验,每组均设置3个重复.
胞外蛋白浓度使用改良型Bradford法蛋白浓度测定试剂盒(产品编号为C503041)进行测量,具体步骤参照该试剂盒说明书.

表1 用于转化子验证及RT-qPCR的引物
2 结果与分析
2.1 mhr1基因RNA干扰质粒的构建
mhr1基因RNA干扰序列经设计、合成及退火后,分别与经NotI和XbaI双酶切后的质粒pUC19-M进行带黏性末端的连接反应,形成环形RNA干扰质粒pUC19-mhr1,质粒结构见图1.

图1 pUC19-mhr1质粒图Fig.1 (Color online) The constructure of plasmid pUC19-mhr1
将质粒pUC19-mhr1转化E.coli, 经过含100 μg/mL氨苄青霉素抗性平板筛选、以Mt-siRNA-mhr1-F和Mt-siRNA-mhr1-R为引物,通过菌液PCR检验载体完整性以及测序,最终确认成功构建载体.其中,1、2、5、6和7号为阳性质粒.对阳性质粒进行测序,2和6号基因序列完全正确,选取6号进行后续实验.
2.2 mhr1RNA干扰菌株的获得与筛选
将RNAi重组质粒pUC19-mhr1和pAN7-1共转入嗜热毁丝霉中,经含潮霉素的PDA平板筛选,获得6株转化子.于普通PDA平板上45 ℃倒置培养7 d后,提取基因组,以引物Mt-siRNA-mhr1-F和Mt-siRNA-mhr1-R进行基因组PCR验证,样品测序结果完全匹配,得到5株mhr1 RNAi阳性转化子,依次命名为MtR1、MtR2、MtR3、MtR4和MtR5.
在液体基本培养基培养条件下,分别提取野生型(wild type,WT)和5个转化子菌株的总RNA,经去基因组DNA处理后反转录为cDNA,分别以引物RTq-tub-F、RTq-tub-R和RTq-mhr1-F、RTq-mhr1-R作为内参基因和mhr1基因的引物,进行RT-qPCR .以WT的mhr1基因mRNA表达量为1作为对照,转化子为MtR1、MtR2、MtR3、MtR4和MtR5,基因mRNA表达量分别为0.090、0.252、0.724、0.061和0.010, 见图2. 由图2可见, 转化子MtR5的mhr1基因mRNA相对表达量最低,表明其RNA干扰效果最好,故选取转化子MtR5进行后续研究.

图2 mhr1基因mRNA表达水平的荧光定量分析Fig.2 RT-qPCR analysis of the expression of mhr1
2.3 转化子MtR5与野生型菌株的差异
从保种库中取出转化子MtR5和WT的保种孢子液,接种等量的孢子液于PDA平板上,生长7 d后,观察平板,发现野生型WT的平板颜色比转化子MtR5的平板颜色更深.
在刮取平板孢子时,发现WT的孢子层较厚,能成片刮取,且残留在平板上的孢子较少;而转化子MtR5的孢子层较薄,很难刮取,且残留在平板上的孢子较多,需要多次重复刮取.刮取等质量的50 mg孢子时,转化子MtR5的孢子层刮取面积为WT的2倍.
2.4 非诱导条件下胞外蛋白浓度与纤维素酶活性分析
在非诱导条件下培养野生型WT与转化子MtR5菌株,分别测定了其从第4天至第8天发酵液的胞外蛋白浓度、滤纸酶活、内切葡聚糖苷酶活和木聚糖酶活.实验结果如图3.培养至144 h时,MtR5的胞外蛋白浓度是WT的1.94倍,差异最大;此时,MtR5与WT的酶活差异也最大,其中,MtR5的FPA是WT的1.52倍,MtR5的内切葡聚糖苷酶活是WT的1.47倍,MtR5的木聚糖酶活是WT的1.20倍.

图3 非诱导条件下MtR5与WT的胞外蛋白浓度和纤维素酶活性Fig.3 The extracellular protein concentration, the cellulase activities in MtR5 and WT under non-inducing conditions
2.5 非诱导条件下主要纤维素酶基因的表达量分析
在非诱导条件下,分别提取MtR5与WT培养至144 h时菌丝的总RNA,对几种主要纤维素酶基因cbh1、cbh2、egl1和egl3, 及调控因子xyr1的表达水平进行RT-qPCR分析.经内参基因beta-tubulin定量校正,设定WT各基因的表达量为1,计算出MtR5中各基因的相对表达量.如图4所示,MtR5中cbh1、egl3和xyr1表达量分别为WT中的6.79、9.53和10.83倍,而MtR5中cbh2和egl1的表达量只比WT略有提高.
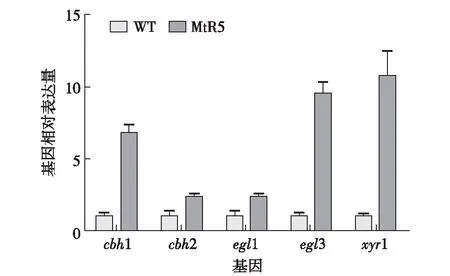
图4 非诱导条件下MtR5中主要纤维素酶基因及xyr1的表达水平Fig.4 Expression of main cellulase genes and xyr1 of MtR5 under non-inducing condition
2.6 诱导条件下胞外蛋白浓度与纤维素酶活性分析
以麦秸秆粉为诱导物的诱导条件下,培养野生型菌株WT与转化子菌株MtR5,分别测定了其从第3天至第5天发酵液的胞外蛋白质量浓度、滤纸酶活、内切葡聚糖苷酶活和木聚糖酶活.如图5所示.在培养至72 h时,MtR5与WT的胞外蛋白浓度和纤维素酶酶活均达到峰值,且MtR5与WT木聚糖酶酶活差异明显,MtR5是WT的1.48倍.而MtR5与WT的滤纸酶活和内切葡聚糖苷酶酶活则在培养第96 h时差异最大,分别是WT的1.33倍和1.65倍.
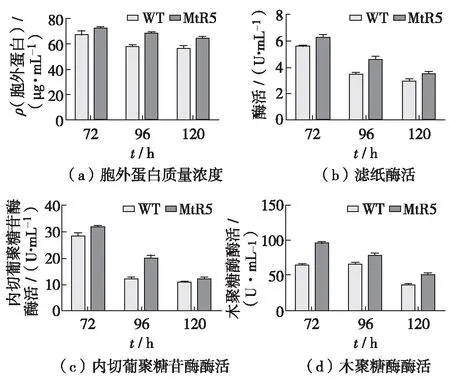
图5 以麦秸秆粉作诱导物条件下MtR5与野生型WT的胞外蛋白浓度和纤维素酶活性Fig.5 The extracellular protein concentration, the cellulase activities in MtR5 and WT under inducing conditions
2.7 诱导条件下主要纤维素酶基因的表达量分析
在诱导条件下,分别提取MtR5与WT培养第72 h时菌丝的总RNA,进行RT-qPCR,分别对几种主要纤维素酶基因cbh1、cbh2、egl1、egl3及调控因子xyr1的表达水平进行分析(图6).MtR5的几种主要纤维素酶基因表达量均明显提高,其中,cbh2的表达量为WT的37.36倍,egl3表达量为WT的56.14倍,xyr1的表达量为WT的28.30倍.

图6 以麦秸秆粉作诱导物条件下MtR5中主要纤维素酶基因表达水平Fig.6 Expression of main cellulase genes of MtR5 under inducing condition
3 讨 论
MHR1蛋白携带有真菌转录因子结构域(fungal_TF_MHR), 因此,可能是广义上的真菌转录因子.本实验结果显示,干扰该转录因子的表达可以增加诱导和非诱导条件下菌株纤维素酶的表达量,表明干扰mhr1基因表达后,主要纤维素酶基因表达量增加,说明MHR1是一种与纤维素酶基因表达调控相关的转录因子.
本研究RT-qPCR结果显示,重组菌株中纤维素酶和木聚糖酶基因的激活因子xyr1表达上调.故木聚糖酶含量的增加可能是mhr1基因的表达沉默引起的,也可能是上调了激活因子xyr1的表达间接造成的.该蛋白的功能目前尚无报道,通过序列比对分析发现数据库中与其有同源性的几个已知蛋白,例如,马杜拉足肿菌(Madurellamycetomatis)的丝状生长调控因子 XXⅦ 和应激基因Ⅰ的激活因子(69%同源性);嗜热毛壳菌(ChaetomiumthermophilumDSM 1495)的一种转录因子(同源性为63%);里氏木霉(TrichodermareeseiRUT C-30)中假定蛋白M419DRAFT_95750 (同源性为44%),上述几种蛋白的功能均未见相关报道.本研究表明转录因子MHR1对纤维素酶基因的表达有一定影响.影响纤维素酶活性的因素很多,除转录因子调控外,一些与表观调控相关的化学物质也会对纤维素酶有一定影响[30].
关于该转录因子仍有诸多问题尚不明确,如它与主要纤维素酶基因的结合位点,与已知的纤维素酶阻遏因子CRE1和ACE1有无关联等,均有待深入研究.
结 语
野生型WT和转化子MtR5无论是在诱导,还是非诱导培养条件下,转化子MtR5的胞外蛋白浓度、维素酶酶活均高于野生型WT,且经RT-qPCR检验,转化子MtR5的主要纤维素酶基因的表达量同样高于WT数倍到几十倍.说明干扰该转录因子可增加嗜热毁丝霉菌株纤维素酶的表达.该研究有助人们深入了解纤维素酶基因诱导表达调控的机制,可为嗜热真菌高产纤维素酶工程菌的构建提供借鉴.
引文:王 娟,龚艳芬,来亚鹏,等.干扰MHR1对嗜热毁丝霉纤维素酶活性的影响[J]. 深圳大学学报理工版,2018,35(1):15-21.
/
[1] CHUNDAWAT S S, BECKHAM G T, HIMMEL M E,et al. Deconstruction of lignocellulosic biomass to fuels and chemicals[J]. Annual Review of Chemical and Biomolecular Engineering, 2011, 2(2): 121-145.
[2] JUTURU V, WU Jinchuan. Microbial cellulases: engineering, production and applications[J].Renewable and Sustainable Energy Reviews, 2014, 33: 188-203.
[3] 来亚鹏,邓婷婷,刘 刚,等. 同源过表达BglR对嗜热毁丝霉β-葡萄糖苷酶活性的影响[J]. 中国生物工程杂志,2017,37(7):64-71.
LAI Yapeng, DENG Tingting, LIU Gang, et al. The influence of homologous overexpression ofBglRon β-glucosidase activities inMyceliophthorathermophila[J]. China Biotechnology, 2017,37(7):64-71.(in Chinese)
[4] MOUCHACCA J. Thermophilic fungi: biodiversity and taxonomic status[J]. Cryptogamie Mycologie,1997,18(1):19-69.
[5] MAHESHWARI R, BHARADWAJ G, BHAT M K. Thermophilic fungi: their physiology and enzymes[J].Microbiology and Molecular Biology Reviews,2000,64(3):461-488.
[6] MORGENSTERN I, POWLOWSKI J, ISHMAEL N,et al. A molecular phylogeny of thermophilic fungi[J].Fungal Biology,2012,116(4):489-502.
[7] BERKA R M, GRIGORIEV I V, OTILLAR R,et al. Comparative genomic analysis of the thermophilic biomass-degrading fungiMyceliophthorathermophilaandThielaviaterrestris[J].Nature Biotechnology,2011,29(10):922-927.
[8] MATSAKAS L, ANTONOPOULOU I, CHRISTAKOPOULOS P. Evaluation ofMyceliopthorathermophilaas an enzyme factory for the production of thermophilic cellulolytic enzymes[J]. BioResources, 2015, 10(3):5140-5158.
[9] VISSER H, JOOSTEN V, PUNT P J,et al. Research: Development of a mature fungal technology and production platform for industrial enzymes based on aMyceliophthorathermophilaisolate, previously known asChrysosporiumlucknowenseC1[J]. Industrial Biotechnology, 2011, 7(3): 214-223.
[10] KOLBUSZ M A, DI FALCO M, ISHMAEL N,et al. Transcriptome and exoproteome analysis of utilization of plant-derived biomass byMyceliophthorathermophila[J]. Fungal Genetics and Biology, 2014, 72: 10-20.
[11] SINGH B.Myceliophthorathermophilasyn.Sporotrichumthermophile: a thermophilic mould of biotechnological potential[J]. Critical Reviews in Biotechnology, 2016, 36(1): 59-69.
[12] LIU Qian, GAO Ranran, LI Jingen,et al. Development of a genome-editing CRISPR/Cas9 system in thermophilic fungalMyceliophthoraspecies and its application to hyper-cellulase production strain engineering[J].Biotechnology for Biofuels,2017,10(1):1.
[13] WANG Juan, WU Yaning, GONG Yanfen,et al. Enhancing xylanase production in the thermophilic fungusMyceliophthorathermophilaby homologous overexpression ofMtxyr1[J].Journal of Industrial Microbiology & Biotechnology,2015,42(9):1233-1241.
[14] KLAUBAUF S, NARANG H M, POST H,et al. Similar is not the same: differences in the function of the (hemi-) cellulolytic regulatorXlnR(Xlr1/Xyr1) in filamentous fungi[J]. Fungal Genetics and Biology, 2014, 72: 73-81.
[15] YANG Fan, GONG Yanfen, LIU Gang,et al. Enhancing cellulase production in thermophilic fungusmyceliophthorathermophilaATCC42464 by RNA interference of cre1 gene expression[J]. Journal of Microbiology and Biotechnology, 2015, 25(7):1101-1107.
[16] 杨 帆. 转录因子CRE1及ACE1在嗜热毁丝霉纤维素酶基因表达调控中的作用[D]. 深圳:深圳大学,2015.
YANG Fan. The role of transcription factorsCRE1 andACE1 in regulation of cellulase gene expression ofMyceliophthorathermophilaATCC42464 [D]. Shenzhen: Shenzhen University, 2015.(in Chinese)
[17] AMORE A, GIACOBBE S, FARACO V. Regulation of cellulase and hemicellulase gene expression in fungi[J]. Current genomics, 2013, 14(4): 230-249.
[18] PORTNOY T, MARGEOT A, SEIDL-SEIBOTH V, et al. Differential regulation of the cellulase transcription factorsXYR1,ACE2, andACE1 inTrichodermareeseistrains producing high and low levels of cellulose[J]. Eukaryotic cell, 2011, 10(2): 262-271.
[19] MORIN R, BAINBRIDGE M, FEJES A,et al. Profiling the HeLa S3 transcriptome using randomly primed cDNA and massively parallel short-read sequencing[J].BioTechniques,2008,45(1):81-94.
[20] CHU Yongjun, COREY D R. RNA sequencing: platform selection, experimental design, and data interpretation[J].Nucleic Acid Therapeutics,2012,22(4):271-274.
[21] WANG Zhong, GERSTEIN M, SNYDER M. RNA-Seq: a revolutionary tool for transcriptomics[J].Nature Reviews Genetics,2009,10(1):57-63.
[22] TRAPNELL C, WILLIAMS B A, PERTEA G,et al. Transcript assembly and abundance estimation from RNA-Seq reveals thousands of new transcripts and switching among isoforms[J]. Nature biotechnology, 2010, 28(5): 511-515.
[23] SONESON C, DELORENZI M. A comparison of methods for differential expression analysis of RNA-seq data[J]. BMC bioinformatics, 2013, 14(1): 91.
[25] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCtmethod[J].Methods,2001,25(4):402-408.
[26] EVELEIGH D E, MANDELS M, ANDREOTTI R, et al. Measurement of saccharifying cellulase[J].Biotechnology for Biofuels,2009,2(1):21.
[27] MANSOUR A A, DA COSTA A, ARNAUD T, et al. Review of lignocellulolytic enzyme activity analyses and scale-down to microplate-based assays[J]. Talanta, 2016, 150: 629-637.
[28] 麦国琴,许晓萍,余翠媚,等.产木聚糖酶和纤维素酶真菌的酶学性质分析[J]. 食品研究与开发,2011, 32(9) :179-183.
MAI Guoqin, XU Xiaoping, YU Cuimei, et al. Screening of cellulase and xylanase-pruducing fungi from mangrove soil and characterization of the enzymatic reactions[J]. Food Research and Development, 2011, 32(9) :179-183.(in Chinese)
[29] BAILEY M J, BIELY P, POUTANEN K. Interlaboratory testing of methods for assay of xylanase activity[J]. Journal of Biotechnology, 1992, 23(3): 257-270.
[30] 周娇娇,佘炜怡,王浩入,等. 5-氮杂-2-脱氧胞苷对里氏木霉产纤维素酶的影响[J]. 深圳大学学报理工版,2017,34(2):122-131.
ZHOU Jiaojiao, SHE Huiyi, WANG Haoru, et al. Effect of 5-Aza-2′-deoxycytidine on the expression of cellulases inTrichodermareesei[J].Journal of Shenzhen University Science and Engineering,2017,34(2):122-131.(in Chinese)
