靶向敲除ck2α基因及其功能分析
2018-01-17金美玲
敖 英,张 杰,李 枫,金美玲
1)深圳大学基础医学院,广东深圳 518060;2)武汉大学基础医学院,湖北武汉 430072
CRISPR-Cas9技术的出现为基因编辑提供了广阔的平台.CRISPR-Cas9是细菌和古细菌在长期演化过程中形成的一种获得性免疫系统,以防御外来遗传物质,如噬菌体和质粒.细菌能通过成簇有规律间隔的短回文重复序列(clustered regularly interspaced short palindromic repeats, CRISPR)与内切酶Cas9组合,在核糖核酸(ribonucleic acid, RNA)的引导下剪切入侵基因.大约40%的细菌和90%的古细菌都拥有该系统[1].CRISPR-Cas第1个子系统负责新的间隔序列的获取,第2个子系统的主要功能是将CRISPR转录后的产物进行加工、识别并降解外源的脱氧核糖核酸(deoxyribonucleic acid, DNA)[2],根据结构分为TypeⅠ、TypeⅡ和Type Ⅲ.这3种类型CRISPR识别和切割DNA有着不同的分子机制,Type I和Type III需要大而复杂的Cas9内切酶蛋白,Type II仅需单个蛋白便能完成RNA介导的DNA识别和切割,因其简易性和可操作性被改造为基因编辑的良好工具[3-4].CRISPR-Cas系统行使功能包括3个阶段:① 新的间隔序列(spacer)的获取,外源遗传物质识别后被切割,外来DNA的一小段以间隔序列的形式插入CRISPR序列中;② CRISPR基因座的表达,当外源DNA再次入侵时,CRISPR的长RNA前体被转录出来(pre-CRISPR RNA),接着被CRISPR-associate(Cas)核酸酶切割其重复序列并得到单个的CRISPR RNA(crRNA),每个crRNA都由一个重复片段和入侵DNA的间隔序列构成;③ CRISPR-Cas系统对外来遗传物质的干扰,通过序列互补的形式,一种小的反式激活的CRISPR RNA(tracrRNA)促使crRNA的转录后加工和成熟,成熟的crRNA与特异性的Cas蛋白形成核糖核蛋白复合物,以核酸酶活性摧毁外源遗传物质[5-6].Cas9蛋白具有内切酶活性的切割域,能特异性识别tracrRNA-crRNA二聚体,在crRNA间隔序列的介导下同源互补识别20碱基对(base pair, bp)的靶序列并进行切割,HNH活性中心切断与crRNA互补的一条链,RuvC活性中心切断非互补链[5,7].JINEK等[7]将tracrRNA-crRNA改造成的单个引导RNA,使其同时具有tracrRNA和crRNA的特性,简称sgRNA(small guide RNA, sgRNA),模拟体内的二聚体结构,它由两个关键部分组成:5′端20 bp的核苷酸序列用于互补识别靶位点,3′端双链发卡结构用于和Cas9蛋白结合.这样,人们便可以针对任何邻近PAM区的位点设计sgRNA进行基因打靶.利用CRISPR-Cas9的特性,MALI等[8-9]率先在《Science》报道采用CRISPR-Cas9技术在哺乳动物细胞上实现了基因敲除,紧接着,科学家们分别在人类和小鼠细胞各个类型的细胞包括诱导多能干细胞上进行了基因敲除工作.
由两个催化亚基(α和α′)和两个调节亚基(β)组成的蛋白激酶CK2(casein kinase 2)主要磷酸化蛋白丝/苏氨酸残基,也可以少部分磷酸化蛋白酪氨酸残基,α和α′亚基具有组织特异性,且单独存在也具有活性[10].CK2参与了多种细胞功能的调控,如基因表达调控、蛋白质合成与降解、DNA损伤修复、细胞增殖与分化和细胞死亡等,它的基因表达方式是管家基因模式,无需第二信使介导其活性,因此在细胞功能调控中处于中心地位[11].以往研究证明,ck2基因在许多原发性肿瘤组织中的表达普遍上调,在肺癌中,ck2α表达量高,抑制CK2酶活性可以降低Notch1的转录活性,抑制肺癌的增殖、运动和侵袭能力[12],而结肠癌死亡率也非常高,在所有癌症中死亡率位居第4,目前对于CK2调节结肠癌发生机制的报道还很匮乏,免疫组化分析104位结肠癌病人和40位结直肠腺瘤病人组织,其中,结直肠癌细胞核中ck2α基因表达量高于结直肠腺瘤[13].综上研究表明,CK2α蛋白在很多疾病的发生发展中起着重要的调控功能,因此,利用新方法在基因水平上敲除或者过表达ck2α基因,有利于深入研究CK2调控相关疾病的分子机制.因此,本研究重点阐述利用CRISPR-Cas9技术构建ck2α基因敲除的细胞系,并初步探讨其功能,以期为深入研究其分子机理提供技术支持.
1 材料和方法
1.1 主要试剂
基因扩增、载体构建各种酶类、RNA提取反转录及实时荧光定量聚合酶链式反应(quantitative real-time polymerase chain reaction,RT-qPCR)试剂盒均购于日本Takara公司;CK2α、P53、P21、PML及actin抗体购于美国Cell Signal Technology公司;辣根过氧化物标记的山羊抗兔二抗及抗鼠二抗购于美国Abcam公司;SA-β-gal染色试剂盒购于美国Cell Signaling Technology公司.细胞增殖检测试剂盒(Owen’s reagent, MTS)购于美国Promega公司.
1.2 研究方法
1.2.1 获得CRISPR-Cas9敲除CK2α稳定转染HCT116细胞株
从GeneBank网站下载CK2α的基因序列(登陆号为NM-177560),设计guide RNA targeting CK2α,委托英潍捷基(广州)生物科技有限公司合成,并将其连接至终载体PX459-g CK2α上,转染至HCT116细胞中,48 h后加入puromycin(1 μg/mL)筛选约5 d,传代细胞待长出单克隆以后挑取克隆进行扩繁并鉴定是否敲除成功.将蛋白水平验证正确的细胞株DNA送公司测序,最终冻存敲除成功的细胞株系.
1.2.2 RNA提取及实时荧光定量PCR
收取细胞样品,利用TRIzol试剂盒按说明提取总RNA.测定RNA浓度及纯度,利用反转录试剂盒逆转录成cDNA;用2 μg的RNA进行逆转录.再利用产物作为模板,荧光定量引物(表1),使用RT-qPCR试剂盒,利用ABI 7500 Fast RT-qPCR仪分析样品中目的基因mRNA含量.RT-qPCR反应体系为20 μL,成分为:cDNA 1 μL(将反转录的产物cDNA稀释,加入约400 ng),上下游引物(10 μmol/L)各0.5 μL,mixture (2×) 10 μL,H2O 8 μL.扩增程序条件为:95 ℃预变性5 min;反应条件为95 ℃ 15 s,55 ℃ 15 s,72 ℃ 40 s,共40个循环.各组扩增结果以beta-actin为内参,利用2-ΔΔCt法做相对定量分析.

表1 RT-qPCR引物序列
1.2.3 Western-blot检测蛋白表达水平
利用蛋白裂解液提取细胞总蛋白,BCA(bicinchoninic acid)法测定蛋白浓度,上样量20 μg,进行聚丙烯酰胺凝胶电泳,采用浓缩电压60 V,分离胶电压120 V.电泳结束后,用湿转法将蛋白转移到聚偏二氟乙烯膜(polyvinylidene fluoride, PVDF)上,湿转条件为恒流300 mA,1.5 h.转膜结束后用含50 g/L脱脂奶粉的杂交膜清洗液 (Tris-buffered saline with tween 20,TBST)室温封闭PVDF膜1 h,再利用相应的一抗4 ℃孵育过夜.孵育完成后,用磷酸盐缓冲液洗膜3次,每次10 min.室温孵育二抗1 h,洗膜3次,每次10 min.利用德国耶拿公司的化学成像系统,加入ECL发光液进行化学发光检测.
1.2.4 MTS检测细胞增殖速率
在96孔板中培养的CK2α敲除细胞及对照细胞,每孔100 μL培养基加20 μL CellTiter 96®AQueous one solution reagent,保证加样准确,每组细胞设置5个附孔,且进行3次生物学重复.在37 ℃,体积分数为5%的CO2环境下孵育2 h.利用酶标仪测定波长为496 nm的光密度值D(496), 以进行统计学分析.
1.2.5 细胞迁移能力的检测
接种细胞之前先用笔在6孔板背面画线标记.接种好细胞后,按文献[14]操作方法进行检测.经划线以后,取不同时间点拍照进行结果分析.
1.2.6 SA-β- gal染色
将培养在6孔板的敲除细胞和对照细胞,用 2 mL 1×PBS 洗1次,去除培养基和杂质;每个孔加入 1 mL 1×细胞固定液(用双蒸水稀释 10×的细胞固定液);弃掉固定液,用 1×PBS 洗3次;),每个孔加入 1 mL 细胞染色工作液(先用 1 mL二甲基甲酰胺稀释 20 mg X-gal,放置于-20 ℃贮存, 1 mL 的工作液包含 930 μL 1×染色液、 10 μL 100×solution A、10 μL 100×solution B、50 μL 20 mg/mL X-gal),37 ℃避光染色 20 h;用1×PBS洗3次,光学显微镜下观察拍照.
1.2.7 统计学方法

2 结果与分析
2.1 pX459-g CK2α重组表达载体的构建和鉴定
根据CRISPR-Cas9的构建原理设计定点敲除ck2α的sgRNA序列,如图1(a)所示.将构建好的表达载体pX459-g CK2α的4个不同克隆利用BamH I 和EcoR I双酶切进行验证,得到预期大小的目的条带,并且测序结果显示正确如图1(b),经测序进一步验证,报告显示正确如图1(c).与预期结果一致,表明表达载体构建成功.

图1 利用CRISPR-Cas9 对ck2α进行基因编辑Fig.1 Gene modification of ck2α by CRISPR-Cas9
2.2 Western blot及RT-qPCR检测ck2α基因敲除效率
将构建好的载体,转染HCT116结肠癌细胞,经过 puromycin抗性筛选以后,扩繁得到单个克隆的细胞系,命名为CK2α-KO,野生型为WT选择其中的两个株系CK2α KO-1和CK2α KO-2进行RT-qPCR检测和免疫印迹实验,结果如图2(a)所示.CK2α-KO的细胞系中,ck2α基因的表达显著降低,相对野生型细胞的16.5% 和19.1%;同时,蛋白水平的检测结果表明CK2α蛋白的表达相对于野生型细胞明显减弱,如图2(b)所示.成功获得了CK2α-KO的HCT116细胞系,可用于后续相关功能探索的实验材料.
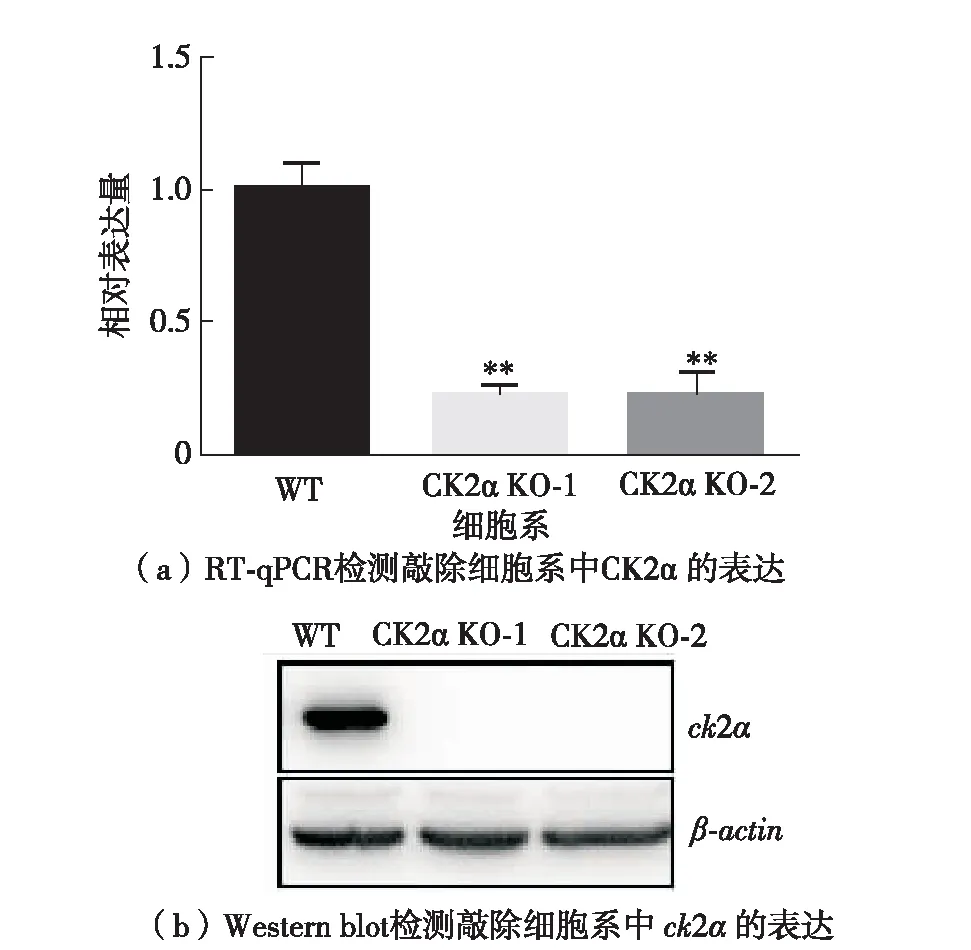
图2 ck2α敲除后mRNA及蛋白表达水平检测Fig.2 Expression of mRNA and protein level in ck2α knockout cells
2.3 ck2α敲除抑制细胞增殖速率和细胞迁移能力
为进一步研究ck2α的功能,首先检测了敲除细胞系中细胞增殖速率的改变.如图3(a),在ck2α敲除的两个细胞系中细胞增殖速率相对于野生型细胞明显减慢;同时通过细胞划痕实现检测HCT116细胞迁移能力的改变,如图3(b),经过12 h后可明显观察到CK2α-KO细胞系的迁移明显比野生型细胞减慢,结果提示,ck2α基因参与了肿瘤细胞增殖和迁移.其作用机制需要进一步探索.

图3 ck2α敲除抑制HCT116细胞增殖和细胞迁移能力Fig.3 Inhibition of HCT116 cell proliferation and cell migration in ck2α knockout cells
2.4 ck2α敲除通过影响衰老相关基因的表达抑制肿瘤细胞的增殖
ck2α敲除抑制细胞增殖速率和细胞迁移能力的研究结果表明,ck2α参与肿瘤细胞增殖和迁移的调控,那么其具体的分子机制如何?由于敲除ck2α后影响了细胞增殖,这是否会导致细胞提前进入老化状态呢?为此,进一步检测了衰老相关基因的表达量变化(图4).由图4可见,参与细胞周期调控的相关基因p53、p21、pml及cdc37在CK2α-KO的细胞系中都有明显的上调表达,并且在蛋白水平也表现为类似的趋势.结果提示,ck2α敲除以后影响了衰老相关基因的上调,从而减缓细胞增殖速率,因此,可能导致肿瘤细胞走向衰老.

图4 ck2α敲除增加衰老相关基因的表达量Fig.4 Senescence-related genes in HCT116 cells up-regulated by ck2α knockout
2.5 ck2α敲除通过影响衰老相关基因的上调表达从而促进肿瘤细胞的衰老
为进一步确定CK2α抑制细胞增殖的具体作用机制,首先检测敲除后是否可以加速细胞衰老.研究表明,细胞出现衰老时,会增加β-半乳糖苷酶的活性.通过SA-β-gal染色实验进一步检测敲除细胞中细胞衰老的β-半乳糖苷酶变化趋势.结果表明,敲除细胞中的衰老细胞比率与野生型细胞明显增加,如图5所示,细胞的蓝染比例明显高于野生型细胞,经过统计学分析由6.21%增加到23.62%.结果表明敲除细胞中,细胞衰老进程加快.

图5 ck2α敲除促进HCT116细胞的衰老比例增加Fig.5 Cell senescence accelerated in ck2α knockout HCT116 cells
3 讨 论
研究表明,在癌症中有许多癌基因和肿瘤抑制基因发生了遗传学和表观遗传学改变,对这些基因修饰进行有效的编辑,改变其遗传特性对模拟疾病发生发展机制和研究基因功能有重要作用[15].在原核生物中发现的CRISPR-Cas9系统打破了传统基因工程技术的种种限制,已经在多种物种的细胞和组织水平实现了有效的基因敲除和基因修饰,将这一技术用于人类疾病尤其是肿瘤的模型中取得了重要的进展.从而为广大科学工作者提供了宝贵的借鉴[16].利用CRISPR-Cas9技术操纵正常和癌症基因组的各种方法已经建立,比如快对肿瘤抑制基因进行定点修饰、快速建立癌症研究模型、对模式生物体细胞基因组进行有效的编辑等[17-18].用这一技术全面分析各类癌症患者,有望建立个性化的细胞模型和动物实验体系,从而为深入研究疾病的发生发展机制提供更好的动物和细胞模型[19].另外,这种方法可以快速地在分子水平上对细胞进行基因敲除和删减,从而考察其分子表型,有利于快速锁定基因的生物学功能,帮助人们找到有效的应对措施[20-21].
目前CRISPR-Cas9技术已广泛用于细胞和动物水平的基因敲除,对疾病的分子机制的研究至关重要,更有利于未来开发各类疾病的基因治疗,对人类的健康功不可没.已有研究证实,这一技术可以在小鼠模型的肝脏中永久性矫正致病突变基因,成功缓解小鼠的患病症状.提示了CRISPR-Cas9也有可能永久矫正肿瘤患者中相关基因的突变.CRISPR-Cas9还可用于免疫细胞治疗,为肿瘤免疫学提供更好的肿瘤免疫疗法[22-23].由此可见,将CRISPR-Cas9技术应用到肿瘤生物学从基础研究、转化医学到临床治疗的各个方面,会给人类的生命健康带来前所未有的机遇,有助于攻克恶性肿瘤这种摧毁性的疾病.
在分子机制研究方面,SiCK2α后不会影响β-catenin的蛋白水平,但是转录因子Snail、Smad2和Smad3的表达降低,E-cadherin表达量增加,从而抑制上皮间质转化过程[13].在HCT116细胞中敲除ck2α明显降低细胞增殖,这与研究报道一致,那么利用CRISPR-Cas9的技术体系构建敲除细胞可快速确定基因功能,相对于传统的SiRNA更为准确可信.研究结果表明,在ck2α敲除细胞系中,观察到与细胞增殖、衰老相关的PML、P53和P21的表达上调,已有研究表明,CK2α通过磷酸化信号转导调节PML蛋白的泛素化降解[24],这种磷酸化是否影响结肠癌中细胞的增殖和迁移过程目前还不清楚,可以利用建立的敲除细胞系进行深入的研究.已有研究表明,P53通路参与多种细胞信号转导过程,比如细胞增殖分化、自噬、凋亡及细胞衰老等[25-26].在ck2α敲除中,P53的上调是直接作用还是通过其他分子介导的间接作用调控结肠癌细胞的增殖和迁移呢?因此,利用CRISPR-Cas9的基因编辑技术对下游分子机制的研究具有重要意义,有助于找到CK2调节相关癌症的下游关键信号分子,为临床治疗肿瘤的药物靶点提供更多的理论支持.
结 语
利用CRISPR-Cas9基因编辑技术可有效地对目的基因进行修饰,在肿瘤的研究中抑制某些促癌基因的表达可成为癌症治疗和其他疾病治疗的药物靶点.本研究发现,靶向敲除蛋白激酶CK2α后能够显著降低细胞的增殖速率和迁移能力,敲除细胞可加速细胞走向衰老的进程.本研究为通过加速细胞衰老而抑制肿瘤细胞增殖提供了新视角,未来可通过深入的机制研究为治疗肿瘤寻找新的作用靶点,从而为临床上治疗癌症提供新的治疗方案.
致谢:衷心感谢深圳大学王子梅教授的悉心指导!
引文:敖 英,张 杰,李 枫,等.靶向敲除ck2α基因及其功能分析[J]. 深圳大学学报理工版,2018,35(1):1-7.
/
[1] WIEDENHEFT B, STERNBERG S H, DOUDNA J A.RNA-guided genetic silencing systems in bacteria and archaea[J].Nature,2012,482(7385):331-338.
[2] GOPHNA U, BRODT A.CRISPR/Cas systems in archaea:What array spacers can teach us about parasitism and gene exchange in the 3rd domain of life[J]. Mobile Genetic Elements,2012,2(1):63-64.
[3] LO T W, PICKLE C S, LIN S,et al. Precise and heritable genome editing in evolutionarily diverse nematodes using TALENs and CRISPR-Cas9 to engineer insertions and deletions[J].Genetics,2013,195(2):331-348.
[4] CHAKRABORTY S, JI H, KABADI A M,et al.A CRISPR-Cas9-based system for reprogramming cell lineage specification[J]. Stem Cell Reports,2014,3(6):940-947.
[5] VAN DER OOST J, WESTRA E R, JACKSON R N,et al.Unravelling the structural and mechanistic basis of CRISPR-Cas systems[J]. Nature Reviews Microbiology,2014,12(7):479-492.
[6] DELTCHEVA E, CHYLINSKI K, SHARMA C M, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III[J].Nature,2011,471(7340):602.
[7] JINEK M, CHYLINSKI K, FONFARA I,et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science,2012,337(696):816-821.
[8] MALI P, YANG Luhan, ESVELT K M, et al.RNA-Guided human genome engineering via Cas9[J].Science,2013,339(6121):823-826.
[9] CRADICK T J, FINE E J, ANTICO C J,et al.CRISPR-Cas9 systems targeting β-globin and CCR5 genes have substantial off-target activity[J].Nucleic Acids Research,2013,41(20):9584-9592.
[10] MONTENARH M. Protein kinase CK2 and angiogenesis[J].Advances in Clinical and Experimental Medicine : Official Organ Wroclaw Medical University,2014,23(2):153-158.
[11] BATTISTUTTA R, LOLLI G. Structural and functional determinants of protein kinase CK2α: facts and open questions[J].Molecular and Cellular Biochemistry,2011,356(1/2):67-73.
[12] AMPOFO E, RUDZITIS-AUTH J, DAHMKE I N,et al. Inhibition of protein kinase CK2 suppresses tumor necrosis factor(TNF)-α-induced leukocyte-endothelial cell interaction[J]. Biochimica et Biophysica Acta,2015,1852(10):2123-2136.
[13] ZOU Jinjin, LUO Hesan, ZENG Qin,et al. Protein kinase CK2α is overexpressed in colorectal cancer and modulates cell proliferation and invasion via regulating EMT-related genes[J].Journal of Translational Medicine,2011,9:97.
[14] SUN Yaohui, SUN Yuxin, ZHU Kan,et al. An experimental model for simultaneous study of migration of cell fragments, single cells, and cell sheets[J].Methods in Molecular Biology,2016,1407:251-272.
[15] KASAP C, ELEMENTO O, KAPOOR T M. Drug target Seq R: a genomics- and CRISPR-Cas9-based method to analyze drug targets[J].Nature Chemical Biology,2014,10(8):626-628.
[16] MADDALO D, MANCHADO E, CONCEPCION C P,et al. In vivo engineering of oncogenic chromosomal rearrangements with the CRISPR-Cas9 system[J].Nature,2014,516(7531):423-427.
[17] GROBARCZYK B, FRANCO B, HANON K, et al. Generation of isogenic human iPS cell line precisely corrected by genome editing using the CRISPR-Cas9 system[J].Stem Cell Reviews,2015,11(5):774-787.
[18] KIM D, BAE Sangsu, PARK J, et al. Digenome-seq: genome-wide profiling of CRISPR-Cas9 off-target effects in human cells[J].Nature Methods,2015,12(3):237-243.
[19] VAN T C, WEBER T, WEFERS B,et al. Increasing the efficiency of homology-directed repair for CRISPR-Cas9-induced precise gene editing in mammalian cells[J].Nature Biotechnology,2015,33(5):543-548.
[20] SHALEM O, SANJANA N E, HARTENIAN E, et al. Genome-Scale CRISPR-Cas9 knockout screening in human cells[J].Science,2014,343(6166):84-87.
[21] ZHANG Feng, WEN Yan, GUO Xiong.CRISPR-Cas9 for genome editing: progress, implications and challenges[J].Human Molecular Genetics,2014,23(R1):R40-R46.
[22] ZHOU Yuexin, ZHU Shiyou, CAI Changzu, et al. High-throughput screening of a CRISPR-Cas9 library for functional genomics in human cells[J].Nature,2014,509(751):487.
[23] CHARI R, MALI P, MOOSBURNER M,et al. Unraveling CRISPR-Cas9 genome engineering parameters via a library-on-library approach[J].Nature Methods,2015,12(9):823-826.
[24] SCAGLIONI P P, YUNG T M, CAI Lufan, et al. A CK2-dependent mechanism for degradation of the PML tumor suppressor[J].Cell,2006,126(2):269-283.
[25] PALAZZO E, KELLETT M, CATAISSON C, et al. The homeoprotein DLX3 and tumor suppressor p53 co-regulate cell cycle progression and squamous tumor growth[J].Oncogene,2016,35(24):3114-3124.
[26] ZHANG Guoxin, XIE Yinyin, ZHOU Ying, et al.p53 pathway is involved in cell competition during mouse embryogenesis[J].Proceedings of the National Academy of Sciences of the United States of America,2017,114(3):498-503.
