青藏高原东南部山丹nrDNA ITS和cpDNA petB/petD序列的分子进化特点
2018-01-09蒋福娟唐道城巨秀婷孙静清
蒋福娟 唐 楠 唐道城 巨秀婷 孙静清
(1.青海大学高原花卉研究中心/青海省园林植物与观赏园艺重点实验室,西宁 810016; 2.青海省高原作物种质资源创新与利用国家重点实验室培育基地,西宁 810016; 3.大通县林业局,大通 810100; 4.西宁市人民公园,西宁 810008)
青藏高原东南部山丹nrDNAITS和cpDNApetB/petD序列的分子进化特点
蒋福娟1,3唐 楠1,2*唐道城1巨秀婷1孙静清4
(1.青海大学高原花卉研究中心/青海省园林植物与观赏园艺重点实验室,西宁 810016;2.青海省高原作物种质资源创新与利用国家重点实验室培育基地,西宁 810016;3.大通县林业局,大通 810100;4.西宁市人民公园,西宁 810008)
采用改良CTAB法提取青藏高原东南部山丹25个居群所有个体的基因组DNA,选取核基因ITS和叶绿体petB/petD区域进行PCR扩增、纯化和测序。对所有序列对位排列,其中ITS序列总长696 bp,变异位点有4处,共产生7种单倍型,变异位点百分率为0.72%,(G+C)含量60.4%;petB/petD序列总长616 bp,仅1处变异位点和2种单倍型,变异位点百分率为0.16%,(G+C)含量34.6%。表明山丹中,petB/petD区域较ITS序列保守,变异速率较慢。对ITS序列单倍型进行失配分布和中性检验分析发现,山丹现有分布范围可能经历了近期居群小范围扩张,AMOVA分析发现山丹居群的遗传变异主要存在于居群内,NST>GST(P>0.01),表明山丹的遗传变异有着不显著的谱系地理结构。因此,山丹ITS序列适合该种的谱系地理学研究。
青藏高原东南部;山丹;ITS;petB/petD;分子进化
山丹(LiliumpumilumRedouté)隶属百合科(Liliaceae)百合属(Lilium)卷瓣组,据Flora of China记载,它广泛分布于中国(尤为西北和东北)、朝鲜、蒙古、俄罗斯(西伯利亚中部和东部)等地。青藏高原东南部的百合属植物主要有山丹、卷丹(Liliumlancifolium)和兰州百合(Liliumdavidii)[1],其中山丹是该地区的唯一野生种,多数分布在海拔1 460.0~3 918.0 m的干旱阳坡草地、林缘及林间沟谷[2]。抗寒,抗旱,喜凉爽,土壤要求疏松肥沃及排水良好。野生植株高5.0~50.0 cm,在适宜条件下可达80.0 cm以上。鳞茎生长较慢,鳞片具有较高的营养价值。叶条形,花单生或数朵排成总状花序,鲜红色,具有较高观赏性。目前山丹的研究多集中在形态解剖学[3~4]、生理生化[5]、资源利用与种质创新[6]、组织培养[7]及种间亲缘关系和遗传多样性[8]等方面,本实验室研究人员Nan Tang等人[2]已利用多种方法对山丹进行了遗传多样性的分析,在前人实验的基础上,对其进行ITS序列和cpDNA序列的研究尚属首次。
随着分子生物学研究的不断深入,用于系统进化研究的分子标记已由过去的RFLP、RAPD、ISSR等过渡到nrDNA(核糖体DNA)、cpDNA(叶绿体DNA)和mtDNA(线粒体DNA)的基因片段。其中应用最广泛的是核基因组nrDNA ITS序列,ITS由18S,5.8S和26S分成两个区段,即ITS1和ITS2。ITS基因保守,且相对进化速度较快[9]。ITS区的可变性为物种鉴定提供了丰富的变异位点,比较适合于研究被子植物种间和种内关系[10]。与nrDNA相比,cpDNA分子量小、多拷贝且结构简单,有利于构建物理图谱、分离与鉴定特定基因和测序。常用的cpDNA片段有编码区和非编码区,两者进化速率不同,故解决的问题也不尽相同。许多物种的研究表明cpDNA片段能很好地表现种间和种内的变异[11],可为系统发育的研究提供具有参考价值的变异位点[12]。
本文以青藏高原东南部山丹25个居群217个个体为研究对象,将ITS和petB/petD两者的序列进行比对分析,从而研究该物种ITS和cpDNA序列的分子特点和遗传多样性,并从这两个序列初步判定该种在青藏高原东南部形成现有分布格局的成因。
1 材料与方法
1.1 实验材料
本试验所用山丹有25个居群217个个体,分布于青海东南部、甘肃西南部、陕西太白山地区(北纬33°59′30″~37°16′54.8″,东经100°56′24.9″~107°12′53″,海拔1 460~3 918 m)。采样时各居群间隔大于10.0 km,以自然山沟或山峰、林区等相对隔离,居群内各单株间距离大于20.0 m,海拔高差5.0 m以上,以尽可能避免居群间采集的样本属于相同或相近的生态区域。每个居群随机采集了3~12个个体,采集的叶片先用ddH2O冲洗,再用70%的乙醇棉球擦洗干净,贴好标签后放入硅胶中快速干燥后带回,于-80℃超低温冰箱中备存,并在青海大学高原花卉研究中心建立山丹居群组培保存体系(表1)。
1.2 实验方法
1.2.1 基因组DNA的提取
使用CTAB改良法提取基因组DNA[13],为减少DNA氧化可加入少量PVP干粉,为提高DNA浓度和质量可加2%的β-巯基乙醇,提取完成后加2 μL RNA酶以消除RNA干扰。使用1.0%的琼脂糖凝胶电泳检测DNA质量,使用核酸蛋白检测仪检测纯度和浓度。
1.2.2ITS和cpDNApetB/petD序列的扩增和测序
采用通用引物ITS4(TCCTCCGCTTATTGATATGC)和ITS5(GGAAGTAAAAGTCGTAACAAGG)[14]以及petB(CTGCCGTATTTATGTTAATG)和petD(GTCTAGCCCCTGTTCTTCCT)[15]分别对山丹ITS和petB/petD序列进行扩增。扩增反应在Bio-Rad MyCycler PCR扩增仪上进行。扩增反应体系[16]为:在25 μL的总反应体系中,含Mg2+2.0 mmol·L-1、dNTPs 0.2 mmol·L-1、引物0.2 μmol·L-1、Taq酶0.25 U、50 ng模板DNA 2 μL、10×PCR Buffer 2.5 μL以及upH2O 15.4 μL。扩增反应程序为:94℃预变性5 min后,进入35个循环:包括94℃变性45 s,ITS-PCR 55℃退火45 s(cpDNA-PCR 53.1℃退火45 s),72℃延伸1 min。随后72℃终延伸10 min,最后-4℃存储备用。琼脂糖电泳检测中条带单一明亮的PCR产物送至生工生物工程(上海)股份有限公司,完成纯化和测序。对各个样品进行双向测序,并将第一次测序所得的有变异位点的样品进行三次测序,以保证变异位点的准确性。为使试验结果更加全面,特选用另外8条cpDNA片段进行PCR扩增与测序,以对照petB/petD序列之结果。

表1 山丹居群采集信息
1.2.3 数据处理
测得的序列用BioEdit和ClustalX软件进行比对[17],并参照峰图进行手工校正。使用DnaSP5.0[18]统计出其核苷酸G+C的含量、变异位点、核苷酸多样性指数和单倍型类型等,失配分析、Tajima’s D和Fu’s Fs分析也在此软件中完成,以检验山丹居群间是否经历了近期居群范围扩张[19~20]。用软件MEGA6.0分析核苷酸组成,并用邻接(Neighbor-Joining,NJ)法以1 000次自展检验构建NJ树[21]。利用软件Arlequin3.5.2.2进行AMOVA(Analysis of Molecular Variance)分析,计算居群内、居群间的变异方差分布,并分析总样本的变异固定于居群间或居群内[22]。使用软件TCS1.21[23]构建ITS各单倍型之间的单倍型中央连接网络图。用程序Permut & CpSSR2.0计算居群间和居群内的平均遗传多样性(HS和HT),并计算居群遗传分化指数GST[24]和NST[25]。
2 结果与分析
2.1 ITS和cpDNA petB/petD目的片段的扩增
利用优化后的扩增体系对ITS和petB/petD片段进行扩增,得出单一、整齐、清晰且稳定的条带,电泳显示基本无非特异性扩增和引物二聚体(图1)。
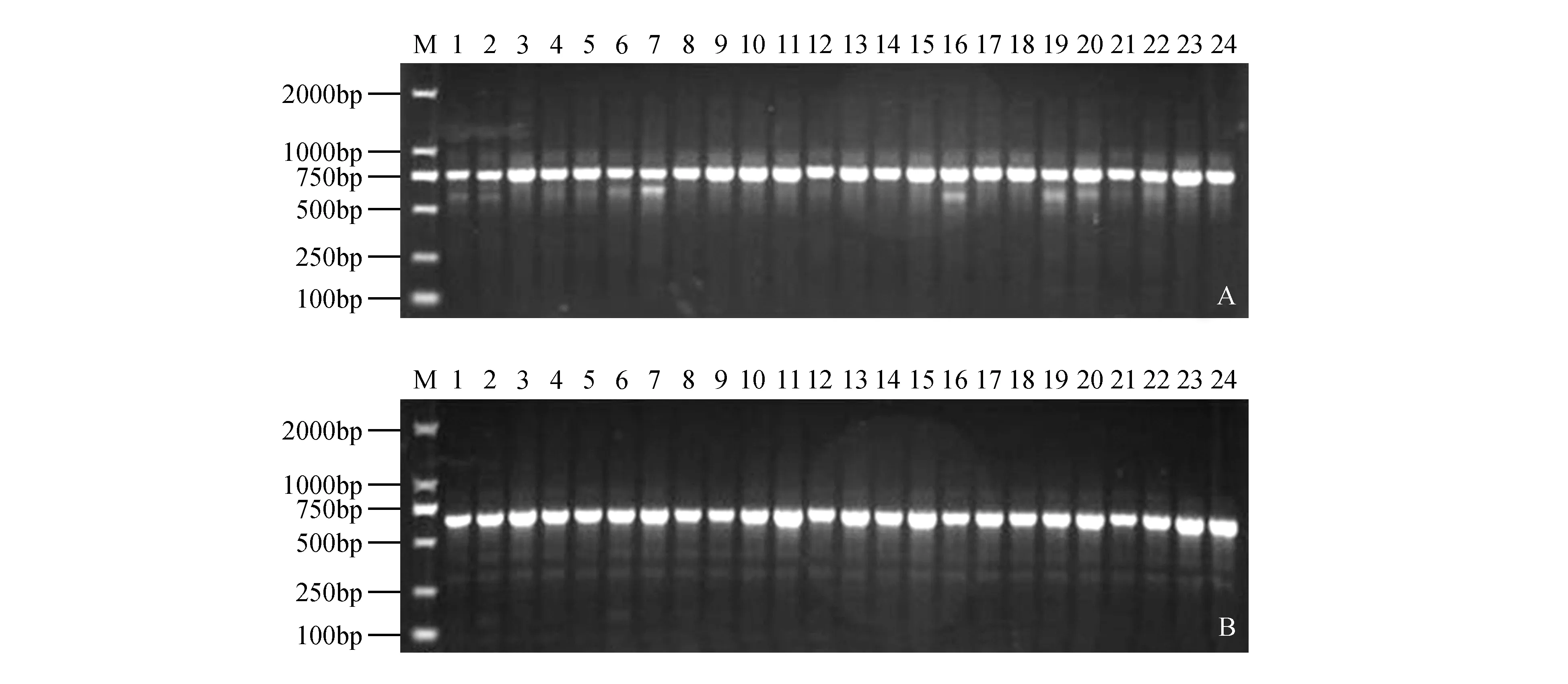
图1 nrDNA ITS区域(A)和cpDNA petB/petD区域(B)PCR扩增电泳结果Fig.1 Results of PCR amplification of ITS(A) and cpDNA petB/petD(B)
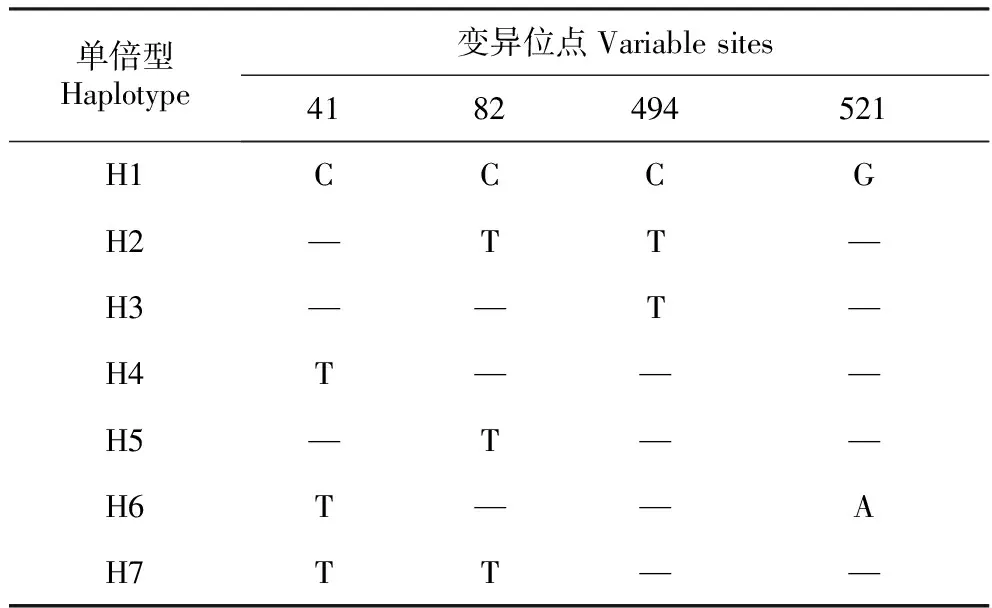
表2 山丹ITS 7种单倍型的变异位点
2.2 山丹各居群ITS和petB/petD序列分析
对所有个体的ITS和petB/petD区域进行双向测序,并利用软件BioEdit和ClustalX进行比对。经排序比对后得到ITS碱基恒定长度为696 bp,G+C含量是60.4%,变异位点百分率为0.72%,变异位点有4处,即在41、82和494处发生C-T转换,在521处发生G-T颠换(表2)。经比对ITS序列共拥有7种单倍型,其中H1为广布单倍型,分布于除SM居群外的其它24个居群中。25个居群中只有YQ、SP、SM 3个居群不存在居群内变异,其余22个居群都存在不同程度居群内个体间变异。
经AMOVA分析,居群内遗传变异占83.65%,居群间占16.35%(表3)。FST=0.163 50(P<0.001),假设研究的ITS4-ITS5序列变异处于漂变—迁移平衡(drift-migration equilibrium),则基于FST值估算的物种水平上居群间的平均基因流Nm=1.279 1,Nm>1,表明群体间的基因流的水平较高,群体间遗传分化较小[26]。居群内平均遗传多样性Hs(se)为0.430(0.048 4),居群间总平均遗传多样性HT(se)为0.518(0.048 8),总的居群遗传分化系数GST(se)值为0.170(0.079 5),NST(se)值为0.218(0.090 9),使用U-统计方法对ITS序列型的变异进行检验后发现NST>GST(P>0.01),表明山丹的遗传变异存在一定的谱系地理结构,但不显著。
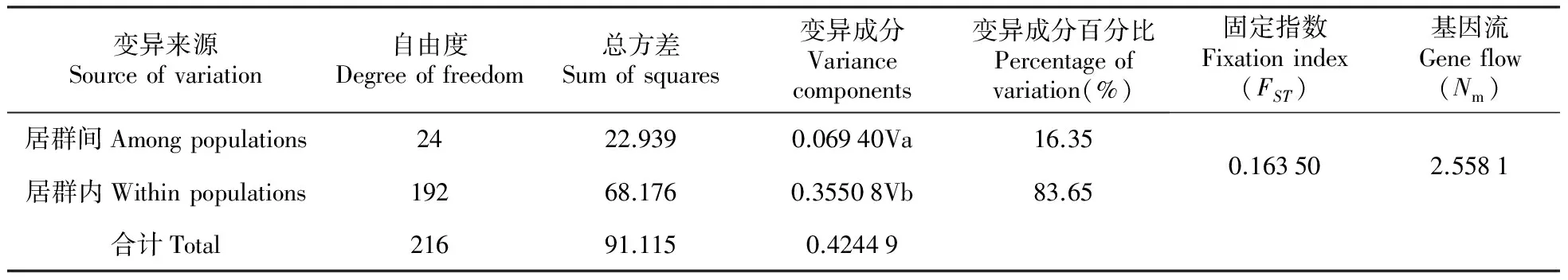
表3 基于ITS4-ITS5序列山丹25个居群的遗传变异分析
运用软件MEGA6.0对7种单倍型进行NJ树构建,并与TCS单倍型网络图进行对照分析。如图2所示,山丹单倍型分布呈典型的辐射状谱系结构,说明存在居群扩张。分布广且频率高的单倍型H1位于中央连接网状图的中心,表示H1可能是较为原始的单倍型,其余的稀有单倍型位于外部节点,表明可能是由原始祖先单倍型衍生而来[23]。
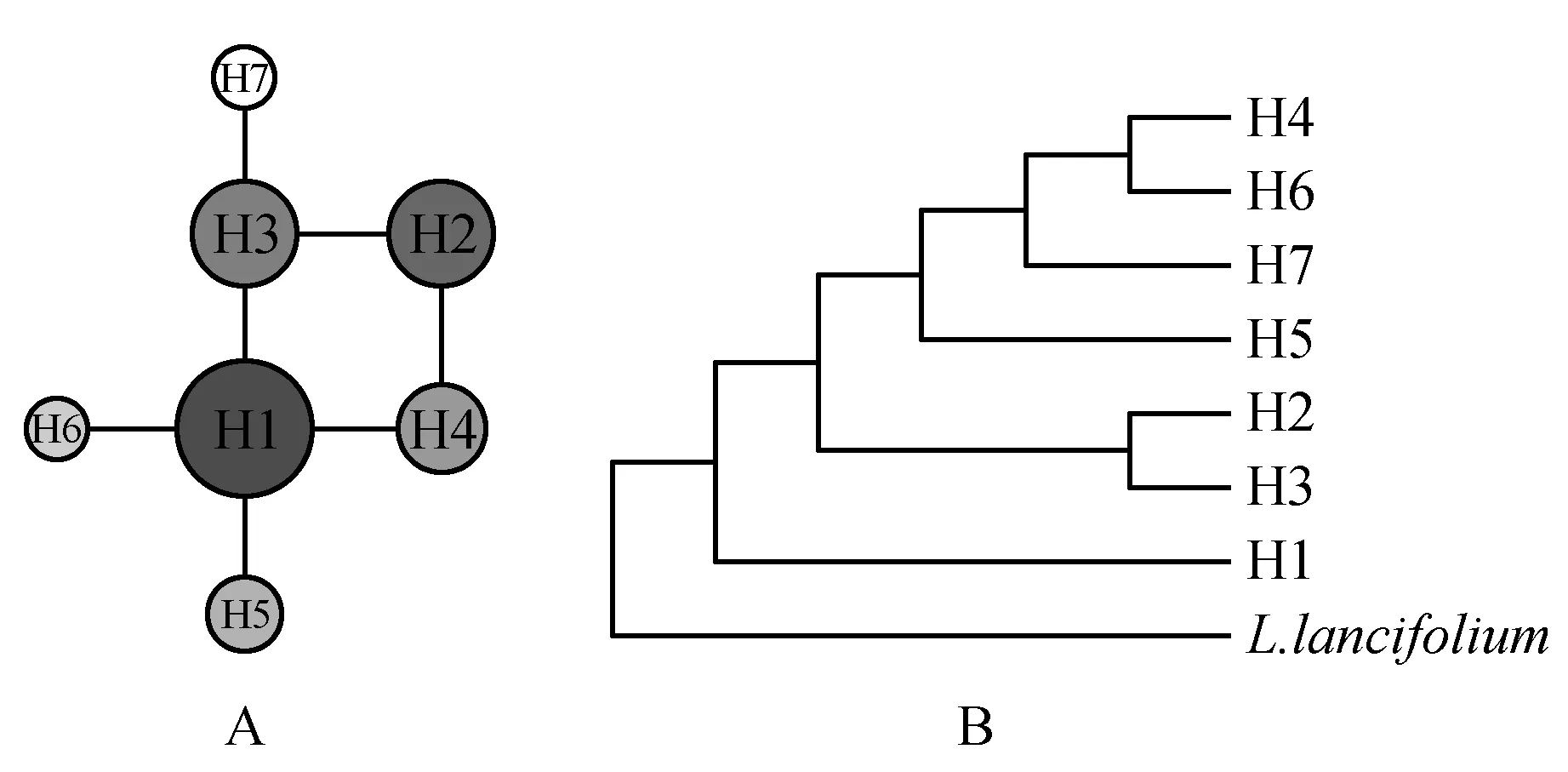
图2 基于ITS4-ITS5序列的山丹单倍型网络图 a.圆圈大小与单倍型频率正相关;b.NJ树Fig.2 Haplotype network of L.pumilum based on ITS4-ITS5 sequence a. Circle size is proportional to the relative frequency of the haplotypes; b. Neighbor-Joining tree
用DnaSP5.0软件对山丹ITS序列进行失配分析(MDA,Mismatch distribution analysis)和中性检验(Neutrality test),失配分析是以可视形式展现群体历史动态的一种方式。若种群经历了扩张,其失配分布曲线为单峰。失配分布图(图3)表明观测值与期望值都为单峰分布曲线,说明青藏高原东南部山丹居群经历了近期范围扩张[19]。中性检验得出较为保守的Tajima’s D检验值为0.468 74(P>0.10),Fu and Li’s D*检验值为0.877 92(P>0.10),Fu and Li’s F*检验值为0.880 31(P>0.10),Fu’s Fs值为-1.537。其中Fu’s Fs值为负值说明与失配分布结果一致,表明其存在居群扩张[27];而其它三个检验值虽为正值,但都不显著,并未背离中性平衡。综合结果表明青藏高原东南部山丹的分布在不背离中性平衡的条件下,可能经历了近期居群的小范围扩张。
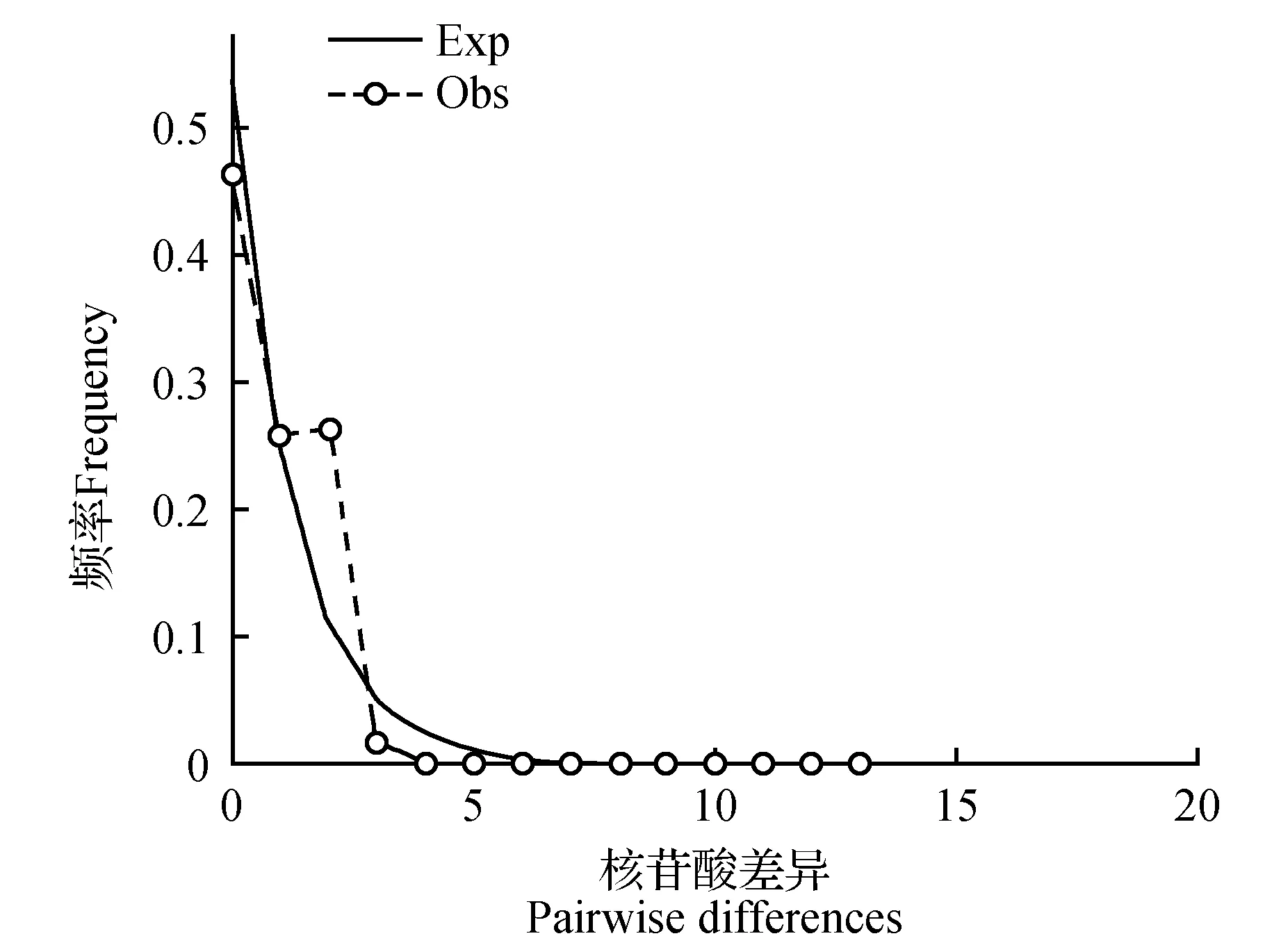
图3 山丹ITS序列数据的失配分布Fig.3 Mismatch distribution of ITS sequences of L.pumilum

表4 山丹ITS 和petB/petD序列的比较
经对位排列,得出petB/petD序列的恒定长度为616 bp,G+C含量为34.6%,这与cpDNA基因G+C含量低[9]的特点相吻合,变异位点百分率0.16%,仅在182 bp处有一个碱基置换(C-T)。合并相同序列后得到2个单倍型,其中T1(表1)分布于除JD居群外的其它24个居群中,说明T1是广布单倍型;T2为JD居群特有,XL居群中两种类型均有,表明XL存在居群内变异。同时,由表4可以看出山丹ITS序列的G+C含量、变异位点百分率和遗传多样性均大于petB/petD序列。后续研究中,针对其它8条cpDNA片段(psbA/trnH、rbcL 1F/rbcL724R、rpl20/rps12、trnQ(UUG)/rps16×1、ndhA×1/ndhA×2、rpL16F71/rpL16R1516、ndhJ/TabE、ndhG/ndhI)的测序结果,尚未提供有用的信息位点。
3 讨论
ITS和cpDNA是被子植物系统进化的主要研究方法。ITS之所以能解决属间及种间的系统发育问题,主要是因其长度的保守和序列的变异[28]。cpDNA在种水平上较为保守,进化速率仅为核基因的1/5[29]。cpDNA之所以成为研究植物种间系统发育的首选方法,是因为其分子量小、多拷贝、结构简单且单亲遗传的特点[29]。
本研究使用PCR产物直接测序法分析了山丹居群间ITS序列和petB/petD序列的碱基差异,初步确定了两套植物基因组的变异速率,即山丹ITS序列的变异速率大于petB/petD序列的变异速率,ITS序列的遗传多样性也大于petB/petD序列的遗传多样性,petB/petD序列较ITS序列保守,这与叶绿体基因较为保守的研究结果一致,但与段义忠等[9]对窄叶鲜卑花、杨路存等[30]对羌活的研究结果不一致,他们认为所研究的物种ITS较cpDNA序列保守,且变异速率较慢。说明不同物种petB/petD和ITS反应的结果不完全一致。
青藏高原东南部山丹ITS序列共产生7种单倍型,其中H1在24个居群中均出现,属于广布单倍型,证实了居群间的遗传保守性,同时25个居群中出现了7种单倍型也说明了青藏高原东南部地区的山丹居群有着丰富的遗传多样性。依据NJ树及TCS单倍型网络图看出H1可能是较为原始的单倍型,也进一步支持了H1所反应的居群间遗传保守性之观点。失配分布图为单峰,证实了青藏高原东南部山丹经历了扩张。由4个中性检验值和失配分布曲线图得知青藏高原东南部山丹在遗传保守的前体下存在近期居群小范围扩张的可能。虽然ITS序列能提供的变异位点较少,但借助其失配分析和中性检验大致可以判断出青藏高原东南部山丹分布的历史演变,因此ITS序列适用于山丹的居群进化历史研究。而对petB/petD序列及其它8条cpDNA片段的研究结果得出,cpDNA序列所能提供的信息位点太少,不能为该种群的进化分析提供有力依据,故不适用于该种的谱系地理学研究。
对ITS序列的AMOVA分析表明山丹居群遗传变异主要在居群内,而居群间具有较小的遗传变异,这说明青藏高原东南部地区的山丹居群间的基因交流比较频繁,这个结果与青藏高原许多植物的遗传多样性是一致的,即基因多样性较高而核苷酸多样性较低;但也与如西川红景天[31]、窄叶鲜卑花等[32]的研究结果存在一定的差异,西川红景天和窄叶鲜卑花表现出居群间变异大于居群内。同时山丹居群间的遗传分化指数NST>GST(P>0.01)差异不显著进一步说明该物种具有不显著的谱系地理结构,这与王昊[33]等人对西藏沙棘的的研究结果类似。一般而言,植物居群间分化程度较高的情况是一年生、异交或风媒传播的种子,而分化程度较低则是多年生、自交的种子[34]。山丹居群内遗传变异高于居群间,这与异交或风媒传播种子和鳞茎多年生繁殖的特性有关,百合的花粉粒径大经风传播受限[35],地理隔离导致居群间遗传多样性低而相对稳定,居群内无性繁殖受青藏高原气候环境复杂性的影响,导致遗传多样性较为丰富。要用山丹居群间和居群内的遗传多样性和遗传分化指数更准确描述分子进化特点,尚需继续扩大采样地范围,在偏离分布中心较远的区域寻找遗传多样性更丰富的地区。本研究仅为青藏高原地区植物区系及系统发育研究提供了又一个新的参考点,对丰富青藏高原地区物种种群历史动态和地理分布格局关系的认识提供了宝贵的理论解释。
1.刘尚武.青海植物志[M].青海:青海人民出版社,1999:280.
Liu S W.Flora of Qinghai[M].Qinghai :Qinghai People’s Press,1999:280.
2.Tang N,Mo G C,Van Tuyl J M,et al.Genetic diversity and structure ofLiliumpumilumDC. in southeast of Qinghai-Tibet plateau[J].Plant Systematics and Evolution,2014,300(6):1453-1464.
3.刘冬云,孟庆瑞,王倩.山丹不同居群花粉形态研究[J].植物遗传资源学报,2014,15(4):765-771.
Liu D Y,Meng Q R,Wang Q.Study on the pollen morphology of differentLiliumpumilumDC. populations[J].Journal of Plant Genetic Resources,2014,15(4):765-771.
4.刘冬云,张晓曼,李艳,等.不同居群山丹的核型分析[J].植物遗传资源学报,2015,16(1):199-204.
Liu D Y,Zhang X M,Li Y,et al.Karyotype analysis of the different population groups[J].Journal of Plant Genetic Resources,2015,16(1):199-204.
5.王仙芝.秦巴山区野生百合及栽培百合3种病毒病发生的研究[D].杨凌:西北农林科技大学,2008.
Wang X Z.Study on the occurency of three kinds of virus onLiliumfrom QinBa Mountains and Lily cultivars under the nursery field[D].Yangling:Northwest A&F University,2008.
6.刘晓丽,李文媛,贾桂霞.11种野生百合杂交亲和性的初步研究[J].北京林业大学学报,2012,34(3):85-90.
Liu X L,Li W Y,Jia G X.Cross compatibility of 11 species of wild lilies[J].Journal of Beijing Forestry University,2012,34(3):85-90.
7.葛蓓孛,杨青杰,吴萍,等.细叶百合组织培养植株再生[J].东北林业大学学报,2010,38(5):54-56.
Ge B B,Yang Q J,Wu P,et al.Tissue culture and plant regeneration ofLiliumpumilum[J].Journal of Northeast Forestry University,2010,38(5):54-56.
8.钟海丰.秦巴山区4个百合野生种种内遗传多样性的RAPD分析[D].杨凌:西北农林科技大学,2008.
Zhong H F.Study on genetic diversity of fourLiliumwild species from Qinba mountain by using RAPD[D].Yangling:Northwest A&F University,2008.
9.段义忠,张得钧,高庆波,等.窄叶鲜卑花(Sibiraeaangustata) nrDNA ITS和cpDNA trnL-F序列分子进化特点的分析[J].植物研究,2010,30(2):146-151.
Duan Y Z,Zhang D J,Gao Q B,et al.Characteristic of molecular evolution ofSibiraeaangustataBased on nrDNA ITS and cpDNAtrnL-F sequence analysis[J].Bulletin of Botanical Research,2010,30(2):146-151.
10.Baldwin B G,Sanderson M J,Porter J M,et al.The ITS region of nuclear ribosomal DNA:a valuable source of evidence on angiosperm phylogeny[J].Annals of the Missouri Botanical Garden,1995,82(2):247-277.
11.Taberlet P,Gielly L,Pautou G,et al.Universal primers for amplification of three non-coding regions of chloroplast DNA[J].Plant Molecular Biology,1991,17(5):1105-1109.
12.Small R L,Lickey E B,Shaw J,et al.Amplification of noncoding chloroplast DNA for phylogenetic studies in lycophytes and monilophytes with a comparative example of relative phylogenetic utility from Ophioglossaceae[J].Molecular Phylogenetics and Evolution,2005,36(3):509-522.
13.陈名红,李玉,刘多,等.利用改良CTAB法提取卷丹百合鳞叶基因组DNA[J].江苏农业科学,2013,41(3):27-29.
Chen M H,Li Y,Liu D,et al.Extraction ofLiliumlancifoliumscale genome DNA using modified CTAB method[J].Jiangsu Agricultural Sciences,2013,41(3):27-29.
14.White T J,Bruns T,Lee S,et al.Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics[M].San Diego,USA:Academic Press 1990:315-322.
15.Shaw J,Lickey E B,Schilling E E,et al.Comparison of whole chloroplast genome sequences to choose noncoding regions for phylogenetic studies in angiosperms:the tortoise and the hare Ⅲ[J].American Journal of Botany,2007,94(3):275-288.
16.蒋福娟,唐道城,唐楠.山丹百合cpDNA-PCR反应体系建立与优化[J].分子植物育种,2017,15(1):213-222.
Jiang F J,Tang D C,Tang N.Construction and optimization of cpDNA-PCR system forLiliumpumilumDC[J].Molecular Plant Breeding,2017,15(1):213-222.
17.Thompson J D,Gibson T J,Plewniak F,et al.The ClustalX windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Research,1997,25(4):4876-4882.
18.Rozas J,Sanchez-Delbarrio J C,Messeguer X,et al.DnaSP,DNA polymorphism analyses by the coalescent and other methods[J].Bioinformatics,2003,19(18):2496-2497.
19.Tajima F.Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J].Genetics,1989,123(3):585-595.
20.Fu Y X,Li W H.Statistical tests of neutrality of mutations[J].Genetics,1993,133(3):693-709.
21.Tamura K,Stecher G,Peterson D,et al.MEGA6:molecular evolutionary genetics analysis version 6.0[J].Molecular Biology and Evolution,2013,30(12):2725-2729.
22.Excoffier L,Lischer H E L.Arlequin suite ver 3.5:A new series of programs to perform population genetics analyses under Linux and Windows[J].Molecular Ecology Resources,2010,10(3):564-567.
23.Clement M,Posada D,Crandall K A.TCS:a computer program to estimate gene genealogies[J].Molecular Ecology,2000,9(10):1657-1659.
24.Bandelt H J,Forster P,Röhl A.Median-joining networks for inferring intraspecific phylogenies[J].Molecular Biology Evolution,1999,16(1):37-48.
25.Forster P,Bandelt H J,Röhl A,et al.Network 4.2.0.1 Software available free at www.fluxus-engineering.com[M].Fluxus Technology Ltda,2004.
26.Millar C L,Libby W J.Strategies for conserving clinal,ecotypic,and disjunct population diversity in widespread species[M].//Fald D A,Holsinger K E.Genetics and Conservation of Rare Plants.New York:Oxford University Press,1991:149-170.
27.Fu Y X,Li W H.Statistical tests of neutrality of mutations[J].Genetics,1993,133(3):693-709.
28.李恩香.黄精叶钩吻属的亲缘地理学及其近缘类群的系统进化研究[D].杭州:浙江大学,2006.
Li E X.Studies on phylogeography ofCroomiaand phylogeny ofCroomiaand its allies[D].Hangzhou:Zhejiang University,2006.
29.龚黄莎.基于ITS和cpDNA江西蕙兰野生居群遗传结构研究[D].南昌:南昌大学,2013.
Gong H S.Population genetic structure ofCymbidiumfaberiRolfe from Jiangxi based on Internal Transcribed Spacers(ITS) and cpDNA[D].Nanchang:Nanchang University,2013.
30.杨路存,刘何春,周学丽,等.羌活(Notopterygiumincisum) nrDNA ITS和cpDNA rpl20-rps12序列分子进化特点的分析[J].植物研究,2016,36(2):291-296.
Yang L C,Liu H C,Zhou X L,et al.Characteristic of molecular evolution ofNotopterygiumincisumbased on nrDNA ITS and cpDNA rpl20-rps12 sequence analysis[J].Bulletin of Botanical Research,2016,36(2):291-296.
31.Gao Q B,Zhang D J,Duan Y Z,et al.Intraspecific divergences ofRhodiolaalsia(Crassulaceae) based on plastid DNA and internal transcribed spacer fragments[J].Botanical Journal of the Linnean Society,2012,168(2):204-215.
32.Duan Y Z,Gao Q B,Zhang F Q,et al.Phylogeographic analysis of the endemic speciesSibiraeaangustatareveals a marginal refugium in the Qinghai-Tibet Plateau[J].Nordic Journal of Botany,2011,29(5):615-624.
33.王昊.西藏沙棘的谱系地理学研究[D].上海:复旦大学,2011.
Wang H.Phylogeography ofHippophaetibetanaSchlecht[D].Shanghai:Fudan University,2011.
34.Nybom H.Comparison of different nuclear DNA markers for estimating intraspecific genetic diversity in plants[J].Molecular Ecology,2004,13(5):1143-1155.
35.Gu X,Zhang Y L,Niu L X.Pollen morphology observation of 15 wild lilies from four provinces in Western China[J].Acta Horticulturae Sinica,2013,40(7):1389-1398.
Natural Science Foundation of China 31660582(Genetic mapping of resistance gene for tulip breaking virus);“Thousand Talents Program”of Qinghai Province;CAS“Light of West China”Program
introduction:JINAG Fu-Juan(1986—),female,master degree students,assistant Engineer,mainly engaged in Landscape Plant Genetic Breeding Research work.
date:2017-05-24
CharacteristicofMolecularEvolutionforLiliumpumilumRedoutéonSoutheastoftheQinghai-TibetanPlateauusingnrDNAITSandcpDNApetB/petDSequenceAnalysis
JIANG Fu-Juan1,3TANG Nan1,2*TANG Dao-Cheng1JU Xiu-Ting1SUN Jing-Qing4
(1.Plateau Flower Research Center of Qinghai University/The key laboratory of landscape plants of Qinghai Province,Xining 810016;2.State Key Laboratory Breeding Base-Key Laboratory of Qinghai Province for Plateau Crop Germplasm Innovation and Utilization,Xining 810016;3.Datong County Forestry Bureau,Datong 810100;4.Xining People’s Park,Xining 810008)
In this study, total DNA was extracted fromLiliumpumilumon southeast of the Qinghai-Tibetan Plateau using modified CTAB method. Using genome DNA as template, nrDNA ITS and cpDNA petB/petD regions were amplified, purified and sequenced. By sequence alignment, it was known that length of nrDNA ITS sequence was 696 bp, of which 4 variable sites with a percentage of 0.72% were found., The G+C content of the ITS sequence was 60.4% and 7 haplotypes were produced. The length of cpDNA petB/petD sequence was 616 bp, of which 1 was variable sites with a percentage of 0.16% was found. The G+C content of the sequence was 34.6% and 2 haplotypes were found. The petB/petD region ofL.pumilumwas more conserved than ITS sequences and evolved more slowly. The present distribution range ofL.pumilumhas probably experienced range expansion by the haplotype analysis of mismatch distribution and neutral test, and AMOVA analysis showed that the genetic variation ofL.pumilummainly existed within populations. TheNST>GST(P>0.01)further showed that there was no significant phylogeographic structure of genetic variation inL.pumilum. Therefore, the nrDNA ITS sequence ofL.pumilumwas more suitable for phylogeographic study of this species.
southeastern of Qinghai-Tibetan Plateau;Liliumpumilum;ITS;petB/petD;molecular evolution
国家自然科学基金项目31660582(Genetic mapping of resistance gene for tulip breaking virus);青海省“高端创新人才千人计划”;中国科学院“西部之光”人才培养计划
蒋福娟(1986—),女,硕士研究生,助理工程师,主要从事园林植物遗传育种研究工作。
* 通信作者:E-mail:natasha_tn@hotmail.com
2017-05-24
* Corresponding author:E-mail:natasha_tn@hotmail.com
Q949.71+8.23
A
10.7525/j.issn.1673-5102.2018.01.014
稿件格式要求
1.稿件可用中英文两种语言撰写。按以下顺序书写:题目(一般不超过20字)、作者姓名(署名不宜超过6位)、作者单位(附所在城市和邮编)、摘要、关键词(3~8个)、中图分类号、文献标识码。英文题目、作者姓名(汉语拼音)、作者单位(英文)、英文摘要、关键词、正文、致谢(可无)、参考文献、图版说明。中英文摘要应详细概括目的、方法、结果和主要结论。
2.来稿首页注脚应注明第一作者简介(包括出生年月、性别、职称、学位及主要从事研究方向);基金资助的研究项目名称和编号。
3.外文字母大小写必须分清,数字或符号的斜体、上下标必须标明。凡文中首次出现的植物属、种须附拉丁文学名(一定用斜体),并核对无误。量和单位按我国法定计量单位以及国际标准中规定书写。外文学术名词术语的译名用通用译名,在文中第一次出现时应注明全称。文中的表格与插图中的所有文字均需具中、英文两种文字,表格用三线表形式。插图须在文中相应处直接给出,并在图下方给出中、英文图题。插图须用硫酸纸、高级白纸黑墨清绘或用600dpi分辨率电脑打印(插图尽可能大些尺寸),插图线条务求准确光洁。插图原件应注明次号。图版照片要求排列整齐紧凑,照片应清晰、层次分明、黑白反差大。按20cm×16cm版心贴于白纸上。图与实物的比例用线段比例尺在图中标注,以便缩放自如。图题、坐标的标目名称、单位要给齐全。新植物种要给出硫酸纸绘制的模式图,标出比例线段。图的大小为:半栏图宽5~6cm,通栏图宽10~13cm。
4.本刊参考文献采用顺序编码制。请作者主要引用国内外同行发表的相关论文。未公开发表的资料请勿列入,但可以脚标形式标注,请参照本样刊。
《植物研究》编辑部
