短期盐胁迫下盐穗木的转录组分析
2018-01-09张丽丽张富春
张丽丽 张富春*
(1.新疆大学生命科学与技术学院,乌鲁木齐 830046; 2.新疆生物资源基因工程重点实验室,乌鲁木齐 830046)
短期盐胁迫下盐穗木的转录组分析
张丽丽1,2张富春1,2*
(1.新疆大学生命科学与技术学院,乌鲁木齐 830046;2.新疆生物资源基因工程重点实验室,乌鲁木齐 830046)
盐穗木(Halostachyscaspica)是荒漠盐碱地广泛分布的盐生植物,具有极强的耐盐性。为揭示盐胁迫下盐穗木基因组层面的基因表达变化特性,通过对300和500 mmol·L-1NaCl胁迫3 h的盐穗木同化枝进行了转录组测序。有效序列组装共得到153 298条平均长度为643 bp的unigenes,进行GO和KEGG功能聚类,分别获得47个GO功能小类和118个KEGG通路。差异表达基因分析显示,短期低盐(300 mmol·L-1)响应基因有4 432个,高盐(500 mmol·L-1)响应基因有2 580个,两个胁迫的共差异基因有1 245个,主要富集在细胞过程、代谢过程和响应刺激等类别中。从短期盐胁迫下盐穗木转录组筛选出渗透调节和活性氧清除的相关基因,大多为上调基因。说明盐穗木能够通过促进渗透调节和增强活性氧清除提高短期的盐胁迫适应能力。
盐穗木;盐胁迫;转录组;差异基因;渗透调节;活性氧
土壤盐渍化不仅是导致生态环境恶化的主要原因,同时也是世界范围内影响农作物生产的主要非生物胁迫因素之一[1]。高浓度的盐会对植物产生包括渗透胁迫、氧化胁迫和离子毒害等的次级伤害,严重地影响了农作物的产量和品质。新疆作为中国最大的盐碱土分布区域,土壤盐渍化面积高达11万km2,占新疆总面积的7%[2],但在新疆荒漠盐碱环境中却生长着种类繁多的盐生植物[3~5],因此,深入研究新疆荒漠盐碱地区生长的盐生植物耐盐的分子机制,筛选和挖掘耐盐功能基因,有助于利用基因工程技术改良植物的耐盐性,培育耐盐能力强的农作物新品种,成为开发盐碱地的重要技术手段。但由于对植物的耐盐分子机制的了解甚少,同时缺乏有效的耐盐基因,其直接影响了植物耐盐基因工程技术的应用和发展[6]。
RNA-seq技术的大规模应用,使得非模式生物在基因组层面基因表达变化的研究有了迅猛的发展。例如利用RNA-Seq技术对盐胁迫下日本结缕草(Zoysiajaponica)的转录组分析发现,大多数的差异表达基因主要富集在响应刺激、响应胁迫等GO功能分类中[7];对盐胁迫下碱蓬(Suaedaglauca)的转录组分析发现,信号转导、转运蛋白以及细胞壁和生长等功能聚类中的差异基因显著富集,这些基因参与了植物的盐胁迫响应[8]。对盐胁迫下盐穗木转录组的分析表明DNA损伤修复相关基因HcRev1、HcRev3基因受盐胁迫诱导表达,参与了DNA损伤修复[9]。
盐穗木(Halostachyscaspica)隶属藜科(Chenopodiaceae)盐穗木属(Halostachys),广泛分布在荒漠盐碱地区,绿色期长,生态价值重要。在长期高盐逆境中,盐穗木进化出抵御盐胁迫独特的抗逆特性。利用高通量测序技术,检测盐胁迫下盐穗木的转录组,是研究盐穗木响应盐胁迫差异基因表达的较好途径。利用RNA-Seq技术对短期(3 h)低盐(300 mmol·L-1)和高盐(500 mmol·L-1)胁迫下盐穗木的转录组进行测序,并对共差异基因进行GO注释分析和验证,为深入探讨盐穗木的耐盐分子机制和筛选相关耐盐功能基因奠定理论基础。
1 材料与方法
1.1 样本来源及处理
盐穗木种子采自野外盐碱地(新疆五家渠103团,东经87.31°北纬44.29°),在室温下,将种子种于蛭石和珍珠岩混合的培养基质中使其萌发,待盐穗木幼苗生长到5个月左右,分别以含有300 mmol·L-1NaCl和500 mmol·L-1NaCl的Hoagland’s营养液浇灌作为处理组,正常的Hoagland’s营养液浇灌作为对照组,处理3 h后,迅速将处理组和对照组植物的同化枝分别置于液氮中,再转移至-80℃保存备用。
1.2 cDNA文库的构建和Illumina HiSeq2000测序
盐穗木同化枝样品的后续处理包括总RNA的提取、纯化、质量分析和构建cDNA文库以及转录组测序工作由武汉生命之美科技有限公司完成,采用Illumina HiSeq2000测序平台进行测序。
1.3 转录组数据分析
每个处理组选取3个样本获得的有效序列(clean reads)用Trinity软件进行双端有方向的组装得到unigenes。将这些unigenes比对到拟南芥蛋白库上,所选参数E-value为1e-5。用Blast2GO软件对unigenes进行GO功能注释。Unigenes的表达量采用RPKM(Reads per kilo base per million mapped reads)的方法计算。本研究以0 mmol·L-1NaCl组的表达量作为对照组,利用edgeR软件进行差异表达基因分析,将变化倍数(fold change)≥2和P值(Pvalue)≤0.01作为差异基因的筛选标准。
1.4 实时荧光定量PCR(qRT-PCR)
为验证盐穗木转录组结果的准确性,挑选了5个基因,用Primer 5.0设计引物,检测高盐胁迫下qRT-PCR与转录组结果的一致性。将各样品3个生物学重复采用植物总RNA提取试剂盒(Omega公司)提取总RNA,用M-MLV反转录酶以Oligo(dT)(Takara公司)为引物合成第一条cDNA链,以cDNA链为模板,Actin为内参,具体操作步骤参照SYBR Green RT-PCR试剂盒(QIAGEN公司)说明书进行,使用7500 Real Time PCR系统(Applied Biosysteme公司)进行实时荧光定量检测。每个样品进行3个技术重复,采用2-ΔΔCt法进行相对表达量计算。
2 结果
2.1 转录组测序数据分析
使用HiSeq2000高通量测序平台对样本进行测序所得的数据为原始测序数据(raw reads)具体值见表1。原始测序序列去除接头序列,去除含有两个N的reads,以及去除低质量值的reads,得到有效序列(表1)。所有有效序列的百分数均大于92.99%。将3个样本测序得到的有效序列合并,用Trinity软件进行双端有方向组装共得到153 298条unigenes,平均长度为643 bp,N50为991 bp,最长的unigene为10 718 bp,最短的为201 bp。
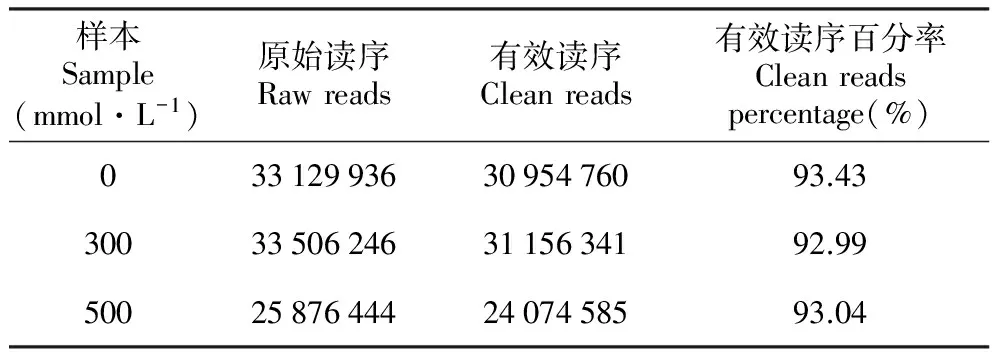
表1 盐穗木转录组测序结果
2.2 Unigene的GO功能注释
将组装所得到的153 298个unigenes进行比对分析,共有71 758个unigenes比对到Nr蛋白库,88 946个比对到拟南芥蛋白库。用blast2go对比对到拟南芥蛋白库的unigenes进行GO注释,有69 526个unigenes(78,17%)有GO注释,富集到3个大类47个小类(图1)。参与生物学过程(biological processes)的基因主要为盐胁迫应答(response to salt stress)、镉离子应答(response to cadmium ion)、损伤应答(response to wounding)、冷胁迫应答(response to cold)等相关基因,这些基因可能与盐穗木耐盐密切相关(图1A)。参与细胞组分(cellular components)的基因在细胞核类别(nucleus)所占比例最高,其次为质膜类别(plasma membrane)和叶绿体类别(chloroplast)(图1B)。在分子功能(molecular functions)分类中,蛋白质结合类别(protein binding)所占比例最高,其次为转录因子活性(transcription factor activity)、ATP结合(ATP binding)和DNA结合(DNA binding)类别(图1C)。

图1 盐穗木转录组数据GO分类注释图 A.生物学过程;B.细胞组分;C.分子功能Fig.1 Gene ontology classification annotation of transcriptome of H.caspica A.Biological process;B.Cellular component;C.Molecular function
2.3 KEGG通路注释结果
将unigenes比对到KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库并注释信息,得到了unigenes的Pathway注释。再结合KEGG数据库, 对盐穗木的unigenes可能参与或涉及的代谢途径进行了统计分析。结果表明9 128条unigenes富集到118个代谢通路中,其中富集前15的通路 (top 15 pathways)如图2所示,富集最多unigenes的通路为代谢通路(Metabolic pathways),共有2 339个unigenes表达,其次为次生代谢物的生物合成(Biosynthesis of secondary metabolites),富集到的unigenes有1 212个。
2.4 盐穗木响应盐胁迫的差异表达基因(DEG)分析
对样本cluster后的unigene进行差异表达基因分析。同化枝中300 mmol·L-1NaCl胁迫组与对照组相比差异基因共4 432个,上调表达的基因有1 343个,下调表达基因有3 089个;而500 mmol·L-1NaCl胁迫组与对照相比差异基因则有2 580个,上调基因有859个,下调表达基因有1 721个。在同化枝中两个盐胁迫处理组的共差异基因共1 245个(图3A),其中共上调基因307个(图3B),共下调基因934个(图3C)。短期盐胁迫下,无论是在高盐还是低盐浓度下,下调unigenes的数量都明显高于上调unigenes的数量。
为深入了解共差异基因的功能,用WEGO在线软件对共差异基因进行功能聚类分析,共差异基因可聚为3个大类40个小类,上调基因和下调基因均在细胞(cell)、细胞部分(cell part)、细胞器(organelle)、结合(binding)、催化(catalytic)、细胞过程(cellular process)、代谢过程(metabolic process)和响应刺激(response to stimulus)等类别中的基因所占比例较高(图5)。分析结果表明上调基因还在电子载体(electron carrier)、结构分子(structural molecule)等聚类中富集;而下调基因则在死亡(death)中富集。
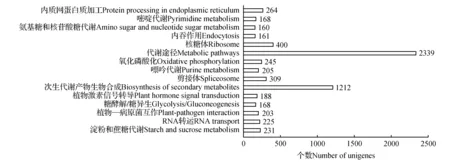
图2 盐穗木转录组的KEGG通路分析Fig.2 KEGG pathway analysis of transcriptome of H.caspica

图3 盐胁迫下盐穗木差异表达基因的维恩图Fig.3 Venn diagram of the differentially expressed unigenes in H.caspica at salt stress
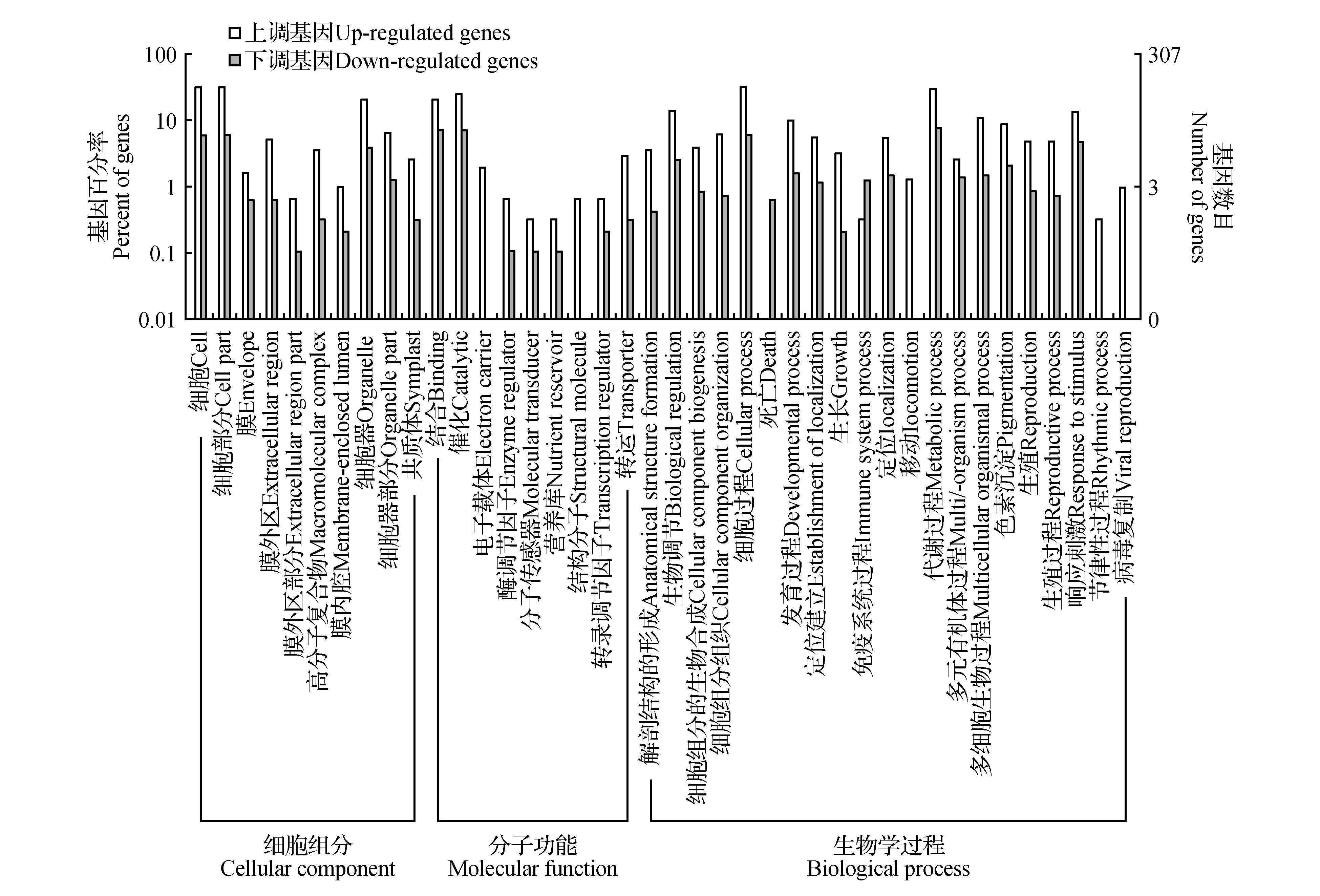
图4 共差异基因的GO功能聚类注释图Fig.4 Gene Ontology classification annotation of co-differentially expressed genes
2.4.1 盐胁迫下渗透调节相关基因的差异表达
在高盐胁迫的条件下,植物细胞中的离子平衡被打破,造成高渗损伤。为了缓解细胞受到的伤害,大多数植物会在体内合成和累积渗透保护剂或渗调剂的小分子化合物,提高细胞的抗逆能力。渗调剂主要有三类:糖类如海藻糖;甜菜碱及其衍生物;醇类及各种氨基酸,如脯氨酸[10~11]。本研究通过转录组分析筛选出海藻糖、甜菜碱以及脯氨酸合成途径中的关键酶基因。低盐胁迫下,筛选出3个上调的渗透调节相关基因,都与海藻糖合成相关,而高盐胁迫下,筛选出6个渗透调节相关基因,1个海藻糖合成关键酶基因和5个甜菜碱合成的关键酶基因(表2)。
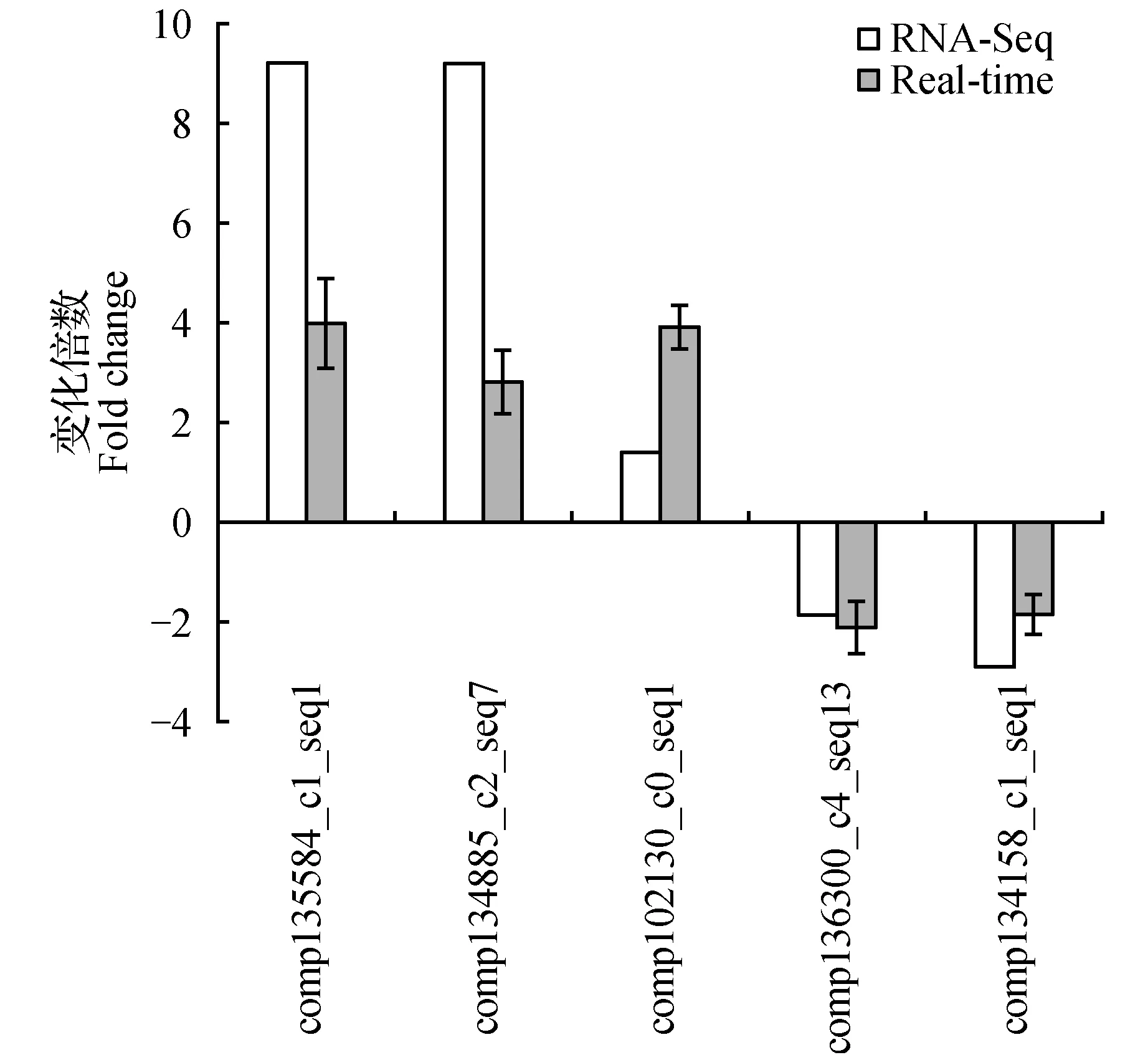
图5 DEGs的qRT-PCR验证Fig.5 Verification of DEGs using qRT-PCR
2.4.2 盐胁迫下活性氧清除相关基因的差异表达
盐对植物生长的影响,大多是由于氧自由基破坏了生物膜的结构,使其丧失功能,从而导致细胞死亡。植物能够通过酶促反应清除系统清除体内的活性氧。本研究选择部分活性氧清除的相关酶,包括超氧化物歧化酶(SOD)、过氧化物酶(POD)、硫氧还蛋白(Trx)、谷胱甘肽S-转移酶(GST)和过氧化氢酶(CAT)等进行分析。在300 mmol·L-1NaCl胁迫下,筛选了13个活性氧清除的相关酶基因,大部分为上调基因,而在500 mmol·L-1NaCl胁迫下,只筛选出7个活性氧清除相关基因,4个上调基因,3个下调基因(表2)。
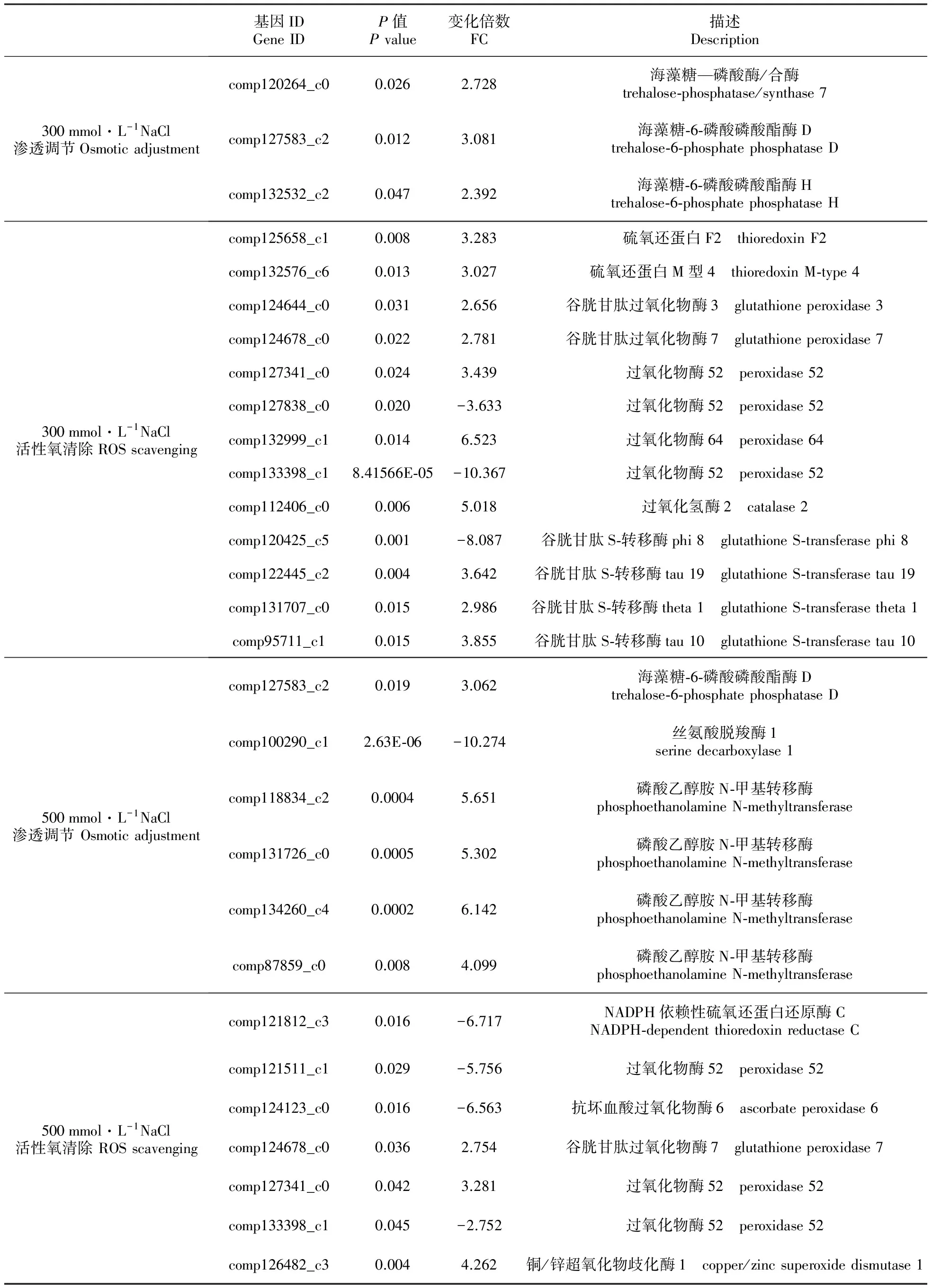
表2 短期盐胁迫响应的基因
2.5 实时荧光定量PCR验证高盐胁迫下基因的表达变化
随机选取5个筛选获得的基因 (高盐胁迫下3个上调基因和2个下调基因),利用实时荧光定量PCR进行对照组和高盐胁迫组基因的表达变化分析。结果显示,实时荧光定量PCR与转录组测序的结果在基因表达变幅上有一定的差异,但基因的表达趋势是一致的(图5),这说明转录组测序结果通过验证是可靠的。
3 讨论
生长在荒漠盐碱地区的盐穗木,由于生存逆境的长期适应,具有极强的耐盐能力。利用RNA-Seq技术,对300和500 mmol·L-1盐胁迫3 h的盐穗木以及0 mmol·L-1NaCl对照组的转录组进行测序,组装得到153 298条unigenes,被注释到47个条目中。KEGG分析显示,这些基因共参与118个通路,其中最显著富集的通路为代谢通路,主要包括脂质代谢、氨基酸代谢和多糖的生物合成和代谢等。脂质和蛋白是植物细胞膜的主要组分,在植物细胞对外界环境胁迫应答中起到主要的防御作用。不同的胁迫因素会扰乱细胞膜上脂质和蛋白的共粘连而影响脂质的代谢。研究表明欧洲油菜中脂质含量会随着NaCl水平的升高而降低[12]。增加土壤的盐浓度会严重影响脂质的生物合成,低不饱和脂肪的程度限制了细胞膜的流动性,从而影响了Na+和Cl-的渗透性[13~14]。而多糖作为另外一种重要的代谢物质,其生物合成代谢不仅为植物的生长提供的能量,同时分解产生的小分子单糖可以调控植物细胞的渗透压以抵抗盐胁迫。次生代谢物的生物合成途径,包括生物碱、帖类化合物和黄酮类化合物等在抵御病原物的入侵方面也发挥着重要的作用,通常还可以作为信号物质参与植物抗胁迫相关的信号转导[15~17]。
共差异基因的GO分析显示,短期盐胁迫下,无论是在高盐(500 mmol·L-1)还是低盐(300 mmol·L-1)浓度下,下调unigenes的数量都明显高于上调的数量。差异表达基因在细胞过程、代谢过程和响应刺激等类别中的基因所占比例较高。这与赵航等人用600 mmol·L-1处理盐穗木12和24 h得到的结果类似[18]。
渗透调节是植物适应逆境胁迫的重要反应。植物的主要渗调剂包括两类:一类是外源的无机离子,通过细胞膜上的离子泵调节胞内外的离子浓度,维持细胞的渗透压;另一类是植物自身合成的小分子有机溶质,包括糖类、甜菜碱及其衍生物、醇类及各种氨基酸。在盐胁迫下的盐离子存在,使植物对部分无机元素的吸收受到影响,因此小分子有机物就起到维持细胞的渗透势和降低盐离子对生物膜损伤的作用[19~23]。本研究从300 mmol·L-1NaCl处理盐穗木早期转录组数据中,筛选出3个渗透调节相关基因,且均为海藻糖合成的关键酶基因,说明海藻糖在低盐胁迫盐穗木早期起关键作用。而在高盐胁迫盐穗木转录组数据中筛选出了6个渗透调节相关基因,其中4个磷酸乙醇胺N-甲基转移酶(PEAMT)基因均上调,而PEAMT的主要作用是催化磷酸乙醇胺发生三次甲基化,最终生成胆碱,胆碱又可以经过催化生成甜菜碱。已有的研究表明,当土壤微生物枯草芽孢杆菌(GB03)与拟南芥共培养,拟南芥的胆碱和甘氨酸甜菜碱的合成提高2~5倍,从而提高甘露醇对拟南芥造成的渗透胁迫的抵抗能力,而GB03与peamt突变体拟南芥共培养时,拟南芥的抗旱能力消失,说明PEAMT是合成胆碱的关键酶,在植物的抗旱中起重要作用[24]。意味着海藻糖和甜菜碱分别在盐穗木低盐和高盐胁迫早期的渗透调节中发挥重要作用。
高浓度的盐对植物体产生渗透胁迫的同时,也可使植物体积累过量的活性氧,产生氧化胁迫,使细胞的正常代谢受到抑制,最终导致细胞产生氧化损伤甚至死亡[25~26]。为快速有效地清除细胞内堆积的大量活性氧,植物在受到盐胁迫后,就会产生一系列的抗氧化酶类来清除活性氧[27]。Chen等人的研究发现盐胁迫后杨树的超氧化物歧化酶、抗坏血酸过氧化物酶和谷胱甘肽还原酶的活性都提高了8~10倍[28]。Vaidyanathan等人发现盐耐受型水稻相比于盐敏感型水稻,其幼苗在用100 mmol·L-1NaCl和300 mmol·L-1NaCl处理后,表现出了更高的活性氧(ROS)清除能力,过氧化氢酶(CAT)、抗坏血酸(ASC)和谷胱甘肽(GSH)等抗氧化剂的水平显著增强,尽管超氧化物歧化酶(SOD)的活性稍低,但在细胞膜损伤程度和细胞内H2O2含量方面都表现出了优势[29]。本研究筛选了活性氧清除的相关酶,包括SOD、POD、Trx、GST和CAT。在低盐处理盐穗木早期转录组数据中筛选出13个,且大部分都是上调基因,而在高盐胁迫下,筛选出7个ROS清除相关基因,4个上调基因,3个下调基因。在低盐胁迫下,植物可能通过合成大量的ROS清除相关的酶类,来降低植物受到的氧化损伤,而高盐可能破坏了植物的活性氧清除相关体系,所以只有少数基因能够响应盐胁迫。
4 结论
利用RNA-Seq技术短期高盐和低盐胁迫的盐穗木进行转录组测序,分析了在低盐和高盐的胁迫下差异表达基因和共差异基因,表明差异表达基因主要富集在细胞过程、代谢过程和响应刺激等类别中。而筛选的渗透调节和活性氧清除的相关基因多为上调基因,证明盐穗木能够通过促进渗透调节和增强活性氧清除提高短期的盐胁迫适应能力。该研究为进一步探讨盐穗木耐盐分子机制和挖掘耐盐关键基因奠定了理论基础。
1.Zhu J K.Plant salt tolerance[J].Trends in Plant Science,2001,6(2):66-71.
2.Hasegawa P M,Bressan R A,Zhu J K,et al.Plant cellular and molecular responses to high salinity[J].Annual Review of Plant Physiology and Plant Molecular Biology,2000,51:463-499.
3.Sahu B B,Shaw B P.Isolation,identification and expression analysis of salt-induced genes inSuaedamaritima,a natural halophyte,using PCR-based suppression subtractive hybridization[J].BMC Plant Biology,2009,9:69.
4.Ventura Y,Eshel A,Pasternak D,et al.The development of halophyte-based agriculture:past and present[J].Annals of Botany,2014,115(3):529-540.
5.郗金标,张福锁,毛达如,等.新疆盐生植物群落物种多样性及其分布规律的初步研究[J].林业科学,2006,42(10):6-12.
Xi J B,Zhang F S,Mao D R,et al.Species diversity and distribution of halophytic vegetation in Xinjiang[J].Scientia Silvae Sinicae,2006,42(10):6-12.
6.Kumari S,Sabharwal V P N,Kushwaha H R,et al.Transcriptome map for seedling stage specific salinity stress response indicates a specific set of genes as candidate for saline tolerance inOryzasativaL.[J].Functional & Integrative Genomics,2009,9(1):109-123.
7.Xie Q,Niu J,Xu X L,et al.Denovoassembly of the Japanese lawngrass(ZoysiajaponicaSteud.) root transcriptome and identification of candidate unigenes related to early responses under salt stress[J].Frontiers in Plant Science,2015,6:610.
8.Jin H X,Dong D K,Yang Q H,et al.Salt-responsive transcriptome profiling ofSuaedaglaucavia RNA sequencing[J].PLoS One,2016,11(3):e0150504.
9.杜驰,张冀,张富春.盐胁迫诱导盐穗木Rev1、Rev3基因的表达分析[J].植物研究,2017,37(2):211-215,226.
Du C,Zhang J,Zhang F C.Expression analysis ofRev1 andRev3 ofHalostachyscaspicaunder salt stress[J].Bulletin of Botanical Research,2017,37(2):211-215,226.
10.Türkan I,Demiral T.Recent developments in understanding salinity tolerance[J].Environmental and Experimental Botany,2009,67(1):2-9.
11.Deinlein U,Stephan A B,Horie T,et al.Plant salt-tolerance mechanisms[J].Trends in Plant Science,2014,19(6):371-379.
12.Benhassaine-kesri G,Aid F,Demandre C,et al.Drought stress affects chloroplast lipid metabolism in rape(Brassicanapus) leaves[J].Physiologia Plantarum,2002,115(2):221-227.
13.Konova I V,Sergeeva Y E,Galanina L A,et al.Lipid synthesis byGeomycespannorumunder the impact of stress factors[J].Microbiology,2009,78(1):42-47.
14.Miled D D B,Zarrouk M,Chérif A.Sodium chloride effects on lipase activity in germinating rape seeds[J].Biochemical Society Transactions,2000,28(6):899-902.
15.Stitt M,Sulpice R,Keurentjes J.Metabolic networks:how to identify key components in the regulation of metabolism and growth[J].Plant Physiology,2010,152(2):428-444.
16.Ritala A,Dong L M,Imseng N,et al.Evaluation of tobacco(NicotianatabacumL.cv.Petit Havana SR1) hairy roots for the production of geraniol,the first committed step in terpenoid indole alkaloid pathway[J].Journal of Biotechnology,2014,176:20-28.
17.Li C Y,Leopold A L,Sander G W,et al.The ORCA2 transcription factor plays a key role in regulation of the terpenoid indole alkaloid pathway[J].BMC Plant Biology,2013,13(1):155.
18.赵航,贾富强,张富春,等.盐胁迫下盐穗木差异表达基因的转录组信息分析[J].生物信息学,2014,12(2):90-98.
Zhao H,Jia F Q,Zhang F C,et al.The transcriptome information analysis of differentially expressed genes ofHalostachyscaspicaunder salt stress[J].Chinese Journal of Bioinformatics,2014,12(2):90-98.
19.黎裕.植物的渗透调节与其它生理过程的关系及其在作物改良中的应用[J].植物生理学通讯,1994,30(5):377-383.
Li Y.The relationship between osmotic adjustment and other physiological processes and its application in crop improvement[J].Plant Physiology Communications,1994,30(5):377-383.
20.Hanson A D,Nelsen C E,Pedersen A R,et al.Capacity for proline accumulation during water stress in barley and its implications for breeding for drought resistance[J].Crop Science,1979,19(4):489-493.
21.Patakas A,Nikolaou N,Zioziou E,et al.The role of organic solute and ion accumulation in osmotic adjustment in drought-stressed grapevines[J].Plant Science,2002,163(2):361-367.
22.王娟,李德全.逆境条件下植物体内渗透调节物质的积累与活性氧代谢[J].植物学通报,2001,18(4):459-465.
Wang J,Li D Q.The accumulation of plant osmoticum and activated oxygen metabolism under stress[J].Chinese Bulletin of Botany,2001,18(4):459-465.
23.Caprioli M,Katholm A K,Melone G,et al.Trehalose in desiccated rotifers:a comparison between a bdelloid and a monogonont species[J].Comparative Biochemistry and Physiology Part A:Molecular & Integrative Physiology,2004,139(4):527-532.
24.Zhang H M,Murzello C,Sun Y,et al.Choline and osmotic-stress tolerance induced inArabidopsisby the soil microbeBacillussubtilis(GB03)[J].Molecular Plant-Microbe Interactions,2010,23(8):1097-1104.
25.Suzuki N,Koussevitzky S,Mittler R,et al.ROS and redox signalling in the response of plants to abiotic stress[J].Plant,Cell & Environment,2012,35(2):259-270.
26.Gill S S,Tuteja N.Reactive oxygen species and antioxidant machinery in abiotic stress tolerance in crop plants[J].Plant Physiology and Biochemistry,2010,48(12):909-930.
27.Jaleel C A,Riadh K,Gopi R,et al.Antioxidant defense responses:physiological plasticity in higher plants under abiotic constraints[J].Acta Physiologiae Plantarum,2009,31(3):427-436.
28.Chen S,Polle A.Salinity tolerance ofPopulus[J].Plant Biology,2010,12(2):317-333.
29.Vaidyanathan H,Sivakumar P,Chakrabarty R,et al.Scavenging of reactive oxygen species in NaCl-stressed rice(OryzasativaL.)-differential response in salt-tolerant and sensitive varieties[J].Plant Science,2003,165(6):1411-1418.
Supported by Special Funds of Xinjiang Key Laboratory(2014KL001)
introduction:ZHANG Li-Li(1993—),female,master student,mainly engages in research of molecular biology of plant.
date:2017-05-08
TranscriptomicAnalysisoftheHalostachyscaspicainResponsetoShort-termSaltStress
ZHANG Li-Li1,2ZHANG Fu-Chun1,2*
(1.College of Life Science and Technology,Xinjiang University,Urumqi 830046;2.Xinjiang Key Laboratory of Biological Resources and Genetic Engineering,Urumqi 830046)
As a halophyte with strong salt tolerance,Halostachyscaspicawidely distributes in desert saline-alkali land. In order to reveal the genomic changes of gene expression under salt stress, transcriptome sequencing ofH.caspicaassimilating branches with treatments of 300 and 500 mmol·L-1NaCl for 3 h was performed. A total of 153 298 unigenes with an average length of 643 bp were obtained with clean reads assembled. The 47 subclasses and 118 KEGG pathways were enriched in the GO terms and KEGG pathways, respectively. Differentially expressed genes analysis showed that there were 4 432 and 2 580 unigenes in response to low salt (300 mmol·L-1) and high salt (500 mmol·L-1) in short-term stress, respectively. The 1 245 unigenes were the common differentially expressed genes in the two salt stresses. They were mainly enriched in cellular process, metabolic process and response to stimulus. The osmotic regulation and reactive oxygen species (ROS) scavenging genes were screened out, and most of them were up-regulated. Therefore,H.caspicacould improve short-term salt stress adaptation by enhancing the osmotic adjustment and ROS scavenging.
Halostachyscaspica;salt stress;transcriptome;differentially expressed genes;osmotic adjustment;reactive oxygen species
新疆重点实验室专项资金资助项目(2014KL001)
张丽丽(1993—),女,硕士研究生,主要从事植物分子生物学研究。
* 通信作者:E-mail:zfcxju@xju.edu.cn
2017-05-08
* Corresponding author:E-mail:zfcxju@xju.edu.cn
S687
A
10.7525/j.issn.1673-5102.2018.01.011
