LOC105375840基因单核苷酸多态性位点与四川地区汉族人群颅内动脉瘤的关联研究
2017-12-08杨驭媒李新丽毛元英
杨驭媒,李新丽,张 可,毛元英
(成都市第六人民医院检验科,四川 成都 610051)
LOC105375840基因单核苷酸多态性位点与四川地区汉族人群颅内动脉瘤的关联研究
杨驭媒,李新丽,张 可,毛元英
(成都市第六人民医院检验科,四川 成都 610051)
目的探讨LOC105375840基因单核苷酸多态性(SNP)位点与四川地区汉族人群颅内动脉瘤(IA)的关联性。方法入选279例IA患者和670例正常对照样本,利用病例-对照关联研究的方法,选取LOC105375840基因内含子区2个SNP位点(rs6473928和rs4738837),并采用单碱基延伸法(SNaPshot)进行基因分型,研究这2个SNP位点与颅内动脉瘤发病是否相关。结果LOC105375840基因rs6473928和rs4738837位点基因型分布均符合哈迪-温伯格平衡(P> 0.05)。两个位点基因型(rs6473928P= 0.0327,rs4738837P= 0.0277)和等位基因(rs6473928P= 0.0106,rs4738837P= 0.0098)频率在IA组与对照组之间差异具有统计学意义。通过Haploview单倍体型分析显示,rs6473928和rs4738837位于8号染色体同一连锁不平衡区,其中:AG为风险单倍体型,可增加IA患病风险54%(OR=1.54,P= 0.0133),而GA则为保护型单倍体型,可降低IA易感性34%(OR=0.66,P= 0.0274)。结论LOC105375840基因内含子区SNP位点(rs6473928和rs4738837)与四川地区汉族人群颅内动脉瘤发病相关。
LOC105375840基因;单核苷酸多态性;颅内动脉瘤;病例-对照关联研究
颅内动脉瘤(intracranial aneurysm,IA)是指颅内动脉血管壁上的气球状异常膨出(balloon-like dilation),主要由动脉管壁局部缺陷和管腔内压力增高所引起,其发病率为(18~23)/100000,而血管造影和尸体解剖评估显示,未破裂动脉瘤的实际发生率为2.7%~6.5%[1~4]。多项研究表明,IA是遗传和环境因素共同作用的复杂疾病[5,6],年龄、性别、高血压以及过量饮酒等均与其发生有关[3],遗传因素也是IA发生发展的关键因子。位于9号染色体9p21区域的单核苷酸多态性(single nucleotide polymorphism,SNP)位点rs10757278是第一个被报道与IA相关的多态位点[7]。随后,不同国家的研究团队先后成功应用全基因组关联分析(Genome Wide Association Study,GWAS)发现多个与IA显著相关的基因和位点,分别位于2q33.1、8q11.12-12.1、9p21.3、18q11.2、13q13.1、10q24.32等染色体区域[8~10]。Bilguvar等[8]的GWAS研究同时包含了欧洲人群(1,818例IA病例和6,038例对照)和日本人群(495例IA病例和676例对照),同时发现2q33.1、8q11.12-12.1、9p21.3三个区域与IA相关。其中,位于8q12.1区域上LOC105375840基因内含子区2个SNP位点(rs6473928和rs4738837)与欧洲和日本人群IA均显著相关,但在中国人群中还未见LOC105375840基因与IA相关性的报道。因此,本研究选取LOC105375840基因2个SNP位点(rs6473928和rs4738837),通过病例-对照关联研究分析其与四川地区中国汉族人群IA发病的相关性。
1 材料与方法
1.1一般资料2010年1月至2016年11月成都市第六人民医院确诊为IA的患者279例为病例组(IA组),平均年龄57.3岁(38~89岁),其中男93例(33.3%);IA组均通过全脑血管造影术(DSA)确诊颅内动脉瘤。同时选取670例健康体检人群作为正常对照组(对照组),平均年龄62.3岁(50~81岁),其中男239例(35.7%);对照组均无心脑血管系统疾病,近期无感染炎症等,并通过CT血管造影(CTA)排除颅内动脉瘤。本项目研究经过医院伦理委员会批准,所有流行病学调查以及样本收集均征得受试者同意并签署知情同意书,IA组与对照组均为无血缘关系的四川地区汉族人群,基本情况见表1。
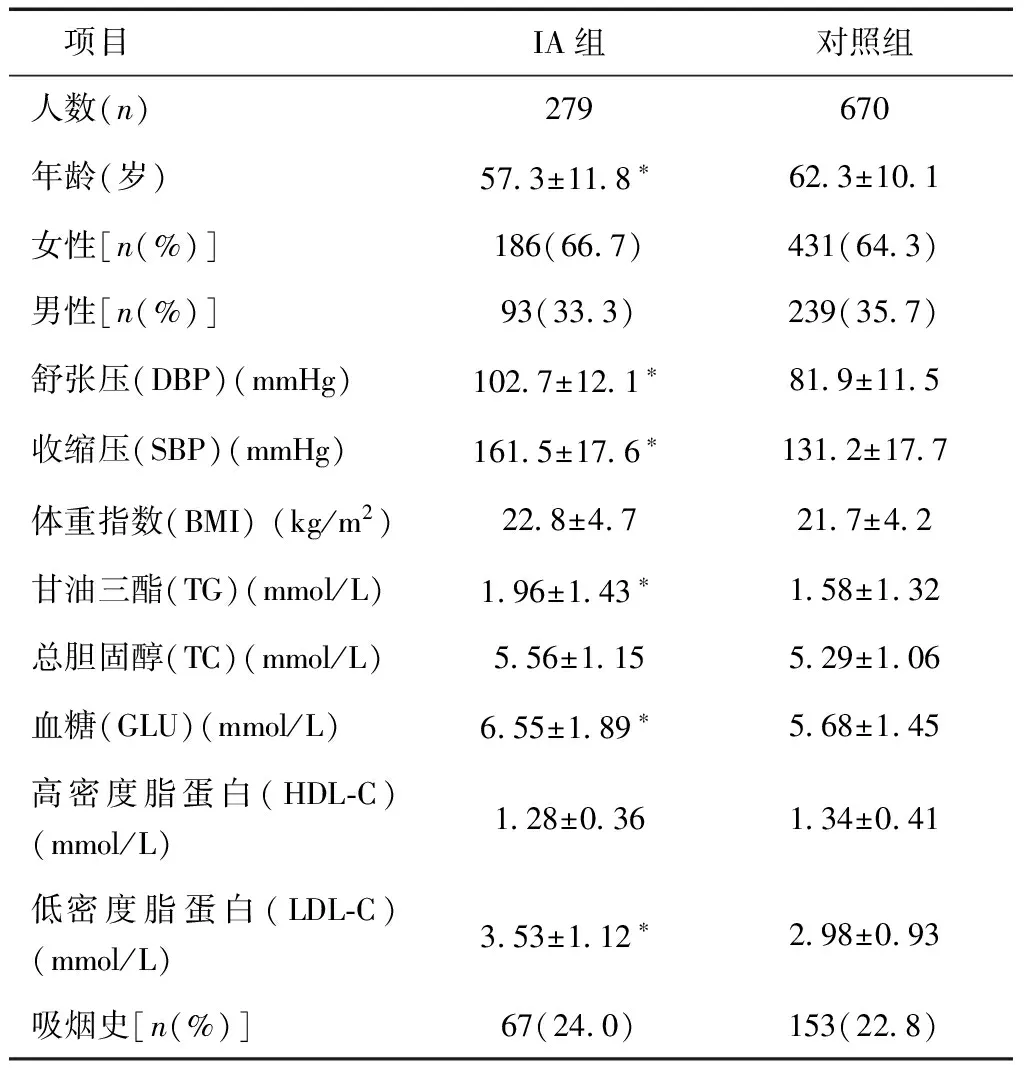
表1 颅内动脉瘤患者(IA)组与对照组基本情况
* 与对照组比较,P< 0.05
1.2相关生物化学指标的检测甘油三脂(TG)、总胆固醇(TC)、血糖(GLU)、高密度脂蛋白(HDL-C)、低密度脂蛋白(LDL-C)检测:研究对象空腹(8小时以上)采集静脉血,相关生化指标的检测均在成都市第六人民医院检验科进行。
1.3基因组DNA提取EDTA抗凝采血管收集受试者外周静脉血5 ml,加入0.2%NaCl,振荡摇匀,放于冰上15分钟;3000 rpm离心30分钟,弃上清,0.2%NaCl洗涤;加入10 mM Tris-HCl和10 mM EDTA (pH=8.0)、10% SDS、25 mg/ml蛋白酶K和10 mg/ml RNaseA,加入量分别为4 ml、16 μl和20 μl,混匀,37 ℃过夜;加入4 ml Tris/酚溶液,混匀,3000 rpm离心10分钟,氯仿抽提两次,采集水相;加入l/10体积的3 M NaAC (pH=5.2),两倍体积的无水乙醇,使DNA沉淀;再用70%乙醇洗涤两次,获得基因组DNA,溶解于TE溶液中。定量测定260 nm和280 nm光吸收率,计算DNA浓度和纯度,DNA工作液浓度稀释至50 ng/ μl,保存于-20 ℃备用。
1.4聚合酶链式反应(PCR)扩增目的片段从NCBI基因组公共数据库网站(https://www.ncbi.nlm.nih.gov/snp/)获取LOC105375840基因内含子区2个SNP位点(rs6473928和rs4738837)周围的DNA序列,采用Primer 3.0(version 0.4.0)在线软件(http://bioinfo.ut.ee/primer3-0.4.0/)辅助设计PCR引物,由生工生物工程(上海)股份有限公司合成(引物序列和PCR产物长度见表2)。PCR反应程序如下:95 ℃预变性 5分钟,(95 ℃ 30秒,56 ℃ 30秒,72 ℃延伸30秒)× 35个循环,72 ℃延伸10分钟,12 ℃保温。

表2 LOC105375840基因2个位点引物序列及PCR产物情况
1.5SNP位点基因分型采用单碱基延伸(SNaPshot)法进行2个SNP位点的基因分型,具体步骤如下:①PCR产物纯化:2个位点各4.5 μlPCR 产物混合,加入1 U碱性磷酸酶(Promega公司)以及1 U核酸外切酶(Biolabs公司),混匀,37 ℃ 30分钟,75 ℃ 15分钟;②SnaPshot反应:上述纯化产物1 μl,加入2个位点的SNaPshot引物各0.3 μl(SNaPshot引物序列见表2),SNPshot Multiplex试剂1 μl(美国ABI公司),双蒸水补足5 μl,反应程序:96 ℃1 分钟,(96 ℃ 10秒,50 ℃ 5秒,60 ℃ 30秒)× 25个循环,12 ℃保温;③二次纯化:5 μl上述产物,加入1 U碱性磷酸酶,混匀,37 ℃ 15分钟,75 ℃ 15分钟;④基因分型:上述产物1 μl,加入HD 9 μl,3730XL测序仪(ABI公司)上机,检测基因型,并通过测序仪自带的Genemap软件读取基因分型情况。
1.6统计学方法采用SPSS 16.0软件进行统计分析。定量资料以均数±标准差表示,组间比较采用t检验;计数资料比较采用χ2检验;应用多因素回归分析进行相关性分析。P< 0.05为差异有统计学意义。
2 结果
2.1IA组与对照组基本情况比较分析如表1所示,IA组的舒张压、收缩压以及血清TG、GLU、LDL-C的水平均高于对照组(P< 0.05)。
2.2LOC105375840基因2个SNP位点的基因型分布如表3中数据显示,2个SNP位点(rs6473928和rs4738837)的基因型分布在IA组和对照组均符合哈迪-温伯格平衡(P> 0.05),说明本研究的研究对象具有群体代表性,没有人群分层的情况。统计结果显示,2个位点的基因型分布在IA组和对照组间差异均具有统计学意义(rs6473928P= 0.0327、rs4738837P= 0.0277);其小等位基因的纯合子对于IA发生具有保护作用(rs6473928OR=0.70、rs4738837OR=0.69),见表3。
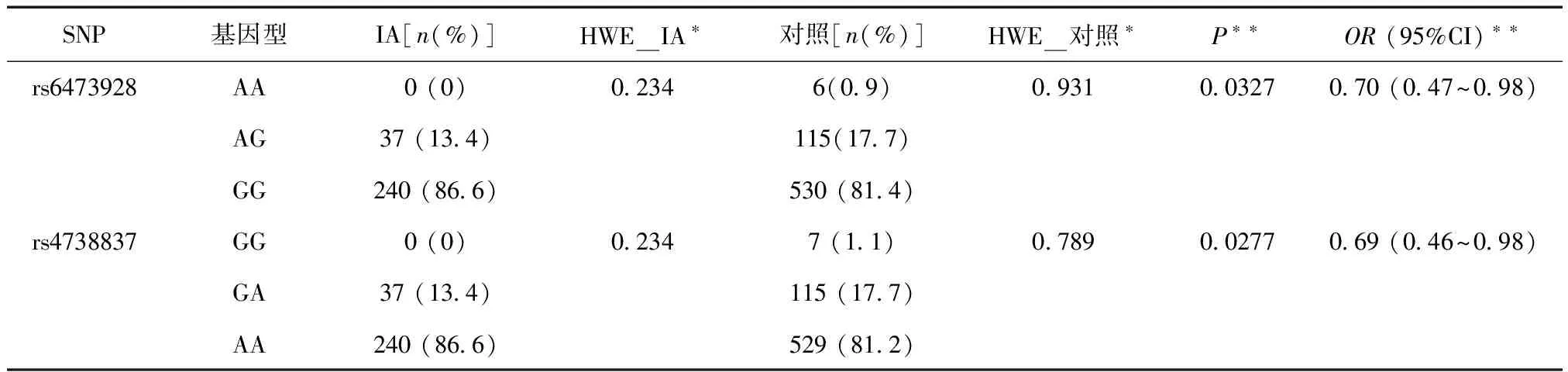
表3 LOC105375840基因2个位点在IA组和对照组中基因型分布比较
*HWE:哈迪-温伯格平衡;**多因素回归分析校正年龄、性别等因素(OR:优势比,95%CI:95%置信区间)
2.3LOC105375840基因SNP位点等位基因频率在IA组和对照组间的比较LOC105375840基因上2个SNP位点的等位基因频率在IA组和对照组之间的差异同样具有统计学意义(rs6473928P= 0.0106、rs4738837P= 0.0098,表4),其小等位基因可以降低对IA的易感性(rs6473928OR=0.66、rs4738837OR=0.65),见表4。
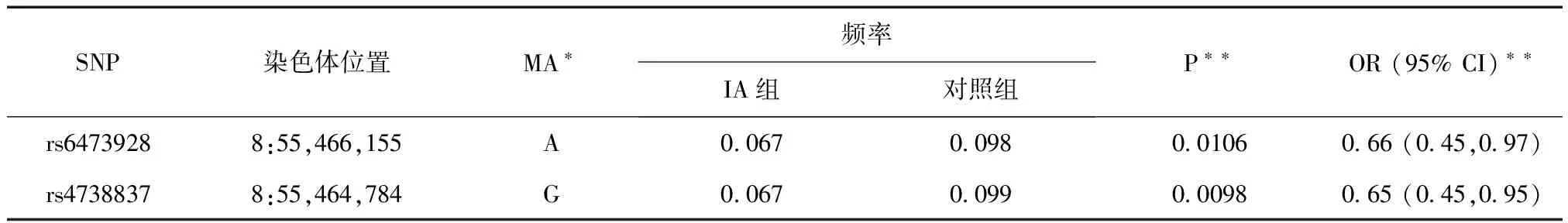
表4 LOC105375840基因2个位点等位基因频率比较分析
*MA:Minor allele(小等位基因);**,P值和OR值为校正年龄、性别等参数之后的结果(OR:优势比,95%CI:95%置信区间)
2.4单倍体型分析通过Haploview 4.2 软件对LOC105375840基因rs6473928和rs4738837位点进行连锁不平衡分析,结果如图1所示,2个SNP位点位于同一个连锁不平衡区域(linkage disequilibrium block,LD)。这2个位点可组合产生3个单倍体型,其中风险单倍体型(H1:AG)可以增加IA患病风险54%(P= 0.013,表5),而保护型的单倍体型(H2:GA)则可降低IA易感性34%(P= 0.027),见表5。
3 讨论
IA发病率高,危害严重,尤其IA破裂所导致的蛛网膜下腔出血(subarachnoid hemorrhages,SAH)是一类非常严重的脑血管意外,具有高致残率和高死亡率[11]。很大一部分IA患者在动脉瘤破裂之前没有任何症状或提示。尽早发现及确诊,尤其是没有相关病史的IA患者,对于降低其致死致残率非常必要。遗传因素在IA发病过程中具有非常重要的作用,能够尽可能多的发现与IA发病相关的基因组标记物。
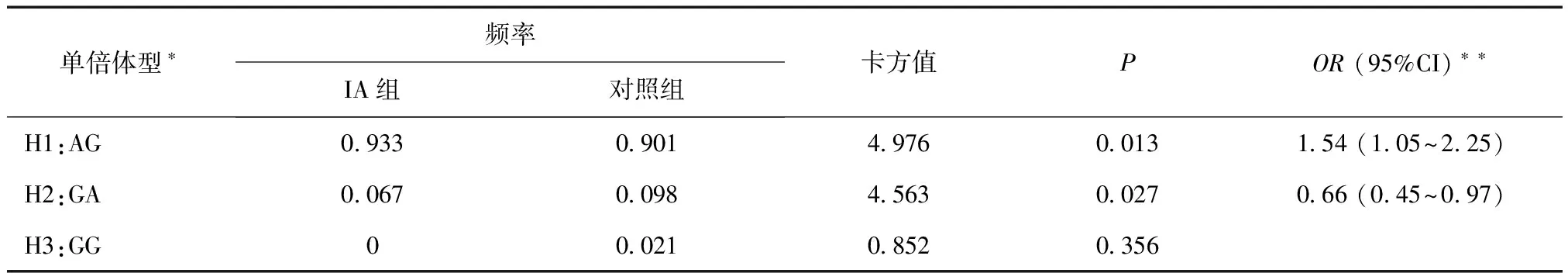
表5 LOC105375840基因两个位点的单倍体型分析
*3个单倍体型(H)由LOC105375840基因2个SNP位点(rs6473928和rs4738837)所产生;**此处只计算P<0.05时的OR值(OR:优势比,95%CI:95%置信区间)
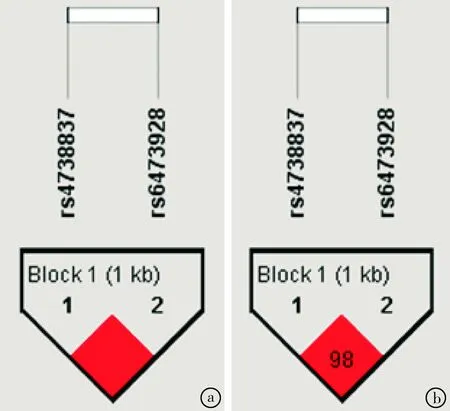
图1 LOC105375840基因2个SNP位点(rs6473928和rs4738837)连锁不平衡图 a:显示D’值(D’=1.0);b:显示r2值(r2=0.98)。
利用全基因组SNP芯片所进行的GWAS研究方法,已经成功鉴定了多种复杂疾病的易感基因,包括青光眼、阿尔海默茨症(AD)、以及多种恶性肿瘤等[12~15]。但是关键在于GWAS所发现的基因/位点能否适用于不同种族人群。本研究针对一项欧洲和日本人群颅内动脉瘤GWAS Meta分析的结果[8],在四川地区汉族人群中进行了的验证。选取了LOC105375840基因内含子区的2个SNP位点(rs6473928和rs4738837),在279例IA患者和670例正常对照中进行病例-对照关联研究,分析其与IA发病的相关性。本研究发现,这3个SNP位点与IA发病具有相关性,其小等位基因可以降低IA的发病风险,我们的结果也证实了欧洲和日本人群的发现。
LOC105375840基因位于染色体8q12.1区域,属于长链非编码RNA(long noncoding RNA,lncRNA)。lncRNA是一类转录本长度大于200 nt的非编码RNA分子,其长度一般介于200~100 000 nt之间,具有高度保守的序列元件和特定的空间二级结构[16]。研究发现lncRNA作为一类重要的基因表达调控因子,本身并不具备编码蛋白的功能,但能以RNA的形式在多个层面上(表观遗传调控、转录调控以及转录后调控等)调控基因的表达,与生长发育、细胞分化及肿瘤发生发展等生物学功能密切相关[17]。目前,lncRNA已成为继microRNA之后又一个热门研究的非编码RNA分子。随着近年来对lncRNA研究的不断深入,越来越多的证据表明lncRNA参与多种疾病的发生发展[18]。
欧洲人群、日本人群以及我们的研究结果都表明,LOC105375840基因SNP位点与IA发病相关,有作为IA早期诊断分子标记物的潜力。而LOC105375840如何在IA的发生发展中发挥作用,及其调控的靶基因和调控方式还有待于进一步的研究,这些研究也将有助于颅内动脉瘤的早期诊断、预防和治疗。
[1] Nasr DM,Brown RD.Management of Unruptured Intracranial Aneurysms[J].Curr Cardiol Rep,2016,18(9):86.
[2] Flemming KD,Lanzino G.Management of Unruptured Intracranial Aneurysms and Cerebrovascular Malformations[J].Continuum (Minneap Minn),2017,23:181-210.
[3] Rinkel GJE,Djibuti M,Algra A,et al.Prevalence and risk of rupture of intracranial aneurysms[J].Stroke,1998,29(1):251-256.
[4] Brown RD,Broderick JP.Unruptured intracranial aneurysms:epidemiology,natural history,management options,and familial screening[J].Lancet Neurol,2014,13(4):393-404.
[5] Ruigrok YM,Rinkel GJ.From GWAS to the clinic:risk factors for intracranial aneurysms[J].Genome Med,2010,2(9):61.
[6] Ruigrok YM,Rinkel GJ,Van’t Slot R,et al.Evidence in favor of the Contribution of genes involved in the maintenance of the extracellular matrix of the arterial wall to the development of intracranial aneurysms[J].Human Molecular Genetics,2006,15(22):3361-3368.
[7] Helgadottir A,Thorleifsson G,Magnusson KP,et al.The same sequence variant on 9p21 associates with myocardial infarction,abdominal aortic aneurysm and intracranial aneurysm[J].Nat Genet,2008,40(2):217-224.
[8] Bilguvar K,Yasuno K,Niemel M,et al.Susceptibility loci for intracranial aneurysm in European and Japanese populations[J].Nat Genet,2008,40(12):1472-1477.
[9] Yasuno K,Bilguvar K,Bijlenga P,et al.Genome-wide association study of intracranial aneurysm identifies three new risk loci[J].Nat Genet,2010,42(5):420-425.
[10]Gretarsdottir S,Baas AF,Thorleifsson G,et al.Genome-wide association study identifies a sequence variant within the DAB2IP gene conferring susceptibility to abdominal aortic aneurysm[J].Nat Genet,2010,42(8):692-697.
[11]Ingall T,Asplund K,Mähönen M,et al.A multinational comparison of subarachnoid hemorrhage epidemiology in the WHO MONICA stroke study[J].Stroke,2000,31(5):1054-1061.
[12]Khor CC,Do T,Jia H,et al.Genome-wide association study identifies five new susceptibility loci for primary angle closure glaucoma[J].Nat Genet,2016,48(5):556-562.
[13]Chen Y,Lin Y,Vithana EN,et al.Common variants near ABCA1 and in PMM2 are associated with primary open-angle glaucoma[J].Nat Genet,2014,46(10):1115-1119.
[14]Apostolova LG.Alzheimer disease:A quantitative trait approach to GWAS pays dividends[J].Nat Rev Neurol,2017,13(6):321-322.
[15]McKay JD,Hung RJ,Han Y,et al.Large-scale association analysis identifies new lung cancer susceptibility loci and heterogeneity in genetic susceptibility across histological subtypes[J].Nat Genet,2017,49(7):1126-1132.
[16]Johnsson P,Lipovich L,Grander D.Evolutionary conservation of long non-coding RNAs;sequence,structure,function.Biochim Biophys Acta[J].2014,1840(3):1063-1071.
[17]Yang L,Froberg JE,Lee JT.Long noncoding RNAs:fresh perspectives into the RNA world[J].Trends in biochemical sciences,2014,39(1):35-43.
[18]Dinger ME.Long non-coding rnas in disease and development[J].Pathology,2014,46(Suppl 1):S26.
AssociationStudybetweenSNPsintheLOC105375840GeneandIntracranialAneurysminaSichuanHanChinesePopulation
YANGYu-mei1,LIXin-li1,ZHANGKe1,MAOYuan-ying1
(ClinicalLaboratory,No.6People’sHospitalofChengdu,Chengdu610051,China)
LIXin-li
ObjectiveTo investigate the association between single nucleotide polymorphisms (SNPs) in the LOC105375840 gene and intracranial aneurysm (IA) in a Sichuan Han Chinese population.MethodsGenomic DNA samples from 279 patients with IA and 670 normal controls were collected.Two SNPs (rs6473928 and rs4738837) in the intron of LOC105375840 gene were genotyped by using SNaPshot method.ResultsAll the genotype frequencies of these two SNPs were in Hardy-Weinberg equilibrium (HWE) (P> 0.05).Significant differences were found in both genotype and allele frequency distribution of these SNPs between IA group and control group (P< 0.05).These two SNPs were in the same linkage disequilibrium (LD) block located in Chromosome 8.The risk haplotype AG could increase the susceptibility to IA by 54% (P= 0.0133),and the protective haplotype GA could decrease the susceptibility to IA by 34% (P= 0.0274).ConclusionOur results suggest that SNPs in the LOC105375840 gene were significantly associated with IA in the Sichuan Han Chinese population.
LOC105375840 gene;Single nucleotide polymorphism;Intracranial aneurysm;Case-control association study
四川省卫生计生委科研基金资助项目(编号:130152)
李新丽
R651.1
A
1672-6170(2017)06-0037-04
2017-10-10)
