急性单核细胞白血病全基因组微小RNA的表达谱分析*
2017-11-28林小聪李宁符伟玉兰柳波张海涛张宇明
林小聪 ,李宁 ,符伟玉 ,兰柳波 ,张海涛 ,张宇明
(1.广东医科大学 生物化学与分子生物学研究所,广东 湛江 524023;2.广东医科大学附属医院 血液科,广东 湛江 524001)
急性单核细胞白血病全基因组微小RNA的表达谱分析*
林小聪1,李宁2,符伟玉1,兰柳波1,张海涛1,张宇明2
(1.广东医科大学 生物化学与分子生物学研究所,广东 湛江 524023;2.广东医科大学附属医院 血液科,广东 湛江 524001)
目的研究急性单核细胞白血病(AMoL)全基因组的微小RNA(miRNA)表达谱。方法应用Illumina高通量测序技术对10例AMoL患者和10例非恶性血液病患者骨髓样本的miRNA表达水平进行检测,分析两者的表达差异,并通过茎环引物实时荧光定量 PCR(Stem-loop qPCR)技术对部分Illumina测序结果进行验证。结果两组样本共发现差异表达的miRNA 285个,其中,199个miRNA在AMoL组呈表达上调,86个miRNA呈表达下调。6个miRNA的Stem-loop qPCR验证结果与其测序结果具有相同的变化趋势。结论miR-126-3p和miR-29b-3p可作为潜在的分子靶点用于AMoL发病机制的进一步研究。
急性单核细胞白血病;微小RNA;测序
急性单核细胞白血病(acutemonocytic leukemia,AMoL)为急性髓性白血病(acute myeloid leukemia,AML)最常见的亚型之一;其病例数约占AML 20%~30%,在各种AML亚型中居第2位[1]。相比于其他的AML亚型,AMoL高白细胞血症、髓外浸润以及不良染色体核型的发生率更高,常规化疗完全缓解率低、容易复发及预后不佳[1-2]。目前,AMoL的发病机制仍未完全明确,可能与电离辐射、染色体异常及基因突变等多种因素有关[3]。为寻找新的治疗靶点,迫切需要对AMoL的发病机制进行进一步研究。微小 RNA(micro ribonucleic acid,miRNA)是一类内源性、具有基因表达调控功能的小分子单链非编码RNA,长度约为 18~25 个核苷酸(nt)[4]。研究表明,miRNA与白血病发生、发展密切相关,可能成为白血病潜在的治疗靶点[5]。为阐明AMoL的miRNA表达谱,本研究应用Illumina高通量测序技术对AMoL与对照组骨髓样本的miRNA表达水平进行对比分析,为研究AMoL相关发病机制和寻找新型的分子靶点提供新的线索。
1 资料与方法
1.1 研究对象
收集来自于广东医科大学附属医院血液科10例AMoL患者(男性6例,女性4例;年龄14~64岁,平均41.2岁)和10例非恶性血液病对照者(骨髓象正常的贫血或发热查因患者,男性6例,女性4例;年龄10~73岁,平均39.0岁)的骨髓样本。所有AMoL患者均为初发病例,在采样前均未接受放疗或化疗,且符合AMoL的诊断标准。本研究已获得广东医科大学附属医院伦理学委员会审核批准,并取得所有患者的知情同意。
1.2 主要试剂
无RNase的DnaseⅠ(购自美国Promega公司),Trizol试剂(购自美国Invitrogen公司),Small RNA测序接头、TruSeq Rapid SR cluster试剂盒以及Tru Seq Rapid SBS试剂盒(美国Illumina公司产品),SuperscriptTMⅢ逆转录酶(购自美国Invitrogen公司),2×SYBR Green PCR混合液(美国 Applied Biosystems公司)。
1.3 方法
1.3.1 总RNA样品的提取 根据Trizol试剂说明书抽提组织总RNA,以无RNase的DnaseⅠ消化以去除基因组DNA污染。20例RNA样品分别以AMoL组和对照组为组单位等质量混合,在230、260及280 nm波长以Nano Drop ND-1000型分光光度计测定其吸光度(A)值,计算RNA样品的浓度并分析其纯度,通过琼脂糖凝胶电泳评估RNA的完整性。
1.3.2 Small RNAs文库构建和Illumina测序RNA样品在T4RNA连接酶的催化下加上3’、5’small RNA测序接头,所得产物经逆转录PCR扩增及聚丙烯胺凝胶电泳纯化生成Small RNAs文库。以Agilent 2100型生物分析仪对Small RNAs文库进行定量分析。应用Illumina cBot簇生成系统,按照TruSeq Rapid SR cluster试剂盒说明书,对文库进行克隆扩增;然后根据TruSeq Rapid SBS试剂盒说明书,在Illumina HiSeq 2000测序系统上行高通量深度测序。
1.3.3 测序数据的处理和注释 以Off-Line Basecaller软件对高通量测序所生成的原始图像进行分析,读取碱基序列信息并转换为相应的数据。然后,经去接头、去冗余、去低质量以及去污染等处理,获得16~30 nt长度的高质量干净序列并进行序列长度分布分析。将干净序列分别与RefSeq数据库、Rfam数据库以及RepBase数据库进行序列比对和分类注释,去除mRNA、重复序列、tRNA、rRNA、snRNA以及sRNA等序列;余下序列再与miRBase数据库中已知的人类pre-miRNA进行序列比对和注释,获取两样本miRNA的表达丰度和染色体分布等信息。
1.3.4 miRNA的差异表达分析 首先对两样本miRNA的表达丰度进行标准化处理 [公式:miRNA的标准化表达丰度(normalization of the calculation of transcript parts per million,TPM)=miRNA 的表达丰度/样本miRNA的总表达丰度×106],然后再计算两样本的miRNA差异表达比值 [公式:miRNA的差异表达比值=(AMoL组miRNA标准化表达丰度值+10TPM)/(对照组miRNA标准化表达丰度值+10TPM)]。如果差异表达比值≤0.5或≥2.0,则为差异有统计学意义;如果0.5<比值<2.0则为差异无统计学意义。
1.3.5 茎环引物实时荧光定量PCR(stem-loop primer-based quantitative real-time polymerase chain reaction,Stem-loop,qRT-PCR)利用 miRNA 的茎环(Stem-loop)逆转录引物,根据SuperscriptTMⅢ反转录酶说明书进行逆转录反应。应用ABI公司Prism7500型qRT-PCR仪,参照2×SYBR Green PCR混合液说明书,以U6 snRNA作为内参照进行qRT-PCR。反应参数为:95℃预反应10 min,使模板完全解链。然后95℃变性10s,60℃退火及延伸60s,重复40个循环。检测结果计算采用常规的2-△△Ct法。
1.4 统计学方法
数据分析采用SPSS 16.0统计软件,计量资料以均数±标准差(±s)表达,比较用t检验,P<0.05为差异有统计学意义。
2 结果
2.1 总RNA的质量分析
提取的组织RNA样品分别以AMoL组和对照组为组单位等量混合后,对其进行纯度分析(见表1)和RNA完整性分析(见图1)。结果表明,其A260/A280比值都在1.8~2.0,且A260/A230比值都>1.8,提示样本RNA的纯度较高、质量良好。电泳结果显示两组总RNA均可见18和28 S 2条清晰的条带,而5 S的条带则比较模糊(见图1);提示RNA的完整性好。上述结果表明,总RNA的质量符合实验要求,可用于后续Small RNAs文库的构建。
2.2 Small RNAs文库的质量评估
以Agilent 2100型生物分析仪对Small RNAs文库进行质量分析(见表2)。结果表明,两样本Small RNAs文库的片段长度峰值均在130~155 nt且其摩尔数>1 fmol,提示Small RNAs文库的质量良好,可用于后续的克隆扩增和测序分析。
2.3 Small RNA干净序列的长度分布和分类注释
Small RNA干净序列(16~30 nt)的长度分布分析表明,对照组及AMoL组的干净序列均大致呈正态分布在22 nt序列两侧(见图2)。其中,符合miRNA片段长度(18~25 nt)的干净序列所占的比例最高,其在对照组与AMoL组分别占93.70%和82.31%。干净序列的序列比对和分类注释表明,miRNA在对照组(占80.05%)及AMoL组(占54.44%)所占的比例最高,两者均>50%(见图3)。上述数据初步验证Illumina测序结果的有效性。
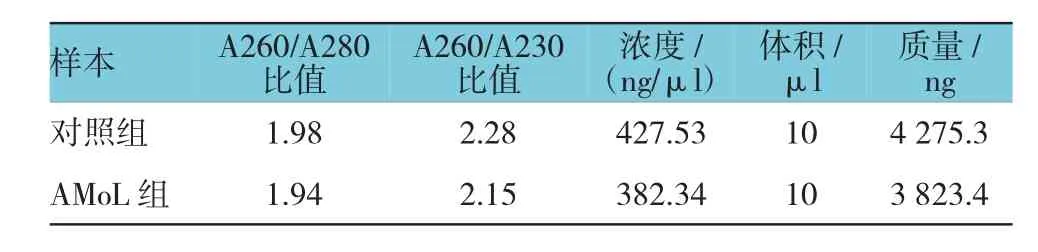
表1 总RNA的纯度分析
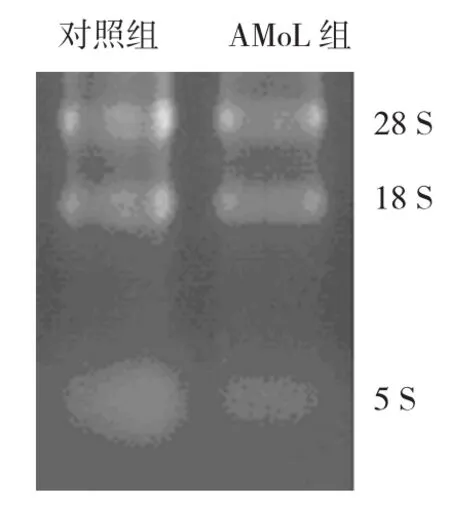
图1 总RNA琼脂糖凝胶电泳图
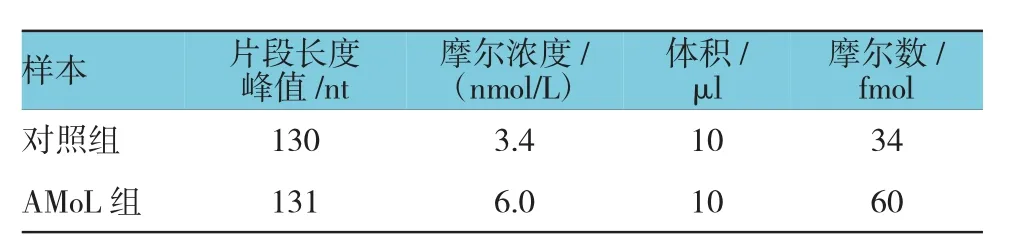
表2 Small RNAs文库的定量分析
2.4 miRNA的差异表达谱分析
两组样本发现差异表达的miRNA 285个。其中,199个miRNA在 AMoL组呈表达上调,86个miRNA呈表达下调。hsa-miR-122-5p在AMoL组表达上调最显著,而hsa-miR-941则表达下调最明显。见表3。
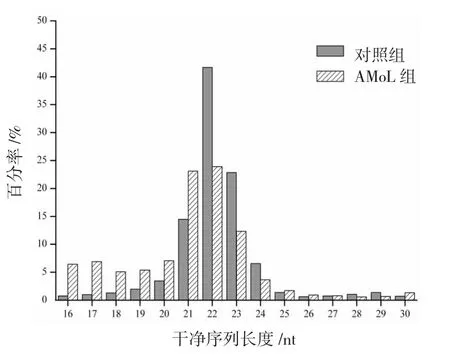
图2 Small RNA干净序列的长度分布
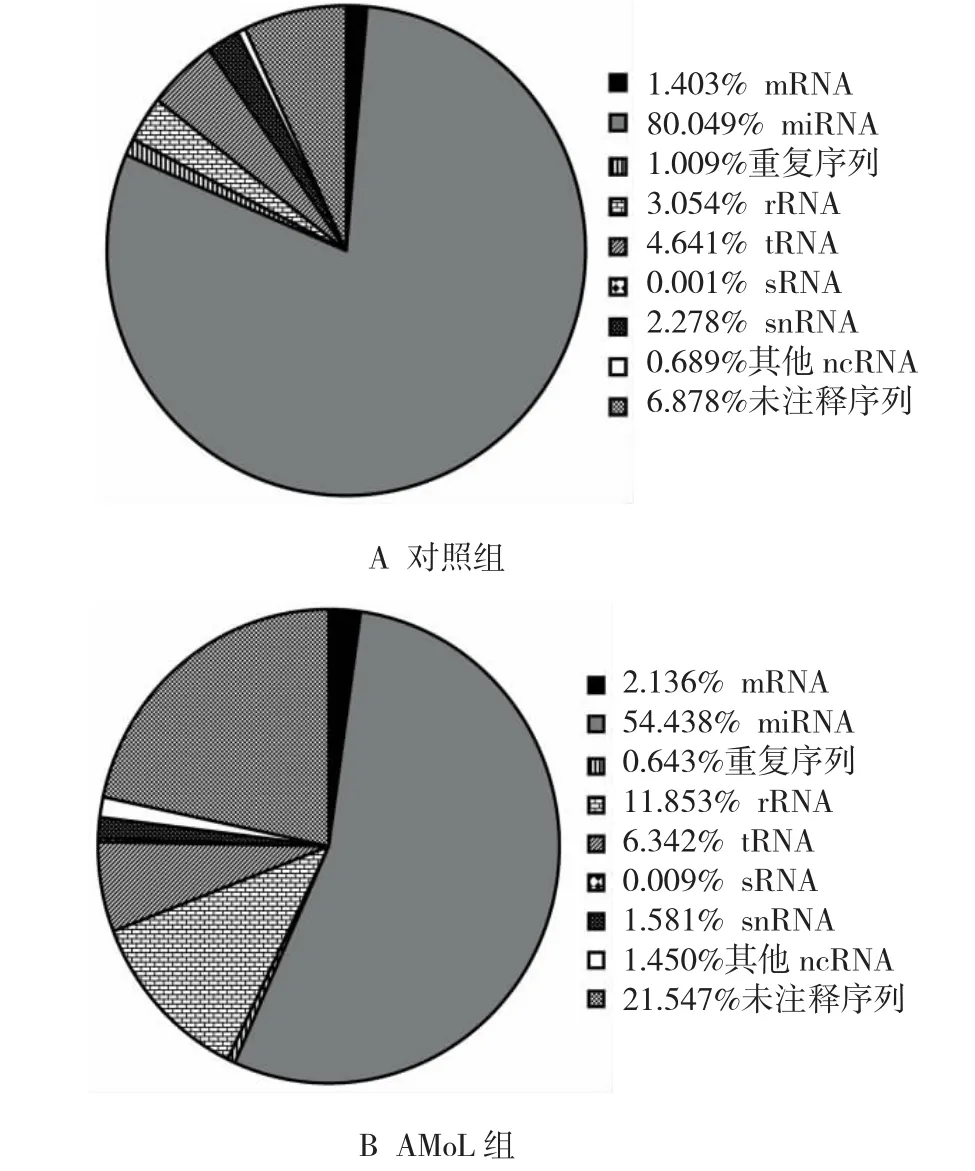
图3 匹配的干净序列的分类注释

表3 前10个表达上调和下调miRNA的差异
2.5 Stem-loop qRT-PCR验证结果
为验证Illumina测序结果的可靠性,笔者选择2个表达上调(miRNA-155-5p和miRNA-221-3p)、2个表达下调(miRNA-106b-5p和miRNA-142-3p)以及2个表达差异无统计学意义miRNA(miRNA-532-5p和miRNA-423-5p)进行Stem-loop qRT-PCR验证。结果表明,上述6个miRNA在两组样本中的变化趋势与Illumina测序结果一致(见表4),说明Illumina测序结果可靠、有效。
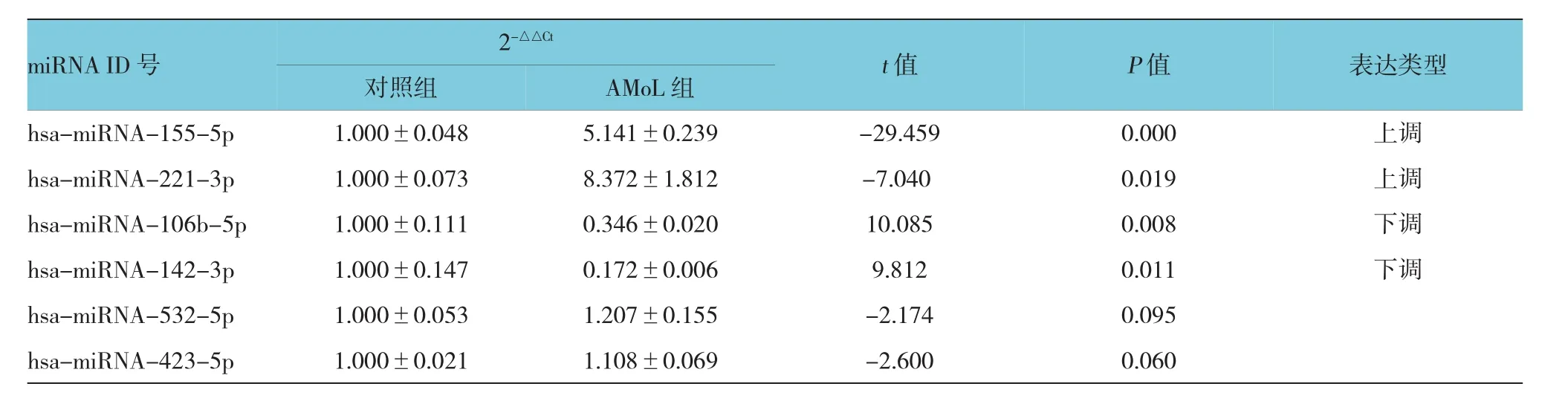
表4 Illumina测序结果的Stem-loop qRT-PCR验证
3 讨论
作为一种具有基因表达调控功能的内源性小片段单链RNA,miRNA与白血病关系密切。研究表明,>60%的miRNA基因存在于白血病相关的染色体易位区,多种miRNA可通过调节细胞增殖、分化及凋亡与耐药等相关基因的表达在白血病发生、发展过程中发挥关键作用,可能成为白血病潜在的诊断标志物和分子靶点[6-7]。但目前尚无通过Illumina高通量测序技术在AMoL患者临床样本中系统性研究miRNA表达谱的报道。本结果表明,两者的miRNA表达谱存在较大的差异,两组样本共鉴定差异表达的miRNA 285个,其中,199个miRNA在AMoL组呈表达上调,86个miRNA呈表达下调。笔者随后选取6个miRNA,应用Stem-loop qRT-PCR技术证实Illumina测序结果的可靠性。
miRNA-29b-3p是一种基因定位于染色体7q32和1q32的miRNA分子,在胃癌、前列腺癌及肺癌等多种实体瘤以及AML中呈低表达;在AML中,染色体7q缺失或CEBPA基因功能障碍可导致miRNA-29b-3p 表达下调[8]。GONG 等[9]报道,miRNA-29b-3p可通过作用于其靶基因CCND2和AKT2,抑制AMoL细胞系THP1增殖并诱导细胞凋亡。本研究表明,miRNA-29b-3p在AMoL患者骨髓样本中表达呈下调,与文献报道基本相符,提示miRNA-29b-3p在AMoL发病机制中可能起一定的作用。相比于其他的AML亚型,miRNA-199b-5p在AMoL临床样本中呈异常低表达,并与AMoL患者预后呈正相关;组蛋白脱乙酰基酶抑制剂AR-42和Panobinostat可通过上调hsa-miRNA-199b-5p表达诱导THP-1细胞凋亡[10]。笔者研究发现,miRNA-199b-5p在AMoL样本中也呈低表达,与文献报道一致,提示miRNA-199b-5p可能与AMoL发病机制相关。
miRNA-126基因在染色体上定位于9q34.3,其转录产物为miRNA-126-5p和miRNA-126-3p。研究表明,miRNA-126-5p和miRNA-126-3p在AML中均呈高表达并与患者预后呈负相关[11]。LEEUW等[12]发现,沉默miRNA-126-3p表达可抑制THP1细胞增殖、诱导细胞凋亡并抑制NOD/SCID小鼠移植瘤的生长。提示miRNA-126-3p在AMoL中可能具有潜在的癌基因活性。本组miRNA-126基因的转录产物miRNA-126-5p和miRNA-126-3p在AMoL中均呈表达上调,与文献报道基本相符。HO等[13]报道,miRNA-135b-5p在依托泊甙以及替尼泊苷耐药的白血病细胞中呈表达上调,其过表达可诱导白血病细胞对依托泊甙以及替尼泊苷产生耐药性。此外,miRNA-135b-5p在AMoL细胞系U937以及THP-1中过表达均可下调Ppm1e并激活AMPK[14]。研究表明,AMPK激活在AMoL细胞凋亡过程中起重要作用;骨髓脂肪细胞β-氧化所介导AMPK的激活可抑制AMoL细胞凋亡并促进细胞增殖[15]。本组miRNA-135b-5p在AMoL样本中也呈高表达,与文献报道基本相符,提示miRNA-135b-5p在AMoL细胞凋亡过程中可能起一定的抑制作用,具有潜在的研究价值。
综上所述,AMoL患者与非恶性血液病对照者骨髓样本的miRNA表达谱存在较大的差异。根据其差异表达趋势与文献报道的符合程度,笔者得到5个与AMoL发病机制有密切关系的miRNA(包括miRNA-29b-3p、miRNA-199b-5p、miRNA-126-3p、miRNA-126-5p 及 miRNA-135b-5p)。其中,miRNA-126-3p的差异表达比值最大,呈明显的表达上调;而miRNA-29b-3p的差异表达比值最小,呈明显的表达下调;再加上已有文献证实两者在AMoL细胞增殖和凋亡过程中具有重要作用。因此,miRNA-126-3p和miRNA-29b-3p可作为潜在的分子靶点用于AMoL发病机制的进一步研究。
[1]胡映歆,仇红霞.急性单核细胞白血病预后因素研究进展[J].国际输血及血液学杂志,2012,35(1):62-65.
[2]张静,秦铁军,秘营昌,等.急性单核细胞白血病的临床研究[J].临床血液学杂志,2010,23(9):536-539.
[3]程译帜,金洁.急性粒-单核及单核细胞白血病研究进展[J].国际输血及血液学杂志,2008,31(3):243-246.
[4]THOMSON D W,BRACKEN C P,GOODALL G J.Experimental strategies for microRNA targetidentification[J].Nucleic Acids Res,2011,39(16):6845-6853.
[5]何冬梅,吴红,高杨军,等.MicroRNA在白血病中的研究新进展[J].暨南大学学报(医学版),2011,32(4):362-368.
[6]王梓,李善妮,刘静.microRNA在急性淋巴细胞白血病中的作用[J].生命科学研究,2012,16(2):172-180.
[7]杨洋,王莉莉,于力.表观遗传调控在白血病发生中作用的研究进展-MicroRNA与白血病 [J].中国实验血液学杂志,2010,18(2):520-524.
[8]LIU H, WANG B, LIN J, et al. microRNA-29b: an emerging player in human cancer[J]. Asian Pac J Cancer Prev, 2014, 15(21):9059-9064.
[9]GONG J N, YU J, LIN H S, et al. The role, mechanism and potentially therapeutic application of microRNA-29 family in acute myeloid leukemia[J]. Cell Death Differ, 2014, 21(1): 100-112.
[10]FAVREAU A J, MCGLAUFLIN R E, DUARTE C W, et al.MiR-199b, a novel tumor suppressor miRNA in acute myeloid leukemia with prognostic implications [J]. Exp Hematol Oncol,2016, 5(1): 4.
[11]SHIBAYAMA Y, KONDO T, OHYA H, et al. Upregulation of microRNA-126-5p is associated with drug resistance to cytarabine and poor prognosis in AML patients[J]. Oncol Rep, 2015,33(5): 2176-2182.
[12]de LEEUW D C,DENKERS F,OLTHOF M C,et al.Attenuation of micro RNA-126 expression that drives CD34+38-stem/progenitor cells in acute myeloid leukemia leads to tumor eradication[J].Cancer Res,2014,74(7):2094-2105.
[13]HO T T, HE X, MO Y Y, et al. Transient resistance to DNA damaging agents is associated with expression of microRNAs-135band-196b in human leukemia cell lines[J]. Int J Biochem MolBiol, 2016, 7(2): 27-47.
[14]LI P, FAN J B, GAO Y, et al. miR-135b-5p inhibits LPS-induced TNF-α production via silencing AMPK phosphatase Ppm1e[J]. Oncotarget, 2016, 7(47): 77978-77986.
[15]TABE Y, YAMAMOTO S, SAITOH K, et al. Bone marrow adipocytes facilitate fatty acid oxidation activating AMPK and a transcriptional network supporting survival of acute monocytic leukemia cells[J]. Cancer Res, 2017, 77(6): 1453-1464.
(王荣兵 编辑)
Whole-genome analysis profile of MicroRNA in acute monocytic leukemia*
Xiao-cong Lin1,Ning Li2,Wei-yu Fu1,Liu-bo Lan1,Hai-tao Zhang1,Yu-ming Zhang2
(1.Institute of Biochemistry and Molecular Biology,Guangdong Medical University,Zhanjiang,Guangdong 524023,China;2.Department of Hematology,Affiliated Hospital of Guangdong Medical University,Zhanjiang,Guangdong 524001,China)
ObjectiveTo investigate the whole-genome MicroRNA (miRNA)profile in acute monocytic leukemia (AMoL).MethodsIllumina high-throughput sequencing technology was utilized to screen differentially expressed miRNA in bone marrow samples of 10 AMoL patients as well as 10 patients with non-malignant hematologic diseases.Stem-loop primer-based real-time quantitative polymerase chain reaction(Stem-loop qPCR)was carried out to validate the sequencing data.ResultsIn total of 285 differentially expressed miRNAs,199 up-regulated and 86 down-regulated miRNAs were identified in AMoL patients compared with the control group.Further stem-loop qPCR results confirmed similar expression of 6 miRNAs identified by sequencing.ConclusionsMiR-126-3p and miR-29b-3p can be potential targets for further study of AMoL.
acute monocytic leukemia;micro ribonucleic acid;sequencing
R394.3
A
10.3969/j.issn.1005-8982.2017.27.007
1005-8982(2017)27-0032-05
2017-05-21
广东省自然科学基金(No:2016A030313677);广东省医学科学技术研究基金(No:A2016193)
张宇明,E-mail:13670982222@163.com
