硒代谢网络与硒蛋白质组的生物信息学研究进展
2017-11-23张茂娜
张茂娜, 姜 亮, 张 焱
深圳大学生命与海洋科学学院, 广东 深圳 518060
硒代谢网络与硒蛋白质组的生物信息学研究进展
张茂娜, 姜 亮*, 张 焱*
深圳大学生命与海洋科学学院, 广东 深圳 518060
硒是大多数生物所必需的微量元素,对维持氧化还原稳态平衡具有重要作用,并与许多重大疾病有着密切联系。一直以来,关于硒的研究工作主要集中于硒代谢机制和硒蛋白功能。近年来快速增长的各类组学数据为硒相关的生物信息学研究工作提供了重要条件与机遇。主要介绍了当前利用生物信息学的理论和方法研究硒的代谢通路、功能和进化等领域的最新进展。通过这些研究,一方面发现了大量新的硒蛋白基因,并确定了众多物种的硒蛋白质组;另一方面揭示了新的硒代谢通路及相关新基因,完善了硒代谢网络。在此基础上,通过比较基因组学分析,深入探讨了硒代谢通路、不同硒蛋白家族乃至硒蛋白质组的分布与进化规律,以期为进一步认识硒研究领域中的重要问题和未来的发展方向提供支持。
硒蛋白;硒蛋白质组;硒代谢;生物信息学;比较基因组学
众多研究指出,硒是许多生物必需的微量营养元素。硒元素在体内发挥抗氧化作用,且和许多重大疾病都有着密切的联系,如肿瘤、糖尿病、心血管疾病和神经退行性疾病等[1]。此外,硒还参与了生物体的生长、发育等基本生命过程,在抗炎、抗病毒和抗衰老等方面都起着重要作用。硒主要以硒代半胱氨酸(selenocysteine,Sec,第21种氨基酸)的形式存在于体内,并进一步插入到硒蛋白(selenoprotein)中而发挥生物学作用[2,3]。在原核生物中,硒还能特异性地合成硒尿苷(selenouridine,SeU)和某种含钼酶中的硒辅因子(Se cofactor)[4]。此外,硒还参与合成硒代甲硫氨酸、硒糖等小分子含硒复合物。
近年来,随着高通量测序技术的蓬勃发展,各种基因组数据不断增加,因此需要开发新的数据分析方法,发现其中隐含的重要信息,于是,生物信息学作为一门新兴交叉学科得到了长足发展。通过各种计算分析,揭示基因的功能,完善代谢调控网络,为研究生物体的各种生命过程提供了重要启示与新的机遇。
本文将主要介绍生物信息学和比较基因组学在硒研究领域中的主要应用,深入阐述硒在不同物种(包括细菌、古菌和真核生物)中的利用、代谢、功能和进化等方面的最新研究进展,尤其是我国科研人员在这方面取得的重要成果,以期为硒代谢与硒蛋白的研究提供参考。
1 硒代半胱氨酸的合成机制
关于Sec的合成与插入硒蛋白的分子机制已基本被阐明,关键步骤是将终止密码子UGA重编码成Sec的密码子[4,5]。在细菌中,这个过程主要包括了编码Sec的UGA(Sec-UGA)、一个顺式作用元件(被称为Sec插入序列元件或SECIS元件)和一些相关酶[4~6]。SECIS元件位于硒蛋白mRNA中紧邻Sec-UGA的下游区域,是一种特殊的RNA茎环状结构。细菌中Sec的生物合成与插入过程如图1A所示。其中SECIS元件与Sec特异性延伸因子SelB结合,并和Sec特异性tRNA[Ser]Sec形成复合物。tRNA[Ser]Sec首先在丝氨酸-tRNA合成酶(SerS)的作用下结合丝氨酸,接着在Sec合成酶(SelA)作用下,以硒磷酸(硒磷酸合成酶SelD的产物)作为硒供体合成Sec-tRNA[Ser]Sec。而真核生物、古菌则与细菌不同,其SECIS元件存在于硒蛋白mRNA的3′非翻译区(3′-UTR)内,而且需要额外的步骤和酶,如激酶PSTK和SECIS结合蛋白SBP2,其基本过程如图1B所示[4 ]。
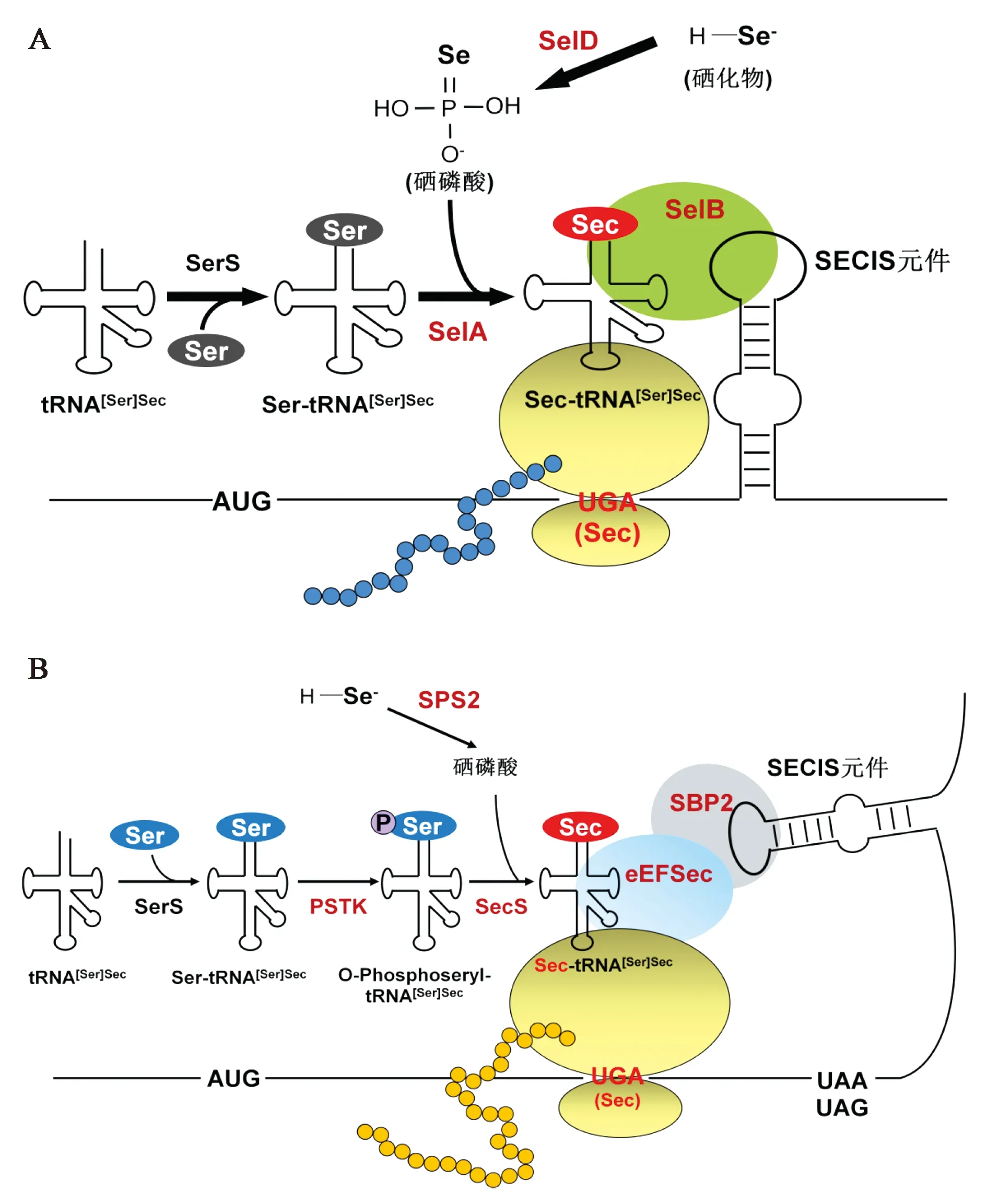
图1 硒代半胱氨酸的合成过程Fig.1 Procedure of selenocysteine biosynthesis.A:细菌;B:真核生物。
在硒的代谢与功能研究方面,基于SECIS元件的重要特征与Sec合成机制,一些生物信息学算法被开发出来,可以有效地预测各种物种基因组中的硒蛋白基因[7~9]。除此之外,鉴于大多数硒蛋白都能找到含有半胱氨酸(cysteine,Cys)的同源蛋白,不依赖于SECIS的硒蛋白预测算法亦被开发,可用来识别环境基因组数据库中的硒蛋白基因[10~12]。通过这些方法,发现了许多新的硒蛋白并得到了实验验证,如现已知的在人和小鼠中各含有25个和24个硒蛋白[13]。另外,一些硒代谢相关的新基因也陆续被发现,从而进一步完善了原核生物和真核生物的硒代谢调控网络。
2 硒蛋白基因识别的生物信息学研究
2.1细菌硒蛋白基因的预测和硒蛋白质组研究
如前所述,在所有硒蛋白基因中,SECIS元件对于Sec插入蛋白质的过程必不可少且具有高度特异性。基于SECIS元件来预测细菌硒蛋白基因本应是一个较为合理的策略,但与真核生物不同,细菌SECIS元件缺乏非常保守的一级序列和二级结构特征,因此长期以来,在预测细菌硒蛋白基因方面缺乏有效的工具。Zhang等[7]通过分析已知细菌硒蛋白基因中各种SECIS元件的组成和结构特征,首次提出了一个细菌SECIS元件的结构模型。在该模型中,SECIS的一级序列并不非常保守,仅发现在SECIS元件顶环的前两个核苷酸中总是存在一个鸟苷酸(G),其后面常跟着一个尿苷酸(U)。此外,Sec-UGA密码子与SECIS元件顶环之间的距离在16~37个核苷酸之间。根据这个模型,Zhang等[7]开发了第一个可用于预测细菌基因组中硒蛋白基因的bSECISearch算法。该算法流程如图2所示,主要原理是通过检查细菌基因组中所有UGA密码子下游是否含有一个类似SECIS元件的结构来预测硒蛋白基因。该算法还运用了一些其他标准,包括开放阅读框约束、基于片段化的SECIS模型打分以及UGA密码子两侧的蛋白序列保守性等。候选硒蛋白基因将进一步分析其UGA密码子的位置、是否具有含Sec或Cys的同源序列,以及包含Sec的直系同源基因中是否也存在SECIS元件等。最后将对确定的硒蛋白基因进行更深入的验证。
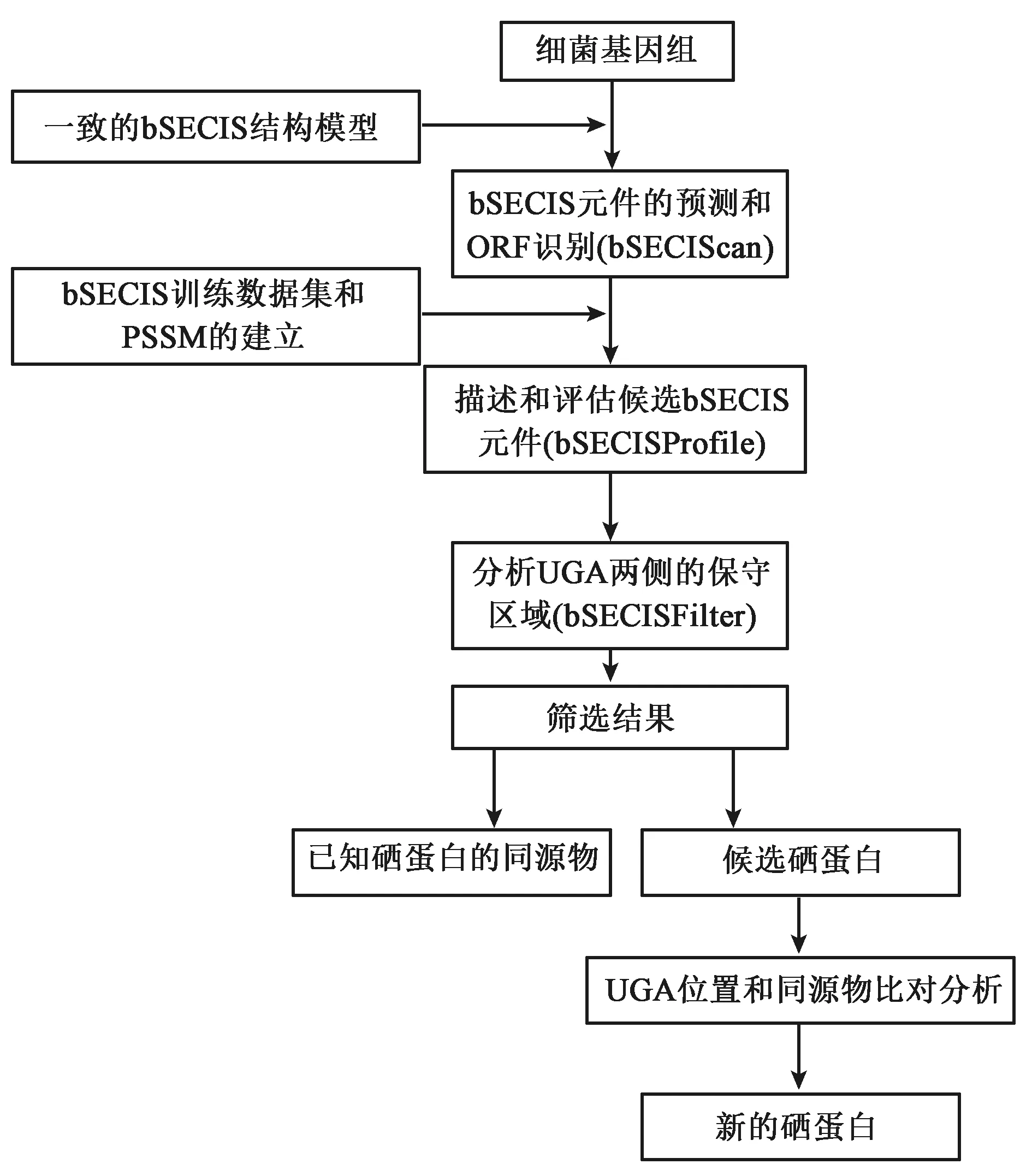
图2 bSECISearch算法流程图Fig.2 A schematic diagram of the bSECISearch algorithm.
除了上述依赖于SECIS的预测方法外,Zhang等[10~12]发现几乎所有的硒蛋白都存在含Cys的同源序列(即Sec的位置被Cys替换),于是又开发出不依赖于SECIS的硒蛋白基因预测方法,主要用于搜索各种环境基因组中的硒蛋白基因。该方法主要利用大型蛋白质数据库(如NCBI的非冗余蛋白序列数据库)中含Cys的蛋白来搜索基因组数据库中潜在编码硒蛋白的核酸序列。该方法的关键步骤在于寻找可能包含Sec-UGA密码子的核酸片段。这些序列经翻译后成为含Cys蛋白的同源序列,保守的Cys残基能与UGA密码子的翻译产物相匹配,且该配对的两侧都比较保守。
利用上述两种方法,在不同细菌基因组和环境基因组中发现了许多新的硒蛋白基因。这些新发现的硒蛋白及其相关信息已经成功整合入目前最大的微量元素相关蛋白数据库dbTEU[14]和国际著名的重编码数据库RECODE[15]。这些生物信息学工具和相关的研究成果为后续开展各种细菌硒蛋白质组的比较基因组学研究提供了重要工具和丰富资源。
Zhang等[16]的一个早期工作对近350个完全测序的细菌基因组进行了分析,确定了所有利用Sec的物种及其硒蛋白质组的组成。该研究发现只有不到四分之一的细菌可以使用Sec,分别含有1~31个硒蛋白基因。富含硒蛋白的生物(含有6个或更多的硒蛋白基因)主要是δ变形菌纲(Deltaproteobacteria)和厚壁菌门/梭状芽孢杆菌属(Firmicutes/Clostridia)。甲酸脱氢酶α亚基(formate dehydrogenase α subunit,FdhA)和SelD是细菌中分布最广泛的硒蛋白家族。大多数硒蛋白似乎都是从含有Cys的祖先蛋白进化而来。然而,在富含硒蛋白物种的许多近缘物种中都发生了特定硒蛋白基因丢失的现象。这些结果表明,Sec的获得和硒蛋白基因丢失之间存在高度动态且微妙的进化平衡,这可部分解释Sec的催化优势与其在自然界中仅被有限使用之间的矛盾。此外,一些环境因素(如氧浓度或温度)可能会影响Sec利用和硒蛋白质组的进化[16]。
另一个比较基因组学研究针对原核生物和真核生物中Sec的利用情况开展了分析[17]。该研究详细分析了近600种细菌和古菌的基因组,发现已测序细菌的硒蛋白质组包含1~39个硒蛋白。但是,迄今为止最大的原核生物硒蛋白质组在一个环境基因组研究中被报道。Zhang等[11]研究发现,某种海洋寡毛纲小蠕虫Olaviusalgarvensis的寄生菌(属于δ变形菌纲)含有60个以上的硒蛋白基因。
最近,Peng等[18]分析了5 200多个细菌的基因组数据。该研究为所有已知的硒代谢途径(包括Sec生物合成和其他硒利用途径)绘制了迄今为止最大的物种分布图谱,展示了细菌中最为详细的硒生物利用的相关信息。和以往的研究结果一致,绝大多数(约80%)测序物种失去了编码Sec的能力。通过对物种硒蛋白质组进行深入分析,发现了一个新的硒蛋白利用非常活跃的门属—互养菌门(Synergistetes)以及一些富含硒蛋白的其他物种。通过对环境因素与Sec利用之间关系的扩展研究,发现了细菌Sec生物利用的新宏观进化特征。在今后的研究工作中,寻找可以影响不同硒蛋白家族进化的其他因素将显得非常重要。
2.2真核生物硒蛋白基因的预测和硒蛋白质组研究
伴随着生物信息学的快速发展,真核生物硒蛋白识别工作取得了很大进展,如一些脊椎动物和哺乳动物(包括人和小鼠)、昆虫、线虫和部分藻类等的硒蛋白被陆续报道。但是当前硒蛋白在动物界中的认识还比较片面,仅局限于某些特定的物种进化分支中。此外,虽然已有的真核生物硒蛋白基因预测算法(如SECISearch[8])被较为广泛地使用,但是其本身固有的缺陷(如需预先建立物种特异性训练集来构建SECIS模型以及针对某些真核生物预测的假阳性较多等)亦限制了这些方法的使用。
为了解决算法普适性的难题,并提高预测准确性,Jiang等[9,19]开发了一个新的硒蛋白基因识别算法。该算法侧重于寻找基因组中所有可能编码Sec的UGA密码子和包含Sec-UGA的开放阅读框。由于真核生物硒蛋白基因结构的特殊性,必须设计特殊的基因组装算法,为此Jiang等[19]开发了新的适合于硒蛋白基因识别的组装算法SelGenAmic。该算法的基本流程如图3。首先从基因组中获得所有可能的外显子,其中包括含有UGA密码子的外显子(称为i-exon)和不含UGA密码子的外显子(称为c-exon),接着利用硒蛋白基因组装算法,进一步得到包含i-exon的特殊基因i-gene。之后进一步分析基因下游的SECIS结构信息和UGA翻译后的Sec残基两侧局部序列的保守性,最后对硒蛋白识别结果进行进一步分析,确定物种的硒蛋白集合。
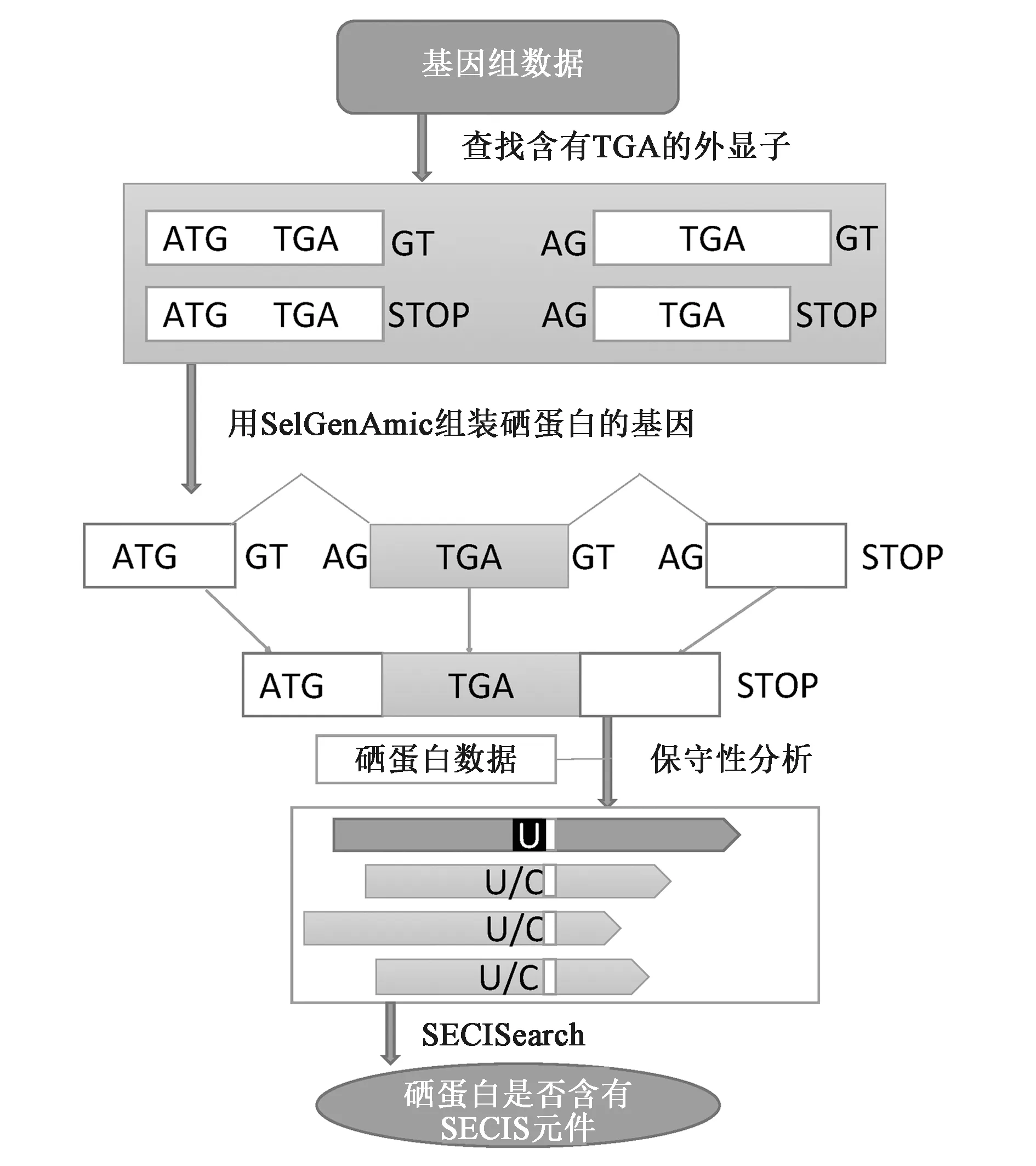
图3 真核生物硒蛋白预测流程图Fig.3 A schematic diagram of eukaryotic selenoprotein prediction.
为了进一步认识动物界,尤其是无脊椎动物中硒蛋白的分布与进化,Jiang等[9,19]利用该算法,选择了6种代表性无脊椎动物——海绵、丝盘虫、海葵、青螺、柱头虫、文昌鱼和玻璃海鞘,结合目前已知的脊椎动物基因组数据,确定了这些物种中的硒蛋白基因,并深入探讨了多细胞动物从诞生、不断进化到出现人类的整个演变过程中硒蛋白家族与硒蛋白质组的变化。其中在玻璃海鞘中,首次发现了真核生物的DsbA硒蛋白,特殊的含双SECIS结构和双Sec结构的SELENOW等硒蛋白[9]。DsbA以前被认为只存在于细菌中,而该研究说明该硒蛋白也可存在于一些海洋无脊椎动物中。此外,多细胞动物具有丰富的硒蛋白种类,并且在主要的进化分支中,硒蛋白基因并没有发生大规模丢失。只是在某些特定的进化分支(如昆虫和线虫)中,由于未知原因导致硒蛋白基因发生了大面积丢失。此外,还发现目前硒蛋白数量和种类最丰富的动物是文昌鱼,并在文昌鱼中找到了一种特殊的硒储存和转运蛋白3NSelP。该硒蛋白的发现,对揭示硒蛋白SELENOP的进化有着重要的意义,也为探索生物有效储存和转运硒的机制提供了重要信息。
Jiang等[20]还对代表性扁形动物(涡虫、血吸虫和猪肉绦虫)的硒蛋白质组进行了分析,研究寄生与自生生活方式对硒蛋白质组的影响。这些扁形动物进化关系较为接近,但生活方式上却存在巨大差异。涡虫是一种在淡水中自由生存的物种,而血吸虫和猪肉绦虫都是对人类和家畜具有较大危害的寄生物种。3个物种的生活方式存在着明显的梯度差异,即从涡虫到血吸虫再到猪肉绦虫,寄生程度越来越高。结果显示,3个物种的硒蛋白质组随着寄生程度的升高而逐渐减小。涡虫中硒蛋白数量最多,而寄生程度最高的猪肉绦虫中硒蛋白数量最少。由此推断,寄生的生活方式可能会显著降低动物对硒的依赖性。
此外,Gobler等[21]在分析有害藻类抑食金球藻(Aureococcusanophagefferens)的基因组时,发现该藻含有目前已知最大的真核生物硒蛋白质组(至少60个硒蛋白基因)。Lobanov等[22]研究发现,脊椎动物、绿藻及一些单细胞生物和纤毛虫等真核生物都富含硒蛋白。在真核生物中,硒蛋白K分布最为广泛,但功能尚不清楚。一些其他硒蛋白分布也比较广,如硒蛋白W、硫氧还蛋白还原酶(TXNRD)和谷胱甘肽过氧化物酶(GPX)等。与细菌比较,可能是由于真核生物的硒蛋白合成过程更为复杂,因此真核生物硒蛋白家族中较少出现Sec和Cys之间的转换[4, 22]。
3 硒代谢调控网络的生物信息学研究
硒主要以Sec的形式出现在蛋白质中发挥功能。此外,在某些细菌中发现,硒还可特异性地合成SeU(存在于某些tRNA的摆动位点)以及硒辅因子(存在于某些含钼酶中)。每一条硒代谢通路都拥有特异的基因群[4]。SelD是参与这些不同硒代谢过程的共有酶也是关键酶,理论上存在于所有可利用硒的物种中。SelD属于一个ATP结合蛋白超家族,其主要功能是催化ATP与硒化物生成硒磷酸,而硒磷酸是合成Sec、SeU和Se cofactor等重要有机硒化合物所必需的硒供体。因此,通过分析SelD的分布,有助于发现新的利用硒的物种和硒代谢途径。
在硒辅因子通路被发现以前,Sec和SeU是仅有的2种硒代谢途径。Zhang等[23]利用比较基因组学和系统发生分析研究了已知硒代谢相关基因在测序细菌中的分布情况,首次发现了一些物种含有SelD、但不含Sec或SeU的代谢通路,提示这些物种中可能存在依赖于SelD的其他硒利用途径。通过进一步分析SelD基因在这些物种基因组中的位置和上下游基因结构,发现了2种新的与SelD关系非常密切的基因(YqeB和YqeC),并且发现这3种基因(SelD、YqeB和YqeC)和一种含钼的氧化还原酶联系紧密。进一步实验表明,这些物种中含有某种不稳定的硒化物,在还原剂处理下可发生分离。Zhang等[23]推测第3种硒代谢通路是某种不太稳定的硒辅因子,可被某含钼酶利用,该通路的标志性基因是YqeB和YqeC。在最近的研究工作中,Peng等[18]发现可能存在第4条依赖于SelD的硒代谢通路。
另外,Li等[24]首次在古菌中发现了某种SelD远缘同源基因(称为SelD-like)的存在。这些SelD-like基因主要存在于泉古菌门中的两个主要目(硫化叶菌目和热变形菌目)中。SelD-like蛋白含有与SelD相同的功能结构域,因此可能具有相似的催化反应机制。SelD-like基因在物种中的分布较为局限(仅存在于部分不能利用硒的古菌中),说明该基因很可能是从某古菌的SelD基因新进化而来,并衍生出与硒无关的新功能。通过对SelD-like基因在古菌基因组中的位置与上下游结构分析,发现了它和一些硫代谢基因高度相关,包括过氧化物氧化还原酶、硫氰酸酶相关硫转移酶、异化亚硫酸还原酶以及Fe-S氧化还原酶等,提示SelD-like蛋白很可能参与这些嗜热古菌的硫代谢,并提出了SelD-like参与硫代谢的两个新模型,从而揭示了硒和硫代谢过程之间的重要关系。
在过去的20年中,虽然参与细菌硒代谢的一些关键基因被陆续报道,但是关于整个硒代谢与调控网络尚有诸多不清楚的地方。最近,Lin等[25]通过比较基因组学和系统发生分析方法,结合所有已知硒代谢通路和相关基因的信息,以期寻找参与硒代谢的新关键基因。通过深入分析各种已知基因的基因组上下游结构,发现了若干可能参与硒代谢调控网络的新基因,包括①YedE:1个与硫转运蛋白具有远缘同源性的膜蛋白,仅在可利用硒的物种中发现,其功能很可能参与硒的跨膜运输;②YedF:含有氧化还原相关的结构域,可能与硒的解毒过程相关;③1个属于LysR超家族的硒特异性转录因子LysR_Se,可能参与Sec生物合成以及其他硒相关基因的表达调控;④1个很小的蛋白家族DUF3343,广泛分布于硒利用物种中,很可能作为一个重要的细胞内硒转运伴侣。最后,Lin等[25]提出了一个硒代谢与稳态调控模型,该成果为进一步完善硒代谢与调控的复杂网络提供了重要信息。
最近,Li等[26]利用果蝇SEPHS2(SelD在真核生物中的同源基因)的基因敲减模型,通过对幼虫与成虫期转录组测序,深入分析了该基因可能影响的代谢通路和相关基因。该研究发现,SEPHS2可以影响许多基因的表达,这些基因多与糖代谢和细胞氧化还原进程密切相关。进一步通过聚类和网络分析,找到与SEPHS2具有强功能关联的新关键基因(包括Nimrod C1和钙调素等),为进一步研究SEPHS2的复杂功能和硒代谢与其他代谢通路之间的关系提供了新线索。
4 硒生物利用的进化规律研究
收集各种物种基因组和环境基因组数据(包括基因组和其他相关信息,如生存环境等),并进一步确定硒代谢通路、不同硒蛋白家族和硒蛋白质组的分布情况,可为深入认识硒生物利用的复杂进化规律及其与环境因素的关系提供重要信息。近年来已陆续开展了若干硒代谢与功能相关的进化研究,并从中发现了一些重要的进化特征。
Zhang等[12]分析了最大的海洋环境基因组计划(Global Ocean Sampling, GOS)项目。通过分析每一个GOS样本(主要是海水样本)的宏基因组数据,共找到了约3 600个硒蛋白基因,这些基因来自于58个硒蛋白家族,并且绝大多数为氧化还原酶。除此之外,还发现了若干新的硒蛋白家族。在已研究过的GOS样本中,SelW和SelD是分布最广泛的硒蛋白。样本所处的不同地理位置对于硒蛋白生物利用的影响很小。然而,温度升高和海水环境(与淡水和高盐度水样相比)可以促进Sec的利用,并且增加了硒蛋白的种类和数量。在海洋环境中,亦发现了一些新的硒蛋白融合基因,往往都具有氧化还原活性。此外,海洋微生物群体中SeU和Sec的生物利用具有不同的进化规律。这些结果首次揭示了不同海洋环境对于硒代谢途径和硒蛋白质组的复杂影响。
在最近Peng等[18]开展的细菌比较基因组学研究中,发现了细菌的栖息环境可以影响不同硒的代谢通路。Sec和硒辅因子通路多见于宿主相关的生存环境,而SeU通路多见于水生生物。氧气和Sec代谢通路的关系并不显著,但低氧环境却可促进硒蛋白的产生,因为大部分富含硒蛋白的物种都是厌氧菌。硒辅因子通路非常偏爱厌氧环境;反之,SeU通路则多见于需氧型物种。这些成果为揭示不同生态环境中微生物对硒利用过程的进化趋势提供了重要依据。
另一个比较基因组学研究确定了44种已测序脊椎动物的硒蛋白质组,共发现了45个硒蛋白家族,其中在哺乳动物中发现28个,在硬骨鱼中发现41个[27]。该研究定义了脊椎动物祖先(28个硒蛋白)和哺乳动物(25个硒蛋白)的硒蛋白质组,并仔细描述了它们是如何通过基因重复(20个事件)、基因丢失(10个事件)和Sec被Cys替代(12个事件)等进化事件而逐渐演变的。此外,还发现了无内含子的SEPHS2基因在早期哺乳动物中产生,并在功能上取代了胎盘类哺乳动物中原始的多外显子基因,然而这2个基因仍然保留在有袋类动物中。哺乳动物的TXNRD1和TXNRD3由一个含有谷氧还蛋白结构域的祖先酶进化而来。SELENOV和GPX6分别从胎盘类哺乳动物的SELENOW和GPX3的基因复制演变而来,GPX6则发生了数次独立地丢失Sec的现象。在硬骨鱼类中,还特征性地发生了一些硒蛋白家族(GPX1、GPX3、GPX4、DIO3、MSRB1、SELENOJ、SELENOO、SELENOT、SELlU1和SELENOW2)的复制。该研究同时确定了若干硒蛋白的新异构体,并报道了异常保守的硒蛋白假基因。该研究是首次对脊椎动物和哺乳动物硒蛋白质组展开较为完整的探索,并着重描述了它们的复杂进化过程,为深入认识这些硒蛋白及其具体形式提供了大量有价值的信息。
5 展望
硒是许多生物所必需的微量元素,其复杂的代谢调控机制与功能一直受到广泛的关注。本文主要讨论了如何利用生物信息学的理论和方法来深入研究硒的生物利用过程及其进化规律,相关的研究成果可以帮助研究人员进一步认识硒在不同生物中利用与代谢过程的共性和个性,深入理解硒蛋白的主要功能和物种硒蛋白质组的组成与进化特征。但应承认,这些研究工作仅仅是个开端。硒在不同生物体中的代谢机制和功能要比此前所认识的更为复杂多样,还有许多未知的问题有待于进一步研究。未来,随着以高通量测序为代表的各种先进技术的普遍应用、各类组学数据(基因组、转录组、蛋白质组、离子组等)急剧增加,将更有利于研究人员运用生物信息学和系统生物学的方法对硒相关的各种生物学问题进行深入研究,为该领域乃至整个微量元素研究领域开拓新的视野并提供重要的研究手段。
[1] Rayman M P. The importance of selenium to human health[J]. Lancet, 2000, 356(9225): 233-241.
[2] Low S C, Berry M J. Knowing when not to stop: Selenocysteine incorporation in eukaryotes[J].Trends Biochem. Sci., 1996, 21(6):203-208.
[3] Stadtman T C. Selenocysteine[J]. Ann. Rev. Biochem., 1996, 65(1): 83-100.
[4] Zhang Y, Gladyshev V N. Comparative genomics of trace elements: Emerging dynamic view of trace element utilization and function[J]. Chem. Rev., 2009, 109(10):4828-4861.
[5] Böck A, Forchhammer K, Heider J,etal.. Selenocysteine: The 21st amino acid[J]. Mol. Microbiol., 1991, 5(3):515-520.
[6] Hatfield D L, Gladyshev V N. How selenium has altered our understanding of the genetic code[J]. Mol. Cell. Biol., 2002, 22(11): 3565-3576.
[7] Zhang Y, Gladyshev V N. An algorithm for identification of bacterial selenocysteine insertion sequence elements and selenoprotein genes[J]. Bioinformatics, 2005, 21(11):2580-2589.
[8] Kryukov G V, Kryukov V M, Gladyshev V N. New mammalian selenocysteine-containing proteins identified with an algorithm that searches for selenocysteine insertion sequence elements[J]. J. Biol. Chem., 1999, 274 (48): 33888-33897.
[9] Jiang L, Liu Q, Ni J. In silico identification of the sea squirt selenoproteome[J]. BMC Genomics, 2010, 11: 289.
[10] Zhang Y, Fomenko D E, Gladyshev V N. The microbial selenoproteome of the Sargasso Sea[J]. Genome Biol., 2005, 6(4): R37.
[11] Zhang Y, Gladyshev V N. High content of proteins containing 21st and 22nd amino acids, selenocysteine and pyrrolysine, in a symbiotic deltaproteobacterium of gutless wormOlaviusalgarvensis[J]. Nucl. Acids Res., 2007, 35(15): 4952-4963.
[12] Zhang Y, Gladyshev V N. Trends in selenium utilization in marine microbial world revealed through the analysis of the global ocean sampling (GOS) project[J]. PLoS Genet., 2008, 4(6): e1000095.
[13] Kryukov G V, Castellano S, Novoselov S V,etal.. Characterization of mammalian selenoproteomes[J]. Science, 2003, 300(5624): 1439-1443.
[14] Zhang Y, Gladyshev V N. dbTEU: A protein database of trace element utilization[J]. Bioinformatics, 2010, 26(5): 700-702.
[15] Bekaert M, Firth A E, Zhang Y,etal.. Recode-2: New design, new search tools, and many more genes[J]. Nucl. Acids Res., 2010, 38(S1): D69-D74.
[16] Zhang Y, Romero H, Salinas G,etal.. Dynamic evolution of selenocysteine utilization in bacteria: A balance between selenoprotein loss and evolution of selenocysteine from redox active cysteine residues[J]. Genome Biol., 2006, 7(10): R94.
[17] Zhang Y, Gladyshev V N. General trends in trace element utilization revealed by comparative genomic analyses of Co, Cu, Mo, Ni, and Se[J]. J. Biol. Chem., 2010, 285(5): 3393-3405.
[18] Peng T, Lin J, Xu Y Z, Zhang Y. Comparative genomics reveals new evolutionary and ecological patterns of selenium utilization in bacteria[J]. ISME J., 2016, 10(8): 2048-2059.
[19] Jiang L, Ni J, Liu Q. Evolution of selenoproteins in the metazoan[J]. BMC Genomics, 2012, 13: 446.
[20] Jiang L, Zhu H Z, Xu Y Z,etal.. Comparative selenoproteome analysis reveals a reduced utilization of selenium in parasitic platyhelminthes[J]. Peer J., 2013, 1: e202.
[21] Gobler C J, Berry D L, Dyhrman S T,etal.. Niche of harmful algaAureococcusanophagefferensrevealed through ecogenomics[J]. Proc. Natl. Acad. Sci. USA, 2011, 108(11): 4352-4357.
[22] Lobanov A V, Fomenko D E, Zhang Y,etal.. Evolutionary dynamics of eukaryotic selenoproteomes: Large selenoproteomes may associate with aquatic life and small with terrestrial life[J]. Genome Biol., 2007, 8(9): R198.
[23] Zhang Y, Turanov A A, Hatfield D L,etal.. In silico identification of genes involved in selenium metabolism: Evidence for a third selenium utilization trait[J]. BMC Genomics, 2008, 9: 251.
[24] Li G P, Jiang L, Ni J Z,etal.. Computational identification of a new SelD-like family that may participate in sulfur metabolism in hyperthermophilic sulfur-reducing archaea[J]. BMC Genomics, 2014, 15: 908.
[25] Lin J, Peng T, Jiang L,etal.. Comparative genomics reveals new candidate genes involved in selenium metabolism in prokaryotes[J]. Genome Biol. Evol., 2015, 7(3): 664-676.
[26] Li G, Liu L, Li P,etal.. Gene expression profiling of selenophosphate synthetase 2 knockdown inDrosophilamelanogaster[J]. Metallomics, 2016, 8(3): 354-365.
[27] Mariotti M, Ridge P G, Zhang Y,etal.. Composition and evolution of the vertebrate and mammalian selenoproteomes[J]. PLoS ONE, 2012, 7(3): e33066.
张焱教授团队介绍
张焱教授及其研究团队长期致力于硒代谢与功能的生物信息学研究。通过开发新算法,在原核和真核生物中发现了大量新的硒蛋白,找到了新的硒代谢通路和新关键基因,并揭示了硒代谢网络与硒蛋白质组的重要进化规律。该团队已发表高水平论文70余篇(总影响因子>370),参与近10部中英文著作的撰写,主持多项国家自然科学基金、中国科学院和省部级项目。
AdvanceonBioinformaticAnalysisofSeleniumMetabolicNetworkandSelenoproteomes
ZHANG Maona, JIANG Liang*, ZHANG Yan*
CollegeofLifeSciences&Oceanography,ShenzhenUniversity,GuangdongShenzhen518060,China
Selenium (Se) is an essential trace element for most organisms and it plays an important role in redox homeostasis and is associated with several major diseases. Previously, much efforts have been devoted to experimental investigation of Se metabolism and functions of different selenoproteins. However, with the rapid increase of omics data in recent years, bioinformatic studies have become more and more important in the research of Se. This review introduced recent advances in investigating the metabolism, functions and evolution of Se by bioinformatic theories and methods. These studies identified a large number of new selenoprotein genes as well as the selenoproteomes in a variety of organisms. On the other hand, new Se metabolic pathways and several new genes involved in Se metabolism and homeostasis were also identified, which greatly improved our understanding of Se metabolic network. Furthermore, comparative genomic studies were carried out, which revealed the general distributions of Se metabolic pathways, selenoprotein families and selenoproteomes as well as their evolutionary patterns. It was expected to provide a foundation for investigating the fundamental questions and future directions of Se research.
selenoprotein; selenoproteome; selenium metabolism; bioinformatics; comparative genomics
2017-07-31;接受日期2017-08-15
国家自然科学基金项目(31771407;31401129);广东省自然科学基金项目(2015A030313555);广东省教育厅科技创新项目(2014KTSCX109)资助。
张茂娜,硕士研究生,主要从事硒蛋白的生物信息学研究。E-mail: 294516606@qq.com。*通信作者:姜 亮,副研究员,主要从事硒蛋白的生物信息学研究。E-mail: jiangliang@szu.edu.cn;张 焱,教授,主要从事微量元素的生物信息学和系统生物学研究。E-mail: zhangyan@szu.edu.cn
10.19586/j.2095-2341.2017.0097

