高效耐盐柴油降解菌的筛选、鉴定及降解基因
2017-11-23胡春辉赵阳国田伟君
胡春辉,于 浩,赵阳国,田伟君,白 洁*
高效耐盐柴油降解菌的筛选、鉴定及降解基因
胡春辉1,2,于 浩2,赵阳国1,田伟君1,白 洁1*
(1.中国海洋大学环境科学与工程学院海洋环境与生态教育部重点实验室,山东青岛266100;2.青岛农业大学生命科学学院,山东青岛266109)
在辽河口湿地油田石油污染土壤中分离到一株耐盐柴油高效降解菌株L7,经过形态学观察、16S rRNA基因序列分析鉴定菌株L7属于不动杆菌属().该菌株能够以柴油、十五烷、十六烷、十七烷、十八烷、十九烷、二十烷和二十二烷为唯一碳源和能源生长.在柴油无机盐培养基中,菌株的最适生长温度为30°C,最适pH值为7.0,菌株L7在pH 6.0~9.0,盐度范围为3%以内都可以生长.与其他的菌株相比较,菌株L7在柴油中生长更加迅速,在最适培养条件下培养4d后菌株的OD600nm就可以达到4.0,通过紫外分光光度法测定培养5d后,柴油的降解效率为61.5%.通过对菌株L7的全基因组测序和分析,在基因组上找到一个烷烃羟化酶基因,将基因克隆到pME6032质粒上,并电转至不依赖柴油生长的恶臭假单胞菌KT2440中,含有该质粒的KT2440菌株能够在柴油无机盐培养基中生长.GC-MS检测确定了菌株L7及其基因对饱和烷烃的降解能力.推测菌株L7中烷烃降解途径为末端氧化途径,是由烷烃羟化酶催化反应生成相应的醇,最终通过β-氧化反应实现完全矿化.
不动杆菌;柴油;烷烃羟化酶;生物降解
柴油的组分以中长链的烷烃为主,是石油中的重要组分,每年有大量的柴油通过石油勘探、生产和运输过程被泄漏到海洋环境中,对海洋生物和海洋环境造成了巨大破坏[1].目前处理柴油污染物的方法有物理法、化学法和生物法,物理法只是将污染物转移,化学法可能会引入新的污染物,生物修复是彻底清除环境中柴油污染物的有效方法.微生物降解菌在柴油污染的生物修复中扮演重要角色[2].环境中存在大量的柴油降解菌株,当环境中柴油含量突然升高时这些菌株会迅速变成该区域的优势菌株[3‑5].已报道的柴油降解菌株分布于不动杆菌属()、假单胞菌属()、诺卡氏菌属(、分支杆菌属()、节杆菌属()、无色杆菌属()、芽孢杆菌属()、微球菌属()、弧菌属()等.不同微生物降解的柴油成分也不尽相同,降解机理差异较大[6].低温高盐条件下石油类有机污染物的降解过程会发生较大的变化.因此研究低温高盐石油降解菌对于生物修复过程有重要的指导意义[7].
不同的柴油降解菌株中烷烃代谢途径差异较大,代谢的烷烃种类也不尽相同[8].微生物中烷烃代谢途径通常是先将烷烃进行羟化变成相应的醇,进一步氧化成酸,最终进入脂肪酸途径或三羧酸(TCA)循环中彻底分解.其中烷烃羟化酶是烷烃代谢途径中的关键酶,决定烷烃代谢的效率和途径[9],同时这些烷烃羟化酶基因可用于探索和寻找环境中的烷烃降解菌株[10].
本文从辽河口湿地石油污染土壤中筛选出一株耐盐高效柴油降解菌株,对菌株进行鉴定和降解性能研究,确定其最适降解条件,同时对其全基因组进行测序,在其基因组上找到一个烷烃羟化酶基因,通过分子生物学技术确定该基因的功能,以期为高盐环境中柴油污染修复技术提供菌株资源和理论研究基础.
1 材料与方法
1.1 样品采集和培养基配制
样品来源:样品采自辽河口湿地油田石油污染土壤.
无机盐(BSN)液体培养基:KH2PO40.5g/L, K2HPO4·3H2O 5.24g/L,KNO31g/L,NaSO40.2g/L, 10%MgCl2·6H2O 2mL,1%CaCl22mL,1%FeSO40.2mL,维生素混合液0.2mL,金属离子混合液(1L的0.1mmol/L盐酸中溶解0.05g CaCl2·2H2O, 0.05g CuCl2·2H2O,0.008g MnSO4·H2O,0.04g FeSO4·7H2O, 0.05g ZnSO4,0.1g Na2MoO4·2H2O, 0.05g Na2WO4·2H2O,0.038g CoCl2·6H2O,0.02g MnCl2·4H2O, 0.0124g H3BO3)5mL,调节pH值为7.0,121℃高压蒸汽灭菌20min.
柴油培养基:无机盐培养基中加入1%的柴油,柴油采用0.22μm滤膜过滤除菌.
饱和烷烃培养基:无机盐培养基中加入0.2% (:)饱和烷烃.
LB液体培养基:蛋白胨10g/L,酵母粉5g/L,氯化钠10g/L,pH7.0,121℃高压蒸汽灭菌20min.
LB固体培养基即为LB液体培养基中添加1.5%的琼脂.
1.2 柴油降解菌株的分离纯化
称取5g土壤样品放入到已灭菌的无机盐培养基中,加入1%的柴油置于170r/min、30℃恒温振荡摇床中培养5d后,取上清液5mL加入到新的柴油培养基中继续培养,重复该过程4~5次后,取上清液1mL依次稀释至10-5,10-6,10-7涂布于LB固体培养基平板上.置于30℃恒温培养箱中培养,挑取单菌落接种到新的柴油培养基中,挑选生长较好的菌株进行后续实验.
1.3 菌株的鉴定
选取一株生长最好的菌株命名为L7,利用基因组提取试剂盒提取菌株的总DNA,以其为模板,用细菌16S rRNA基因通用引物27F和1492R(引物序列见表1)扩增其16S rRNA基因.PCR反应体系:基因组DNA 0.5μL, 2×Pfu Mix 12.5μL,引物27F 1μL,引物1492R 1μL,用ddH2O补足至25μL.PCR反应条件为:94℃预变性5min, 94℃变性1min,55℃退火1min,72℃延伸2min,共30个循环;最后72℃延伸5min,4℃保温.将PCR产物经琼脂糖凝胶电泳检测条带单一、大小正确后,送至上海生工生物工程股份有限公司测序,测序结果递交至NCBI数据库进行比对,选取同源性较高的序列利用MEGA6.0软件,采用Neighbor Joining法构建系统发育进化树[11].
1.4 菌株的最适生长条件优化和降解效率测定
微生物对柴油的降解效率受温度、pH值、盐浓度等的影响.将菌株L7以1%的接种量接种到柴油无机盐培养基中,分别置于25,30,37℃摇床中培养,摇床转速为170r/min,每隔12h取样1mL,12000r/min离心1min,用蒸馏水重悬后,利用分光光度计测定波长在600nm处的吸光度值(OD600nm)确定微生物的生长量.将菌株L7以1%的接种量接种到不同初始pH值的柴油无机盐培养基(5.0、6.0、7.0、8.0、9.0)中,置于170r/min,30℃摇床中培养,每隔12h取样测OD600nm处的吸收值绘制生长曲线.将菌株L7以1%的接种量接种到不同盐浓度的柴油培养基中(氯化钠浓度依次为0%、1%、2%、3%、5%),置于170r/min,30℃摇床中培养,每隔12h取样测定生长情况并绘制生长曲线.
确定菌株的最适生长温度、pH值和盐浓度后,在最适生长条件下培养菌株L7,培养至稳定期后,每个实验做3组平行.以不接菌的培养基作为空白对照,取样并测定柴油降解效率.
通过测定不同柴油浓度的紫外吸收值,绘制标准曲线,根据标准曲线拟合得到方程:
=0.01414-0.0446,2=0.99318 (1)
式一中:为柴油浓度,为OD225nm处的吸收值.
柴油降解率的计算公式为:
=(0-1)/0×100% (2)
式二中:0为对照组柴油浓度,μL/L;1为实验组柴油浓度,μL/L.
1.5 菌株L7的底物谱测定
柴油是一种成分复杂的混合物,主要组成成分是C10~C22的饱和烷烃.而每个菌株降解的柴油成分差异较大,因此将菌株L7分别接种到正己烷、正癸烷、十一烷、十二烷、十三烷、十四烷、十五烷、十六烷、十七烷、十八烷、十九烷、二十烷、二十一烷、二十二烷的烷烃培养基中,观察其生长情况,对于能够生长的培养基每隔12h取样测定OD600nm,绘制生长曲线.
1.6 基因组测序和烷烃羟化酶基因分析
利用上海生工生物基因组提取试剂盒提取菌株L7的全基因组DNA,送至上海锐翌生物科技有限公司,采用Illumina Hi-Seq4000测序技术对菌株进行全基因组测序,将获得的基因组序列提交到Rapid Annotation using Subsystem Technology(RAST)网站上进行自动注释,获得L7全基因组的注释信息.将注释得到的烷烃羟化酶基因跟已有的烷烃羟化酶进行Blast同源性分析,利用ClustalX软件对蛋白序列进行比对.
1.7 烷烃羟化酶基因的功能验证
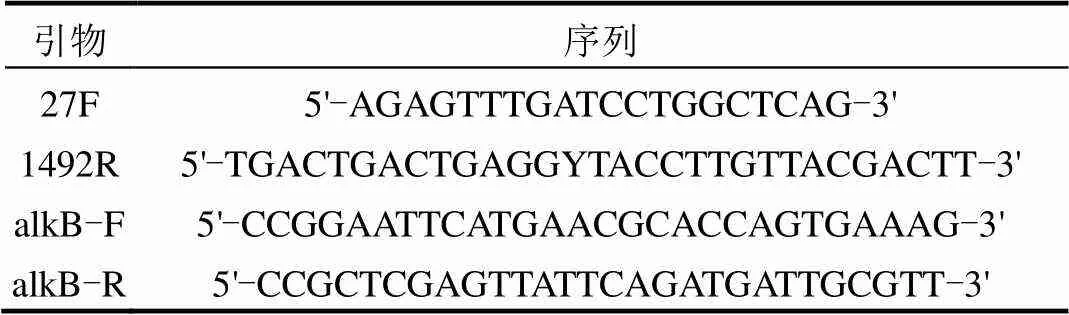
表1 本研究中所使用的PCR引物序列
1.8 直链烷烃含量的测定方法
样品处理方法:准确量取培养液的体积,加入等体积的正己烷,超声10min后8000r/min离心10min,静置10min待其分层后,去掉水分,重复该过程.正己烷萃取两次后,向分液漏斗中加入2g无水硫酸钠,去除多余水分.
紫外分光光度法测定柴油含量[13]:柴油的最大吸收波长为225nm,分别配置不同浓度梯度的柴油溶液测定其吸光度,每个实验做3组平行,绘制柴油标准曲线.
GC-MS检测柴油的方法[14]:气相色谱质谱联用仪为美国安捷伦公司生产(5975B/6890N),色谱柱为DB-5MS Capillary column(0.32mm× 30m×0.25μm),FID检测器,载气为He,气体流速为1.5mL/min,进样器温度为270℃,连接线温度为280℃,离子源温度为230℃,四级杆温度为150℃,升温程序为:150℃保持2min,然后以5℃/min的速度升温至200℃,再以5℃/min的速度升温至290℃,进样量为1μL[14].
2 结果与分析
2.1 菌株L7的筛选与鉴定
从辽河口湿地石油污染的土壤中筛选到一株高效柴油降解菌通过革兰氏染色和扫描电镜观察(图1),确定其为革兰氏阴性菌,在LB固体平板上的菌落形态为白色、半透明、边缘整体、表面湿润,菌体为球杆状,直径约为0.4~0.5μm,长度约为0.6~0.8μm.将该菌株命名为L7.将菌株L7的16S rRNA基因序列(GenBank accession number:KY817316)与标准菌株的16S rRNA基因序列(从LPSN数据库下载)进行序列比对和进化树分析,结果表明菌株L7的16S rRNA基因的序列与B2的序列相似性最高为~97%.选取了部分菌株的16S rRNA基因序列与菌株L7进行了系统发育树分析(图2).通过生理生化、形态学性状和16S rRNA基因序列分析可以确定菌株L7属于属,并名为sp. L7.
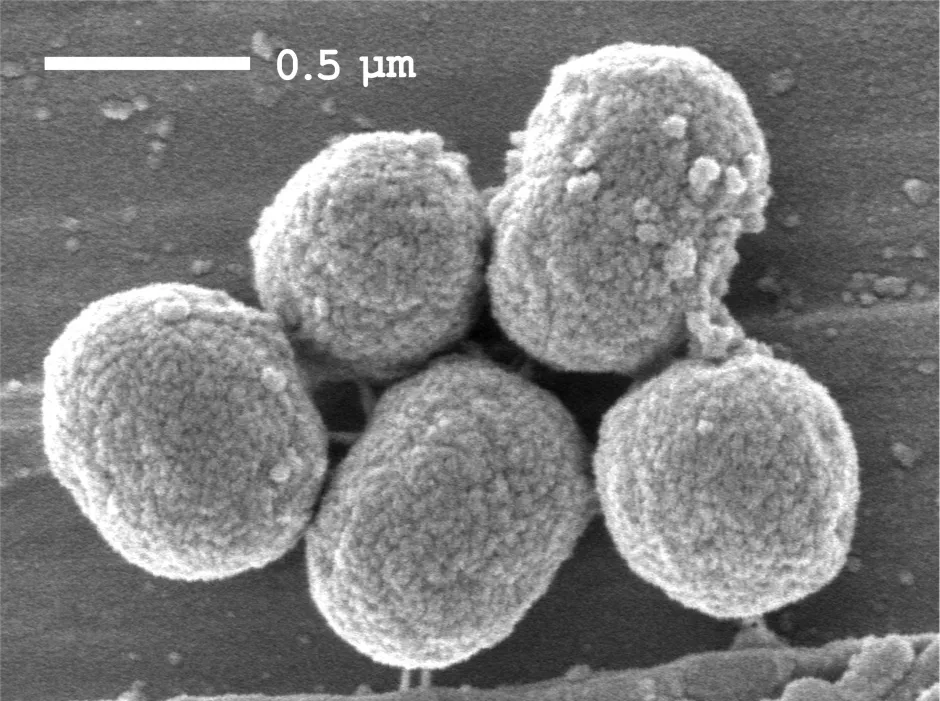
图1 菌株Acinetobacter sp. L7扫描电镜细胞形态观察
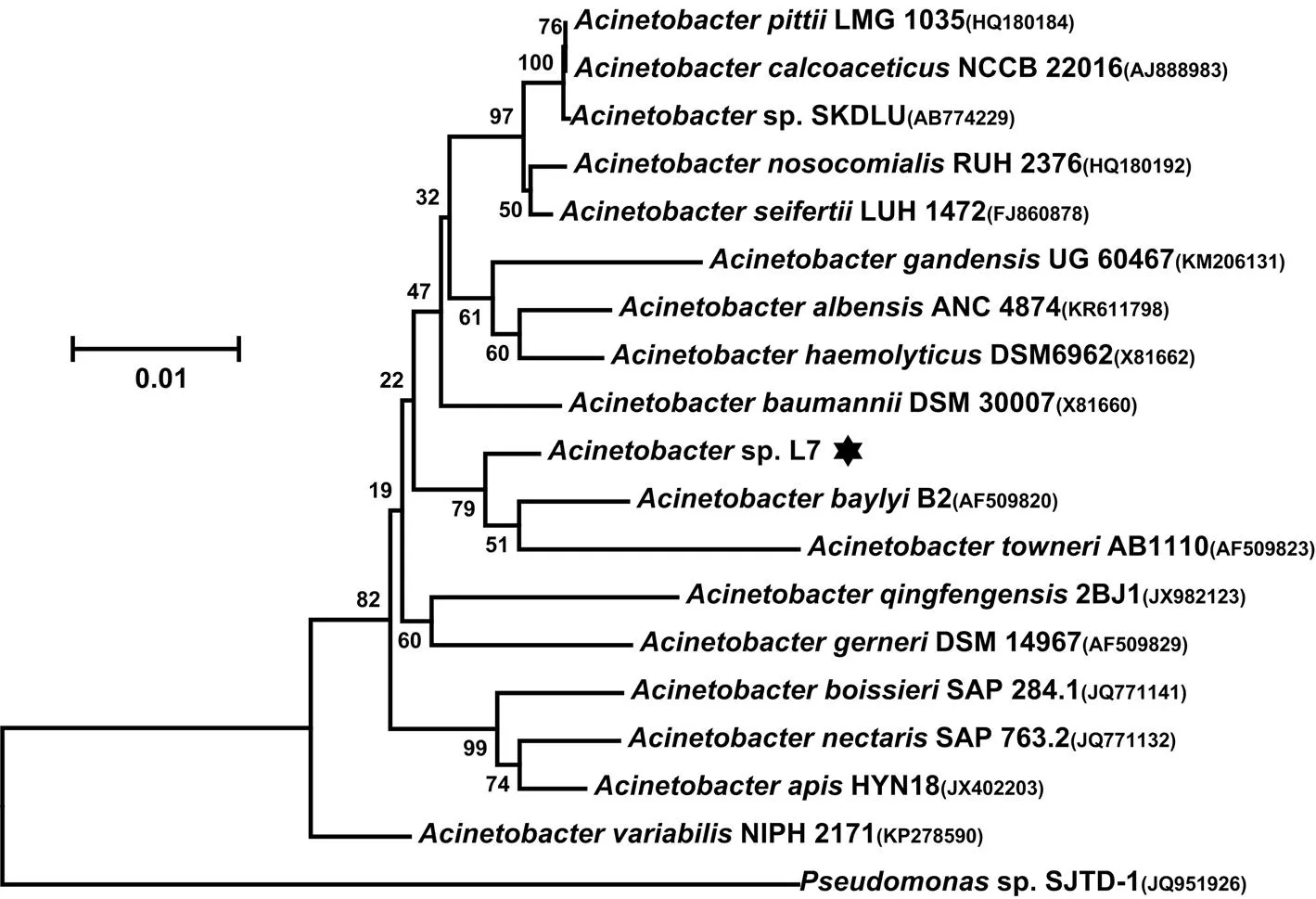
图2 菌株L7的系统发育进化树
2.2 菌株的最适生长条件
如图3a所示,菌株在30℃生长最好,其次为25℃,在37℃以上生长缓慢,且稳定期的生物量(最大OD600nm吸收值)也依次减少.菌株L7是从辽河口石油污染土壤中筛选得到的,辽河口位于高纬度地区当地年最高气温常年等于或者低于30℃,因此菌株在该温度下具有较强的柴油分解能力.
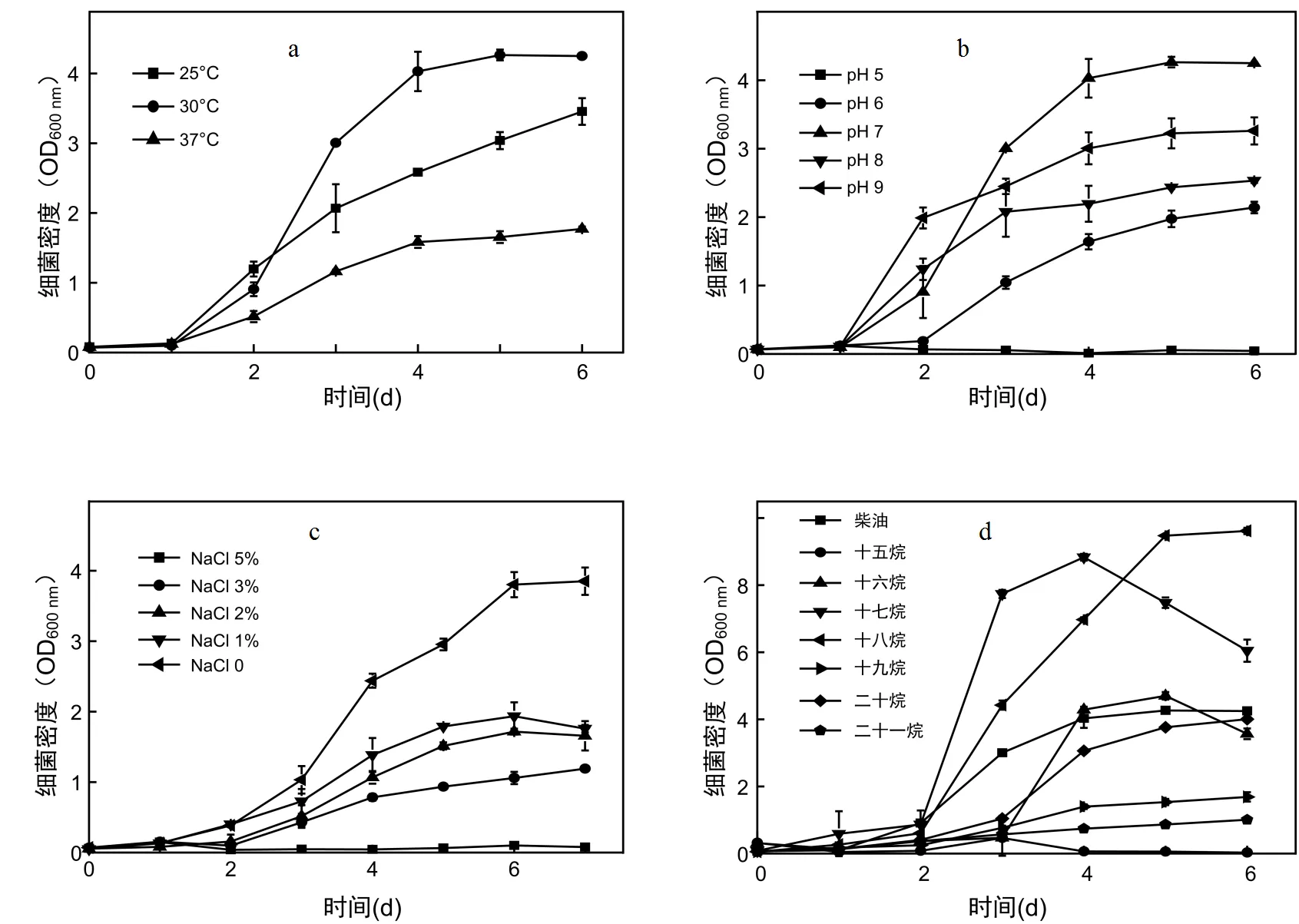
图3 菌株L7在不同生长条件下的生长曲线
如图3b所示,菌株L7的最适pH值为7.可以良好的适应弱碱性环境,在pH值为9的情况下仍能够保持较好的生长,但酸性条件下菌株L7生长受到明显抑制,培养基中pH值为5时基本不能够生长.
如图3c所示,在无机盐培养基中,不添加NaCl时,菌株生长最好,随着培养基中氯化钠浓度的升高,生长受到抑制,但在1%氯化钠条件下终OD600nm值仍能达到2.0左右,在3%氯化钠条件下终OD600nm值仍能达到1.0以上.说明菌株L7具备一定的耐盐能力.
如图3d所示,菌株L7能够利用十五烷、十六烷、十七烷、十八烷、十九烷、二十烷和二十二烷为唯一碳源进行生长,在正癸烷、十一烷、十二烷、十三烷、十四烷和二十一烷为唯一碳源的无机盐培养基中不能生长,其中在柴油培养基中L7的适应能力最强,能够在2d时间达到最大生物量,另外在十七烷和十八烷无机盐培养基中L7菌体稳定期细胞密度(OD600nm值)明显高于柴油培养基中的最大值.说明菌株L7对中长链烷烃有较好的利用和降解效果.
通过当前表格可以看出,实施不同的护理模式以后,研究组患者的护理满意度为95.92%,对照组患者的护理满意度为81.63%,研究组患者的护理满意度高于对照组,差异有统计学意义(P<0.05)。说明相较于常规骨科护理模式,在骨科护理中应用临床护理路径,患者的护理满意度更高。见表1。
2.3 烷烃羟化酶基因的分析
对菌株L7的全基因组进行测序,基因组已经递交到DDBJ/ENA/GenBank数据库,序列号为NBNP00000000.基因组(草图)全长3388376bp, GC含量为42.89%,通过基因功能注释(RAST)预测到3144个蛋白编码基因(CDSs).在基因组上发现一个烷烃羟化酶基因(),该AlkB蛋白的氨基酸序列跟已报道的该家族蛋白氨基酸序列比对如图4所示,跟不动杆菌ADP1菌株中AlkB蛋白的氨基酸相似性最高,为89%.进一步分析基因组信息在该基因上游发现一个反向AraC家族的转录调控蛋白基因,跟已有的不动杆菌ADP1中基因簇的排列顺序一致[15].
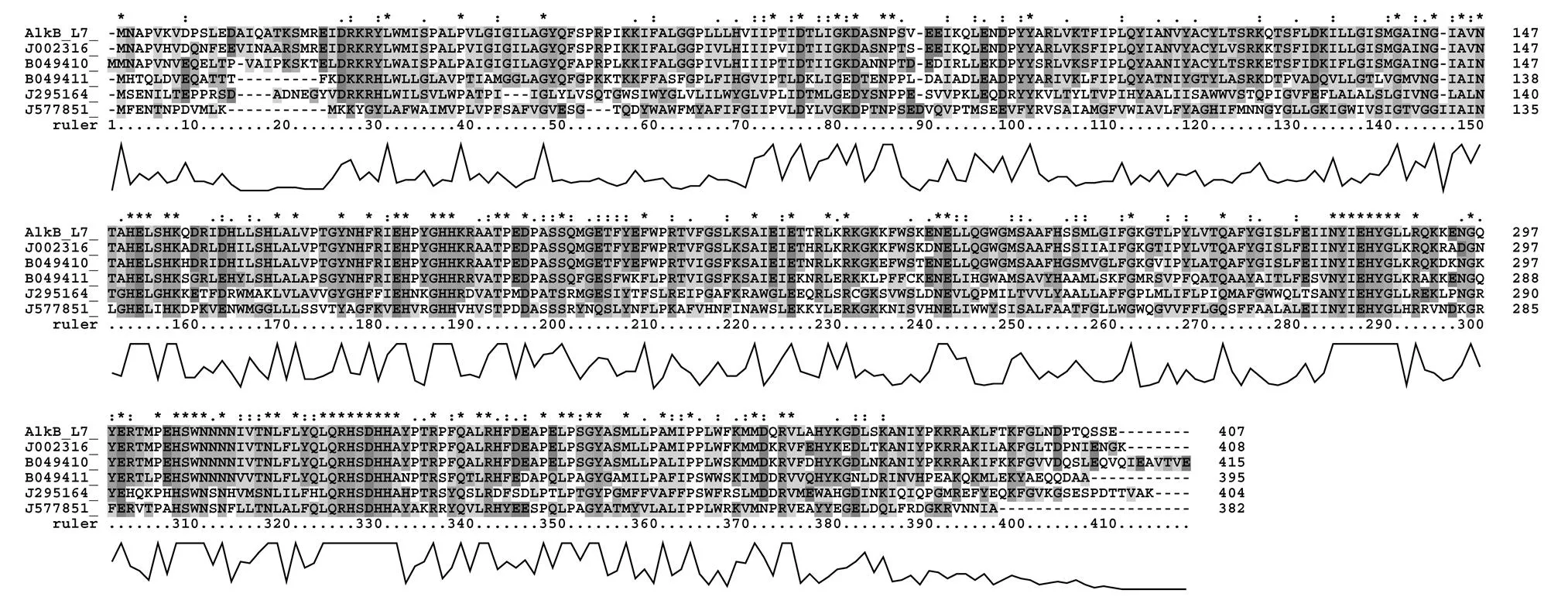
图4 L7中烷烃羟化酶的氨基酸序列比对
2.4 烷烃羟化酶基因的功能验证
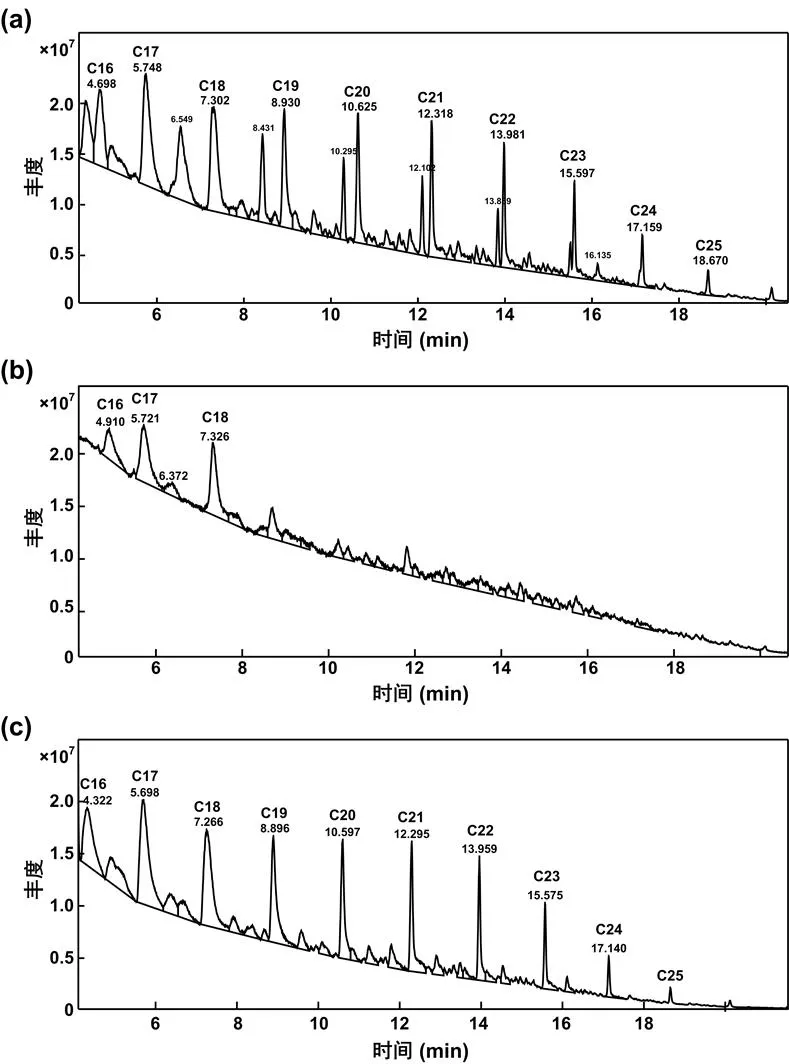
图5 Acinetobacter sp. L7对饱和烷烃降解的GC-MS分析
(a)柴油样品;(b)菌株r sp. L7对柴油的降解;(c)含有pME6032-质粒的KT2440对柴油的降解
利用正己烷萃取样品,紫外分光光度法检测在最适生长条件下(温度30℃,pH值为7.0,盐度0%,170r/min)OD225nm处的吸收值,菌株L7对柴油的降解效率为61.5%.
利用GC-MS法对碳链长度C16~C25的饱和烷烃的降解情况进行检测.与未接种L7菌株的对照组相比,5d后L7菌株对柴油中的十六烷到二十五烷均有较好的降解效果.发现菌株L7对十九烷到二十五烷的降解效率接近100%(图5).
2.5 烷烃羟化酶基因的功能验证
为了验证从L7基因组中比对获得的基因的功能,将长度为1224bp的基因通过RI和I酶切位点连接到pME6032质粒上.将重组质粒pME6032-通过电击转化的方法转化到恶臭假单胞菌KT2440中.将含有pME6032-的KT2440菌株接种到四环素抗性柴油培养基中,观察其生长情况并检测对柴油的降解,以进一步验证基因的功能.
菌株KT2440在以柴油作为唯一碳源的无机盐培养基中不能生长,而含有pME6032-质粒的KT2440菌株能够在柴油无机盐培养基中生长.对培养5d后的样品用正己烷萃取后,用GC-MS检测,结果如图5c所示,含有基因的KT2440培养液中十八烷~二十三烷的含量均出现了下降,说明菌株L7中的AlkB蛋白参与催化十八烷到二十三烷的降解过程.这说明了该基因的确参与了柴油的降解.
3 讨论
根据底物谱实验和GC-MS图谱分析,菌株L7对C19~C25的饱和烷烃的降解效率几乎达到100%.对菌株L7基因组中的基因进行体外功能验证,通过基因克隆表达技术和GC-MS确定AlkB蛋白参与催化十八烷、十九烷、二十烷、二十一烷、二十二烷和二十三烷的降解反应过程,但是跟L7菌株相比降解效果较差,原因可能是烷烃代谢过程还需要其他蛋白的参与,菌株KT2440中没有相关蛋白,导致降解效率不高.菌株L7对碳链长度C14~C25的饱和烷烃均有一定的降解作用,说明菌株L7中还有存在有其他催化中长链烷烃的羟化酶基因.基因比对并没有发现其他功能基因,说明该基因的序列可能跟已有的烷烃羟化酶基因相比相似性较低,这些有待于进一步研究.
不动杆菌属在有氧条件下代谢饱和烷烃的途径主要有2条:一是在末端氧化酶的催化下生成相应的醇,进一步氧化成醛,然后转化成相应的脂肪酸通过β-氧化反应被完全降解[16];二是在双加氧酶的催化下将饱和烃氧化为相应的过氧化物,然后生成醛和脂肪酸进入β-氧化反应[17].同时有大量的烷烃羟化酶被发现,如sp. DSM17874中降解长链烷烃的基因[18]、[19]和sp. BCP1[20]中的基因;这些基因的表达都受到烷烃的诱导.在L7基因组发现的烷烃羟化酶基因,跟已有的基因的序列相似性较高,并在该基因上游发现一个AraC家族的调控蛋白基因,基因结构跟已经报道的基因排列顺序类似,说明菌株L7中的烷烃羟化酶基因也是受到调控蛋白的调控是诱导表达的.推测L7跟其他菌株一样也是通过末端氧化途径生成伯醇,伯醇经过氧化最终通过脂肪酸β-氧化途径被完全分解.
与已经报道的柴油降解菌株相比较,当以柴油作为唯一碳源进行生长的时候L7可以获得更高的细胞密度.菌株SJTD-1在柴油培养基中培养时终OD600nm在0.8左右[21],菌株SY-1在柴油培养基中培养时终OD600nm在0.4左右[22],而菌株L7在柴油培养基中培养时终OD600nm可达到4.0以上,这说明了L7对于柴油的高效利用能力和高效的烷烃降解能力.即使是在盐浓度为3.0%的情况下,菌株L7的菌体密度仍然可以达到1.0以上,可以达到其他的柴油降解菌株在最适生条件下的菌体密度[21,22].这说明了菌株L7不仅仅适合在淡水环境中用于柴油污染的去除,也可以适应海洋环境中柴油的去除工作,是一株优秀的耐盐柴油污染物生物修复菌株.
4 结论
4.1 从石油污染土壤中筛选到一株以柴油为唯一碳源,氮源和能源生长的高效降解菌株L7,经过形态学观察和16S rRNA基因序列比对鉴定为不动杆菌(sp.).菌株L7降解柴油的最适温度为30℃,最适pH值为7.0,能够耐受3%盐度的生长条件,在最适条件下培养5d后,对柴油的降解效率为61.5%.
4.2 菌株L7具有高效降解中长链烷烃的能力,对碳链长度在19~25的饱和烷烃降解效率接近100%.
4.3 对菌株L7的全基因组测序分析,通过序列比对找到一个烷烃羟化酶基因,通过对基因进行体外功能验证确定基因的功能,为烷烃代谢相关基因的研究提供理论参考,并推测出菌株L7降解烷烃的途径为末端氧化途径.本研究可为柴油污染的生物修复提供降解效率高、耐盐、耐碱的优质菌株.
[1] Hassanshahian M, Emtiazi G, Cappello S. Isolation and characterization of crude-oil-degrading bacteria from the Persian Gulf and the Caspian Sea [J]. Marine Pollution Bulletin, 2012, 64(1):7‑12.
[2] 黄 艺,礼 晓,蔡佳良.石油污染生物修复研究进展[J]. 生态环境学报, 2009,18:361‑367.
[3] Rojo F. Degradation of alkanes by bacteria [J]. Environmental Microbiology, 2009,11(10):2477‑2490.
[4] 林莹莹,聂麦茜,王 琰,等.生物膜处理高含油废水及膜表面微生物群落特性研究 [J]. 中国环境科学, 2016,36(9):2800‑2806.
[5] 张 楠,初里冰,丁鹏元,等.A/O生物膜法强化处理石化废水及生物膜种群结构研究[J]. 中国环境科学, 2015,35(1):80‑86.
[6] Huan L, Jing X, Rubing L, et al. Characterization of the medium- and long-chain n-alkanes degradingstrain SJTD-1and its alkane hydroxylase genes [J]. PLOS ONE, 2014,9(8):e105506.
[7] 刁 硕,王红旗,许 洁,等.低温耐盐芘降解菌的筛选鉴定及降解特性研究[J]. 中国环境科学, 2017,37(2):677‑685.
[8] Shigeaki H, Yuki K, Akihiro H. Microbial communities in oil-contaminated seawater [J]. Current Opinion Biotechnology, 2004,15(3):205‑214.
[9] Van Beilen J B, Funhoff E G. Alkane hydroxylases involved in microbial alkane degradation [J]. Applied Microbiology and Biotechnology, 2007,74(1):13‑21.
[10] Diogo J, Vanessa M A, Raquel P, et al. The use of a combination ofprimers to better characterize the distribution of alkane-degradign bacteria [J]. PLOS ONE, 2013,8(6):e66565.
[11] Pearson W R, Lipman D J. Improved tools for biological sequence comparison [J]. Proceedings of the National Academy of Science of the United States of America, 1988,85(8): 2444‑2448.
[12] Shan Z Y, Xu H J, Shi X Q, et al. The study of optimal conditions of electroporation in[J]. Acta Genetica Sinica, 2004,31(3):311‑316.
[13] 黄延林,宁亚平,柴蓓蓓.石油高效降解菌的筛选及其降解特性[J]. 西安建筑科技大学学报(自然科学版), 2006,38(6):795‑803.
[14] Wang X B, Chi C Q, Nie Y, et al. Degradation of petroleum hydrocarbons (C6-C40) and crude oil by a novelstrain [J]. Bioresource Technology, 2011,102(17):7755‑7761.
[15] Andreas R, Walter G, Wolfgang H. Alkane hydroxylase fromsp. ADP1is encoded byand belongs to a new family of bacterial integral-membrane hydrocarbon hydroxylases [J]. Applied and Environment Microbiology, 1998,64(4): 1175‑1179.
[16] Van Hamme J D, Singh A, Ward O P. Recent advances in petroleum microbiology [J]. Microbiology and Molecular Biology Reviews, 2003,67(4):503‑549.
[17] Maeng J H, Sakai Y, Tani Y. Isolation and characterization of a novel oxygenase that catalyzes the first step of n-alkane oxidation inM-1 [J]. Journal of Bacteriology, 1996,178(13):3695‑3700.
[18] Mimmi T H, Alexander W, Trond E, et al. Identification of novel genes involved in long-chain n-alkane degradation bysp. strain DSN 17874 [J]. Applied and Environmental Microbiology, 227,73(10)3327‑3332.
[19] Mercedes M M, Luis Y, Fernando R. Differential expession of the components of the two alkane hydroxylases from[J]. Journal of Bacteriology, 2003,185(10): 3232‑3237.
[20] Cappelltti M, Fedi S, Frascari D, et al. Analyses of both thegene transcriptional atart site andpromoter-inducing properties ofsp. strain BCP1grown on n-alkanes [J]. Applied and Environment Microbiology, 2011,77(5):1619‑1627.
[21] Liu H, Xu J, Liang R B, et al. Characterization of the medium- and long-chain n-alkanes degradingstrain SJTD-1and its alkane hydroxylase genes [J]. PLOS ONE, 2014,9(8):e105506.
[22] 陆洪省,魏文超,王厚伟,等.高效石油烃降解菌SKD-1的分离、筛选及其降解性能[J]. 环境工程学报, 2013,7(10):4116‑4120.
Isolation and identification of a high-efficient diesel degrading bacterial strainsp. L7.
HU Chun-hui1,2, YU Hao2, ZHAO Yang-guo1, TIAN Wei-jun1, BAI Jie1*
(1.Key laboratory of Marine Environmental Science and Ecology, Ministry of Education, Ocean University of China, Qingdao 266100, China;2.College of Life Science, Qingdao Agricultural University, Qingdao 266109, China)., 2017,37(11):4251~4258
A high-efficient diesel degrading bacterial strain isolated from oil-contaminated soil in Liaohe oil field was identified assp. and named as L7. The strain could efficiently grow with diesel, n-tridecane, n-hexadecane, n-heptadecane, n-octadecane and n-nonadecane as its sole carbon and energy source. The cultivation conditions of the strain L7 in diesel basic salt medium were investigated. The suitable pH value for the growth of the strain ranged from 6.0~9.0 with the optimum value of 7.0. The optimum temperature and salinity for the cultivation of the strain was 30°C and 1%, respectively. The degradation efficiency of diesel oil was 61.5% under the optimum conditions. Based on the whole-genome DNA sequence analysis of the bacterial strain, an alkane hydroxylase gene,, was identified. To study the function of thegene in n-alkane utilization by the bacterial strain, the-plasmid was constructed and electroporated into another bacterial strainKT2440, which could not grow in diesel. The electroporated bacteriaKT2440 could grow in diesel basic salt medium. Through gas chromatography-mass spectrometric analysis, the degradation capacity of the bacterial strain L7 for n-alkanes and the roles of thegene were determined. The results indicated that the n-alkanes were degraded by the strain L7 via the terminal oxidation and β-oxidation pathway.
;diesel;alkane hydroxylase;biodegradation
X703.5
A
1000-6923(2017)11-4251-08
胡春辉(1987-),女,山东青岛人,实验师,中国海洋大学博士研究生,主要从事环境中石油污染物降解.发表论文10篇.
2017-04-05
国家水体污染控制与治理科技重大专项(2013ZX07202- 007)
* 责任作者, 教授, baijie@ouc.edu.cn
