低温胁迫下杜鹃杂交后代优良株系的转录组分析
2017-11-04吴月燕何静雯沈梓力吴燕燕谢晓鸿
吴月燕,明 萌,2,何静雯,2,卢 丹,沈梓力,邱 甜,吴燕燕,谢晓鸿
(1.浙江万里学院 生物与环境学院,浙江 宁波 315100;2.上海海洋大学 水产与生命学院,上海 201306)
低温胁迫下杜鹃杂交后代优良株系的转录组分析
吴月燕1,明 萌1,2,何静雯1,2,卢 丹1,沈梓力1,邱 甜1,吴燕燕1,谢晓鸿1
(1.浙江万里学院 生物与环境学院,浙江 宁波 315100;2.上海海洋大学 水产与生命学院,上海 201306)
为探索杜鹃抗寒的分子机制,筛选抗寒相关基因,利用人工模拟低温,以杜鹃杂交后代优良株系繁景-6 ℃(T)处理和25 ℃(CK)对照为试验材料,进行转录组测序和数据从头组装及生物信息学分析,通过组装得到 Unigenes序列122 654个,功能注释结果显示,有大量Unigenes可以注释到基因本体(GO)和蛋白质直系同源数据库(COG) 等数据库上,部分Unigenes无匹配信息,可能为杜鹃特有的基因序列。按GO功能分类,繁景杜鹃中的 Unigenes分为细胞组分、分子功能以及生物学过程3类,包括51个分支,其中有大量的Unigenes与代谢进程、催化活性和细胞进程相关。上调差异表达基因有6 347个,下调差异表达基因4 482个。通过对转录结果中差异表达显著的4个转录因子家族中的24个基因,其中,上调12个,下调12个,在低温胁迫下的表达水平验证,发现其与转录结果基本一致,证明该转录组数据可靠。
杜鹃;低温胁迫;转录组;抗寒基因
杜鹃花(Rhododendron),是杜鹃花科(Ericaceae)杜鹃花属(RhododendronL.)多种植物的总称,又称山踯躅、山石榴、映山红,是常绿或落叶灌木[1-2],为浙江省重要的绿化植物。但由于受气候条件等影响,只能用品种较单一,花色较为单调,花期大多在4-5月中下旬较为耐寒的杜鹃品种。以毛鹃春如意为母本、毛鹃琉球红为父本进行杂交育种,获得246株杂交后代,筛选后得到一杂交后代的优良株系,暂命名为繁景,其花期为4月上旬,略早于大部分浙江省绿化杜鹃品种,花4~6朵簇生于枝端,花朵丰富,花色为深粉红色,花色较为少见。但是,大多数年份,浙江部分地区年极端低温为-2~-5 ℃,部分品种杜鹃会出现冻害现象。因此,繁景杜鹃抗寒性的研究,对其栽培和生产具有重要的理论指导意义。
目前,对于杜鹃耐寒的研究报道比较少,主要研究在低温胁迫下叶片的相对电导率、丙二醛(MDA)含量、过氧化物酶(POD)活性等生理指标的变化情况[3-7],还没有相应杜鹃转录组测序等方面的研究报道。转录组测序是指生物在特定环境和状态下总RNA的变化情况,是连接基因组遗传信息与生物功能蛋白质组的纽带[8],对于缺乏基因组信息的物种,采用转录组测序可获得大量的数据信息,有助于挖掘重要功能基因和植物优良性状研究的开展,因此,对杜鹃转录组的测序研究具有重要的意义。本试验以一年生繁景植株为材料,通过对低温胁迫下繁景杜鹃转录组的分析,为该杜鹃杂交后代优良株系,作为绿化材料在浙江以及环境相似地区的推广、新品种的选育和栽培模式提供理论依据,为杜鹃的分子育种及品种改良提供了一定的理论基础。
1 材料和方法
1.1试验材料
供试杜鹃(Rhododendronsp.)材料来源于宁波市北仑区柴桥镇杜鹃花基地。试验于2015年12月-2016年5月在浙江万里学院实验室进行。试验随机选择了36株生长健壮、形态大小基本一致的繁景杜鹃。通过之前生理指标检测及对超微结构的观察,确定测序条件和时间点,繁景杜鹃耐受的最低温度为-6 ℃,且在-6 ℃处理24 h后各项生理指标达到最大值,因此以-6 ℃低温为处理(T),以25 ℃处理为对照(CK),处理24 h,每个处理6株,共设3组生物学重复。2016年1月12-14日,放入低温生化培养箱进行人工低温处理,生化培养箱内处理与对照除温度不同,光照和水分等环境因素保持一致,空气相对湿度为70%~80%[9],光照为216 μmol/(m2·s),光周期12 h/12 h (昼/夜),处理24 h后进行采样。每次采样随机选取数片顶端叶片,置于超低温冰箱中备用。
1.2试验方法
1.2.1 RNA的质量检测 按OMEGA试剂盒说明书进行杜鹃总RNA的提取。浓度检测方法:用NanoDrop分光光度计测定所抽提RNA样品的A260、A280值,计算样品浓度;样品完整性检测方法:1.0%变性琼脂糖凝胶电泳定性检测。
1.2.2 All-unigenes的统计与组装 转录组测序由杭州晶佰生物科技有限公司完成,每个测序样品重复一次,经测序质量控制,各样品碱基准确Q20的百分比均大于98%,GC含量不超过50%,测序数据可用于组装。
1.2.3 All-unigenes的功能注释 通过转录组测序平台,对不同样品的Unigenes进行差异表达基因分析,并将差异表达基因与GO、COG、KEGG、COG等数据库进行比对,整理出对应基因的功能、通路和富集情况等。
1.2.4 低温胁迫下基因的表达 根据|log2ratio|≥1,FDR≤0.001基因在样品间的筛选标准,分别找出在不同样本间表达量上调高和表达量下调高的基因,基因的表达量代表低温胁迫下的基因表达差异。
1.2.5 实时荧光定量PCR分析 定量检测利用STBR试剂盒,选择差异表达显著的4个转录因子ALDH、ERF、MYB和bHLH家族中的24个转录因子(上调和下调各12个)和内参基因EF-1-α(GenBank Accession为LN829627.1),用Primer Premier 5软件设计引物,如表1所示,进行实时荧光定量PCR分析。用2-ΔΔCT算法计算待测基因的相对表达量[10]。每组样品3个生物学重复,每个反应各设一组空白对照(cDNA模板空白)。
2 结果与分析
2.1繁景杜鹃叶片处理和对照的表型变化
观测CK与T的36株繁景杜鹃,随机摘取若干片顶端叶片,如图1所示,在低温处理下,叶片的颜色加深,并出现轻微的褶皱。
2.2RNA的质量检测
按OMEGA试剂盒说明书提取总RNA,用Nano Drop核酸蛋白仪测定RNA样品的浓度及样品在260,280 nm波长下OD值的比值,并用琼脂糖凝胶电泳定性检测所抽提的RNA质量。如表2所示,2组RNA完整度均较好,均符合文库构建要求。处理与对照各3个生物学重复,用于RNA-Seq转录组测序。

表1 实时荧光定量PCR所用的引物Tab.1 Reaction sequences of primers used in Real-time PCR

图1 繁景杜鹃叶片处理和对照的表型变化Fig.1 The phenotypic changes of leaf treatment and control in Fanjing Rhododendron
2.3测序数据的产出与组装
每个测序样品经测序质量控制,如表3所示各样品质量不低于20的碱基(Q20)的百分比均大于98%,碱基G和C数占总碱基数的比例(GC含量)不超过50%,测序数据可用于组装[11]。如表4所示,组装结果总Unigenes 122 654个,总长78 017 305 nt,平均长度636 nt,N50为1 018 nt。
2.4Contig和Unigenes的长度分布
如图2所示,通过转录组测序获得大量的序列,且组装后Conting和Unigenes的长度增加,表明组装的效果较好,可进一步开展后续分析。
2.5功能注释和分类
基因本体论(简称GO)是一种国际标准化的基因功能分类系统,用来定义生物体中基因和基因产物。通过GO 数据库对繁景杜鹃的 Unigenes进行功能分类,从宏观上了解杜鹃低温胁迫下,表达基因的功能分布特征[12]。GO分为3个本体,分别是基因的分子功能、所处的细胞位置、参与的生物过程。研究结果表明,注释到GO数据库的44 534个Unigenes可划分为 51个功能组,并对每一个功能组涉及的Unigenes进行了归类分析。如图3所示,注释到GO数据库的44 534个Unigenes可划分为 51个功能组, 21个功能组共13 516(47.8%)个Unigenes归属于参与的生物过程, 16个功能组共9 460(33.5%)个Unigenes归属于细胞组分, 14个功能组共5 294(18.7%)个Unigenes归属于分子功能。其中,在上调和下调表达的基因中,细胞进程、代谢进程、单生物进程、催化活性、结合活性和糖酵解功能组中涉及的Unigenes较多,而生物黏附、胞外基质部分、类核、鸟嘌呤核苷酸交换因子活性、营养库活性和蛋白标签功能组中涉及的Unigenes较少。

表2 繁景杜鹃RNA质检结果Tab.2 RNA quality inspection results of Fanjing Rhododendron
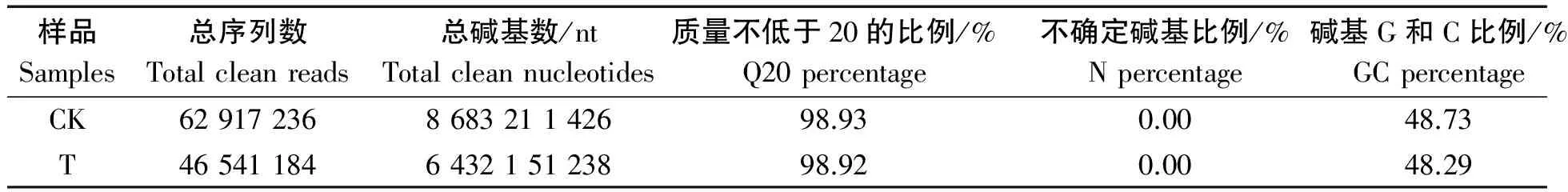
表3 繁景杜鹃转录组测序的产出量Tab.3 Transcriptome sequencing yield statistics of Fanjing Rhododendron
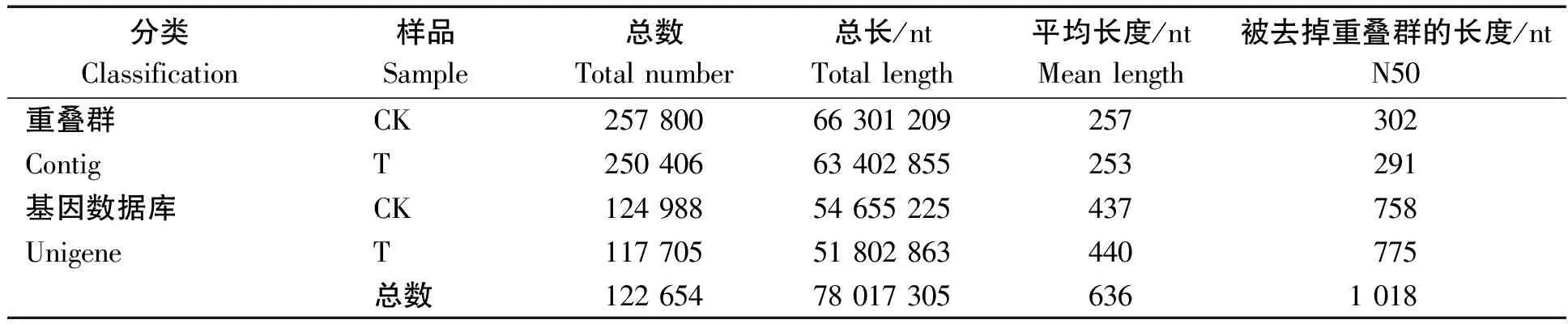
表4 繁景杜鹃基因序列组装统计结果Tab.4 Statistical results of gene sequence assembly of Fanjing Rhododendron

图2 繁景杜鹃基因序列组装的Contigs和Unigenes的长度分布Fig.2 Length distribution of Contigs and Unigenes in gene sequence assembly of Fanjing Rhododendron
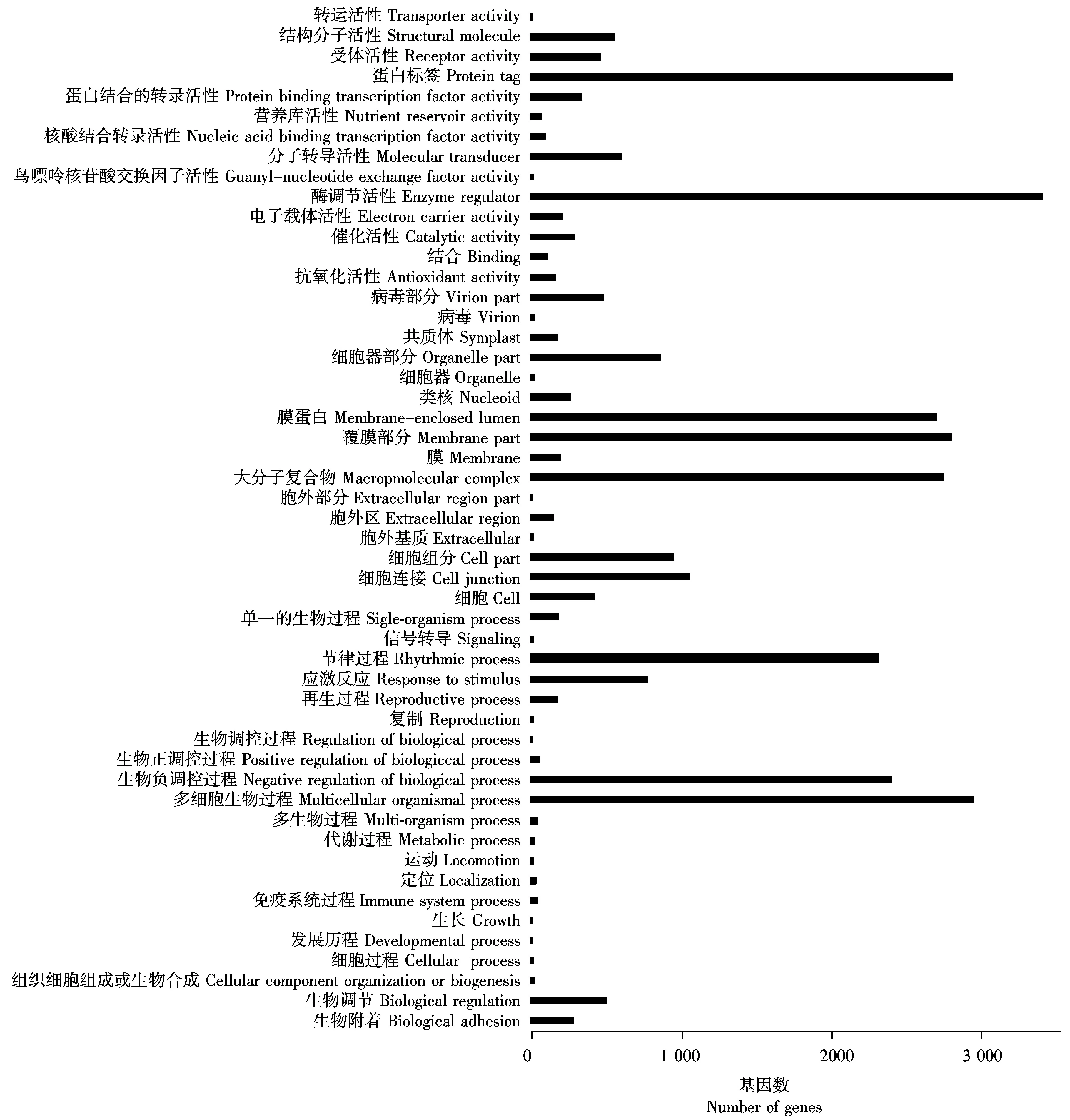
图3 繁景杜鹃Unigenes的GO分类Fig.3 Fanjing Rhododendron GO classification of Unigenes
2.6Unigenes的COG功能分类
蛋白质直系同源数据库(Cluster of orthologous groups,COG)是对基因产物进行直系同源分类的数据库。所有组成COG的蛋白都被暂且认为源于同一个蛋白,这个源蛋白可能是orthologs也可能是paralogs[13-14]。将繁景杜鹃Unigenes比对到COG 数据库上,对Unigenes的功能进行预测和分类统计。得知,繁景杜鹃的 81 850 个Unigenes根据其功能大致可分为 25 类,并对每一类的Unigenes进行了统计分析。如图4所示,Unigenes涉及的COG功能类别比较全面,涉及了大多数的生命活动。其中一般功能预测类基因最多(12 084个),翻译、核糖体结构与生物合成类基因(8 682个)等也较多;而核酸结构类基因(16个)、胞外结构基因(89个)较少;其他类别基因表达各不相同。
2.7KEGG通路分析
KEGG是系统分析基因产物在细胞中的代谢途径以及基因产物功能的数据库。根据 KEGG 数据库的注释信息能进一步得到Unigenes的Pathway注释[15]。结合KEGG数据库,对繁景杜鹃的52 057个Unigenes参与或涉及的代谢途径进行了统计分析。如图5所示,可将繁景杜鹃Unigenes归属于 5大类的代谢途径,主要包括代谢途径和次生代谢产物合成、能量代谢;蛋白折叠、脂类物质代谢;碳水化合物代谢;氨基酸代谢;分类和降解、转录与翻译、信号转导等。
将 KEGG pathway 数据库作为参考,可将Unigenes定位到 128 个具体的代谢途径分支。其中,在上调和下调表达的基因中,主要涉及能量代谢的基因,蛋白折叠分类和降解途径的基因,核苷酸代谢途径基因,转录与翻译的基因以及运输和代谢途径的基因等。
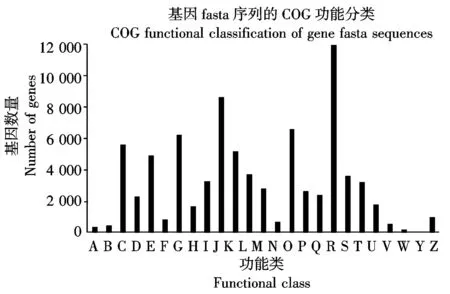
A.RNA的加工与修饰;B.染色体结构与动力学;C.能量产生和转换;D.细胞周期调控与分裂、染色体重排;E.氨基酸运输与代谢;F.核苷酸运输与代谢;G.碳水化合物运输与代谢;H.辅酶运输与代谢;I.脂质运输与代谢;J.翻译、核糖体结构与生物合成;K.转录;L.复制、重组与修复;M.细胞壁、膜生物合成;N.细胞运动;O.翻译后修饰、蛋白质折叠和分子伴侣;P.无机离子运输与代谢;Q.次生代谢物合成、运输和代谢;R.一般功能预测;S.未知功能;T.信号转导机制;U.细胞内转运、分泌和囊泡运输;V.防御机制;W.胞外结构;Y.核酸结构;Z.细胞骨架。
A.RNA processing and modification; B.Chromatin structure and dynamics; C.Energy production and conversion; D.Cell cycle control,cell division,chromosome partitioning; E.Amino acid transport and metabolism; F.Nucleotide transport and metabolism; G.Carbohydrate transport and metabolism; H.Coenzyme transport and metabolism; I.Lipid transport and metabolism; J.Translation,ribosomal structure and biogenesis; K.Transcription; L.Replication, recombination and repair; M.Cell wall/membrane/envelope biogenesis; N.Cell motility; O.Posttranslational modification,protein turnover,chaperones; P.Inorganic ion transport and metabolism; Q.Secondary metabolites biosynthesis,transport and catabolism; R.General function prediction only; S.Function unknown; T.Signal transduction mechanisms; U.Intracellular trafficking,secretion,and vesicular transport; V.Defense mechanisms; W.Extracellular structure;Y.Nucleic acid structure;Z.Cytoskeleton.
图4繁景杜鹃Unigenes的COG功能分类
Fig.4FanjingRhododendronCOGclassificationofUnigenes
2.8差异表达基因分析
如图6所示,差异表达的基因共有10 829个,其中上调表达基因6 347个,下调表达基因4 482个,上调与下调的基因相差较大,这些上调和下调的基因,为揭示杜鹃抗寒机理,及其相关代谢途径分析提供了大量和可靠的数据。
2.9差异表达显著的基因在低温胁迫下的表达分析
分别取低温胁迫处理后,上调表达和下调表达显著的4个转录因子ALDH、ERF、MYB和bHLH家族中的24个基因,上调12个,下调12个(其中ALDH家族全部为上调表达的基因,ERF、MYB和bHLH家族中既有转录水平上调的也有转录水平下调的),利用实时荧光定量PCR对其低温胁迫处理前后表达变化进行分析。如图7所示,荧光定量结果虽然在表达变化幅度上有些差异,但从24个基因的表达趋势来看,除了20776基因相对表达量较低(0.2),其余23个基因相对表达量均比较高,与转录组结果基本一致,且31795、10108、937 3个基因的相对表达量最高。实时荧光定量分析结果与转录组结果基本一致,说明本次Illumina测序所获得的转录组数据可靠。
3 讨论与结论
随着新一代高通量测序技术的广泛应用,植物基因组研究得到快速发展,但杜鹃基因组研究还很少。Illumina 高通量测序的数据量大、速度快、成本低、效率高,适合于没有参考基因组信息的杜鹃展开转录组研究。本研究中通过组装得到 Unigenes序列122 654个,说明本研究所得的数据量和数据信息较为丰富。前人应用转录组测序技术,分别对拟南芥在不同光强和温度之下,分析了其分子机制及拟南芥中 872 个基因在其花粉突变体中的差异表达情况[16-17]。Liu等[18]运用Illumina 高通量测序的方法,发现了控制拟南芥发育出根的关键基因WOX11和WOX12。Li等[19]利用Illumina 测序技术分析了差异基因在越橘果皮和果肉中的表达情况,结果表明,核苷酸序列为4.8 Gb,Unigenes的平均长度为 735 bp,能够进行基因功能注释信息的有 25 376 个。Rowland 等[20]利用高通量测序技术研究了植物各组织之间的转录组信息与低温锻炼等的相关性,结果表明,所预测基因的表达情况和结果是一致的。根据张振亚等[21]的转录组数据的分析,发现其中有25个差异表达的基因在盐胁迫下苜蓿和蛋白质表达的变化趋势相同,上调表达基因有14个,下调表达基因有11个,这些基因参与代谢、信号转导、蛋白质降解、防御、抗氧化、细胞骨架、膜转运、转录等及一系列未知功能。孙颖等[22]利用 RNA-seq 技术对油桐 2 个不同发育时期的花芽的转录组进行比较分析,获得大量差异表达的Unigenes序列,在一定程度上解析油桐花芽形态分化的分子调控模式与机制。陈娜等[23]利用高通量测序技术,通过低温处理后芯片杂交实验,筛选花生叶片中低温胁迫响应转录因子基因。结果表明,175 个具有转录调控活性的基因在低温胁迫的花生叶片中表达变化量达到 2 倍以上,其中 92 个为上调基因,83 个为下调基因。因此,通过转录组测序结合生物信息学分析及定量表达分析的方法寻找与杜鹃抗寒相关的基因也是可行的。
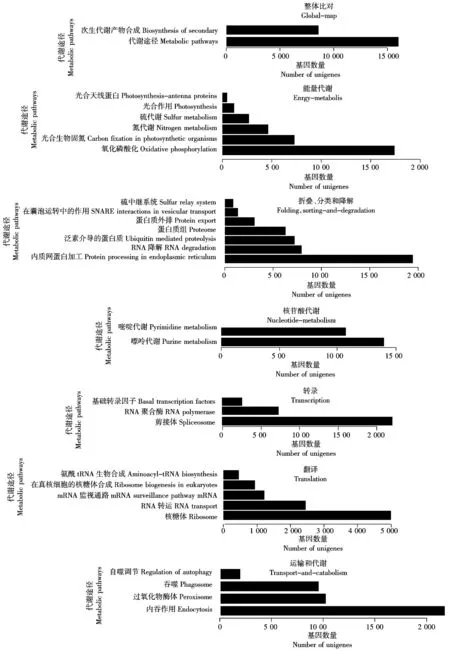
图5 繁景杜鹃Unigenes的部分KEGG 分类Fig.5 Fanjing Rhododendron KEGG classification of Unigenes
本研究中应用Illumina高通量测序技术对杜鹃叶片转录组进行测序,研究其基因表达谱和挖掘低温胁迫下的重要表达基因。本研究通过组装得到 Unigene 序列122 654个,将测序结果与nr、Swiss-Prot、GO、KEGG等蛋白数据库进行BlastX比对,有部分Unigenes无匹配信息,可能是片段过短或基因注释信息匮乏等原因,也可能为杜鹃特有的基因序列。经前人研究发现,GO在结构设计上的存在缺陷,同时基因的大部分功能目前并不明确,因此导致了这种基因注释信息并不完整[24]。因此,在本研究中,繁景杜鹃通过 GO 数据库对Unigenes所做的有关功能方面的分类等数据还不完善,仍有较多的Unigenes没有对应相关的 GO 注释,需要借助另外的生物信息学分析对 Unigenes功能进行进一步的分类和完整。将繁景杜鹃的Unigenes比对到 COG 数据库,对基因的功能进行统计和归类,从基因组层次,为其对铜元素的追踪和预测未知开放阅读框(ORF)的生物学功能提供了可能和便利,这使得基因功能的注释更加精确。将以上Unigenes比对到KEGG 数据库中,并对其代谢途径进行分析,发现共涉及128个具体的代谢途径分支,参与到繁景杜鹃体内的碳水化合物代谢、能量代谢、转录与翻译、信号转导等代谢途径中,为进一步大量挖掘杜鹃低温胁迫下的重要表达基因,开展杜鹃的基因克隆及功能分析等研究提供了基础数据。

图6 繁景杜鹃差异表达基因统计Fig.6 Differential expression gene of Fanjing Rhododendron

图7 低温胁迫下差异表达显著基因的相对表达量Fig.7 The relative expression of the differentially expressed genes under low temperature stress
通过荧光定量 PCR 结果分析发现,在低温胁迫下,上调和下调表达显著的4个转录因子家族中的24个基因,除了其中的20776基因相对表达量较低(0.2),其余23个基因相对表达量均比较高,与转录组结果基本一致。其中31795、10108、937 3个基因的相对表达量较高,推测这3个基因可能在杜鹃低温胁迫的过程中,行使着促进耐寒相关的功能。其中相对表达量最高的31795为醛脱氢酶(ALDH)转录家族中的基因,通过前人对该相关基因的研究,笔者发现,第一个在植物中被克隆出来的醛脱氢酶基因来自于玉米雄性不育恢复基因Rf2a,它在线粒体乙醛脱氢酶的合成中起关键作用[25-26];而类似的是,通过对拟南芥AtALDH3I1基因的研究,发现该基因表达受多种胁迫的诱导,如干旱胁迫、高浓度盐胁迫、重金属胁迫以及脱落酸胁迫等[27];Gao等[28]发现20个醛脱氢酶基因在水稻中,分属于10个亚科。值得注意的是,约有12个与干旱胁迫或高浓度盐胁迫相关的基因,表明植物中的醛脱氢酶积极地参与了植物的各类抗逆反应。目前,31795基因的生物学功能只是通过荧光定量表达分析以及通过对拟南芥、玉米和水稻中该基因功能的研究进行推断,究竟它的功能是否如此,以及是否还参与其他生命和代谢活动,仍需进一步研究。
在本研究中,第一次利用 Illumina高通量测序技术构建了繁景杜鹃转录组数据库,获得了大量与转录本相关的信息,并通过序列组装、功能注释和分类、代谢途径等对该基因的表达进行分析,挖掘出杜鹃低温胁迫下转录组的整体表达特征。为深入研究杜鹃的基因克隆、抗逆机理以及分子标记开发提供了极大的方便,为杜鹃花科植物的分子生物学研究提供了丰富的数据资源。
[1] 周 泓. 杜鹃花品种资源多样性研究及品种分类体系构建[M]. 杭州:浙江大学,2012.
[2] 吴福建,李凤兰,黄凤兰,等. 杜鹃花研究进展[J]. 东北农业大学学报,2008,39(1):139-144.
[3] 余丽玲,何天友,陈凌艳,等. 人工低温胁迫下西洋杜鹃生理生化指标的变化[J]. 农学学报,2014,4(1):48-53.
[4] 刘 冰,曹 莎,周 泓,等. 杜鹃花品种耐寒性比较及其机制研究[J]. 园艺学报,2016,43(2):295-306.
[5] 鲍思伟. 云锦杜鹃杜威半致死温度对自然降温的适应[J]. 西南民族大学学报:自然科学版,2005,31(1):99-102.
[6] 张艳红,沈向群. 辽宁园林杜鹃花耐寒能力研究[J]. 江苏农业科学,2009,10(3):220-222.
[7] 兰 熙,张乐华,张金政,等. 杜鹃花属植物育种研究进展[J]. 园艺学报,2012,39(9):1829-1838.
[8] 费 元,韩 雪,余 红,等. 大岩桐花萼和幼叶转录组研究[J]. 园艺学报,2015,42(12):2519-2525.
[9] 陶巧静,付 涛,项锡娜,等. 模拟酸雨对西洋杜鹃生理生态特性的影响[J]. 生态学报,2014,34(8):2020-2027.
[10] Livak K J,Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method[J]. Methods,2001,25(4):402-408.
[11] 陈嘉贝,张芙蓉,黄丹枫,等. 盐胁迫下两个甜瓜品种转录因子的转录组分析[J]. 植物生理学报,2014,50(2):150-158.
[12] 宋 杨,刘红弟,张红军,等. 越橘果实转录组及R2R3-MYB转录因子分析[J]. 园艺学报,2015,42(12):2383-2394.
[13] 王海波,程来亮,常源升,等. 苹果矮化砧“71-3-150”对冷胁迫的生理与转录组响应[J]. 园艺学报,2016,43(8):1437-1451.
[14] 杨 伟,龚荣高,石佳佳,等. 低温胁迫下枇杷幼果转录组的 Denovo 组装和功能注释[J]. 西北农林科技大学学报:自然科学版,2014,42(8):138-146.
[15] 江香梅,伍艳芳,肖复明,等. 樟树5种化学类型叶片转录组分析[J]. 遗传,2014,36(1):58-68.
[16] Szymanski J,Brotman Y,Willmitzer L,et al. Linking gene expression and membrane lipid composition ofArabidopsis[J]. The Plant Cell,2014,26(3):915-928.
[17] Zhang D,Liu D,Lü X,et al. The cysteine protease CEP1,a key executor involved in tapetal programmed cell death,regulates pollen development inArabidopsis[J]. The Plant Cell,2014,26(7):2939-2961.
[18] Liu J,Sheng L,Xu Y,et al. WOX11 and 12 are involved in the first-step cell fate transition during de novo root organogenesis inArabidopsis[J]. The Plant Cell,2014,26(3):1081-1093.
[19] Li X,Sun H,Pei J,et al. De novo sequencing and comparative analysis of the blueberry transcriptome to discover putative genes related to antioxidants[J]. Gene,2012,511(1):54-61.
[20] Rowland L J,Alkharouf N,Darwish O,et al. Generation and analysis of blueberry transcriptome sequences from leaves,developing fruit,and flower buds from cold acclimation through deacclimation[J]. BMC Plant Biology,2012,12(1):46.
[21] 张振亚,裴翠明,马 进. 基于转录组和蛋白质组关联研究技术筛选紫花苜蓿耐盐相关候选基因[J]. 植物生理学报,2016,52(3):317-324.
[22] 孙 颖,谭晓风,罗 敏,等. 油桐花芽 2 个不同发育时期转录组分析[J]. 林业科学,2014,50(5):70-74.
[23] 陈 娜,迟晓元,程 果,等. 花生中低温胁迫相关转录因子基因的筛选[J]. 核农学报,2016,30(1):19-27.
[24] 贾新平,孙晓波,邓衍明,等. 鸟巢蕨转录组高通量测序及分析[J]. 园艺学报,2014,41(11):2329-2341.
[25] Cui X,Wise R P,Schnable P S. The rf2 nuclear restorer gene of male-sterile T-cytoplasm maize[J]. Science,1996,272(5266):1334-1336.
[26] Liu F,Cui X,Horner H T,et al. Mitochondrial aldehyde dehydrogenase activity is required for male fertility in maize[J]. The Plant Cell,2001,13(5):1063-1078.
[27] Kirch H H,Schlingensiepen S,Kotchoni S,et al. Detailed expression analysis of selected genes of the aldehyde dehydrogenase (ALDH) gene superfamily inArabidopsisthaliana[J]. Plant Molecular Biology,2005,57(3):315-332.
[28] Gao C,Han B.Evolutionary and expression study of the aldehyde dehydrogenase. (ALDH)gene superfamily in rice(Oryzasativa) [J]. Gene,2009,431(1/2):86-94.
TranscriptomeAnalysisofSuperiorStrainsofRhododendronHybridsunderLowTemperatureStress
WU Yueyan1,MING Meng1,2,HE Jingwen1,2,LU Dan1,SHEN Zili1,QIU Tian1,WU Yanyan1,XIE Xiaohong1
(1.College of Biological and Environmental Sciences,Zhejiang Wanli University,Ningbo 315100,China;2.College of Fisheries and Life Science,Shanghai Ocean University,Shanghai 201306,China)
To explore the molecular mechanism of cold resistance ofRhododendron,screening of cold resistant genes,using artificial low temperature,excellent strains withRhododendronFanjing-6 ℃(T)and 25 ℃(CK)control as test materials,conduct transcriptome sequencing and de novo assembly data and bioinformatics analysis,the data obtained by assembling 122 654 Unigenes sequences,functional annotation showed that a large number of Unigenes could be annotated with GO and COG database,information,some of Unigenes,might be the unique gene sequence ofRhododendron.RhododendronFanjing transcription in the Unigenes group according to the GO function can be divided into cellular component,molecular function and biological process of 3 kinds of 51 branches,including Unigenes and metabolic process,a large number of catalytic activity and cell process. There were 6 347 up-regulated differentially expressed genes and down regulated expression genes of 4 482. The expression of 24 genes in the four transcription factors,with 12 up-regulated and 12 down-regulated at the transcription level,which were significantly different in the transcriptional results,was verified by the expression level under low temperature stress. It was found that the transcriptional data were reliable.
Rhododendron;Low temperature stress;Transcriptome;Cold-resistant genes
2017-08-16
浙江省重中之重学科“生物工程”开放基金项目(KF2015008);宁波市重大科技专项(2014C11002)
吴月燕(1963-),女,浙江金华人,教授,硕士,主要从事植物生理生化与分子生物学研究。
S685.21;Q78
A
1000-7091(2017)05-0052-09
10.7668/hbnxb.2017.05.009
