贝类包纳米虫病焦磷酸测序检测方法的建立与应用*
2017-11-01王彩霞吴绍强林祥梅王素华冯春燕
王彩霞, 吴绍强**, 林祥梅, 王素华, 冯春燕, 徐 赓
(1.中国检验检疫科学研究院 动物检疫研究所,北京 100029; 2.浙江省温州出入境检验检疫局,浙江 温州 325027;3.山东省沂南县畜牧兽医局, 山东 沂南276300)
贝类包纳米虫病焦磷酸测序检测方法的建立与应用*
王彩霞1, 吴绍强1**, 林祥梅1, 王素华2, 冯春燕1, 徐 赓3
(1.中国检验检疫科学研究院 动物检疫研究所,北京 100029; 2.浙江省温州出入境检验检疫局,浙江 温州 325027;3.山东省沂南县畜牧兽医局, 山东 沂南276300)
以焦磷酸测序技术为检测平台,在研究包纳米虫基因特性的基础上,选择包纳米虫(Bonamiaspp.)DNA序列的保守区域,利用焦磷酸测序软件设计专用引物。以OIE推荐的PCR方法构建的阳性质粒作为模板,建立了包纳米虫的PCR-焦磷酸测序检测方法,确定测得序列为包纳米虫序列,经酶切位点分析可鉴别感染种类。以建立的包纳米虫PCR-焦磷酸测序方法对进口牡蛎(Ostrea gigas thunberg)样品进行检测,同时以OIE推荐的PCR-RFLP方法进行对照检测,检测结果表明,本研究建立的PCR-焦磷酸测序检测方法可以在基因序列水平上准确鉴定样品是否为包纳米虫感染以及感染种类,检测结果与OIE推荐方法的检测结果一致。研究结果表明,本研究建立的检测方法可应用于口岸包纳米虫感染情况的检测鉴定。
牡蛎;包纳米虫;聚合酶链式反应;焦磷酸测序;检测;鉴定
牡蛎包纳米虫病是由牡蛎包纳米虫(Bonamiaostreae)、杀蛎包纳米虫(B.exitiosa)等在牡蛎(Oyster)血细胞内寄生或者游离在结缔组织、鳃、内脏或套膜上皮中而引起贝类的一种严重的寄生虫病。现发现的包纳米虫包括5个种,除主要寄生在欧洲牡蛎体内的牡蛎包纳米虫(B.ostreae)、杀蛎包纳米虫(B.exitiosa)[1]外,还有寄生在悉尼牡蛎体内的鲁道夫包纳米虫(B.roughleyi)[2]、美国牡蛎体内的成孢包纳米虫(B.perspora)[3]和澳大利亚牡蛎体内的包纳米虫未定种(Bonamiasp.)[4]。目前,除牡蛎包纳米虫、杀蛎包纳米虫为世界动物卫生组织(Office International Des Epizooties,OIE)规定的检疫性疫病外,其它3种均为非检疫性寄生虫[5]。1980年,Pichot等[6]指出,牡蛎包纳米虫是造成欧洲牡蛎大量死亡的重要原因之一,此后,人们对牡蛎包纳米虫进一步研究,发现牡蛎包纳米虫是一种软体动物血液原虫。1987年爱尔兰科克港(Cork)发生牡蛎包纳米虫病,死亡率达90%;1989年,爱尔兰戈尔韦海峡(Galway)的牡蛎感染牡蛎包纳米虫,死亡率达70%~80%[7]。1995年,牡蛎包纳米虫与折光马尔太虫(Marteiliarefringens)同时爆发,使法国贝类产品从1970年的20 000 t下降至1 800 t[8]。阿根廷圣安东尼奥(San Andonio)海湾自1995年初次报道以来,1996年导致33%的牡蛎死亡,1997年的牡蛎累计死亡率高达95%,使得牡蛎养殖业遭到重创[9],近十几年来,牡蛎包纳米虫病的流行趋于缓和,虽未出现重大的流行,但在美国、英国、爱尔兰、加拿大等地时有发生,流行率从百分之零点几到百分之三、四十不等。2004年英国大不列颠牡蛎养殖场牡蛎大量死亡[10],经证实,导致此次牡蛎大量死亡的原因正是牡蛎包纳米虫,牡蛎包纳米虫病已成为对欧洲和美国牡蛎养殖业影响重大的主要疾病之一。2008年3月13日,英国向OIE报告,本国自2007年11月以来,在北肯特(North Kent)海湾发生包纳米虫感染,受感染贝类数量达500万,其中发病10万[11]。2009年,挪威的东阿格德尔郡(AUST-AGDER)在野生牡蛎中发生包纳米虫病,这是挪威首次发生包纳米虫病,感染来源尚不清楚。2016年澳大利亚首次发生杀蛎包纳米虫病,这是OIE关于该病最近一次的疫情报道。
包纳米虫能直接在牡蛎间通过呼吸、食物而传播,但其生活史尚不清晰,是否存在中间宿主或携带者尚未研究清楚。包纳米虫的检测方法主要包括一些形态学、免疫学及分子生物学检测方法。形态学检测方法能直接观察到虫体,操作相对比较简单,检测费用低,但检出率较低,主要有虫体浓缩法、病理切片技术、组织印记法、组织细胞学等;而分子生物学方法敏感性、特异性较高,能检测到牡蛎包纳米虫感染的潜伏期,目前主要有原位杂交法、PCR技术、实时荧光定量PCR技术[12]以及鉴别物种类别的PCR-限制性片段长度多态性分析方法(PCR-RFLP)技术。但是PCR技术以及实时荧光PCR无法获得基因的序列信息,不能从基因序列水平进行鉴定,且容易造成假阳性、假阴性结果的出现,DNA测序技术虽然能解决基因序列水平上的检测问题,但是存在取样、送样、结果反馈等手续繁琐以及检测速度慢、成本高的缺点,不利于大规模样本的快速检测。相关报道指出,在实际工作中,如果引物对选择得好,很短的一段保守/特异序列的测定就可满足对病原分子诊断的需要[13]。焦磷酸测序技术是1987年发展起来的一种能够进行定量序列测定的新型DNA测序技术[14],具有高通量、快速、敏感等特点,该技术利于大批量样本的检测[15]。因此,本研究拟利用焦磷酸测序技术建立包纳米虫基因序列水平上的检测方法,并应用于进口牡蛎样品的包纳米虫检测鉴定。
1 材料与方法
1.1 材料
样品 2016年3月,温州出入境检验检疫局从荷兰和法国进口的牡蛎样品。
阳性对照 牡蛎包纳米虫(BonamiaOstreae)DNA由法国软体动物疾病参考实验室的Isabelle Arzul教授惠赠。
阴性对照 实验室保存的采自中国东部沿海的牡蛎,已经OIE方法[16]检测确认无包纳米虫感染。
1.2 方法
1.2.1 包纳米虫焦磷酸测序方法的建立
1.2.1.1 阳性克隆质粒的构建 以法国软体动物疾病参考实验室Isabelle Arzul教授惠赠的牡蛎包纳米虫DNA为模板,采用OIE发表的根据牡蛎包纳米虫核糖体DNA(rDNA)设计的特异性引物Bo:5’-CATTTAATTGGTCGGGCCGC-3’, Boas:5’-CTGATCGTC TTCGATCCCCC-3’,按照OIE推荐的反应条件进行聚合酶链反应,预期扩增片段长度为300bp[16]。PCR产物电泳后,采用Agarose Gel DNA Purification Kit(OMEGA)切胶回收目的条带,并克隆到pGEM-T载体,转化至大肠杆菌E.coliJM109感受态细胞。经蓝白斑筛选、PCR鉴定获得含有目的基因片段的重组质粒并纯化,测浓度后-20 ℃保存备用。
1.2.1.2 引物设计 根据GenBank中包纳米虫基因组DNA序列AF262995.1的保守区域片段,利用焦磷酸测序检测系统的自带软件设计引物。上游引物BoF:5’-CGCTGGTCCTGATCCTTTACT-3’;下游引物BoR:5’-Biotin-TACTAGCACCCCCAATTGTTTC-3’,产物长度为168 bp,测序引物BoS:5’-GAATGCATTAGCATGG-3’。引物由上海英维捷基生物技术有限公司合成。灭菌水稀释引物为10μmol/L,分装,-20 ℃保存备用。
1.2.1.3 焦磷酸测序——PCR扩增 以构建的包纳米虫阳性质粒为模板,以焦磷酸测序引物(BoF/BoR)进行PCR扩增。25 μL的PCR反应体系:10×PCR Buffer 2.5 μL,2.5 mmol/L dNTP 2μL,10 μmol/L BoF 1 μL,10 μmol/L BoR 1 μL,5 U/μL rTaq 0.5 μL,质粒DNA1 μL,ddH2O补充至25 μL。PCR反应条件为:95 ℃预变性5 min,95 ℃变性30 s,58 ℃退火30 s,72 ℃延伸30 s,扩增50个循环;最后72 ℃补充延伸5 min。扩增完毕取5 μL PCR产物以2%的琼脂糖凝胶电泳检测。
1.2.1.4 单链模板制备及焦磷酸测序 取PCR产物10 μL加ddH2O补至50 μL,在产物中分别加入磁珠3 μL和结合缓冲液47 μL,常温震荡混匀10 min;打开真空泵,将真空预装工具在超纯水中清洗30 s后抓取磁珠,分别在70%乙醇、变性缓冲液、洗涤缓冲液中清洗5~10 s,将磁珠移至预先加入0.3 μmol/L测序引物BoS和退火缓冲液的PSQ96孔板中;将此PSQ 96板放在ThermoPlate上在80 ℃放置3 min,取出自然冷却至室温;设定测序程序及碱基投放顺序和循环数,根据程序给定的剂量,在试剂舱中相应的孔中加入酶混合物、底物混合物以及4种碱基,将试剂舱和96孔测序板放入机箱进行测序反应。反应完毕,仪器自动给出测序结果,对测序结果进行在线Blast分析。
1.2.1.5 包纳米虫种类的确定 根据OIE手册[16],B.ostreae和B.exitiosa能被限制性内切酶HaeII酶切为115和189bp,B.roughleyi不能被酶切;B.ostreae被限制性内切酶BglI酶切为120和180bp,B.exitiosa和B.roughleyi不被酶切。对测得序列进行酶切位点分析,如果测得序列中同时含有HaeII和BglI两个酶切位点,则可以判断感染的包纳米虫种类为B.ostreae;如果测得序列中只含有HaeII酶切位点,不含有BglI酶切位点,则可以判断感染的包纳米虫种类为B.exitiosa;如果测得序列中不含有HaeII和BglI两个酶切位点,则可以判断感染种类为B.roughleyi或其他非检疫性包纳米虫。
1.2.2 DNA的提取 按照试剂盒DNeasy Blood and Tissue kit(QIAGEN)操作步骤提取荷兰、法国以及阴性牡蛎鳃组织样品的DNA,-20 ℃保存备用。
1.2.3 焦磷酸测序方法检测进口牡蛎样品 以所建立的包纳米虫焦磷酸测序方法对荷兰和法国进口牡蛎样品进行检测,同时设立阴阳性对照。
1.2.4 PCR-RFLP方法检测进口牡蛎样品 以OIE推荐的PCR-RFLP方法[16]对荷兰和法国进口牡蛎样品进行检测,并比较焦磷酸测序方法和PCR-RFLP方法的检测结果。
2 结果
2.1克隆及测序结果
将克隆的阳性质粒送北京诺赛基因组研究中心有限公司测序,结果经在线BLAST分析,与B.ostreae相关基因的序列一致性达100%、Bonamiasp.相关基因的序列一致性达99%。提取质粒,用核酸蛋白测定仪(NanoDrop ND-100)进行测定,拷贝数为1.13×1011拷贝/μL,-20 ℃保存备用。
2.2 包纳米虫焦磷酸测序方法的建立

(M:DL-2000 DNA Marker;1、2:B.ostreae阳性重组质粒的PCR扩增产物。M:DL-2000 DNA Marker;1、2:The PCR products ofB.ostreaerecombinant plasmid.)
图1 重组质粒焦磷酸测序PCR扩增产物的电泳结果
Fig.1 The PCR products electrophoresis result of recombinant plasmid

图2 重组质粒PCR产物的焦磷酸测序结果
以构建的包纳米虫阳性质粒为模板,以焦磷酸测序引物进行PCR扩增,扩增完毕以2%的琼脂糖凝胶电泳检测,电泳结果见图1。取适量PCR产物按照焦磷酸测序的具体操作说明进行试验,可很好的测得重组质粒PCR产物的序列,测序结果见图2,测得的序列为:AATAATAAGACACGACTTCGGCGCCGCCT-CGGCGGTTGTTTT。将此序列在网上进行Balst比对分析结果见图3,从图中可以看出序列一致性在66%以上的均为包纳米虫的序列,而且与参照序列的一致性可达100%,因此可以通过此方法来确定样品是否为包纳米虫感染。对测得序列进行酶切位点分析,该段测得序列中既含有BglI酶切位点,又含有HaeII酶切位点,由此可以判定感染种类为B.ostreae。

图3 测得序列在线Blast分析结果
2.3 包纳米虫PCR-焦磷酸测序方法的样品检测结果
采用包纳米虫焦磷酸测序PCR方法对荷兰和法国牡蛎样品DNA进行扩增,并进行电泳检测(见图4,法国牡蛎未见扩增条带电泳图省略)。抽取有目的条带的样品和几个没有目的条带的样品进行焦磷酸测序试验。有目的条带的样品测得序列均为:AATAATAAGACACGACTTCGGCGCCGCCTCGGCGGTTGT-TTT;而没有目的条带的样品则没有测到序列。经焦磷酸测序检测,所检样品中有6个样品有目的条带,测序后进行酶切位点分析,可见测得序列中均含有BglI和HaeII两个酶切位点,由此可判定6个样品均为B.ostreae感染。
2.4 样品的包纳米虫PCR-RFLP检测结果

(1~12为12个荷兰牡蛎样品;M:DL-2000 DNA Marker;阳性:B.ostreaeDNA;阴性:无B.ostreae感染的牡蛎DNA。1~12:12 oysters from Netherlands;M:DL-2000 DNA Marker;Positive control:B.ostreaeDNA; Negative control: oyster DNA withoutB.ostreaeinfection.)
图4 荷兰牡蛎的焦磷酸测序PCR扩增产物电泳结果
Fig.4 The electrophoresis result of pyrosequencing PCR products of oysters from Netherlands
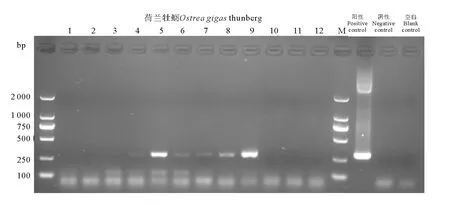
(1~12为12个荷兰牡蛎样品;M:DL-2000 DNA Marker;阳性:B.ostreaeDNA;阴性:无B.ostreae感染的牡蛎DNA;空白:双蒸水。1~12:12 oysters from Netherlands; M:DL-2000 DNA Marker; Positive control:B.ostreaeDNA; Negative control: oyster DNA withoutB.ostreaeinfection; Blank control: ddH2O.)
图5 荷兰牡蛎的PCR产物电泳检测结果
Fig.5 The electrophoresis result of PCR products of oysters from Netherland
经检测,荷兰牡蛎有6份样品出现了300bp的阳性条带(见图5),而法国牡蛎的17个样品均未见预期大小的目的条带(图略)。6份出现目的条带的荷兰样品PCR扩增产物分别用限制性内切酶BglI和HaeII进行酶切,2%琼脂糖凝胶电泳结果表明:目的基因的扩增产物可同时被限制性内切酶BglI和HaeII酶切,产生特异的酶切片段(见图6)。根据OIE手册中的描述[16]可将目的基因鉴定为B.ostreae,该检测结果与焦磷酸测序结果一致,2种方法的样品检测结果均显示4、5、6、7、8、9号样品为牡蛎包纳米虫感染阳性。

(M:DL-2000 DNA Marker; 4B:4号样品PCR产物的BglI酶切结果;4H:4号样品PCR产物的HaeII酶切结果;5B:5号样品PCR产物的BglI酶切结果;5H:5号样品PCR产物的HaeII酶切结果;6B:6号样品PCR产物的BglI酶切结果;6H:6号样品PCR产物的HaeII酶切结果;7B:7号样品PCR产物的BglI酶切结果;7H:7号样品PCR产物的HaeII酶切结果;8B:8号样品PCR产物的BglI酶切结果;8H:8号样品PCR产物的HaeII酶切结果;9B:9号样品PCR产物的BglI酶切结果;9H:9号样品PCR产物的HaeII酶切结果。M: DL-2000 DNA Marker; 4B: TheBglI enzyme digestion result of the PCR product No.4; 4H: TheHaeII enzyme digestion result of the PCR product No.4; 5B: TheBglI enzyme digestion result of the PCR product No.5; 5H: TheHaeII enzyme digestion result of the PCR product No.5; 6B: TheBglI enzyme digestion result of the PCR product No.6; 6H: TheHaeII enzyme digestion result of the PCR product No.6; 7B: TheBglI enzyme digestion result of the PCR product No.7; 7H: TheHaeII enzyme digestion result of the PCR product No.7; 8B: TheBglI enzyme digestion result of the PCR product No.8; 8H: TheHaeII enzyme digestion result of the PCR product No.8; 9B: TheBglI enzyme digestion result of the PCR product No.9; 9H: TheHaeII enzyme digestion result of the PCR product No.9.)
图6 4-9号荷兰牡蛎样品PCR扩增产物的酶切分析结果
Fig.6 The enzyme digestion result of PCR products of positive oysters from Netherlands
3 讨论
Cochennec等[17]报道被牡蛎包纳米虫感染的牡蛎大部分表现正常,一般不出现临床症状,但影响贝类的生长速度。我国是世界最大的水产贝类养殖国和出口国,虽然目前尚未报道有牡蛎包纳米虫病的发生,但其一旦发生,无法用药物根除,在感染的前6年,会造成很高的流行率和死亡率,将会对我国的贝类养殖造成很大的经济损失,而且我国对牡蛎包纳米虫的研究尚处于初级阶段,存在着包纳米虫潜在入侵的极大风险。焦磷酸测序技术是近几年来发展起来的一种能够进行定量序列测定的新技术,其基本原理是通过PCR制备待测序DNA模板,其中PCR的一条引物用生物素标记。PCR产物和偶联抗生物素蛋白的磁珠孵育,DNA双链经碱性分开,纯化得到含生物素标记引物的待测序单链,并和测序引物结合成杂交体,然后进行焦磷酸测序[18]。为做到防患于未然,本研究根据焦磷酸测序的原理,根据GenBank中包纳米虫的保守序列利用焦磷酸测序仪携带的引物设计软件设计测序专用的PCR扩增引物(生物素标记)和测序引物,对样品进行PCR扩增,然后对PCR产物进行单链分离、上机进行焦磷酸测序,结果显示,测得序列为AATAATAAGA CACGACTTCG GCGCCGCCTC GGCGGTTGTT TT的42个碱基,网上Blast分析比对,序列一致性在66%以上的均为包纳米虫序列,可以确定这42个碱基为包纳米虫序列,因此可以此判断是否为包纳米虫感染,进一步分析测得序列中是否含有BglI和HaeⅡ两个酶切位点,根据OIE标准[16]可以区分包纳米虫的感染种类。因此,本研究首次建立了包纳米虫的PCR-焦磷酸测序检测方法。
本研究采用新建立的包纳米虫PCR-焦磷酸测序检测方法对温州出入境检验检疫局提供的荷兰和法国进口牡蛎样品进行包纳米虫检测,同时辅以OIE推荐的PCR-RFLP方法检测,检测结束后对比2种方法的检测结果。结果发现,2种方法均在荷兰牡蛎中检测到了包纳米虫感染,而法国牡蛎中未检测到包纳米虫感染。焦磷酸测序方法通过对测得序列的酶切位点分析可以确定感染种类为牡蛎包纳米虫,同时PCR-RFLP方法也证实感染种类为牡蛎包纳米虫,2种方法的检测结果一致。
综上,本研究将焦磷酸测序原理应用到水产寄生虫病的检测研究中,建立了包纳米虫的PCR-焦磷酸测序检测方法,并应用于口岸疫病的监测中,不但为水产寄生虫的研究提供了一个新思路,而且为口岸疫病的高通量检测提供了新技术,相信随着不断的研究与完善,焦磷酸测序技术的应用将会越来越广泛,该方法的高通量和简便快速将会为日益纷繁复杂的检测业务提供很大帮助。
[1] Hine P M, Cochennec-Laureau N, Berthe F C J.Bonamiaexitiosusn. sp. (Haplosporidia) infecting flat oystersOstreachilensis(Philippi) in New Zealand [J]. Dis Aquat Org, 2001, 47: 63-72.
[2] Cochennec N, Reece K S, Berthe F C J, et al. RevisitingMikrocytosroughleyitaxonomic affiliation points to the genusBonamia(Haplosporidia)[J]. Dis Aquat Org, 2003, 54: 209-217.
[3] Carnegie R B, Burreson E M, Hine P M, et al.Bonamiapersporan. sp. (Haplosporidia), a parasite of the oysterOstreaolaequestris, is the firstBonamiaspecies known to produce spores [J]. J Eukaryot Microbiol, 2006, 53: 232-245.
[4] Kroeck M A, Montes J. Occurrence of the haemocyte parasiteBonamiasp. In flat oystersOstreapuelchanafarmed in San Antonio Bay (Argentina) [J]. Dis Aquat Organ, 2005, 63(2-3): 231-235.
[5] Narcisi V, Mascini M, Perez G, et al. Electrochemical genosensors for the detection ofBonamiaparasite. Selection of single strand-DNA (ssDNA) probes by simulation of the secondary structure folding [J]. Talanta, 2011, 85 (4): 1927-1932.
[6] Pichot Y, Comps M, Tigé G, et al. Recherches surBonamiaostreaegen. n., sp. n., parasite nouveau de l’hutre plateOstreaedulisL[J]. Rev Trav Inst Pêche Marit, 1980, 43: 131-140.
[7] Culloty S C, Mulcahy M F. Living with bonamiasis: Irish research since 1987[J]. Hydrobiologia, 2001, 465: 181-186.
[8] Boudry P, Chatain B, Naciri-Graven Y, et al. Genetical improvement of marine fish and shellfish: A French perspective[J]. Proceedings of FOID, 1996, 96(5): 141-150.
[9] Culloty S C, Mulcahy M F. Season, age, and sex-related variations in the prevalence of bonamiasis in flat oyster (OstreaedulisL. ) on the south coast of Ireland [J]. Aquaculture, 1996, 144(144): 53-63.
[10] Carnegie R B, Cochennec-Laureau N. Microcell parasites of oysters: Recent insights and future trends [J]. Aquat Living Resour, 2004, 17(4): 519-528.
[11] Bower S M, Mcgladdery S E, Price I M. Synopsis of infectious diseases and parasites of commercially enphited shellfish[J]. Annu Rev Fish Dis, 1994, 4(94): 1-199.
[12] 李海燕, 吴绍强. 牡蛎包拉米虫病的研究进展[J]. 中国畜牧兽医, 2008, 35(10): 104-107.
Li H Y, Wu S Q. Research progress of Bonamiasis caused byBonamiaOstreae[J]. Chin Anim Husb Vet Med, 2008, 35(10): 104-107.
[13] Zongzhi Liu, Catherine Lozupone, Micah Hamady, et al. Short pyrosequencing reads suffice for accurate microbial community analysis [J]. Nucleic Acids Res. 2007, 35(18): 120.
[14] Nyrén P. Enzymatic method for continuous monitoring of DNA polymerase activity [J]. Anal Biochem, 1987, 167(2): 235-238.
[15] 倪红兵, 王惠民. 焦磷酸测序技术及其应用进展[J]. 医学检验与临床, 2006, 17(2): 1-3.
Ni H B, Wang H M. Pyrosequencing and its application progress [J]. Med Lab Sci Clin. 2006, 17(2): 1-3.
[16] OIE. Infection withBonamiaostreae[S]. Paris: Manual of Diagnostic for Aquatic Animals, 2016.
[17] Cochennec N, Renault T, Boudry P. et al. Bonamia-like parasite found in the Suminoe oyster Crassostrea rivularis reared in France[J]. Dis Aquat Organ, 1998, 34(3): 193-197.
[18] 刘华雷, 吕艳, 黄伟坚, 等. 应用焦磷酸测序技术快速检测猪流感病毒金刚烷胺耐药性的分子标签[J]. 微生物学报, 2010, 50(3): 395-399.
Liu H L, Lv Y, Huang W J, et al. Detection of molecular markers of amantadine resistance in swine influenza viruses by pyrosequencing[J]. Acta Microbiologica Sinica, 2010, 50(3): 395-399.
DevelopmentandApplicationofPyrosequencingMethodDetectingBonamiaspp.CausedDiseaseofBivalves
WANG Cai-Xia1, WU Shao-Qiang1, LIN Xiang-Mei1, WANG Su-Hua2, FENG Chun-Yan1,XU Geng3
(1.The Institute of Animal Quarantine, Chinese Academy of Inspection and Quarantine, Beijing 100029, China; 2.Wenzhou Entry-Exit Inspection and Quarantine Bureau, Wenzhou 325027, China; 3. Bureau of Animal Husbandry and Veterinary in Yinan County of Shandong Province,Yinan 276300, China)
One pair of specific primers were designed and used to pyrosequencing the conserved region ofBonamiaDNA with PyroMark software basing on pyrosequencing platform andBonamiagene characteristics. PCR-pyrosequencing method was established with the recombinant plasmid ofBonamiaspp. constructed with OIE PCR method as the template. The obtained sequence can be identified as the sequence ofBonamiaspp. through Blast analysis, and enzyme digestion site analysis identified the specie asBonamiaspp..The imported oysters were detected with the newly developed method and PCR-RFLP as well as was recommended by OIE. The result showed that the developed PCR-pyrosequencing method can be used to correctly identifyingBonamiaspp. infection and species. The detection results were consistent with that yielded with OIE method. Therefore, this method can be applied to the detection and identification ofBonamiaspp. infection in port.
Oyster;Bonamiaspp.; PCR; pyrosequencing; detection; identification
S944
A
1672-5174(2017)12-053-06
责任编辑 朱宝象
10.16441/j.cnki.hdxb. 20160408
王彩霞, 吴绍强, 林祥梅, 等. 贝类包纳米虫病焦磷酸测序检测方法的建立与应用[J]. 中国海洋大学学报(自然科学版), 2017, 47(12): 53-58.
WANG Cai-Xia, WU Shao-Qiang, LIN Xiang-Mei, et al. Development and application of pyrosequencing method detectingBonamiaspp. caused disease of bivalves [J].Periodical of Ocean University of China, 2017,47(12): 53-58.
国家科技支撑计划项目(2013BAD12B02)资助
Supported by National Key Technologies R&D Program(2013BAD12B02)
2016-12-11;
2017-03-08
王彩霞(1982-),女,助理研究员。E-mail:friday128@sina.com
** 通讯作者:E-mail:sqwu@sina.com
