实时定量PCR检测新生儿全血中肺炎克雷伯菌方法的建立和应用
2017-10-24章晟庄璐李秋平宋婕张玉佩朱丽敏赵丹华王瑞娟封志纯
章晟,庄璐,李秋平,宋婕,张玉佩,朱丽敏,赵丹华,王瑞娟,封志纯
陆军总医院 附属八一儿童医院;出生缺陷防控关键技术国家工程实验室;儿童器官功能衰竭北京市重点实验室;北京 100007
实时定量PCR检测新生儿全血中肺炎克雷伯菌方法的建立和应用
章晟,庄璐,李秋平,宋婕,张玉佩,朱丽敏,赵丹华,王瑞娟,封志纯
陆军总医院 附属八一儿童医院;出生缺陷防控关键技术国家工程实验室;儿童器官功能衰竭北京市重点实验室;北京 100007
目的:建立实时定量PCR(RQ-PCR)快速检测新生儿血液标本中肺炎克雷伯菌基因组DNA的方法,并进行初步临床应用。方法:选择肺炎克雷伯菌高度保守的二鸟苷酸环化酶基因作为靶基因,设计特异性引物和TaqMan探针,建立RQ-PCR反应体系;采用含靶基因扩增片段的重组质粒建立标准曲线,提取血液中的基因组DNA,对500份新生儿血液标本进行RQ-PCR检测。结果:设计的引物和探针特异性高,检测限为1个拷贝数;500份新生儿血液标本中,RQ-PCR检测为阳性的有18个病例,阳性率为3.6%,明显高于血培养的阳性率2.4%(12个病例),差异具有统计学意义(P<0.01)。结论:RQ-PCR可用于检测新生儿血液标本中的肺炎克雷伯菌,此方法快速、灵敏、特异性强。
实时定量PCR;新生儿;肺炎克雷伯菌;血标本
新生儿重症监护病房收治出生小于28 d的新生儿,包括(极)早产儿、极(超)低出生体质量儿、足月重症新生儿等病情严重的患儿,新生儿本身刚脱离子宫内环境骤然转变至宫外,对外界适应性差,自身免疫系统不够完善,加上患儿病情较重、住院时间长,并且经常进行侵入性操作,极易并发院内感染,往往导致患儿病情加重,甚至是死亡[1-3]。在新生儿重症监护病房中血液感染是最主要的感染途径,而肺炎克雷伯菌占到很高的比例[4-6],因此快速检测出血液中的肺炎克雷伯菌,对于指导临床抗感染治疗具有重要意义。随着分子生物学技术的快速发展,实时定量PCR(RQ-PCR)技术在细菌、病毒的快速检测方面得到广泛应用[7-8]。基于此,我们研究建立了能够快速、高效地检测新生儿全血中肺炎克雷伯菌基因组DNA的RQ-PCR方法,可为新生儿感染的早期诊断和治疗提供参考。
1 材料与方法
1.1 材料
肺炎克雷伯菌(NCTC 5056)及其他常见病原菌[大肠埃希菌(NCTC 10538)、金黄色葡萄球菌(ATCC 29123)、表皮葡萄球菌(ATCC 12228)、溶血葡萄球菌(X03)、单核增生李斯特菌(ATCC 19115)、鲍曼不动杆菌(ATCC 19606)、铜绿假单胞菌(ATCC 9027)、屎肠球菌(ATCC 6057)]均购自中国普通微生物菌种保藏管理中心。大肠埃希菌以LB培养基培养,肺炎克雷伯菌、金黄色葡萄球菌、表皮葡萄球菌、溶血葡萄球菌、鲍曼不动杆菌、铜绿假单胞菌以营养肉汁琼脂培养,单核增生李斯特菌、屎肠球菌以PYG培养基培养。
细菌基因组DNA提取试剂盒(天根生化科技有限公司);质粒小量提取试剂盒、凝胶回收试剂盒(Omega Bio-Tek公司);QIAamp DNA Blood Mini Kit(Qiagen公司);pMD 19-T载体、Ex Taq DNA聚合酶、Premix Ex Taq(Probe qPCR)(大连宝生物工程有限公司);ABI 7500荧光实时定量PCR仪(ABI公司);引物和探针由Invitrogen公司合成。
1.2 特异性引物及TaqMan探针设计
依据GenBank收录的肺炎克雷伯菌二鸟苷酸环化酶(diguanylate cyclase)基因全序列(NC_023333.1)。将二鸟苷酸环化酶基因序列在Gen⁃Bank基因库中进行比对,结果显示其具有良好的种间特异性。参照TaqMan探针和引物设计原则,将RQ-PCR反应的引物(上游引物:GCAGATAAT TCACGCCCAGC;下游引物:CCCGCTGGACGCCA TC;扩增片段长113 bp)和探针(FAM-CCACCAC GCTCATCTGTTTCGCC-BHQ1)选取在二鸟苷酸环化酶基因的保守区,并将PCR扩增序列同样在基因数据库中进行比对,结果显示引物和探针具有良好的特异性。
1.3 细菌基因组DNA提取
采用天根公司的细菌基因组DNA提取试剂盒分别提取大肠埃希菌、金黄色葡萄球菌、表皮葡萄球菌、溶血葡萄球菌、单核增生李斯特菌、鲍曼不动杆菌、铜绿假单胞菌、屎肠球菌的基因组DNA,其中革兰阳性细菌(金黄色葡萄球菌、表皮葡萄球菌、溶血葡萄球菌、单核增生李斯特菌、屎肠球菌)先用溶菌酶处理4 h后再进行提取。
1.4 引物有效性及特异性验证
以提取的细菌基因组DNA及健康人基因组DNA 为模板进行 PCR,反应体系为 50 μL(10×PCR 缓冲 液 5 μL,10 μmol/L 上、下游引物 各1.25 μL,10 mmol/L dNTP 1 μL,5 U/μL Ex Taq DNA 多聚酶 0.2 μL,模板 DNA 2 μL,灭菌去离子水 39.3 μL)。反应条件:94℃ 3 min;94℃ 60 s,56℃ 30 s,72℃ 30 s,35 个 循 环 ;72℃ 5 min。PCR反应结束后,经琼脂糖凝胶电泳检测。
1.5 探针有效性及特异性验证
以提取的细菌基因组DNA及健康人基因组DNA为模板进行RQ-PCR,反应体系为20 μL(Premix Ex Taq 10 μL,10 μmol/L上、下游引物各 0.5 μL,0.2 μmol/L TaqMan 探针 1 μL,模板DNA 5 μL,ROX Reference DyeⅡ 0.2 μL,灭菌去离子水2.8 μL)。采用ABI 7500荧光定量PCR仪进行检测。反应条件:95℃ 2 min;95℃ 5 s,60℃ 40 min,40个循环。
1.6 重组质粒标准品的构建及提取
以肺炎克雷伯菌基因组为模板进行普通PCR,扩增产物用凝胶回收试剂盒纯化,纯化后的DNA片段与pMD 19-T载体连接,将连接产物转化大肠杆菌DH5α感受态细胞,进行蓝白斑筛选,挑选白色的单克隆菌落增菌培养、测序,测序结果经比对与靶基因片段序列一致后再次增菌培养,用质粒小量提取试剂盒提取重组质粒pMD 19-T/KP,用紫外分光光度计测定重组质粒的纯度(D260nm/D280nm)及浓度。根据pMD 19-T/KP质粒在溶液中的浓度与拷贝数换算方法,按1D=2.65×1010拷贝/μL,计算所提取质粒DNA的单位拷贝数,分装后于-70℃保存。
1.7 灵敏度检测
取106~100拷贝/μL连续浓度梯度的标准品作为模板,每个反应体系中加入1 μL模板,即每个反应检测的拷贝数为106~100拷贝,同时设立灭菌去离子水和健康人全血基因组2种阴性对照,选定标准曲线任务进行RQ-PCR反应,反应结束后用SDS软件自动绘制标准曲线,根据相关系数、扩增效率等参数,确定标准曲线范围,以上实验经多次重复,验证其准确性。
1.8 RQ-PCR检测血液中肺炎克雷伯菌预实验
在 200 μL 健康人全血中分别加如 105、103、10 cfu的肺炎克雷伯菌,模拟感染肺炎克雷伯菌的全血标本。用QIAamp DNA Blood Mini Kit提取全血标本基因组DNA,通过RQ-PCR反应检测肺炎克雷伯菌的基因组DNA,每个反应中加入模板5 μL。以上实验重复3次,以验证其准确性。
1.9 RQ-PCR检测临床标本中的肺炎克雷伯菌
对500份取自本院新生儿监护病房的疑似感染的全血标本进行肺炎克雷伯菌基因组DNA检测,方法同前,每个RQ-PCR反应中加入模板5 μL,阳性结果的样本复检1次,复检一致的样本,根据标准曲线推算样本拷贝数。对RQ-PCR结果与同时采集的血培养、常规培养鉴定结果进行平行比较,其中常规鉴定方法使用VITEK-2全自动微生物分析系统。
1.10 统计分析
所收集的数据采用SPSS 20.0软件进行分析处理,统计学差异分析采用Fisher精确检验,P<0.05为差异有统计学意义。
2 结果
2.1 引物特异性检测
经过琼脂糖电泳检测,仅在肺炎克雷伯菌泳道的110 bp处可见目标条带,其余各泳道未见扩增条带(图1),说明PCR引物只能特异性地扩增肺炎克雷伯菌的基因组DNA。
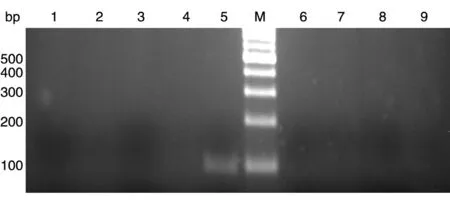
图1 肺炎克雷伯菌引物PCR扩增产物电泳图
2.2 探针特异性检测
以9种细菌基因组DNA、人基因组DNA为模板进行RQ-PCR,结果只有加入肺炎克雷伯菌基因组DNA的反应孔中才有荧光信号出现,其他各反应孔均无荧光信号(图2),说明引物和探针能特异性地检测肺炎克雷伯菌基因组DNA。

图2 肺炎克雷伯菌RT-PCR特异性检测结果
2.3 重组质粒的构建及测序
将筛选的质粒pMD 19-T/KP测序结果与GenBank所收录的序列进行比对,结果显示pMD 19-T/KP插入序列与靶基因序列一致(图3)。
图3 pMD 19-T/KP插入序列色谱图
2.4 灵敏度检测
多次RQ-PCR结果显示,106~100拷贝的pMD 19-T/KP质粒均能被检测到,Ct值随拷贝数的减少而递增,且曲线基本符合线性关系,而以灭菌去离子水和健康人全血基因组为模板的阴性对照未检测到荧光信号,因此RQ-PCR检测肺炎克雷伯菌基因组DNA的最低检测限为1个拷贝数(图4)。
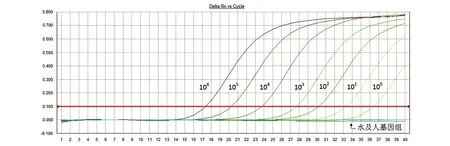
图4 肺炎克雷伯菌RT-PCR灵敏度检测结果
2.5 RQ-PCR检测血液中肺炎克雷伯菌预实验
将模拟感染肺炎克雷伯菌的全血标本提取基因组DNA,通过RQ-PCR检测,加入不同浓度肺炎克雷伯菌的标本均能检测到荧光信号,且Ct值随浓度的减少而递增,而提取健康人的全血标本基因组DNA为模板的反应中未检测到荧光信号(图5)。多次实验结果均一致。
2.6 RQ-PCR检测临床标本中的肺炎克雷伯菌
采用RQ-PCR对500份疑似感染的全血标本进行肺炎克雷伯菌基因组DNA检测,检出阳性标本18份,通过公式推算,检测到的拷贝数为2~89;常规培养鉴定结果为阳性的标本有12份。500份标本中,2种方法鉴定均为阳性的10份,RQ-PCR结果为阳性、常规培养鉴定为阴性的标本有8份,RQ-PCR结果为阴性、常规培养鉴定为阳性的标本有2份,2种方法鉴定均为阴性的标本有480份。因为有理论频数小于1,所以采用Fisher精确检验进行统计学分析,P=0.000<0.05,2种检测方法结果之间存在统计学差异(表1)。

图5 RT-PCR检测血液中肺炎克雷伯菌预实验结果
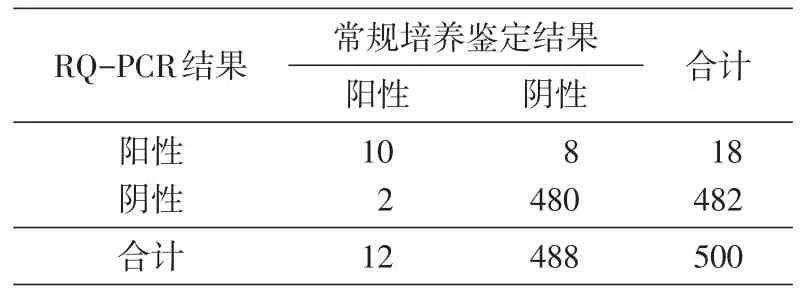
表1 RT-PCR与常规培养鉴定检测结果比较
3 讨论
近年来随着围产医学,尤其是新生儿重症监护病房(NICU)的快速发展,危重新生儿的存活率不断提高。但是由于新生儿的免疫功能低下、住院时间长、侵入性操作多、广谱抗生素广泛应用,导致NICU中感染发生率较高,其中肺炎克雷伯菌的感染发生率不断提高,已成为NICU院内感染的主要菌株之一。肺炎克雷伯菌侵入新生儿血液后,如果不能得到及时的治疗,极易导致新生儿败血症的发生,可迅速出现多脏器功能衰竭,并发休克,病死率高,严重威胁新生儿的生命健康[9-10]。
细菌培养鉴定是目前检测肺炎克雷伯菌的最常用方法,一般需要经过培养、分离、生化反应或质谱鉴定等步骤,虽然目前已有多种半自动或全自动微生物培养、鉴定系统,但整个过程至少需要48 h,耗时耗力,而且细菌检出阳性率不高。但是新生儿病情发展快,如何快速鉴定病原菌,对于临床治疗具有重要意义。核酸检测是目前细菌快速检测的发展方向,它不需要进行细菌培养,而是直接检测样本中的细菌基因组DNA,可大大缩短检测时间;并且不再局限于细菌形态结构及生理特性,具有较高的准确性[11]。目前细菌核酸检测方法包括普通PCR检测[12]、实时荧光PCR检测[13]、基因芯片检测[14]、基因组测序[15]。普通PCR检测准确度不够、特异性差,而基因芯片检测和基因组测序技术要求高、检测成本高。相比较而言,实时荧光PCR检测具有较高的敏感性、特异性和稳定性,更适合医院开展临床细菌检测工作。此外,实时荧光PCR检测全血标本时需要的全血量较少,200~500 μL 即可,而普通血培养需要3~5 mL,针对新生儿体重轻、血量少的特点,减少采血量更有意义[16]。
本研究根据肺炎克雷伯菌二鸟苷酸环化酶基因的保守序列设计了特异性引物和探针,在RQ-PCR反应中,加入肺炎克雷伯菌基因组DNA的反应孔能检测到荧光信号,而加入其他几种常见病原菌基因组DNA的反应孔中均无荧光信号,说明引物和探针具有较高的特异性。用克隆到pMD 19-T载体上的二鸟苷酸环化酶基因DNA片段作为标准DNA模板建立标准曲线,多次结果显示本方法的Ct值与质粒拷贝数呈线性相关,最低检测限为1个拷贝,说明引物和探针具有较高的敏感性和稳定性。
对500例临床标本进行RQ-PCR检测,阳性率为3.6%,而常规培养鉴定方法的阳性率为2.4%,通过统计学计算,2种方法检测结果之间存在统计学差异,说明RQ-PCR的灵敏度优于常规培养鉴定方法。有8个标本RQ-PCR结果为阳性而常规培养鉴定未检出,可能的原因是:①菌体量较少会导致无法培养出细菌;②经过抗生素治疗后,血液内还残留有较少的死菌,可以通过RQ-PCR检测出来,但无法培养;③由于污染导致出现假阳性。有2个标本常规培养鉴定结果为阳性而RQ-PCR结果为阴性,可能的原因是:①提取过程中细菌基因组DNA未结合到吸附柱上,导致细菌基因组DNA丢失;②提取的细菌基因组DNA溶液中混有血红蛋白等杂质,抑制RQ-PCR的反应;③常规培养过程中由于污染导致结果出现假阳性。
综上所述,我们建立的检测血液中肺炎克雷伯菌细菌基因组DNA的RQ-PCR方法,具有快速、灵敏度高、特异性强等优势,检测时间比常规细菌鉴定方法缩短48~72 h,检测灵敏度高于常规细菌培养结果。血液培养、常规培养鉴定是血液感染检测最为重要的金标准,而RQ-PCR检测方法可以作为有益补充,尤其是重症新生儿病情变化快,RQ-PCR能够快速得到检测结果,对于临床诊断、治疗具有重要参考价值。
[1]耿庆红,文海燕,郭瑞霞.NICU患儿感染病原菌分布及药敏试验分析[J].中华医院感染学杂志,2015,25(6):1276-1280.
[2]钟巧,李晖,高晓玲,等.NICU早产与足月新生儿败血症特点对比研究[J].中华医院感染学杂志,2014,24(1):211-213.
[3]Blencowe H,Vos T,Lee A C,et al.Estimates of neo⁃natal morbidities and disabilities at regional and glob⁃allevels for2010:introduction,methods overview,and relevant findings from the Global Burden of Dis⁃ease study[J].Pediatr Res,2013,74(Suppl 1):4-16.
[4]许亚茹,关毅,王淑娟,等.NICU医院感染病院菌传播途径与预防控制研究[J].中华医院感染学杂志,2014,24(13):3167-3171.
[5]王兆莉,谢建宁,戴怡蘅,等.新生儿医院感染败血症临床分析[J].中华医院感染学杂志,2014,24(21):5390-5392.
[6]邵波,胡小红,陆国平,等.NICU早产儿肺炎克雷伯菌医院感染临床分析[J].中华医院感染学杂志,2014,24(19):4882-4884.
[7]Duffy T,Cura C I,Ramirez J C,et al.Analytical per⁃formance of a multiplex real-time PCR assay using TaqMan probes for quantification of Trypanosoma cru-zi satellite DNA in blood samples[J].PLoS Negl Trop Dis,2013,7(1):e2000-2010.
[8]Yang S,Lin S,Kelen G D,et al.Quantitative multi⁃probe PCR assay for simultaneous detection and identi⁃fication to species level of bacterial pathogens[J].J Clin Microbiol,2002,40(9):3449-3454.
[9]吕媛,任静,缪珀,等.新生儿肺炎克雷伯菌败血症临床分析[J].中国新生儿科杂志,2013,28(2):76-79.
[10]刘晓翠,元小冬,许亚茹,等.NICU患者感染肺炎克雷伯菌与耐药性研究[J].中华医院感染学杂志,2015,25(22):5066-5068.
[11]孙长贵,成军.分子诊断技术在临床微生物学检验中的应用[J].中华检验医学杂志,2013,36(2):109-112.
[12]Muyzer G,Waal E D,Uitterlinded A G.Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reac⁃tion-amplified genes coding for 16S rRNA[J].Appl En⁃viron Microbiol,1993,59(3):695-700.
[13]Tuomisto S,Karhunen P J,Vuento R,et al.Evalua⁃tion of postmortem bacterial migration using culturing and real-time quantitative PCR[J].J Forensic Sci,2013,58(4):910-916.
[14]Mancini N,Infurnari L,Ghidoli N,et al.Potential im⁃pact of a microarray-based nucleic acid assay for rap⁃id detection of gram-negative bacteria and resistance markers in positive blood cultures[J].J Clin Microbi⁃ol,2014,52(4):1242-1245.
[15]Hasman H,Saputra D,Sicheritz-Ponten T,et al.Rap⁃id whole-genome sequencing for detection and charac⁃terization of microorganisms directly from clinical sam⁃ples[J].J Clin Microbiol,2014,52(1):139-146.
[16]Pusterla N,Mapes S,Byrne B A,et al.Detection of bloodstream infection in neonatal foals with suspected sepsis using real-time PCR[J].Vet Rec,2009,165(4):
Establishment and Application of TaqMan Real-Time PCR for Detection ofKlebsiella pneumoniaein Neonatal Blood Sample
ZHANG Sheng,ZHUANG Lu,LI Qiu-Ping,SONG Jie,ZHANG Yu-Pei,ZHU Li-Min,ZHAO Dan-Hua,WANG Rui-Juan,FENG Zhi-Chun*
Affiliated Bayi Children's Hospital,Chinese People's Liberation Army General Hospital;National Engineering Labo⁃ratory for Birth Defects Prevention and Control of Key Technology;Beijing Key Laboratory of Pediatric Organ Fail⁃ure;Beijing 100007,China
Objective:To establish a real-time quantitative PCR(RQ-PCR) assay for detectingKlebsiella pneu⁃moniaegenomic DNA from neonatal blood sample,and preliminarily apply it in clinical test.Methods:The prim⁃ers and TaqMan probe were designed targeting the highly conserved diguanylate cyclase gene ofK.pneumoniae,and a amplification reaction system was established.The standard curve was built based on the recombinant plas⁃mid DNA containing the amplification of the target gene.The genomic DNA was extracted using QIAamp DNA Blood Mini Kit from five hundred neonatal blood sample,followed by RQ-PCR detection.Results:The specificity of primers and probe were high,the detecting limit was 1 copy.For five hundred neonatal blood sample,the posi⁃tive rate of RQ-PCR was 3.6%(18 patients),which was significantly higher than 2.4%(12 patients) by blood cul⁃ture.Conclusion:RQ-PCR assays is a rapid,sensitive,and specific method and can be used in the detection ofK.pneumoniaein neonatal blood samples.
real-time quantitative PCR;neonate;Klebsiella pneumoniae;blood sample
Q78
A
1009-0002(2017)05-0665-06
10.3969/j.issn.1009-
*Corresponding author,E-mail:fengzc81@sina.com
2017-03-01
中国博士后科学基金(2013M542472)
章晟(1980- ),男,副主任技师,(E-mail)ahswzs@sina.com
封志纯,(E-mail)fengzc81@sina.com
