lncRNA在食管鳞癌中的研究进展
2017-10-16常卫东赵盼盼王俐峰王林嵩
常卫东, 王 丽, 赵盼盼, 王俐峰, 王林嵩
(河南师范大学 生命科学学院, 新乡 453007)
lncRNA在食管鳞癌中的研究进展
常卫东, 王 丽, 赵盼盼, 王俐峰, 王林嵩
(河南师范大学 生命科学学院, 新乡 453007)
食管鳞癌(Esophageal squamous cell carcinoma, ESCC)是消化系恶性肿瘤中最致命的类型之一。长链非编码RNA(Long non-coding RNA,lncRNA)由于在多种水平参与基因表达调控,影响细胞增殖、分化、代谢和凋亡,其作用备受关注。首先简要介绍lncRNA的结构,并对近年来有关lncRNA在食管鳞癌中的研究进行综述。lncRNA通过表观遗传修饰、剪接调控和共表达网络等调控食管鳞癌相关基因表达,影响食管鳞癌发生发展,并有可能作为食管鳞癌的诊断和预后标志物以及治疗靶点。而lncRNA的二级结构预测有助于推进lncRNA的研究。
食管鳞癌; 长链非编码RNA;作用机制;结构预测
AbstractEsophageal squamous cell carcinoma(ESCC)is one of the most fatal malignant tumors in digestion system. Long non-coding RNA(LncRNA)has become a research hotspot since it is not only involved in gene expression and regulation in different levels, but also affects cell proliferation, differentiation, metabolism and apoptosis. In this review, we first briefly introduce the structure of lncRNA and then summarize the recent research progresses of lncRNAs in the field of ESCC. LncRNAs regulate gene expression of esophageal squamous carcinoma associated genes by epigenetic modification, alternative splicing and co-expression network,furthermore, affect the development and progression of esophageal squamous cell carcinomas. Therefore, LncRNAs may serve as markers for ESCC diagnosis and prognosis and even serve as therapeutic targets. Secondary structure prediction of lncRNA is helpful to promote the research of lncRNA.
Keywordsesophageal squamous cell carcinoma; long non-coding RNA; mechanism; structure prediction
食管鳞癌是消化道肿瘤中最常见的类型,占全球癌症死亡率的第6位[1]。食管癌早期手术治疗切除率可达100%,5年生存率可达90%以上。因此,近年来人们一直致力于寻找食管鳞癌的早期诊断标志物,如癌胚抗原、miRNA等。但人类基因中只有2%的基因序列编码蛋白质,其余的则被转录成非编码RNA。而其中microRNA和lncRNA越来越受到人们的重视。本文就近几年lncRNA在食管鳞癌中的研究进展做一综述,希望能为今后食管鳞癌的诊断和治疗等提供新的思路。
1 lncRNA的结构
1990年Brannan等首次报道了lncRNA H19[2],开启了lncRNA的研究。长链非编码RNA 是一种内源性细胞RNA,长度>200个核苷酸[3]。在细胞质和细胞核中均有lncRNA,但是大部分lncRNA位于细胞质。lncRNA在不同物种之间保守性较低,只有约12%的lncRNA存在于人类之外的其他生物。随着对lncRNA越来越多的研究,lncRNA的数量也从7000显著增加到23 000[4]。结构是功能的基础,任何物质的功能发挥都离不开其特有的分子结构,研究一个物质的结构是了解其功能及其作用机制的必要前提。因此,lncRNA分子的结构解析对lncRNA的功能及作用机制研究同样具有非常重要的作用。不同于mRNA,lncRNA的基因内含子数量较少,有些缺乏内含子,CG含量低,缺乏起始密码子和开放阅读框[5]。poly(A) lncRNA的内含子和外显子的结构简单,将近一半的lncRNA只有两个外显子。尽管lncRNA有精确的组织特异性,但是和编码蛋白质的基因相比,lncRNA的表达水平明显偏低[4]。最近,基因组规模的方法被用于测量RNA的二级结构。研究发现,许多lncRNA都有一个重要的二级结构,对于它们的特异性结合和功能有重要作用[5]。lncRNA的二级结构包括双茎环和3′末端的三叶草结构等[6-7],这些复杂的结构使lncRNA具有调节能力。但是,迄今为止,还没有确定lncRNA的三级结构。
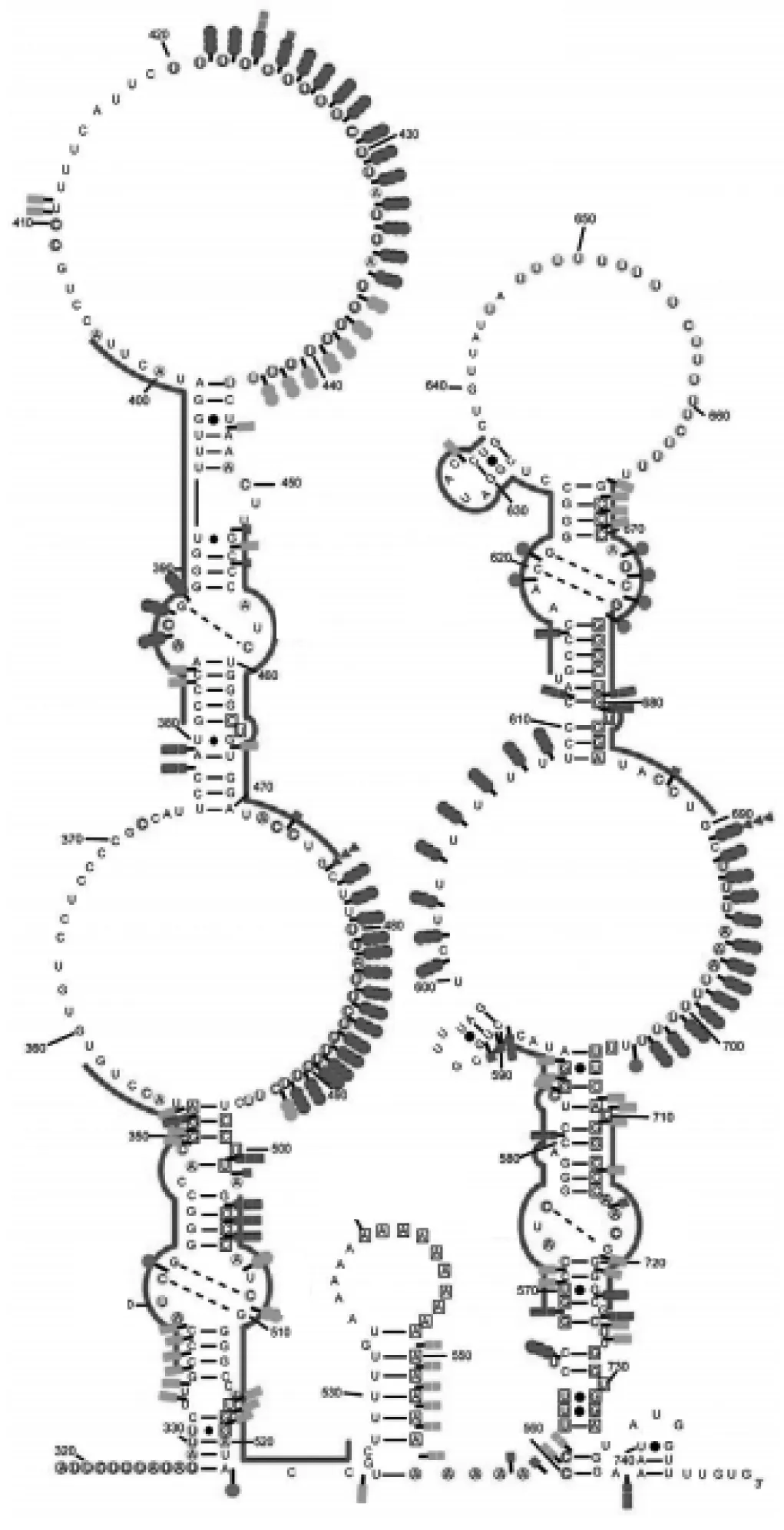
图1 lncRNA Xist的双茎环结构[7]
lncRNA的研究方法主要是分为生物方法和计算方法两种。生物方法主要包括微阵列(microarray)[8]、转录组测序(RNA-seq)[9]、荧光原位杂交 (fluorescence in situ hybridization,FISH)[8]以及CHART (capture hybridization analysis of RNA targets) 和 ChIRP (chromatin isolation by RNA purification)等技术,能够用于发现、鉴定lncRNA。计算方法主要是通过分析lncRNA发挥作用的理论研究,结合lncRNA的序列信息,利用动态规划算法、热力学最小自由能法、碱基最大配对法、比较序列分析法、组合优化法等[10]方法对lncRNA的二级结构、作用位点、功能等进行预测。但是生物方法需要消耗大量人力、物力和财力;计算方法太过复杂,同时预测结果的准确性还有待提高,需要考虑加入更多的lncRNA信息,完善预测模型。随着测序技术的飞速发展,lncRNA的数据量已经出现倍数甚至是指数级的增长,单一地采用生物方法或者计算方法已经不能满足现实的需求,计算方法和生物方法结合将是未来lncRNA预测的研究方向。
2 lncRNA在食管鳞癌中的作用机制
2.1 lncRNA参与染色质甲基化
lncRNA HOTAIR作为第一个被发现具有反式转录调控作用的lncRNA,在多种癌症中发挥关键作用。HOTAIR[11-12]作为一个分子支架,5′端结合多梳抑制复合体2(Polycomb repressive complex 2,PRC2),而其 3′端则能够和赖氨酸特异性组蛋白去甲基化酶l(Lysine specific demethylasel, LSD1)结合。HOTAIR介导这2种复合体结合到位于染色体2 的PRC2的靶点HOXD处,使HOXD位点上一段40 kb的区域发生转录沉默进而促进癌症的转移。
然而还有人提出了lncRNA HOTAIR在ESCC中作用的不同的机制。Ge等[13]和Li等[14]在对ESCC中HOTAIR进行分析之后发现了HOTAIR/WIF-1轴这种新的分子机制。由于WIF-1在Wnt /β-catenin 信号通路中的发挥重要作用,而HOTAIR可以直接减少WIF-1的表达,促进了组蛋白H3K27启动子区域甲基化,激活Wnt/β-catenin信号通路。解释了ESCC细胞转移的分子机制。但是WIF-1是如何在Wnt /β-catenin 信号通路中发挥作用,Wnt/β-catenin信号通路如何具体作用于ESCC细胞转移等方面还需要进一步的探索。
EZH2与lncRNA POU3F3也有类似的作用。首先,lncRNA POU3F3与EZH2(enhancer of zeste homolog 2)相互作用将DNA甲基转移酶募集到POU3F3的启动子区,对启动子进行甲基化。使POU3F3的表达显著降低,从而抑制其下游靶基因DLL1的转录。DLL1不仅能够影响细胞的凋亡,还是NOTCH通路的重要一员。至此,lncRNA POU3F3的调控信号经过复杂的过程最终通过DLL1传递到NOTCH通路,抑制NOTCH的表达。不仅能够促进食管内皮细胞向促血管生成因子迁移并增殖,之后内皮萌发形成管腔,而且还能够参与到ESCC的微血管和淋巴管的新生,促进ESCC的浸润转移。
除此之外,一些研究表明EZH2还可以和lncRNA HOTAIR结合,定位至HOXD位点,影响染色体组蛋白甲基化,重编程染色体状态,从而使该位点发生表观遗传学沉默[15]。由此可见,lncRNA通过调节甲基化酶的活性,改变DNA和组蛋白的甲基化水平,从而在染色质结构上调控基因转录,影响ESCC的发生发展。
2.2 lncRNA参与mRNA的选择性剪接
lncRNA不但在转录调控中发挥作用, 而且是mRNA前体剪接的调控因子。lncRNA MALAT1在剪接调控方面的作用已经在很多癌症中有报道,它与丝氨酸/精氨酸(Serine/arginine, SR)剪接因子相互作用, 并调控剪接因子在剪接点中的分布和磷酸化水平, 从而改变 mRNA 前体的选择性剪接模式。lncRNA MALAT1缺失引起SR剪接因子的一个重要部分脱磷酸化,使磷酸化和脱磷酸化的SR蛋白比例发生改变,进而通过两条途径影响剪接。一是影响 SR 剪接因子在核小点和转录部位间的分布,剪接因子在细胞的不同定位会使 mRNA 前体进行不同形式的剪接。二是SR蛋白的活性发生改变,连续的磷酸化和脱磷酸化可影响SR蛋白活性,进而决定mRNA前体是否进行剪接及进行何种剪接。在食管鳞癌中,lncRNA uc002yug.2[16]能够通过招募剪接因子SR选择性剪接调控CEBPα的表达。由于剪接因子SR含有RUNX1的结合位点,能够促进RUNX1的pre-mRNA的选择性剪接,从而产生更多的RUNX1a。相比于RUNX1的另两个剪接体,RUNX1a与靶基因CEBPα的亲和力更强。且RUNX1a能够发挥促癌因子的作用,通过和CEBPα的启动子和增强子序列的结合抑制CEBPα的表达,促进ESCC细胞的增殖和肿瘤的生长。
Wang等[17]发现lncRNA MALAT1通过与Ago2蛋白(Argonaute 2)结合,能够抑制MALAT1的功能,并导致下游基因P21和P27的表达水平升高,而B-MYB的表达水平下降,造成ESCC肿瘤细胞停滞在 G2/M 期。另外,还有众多研究表明lncRNA能够参与到p53沉默,促进细胞完成G1/S期向G2期的转化,并能够特异性地影响B-MYB和CENPE的表达,调节有丝分裂,控制细胞周期。
2.3 lncRNA的共表达网络
lncRNA作为近几年在肿瘤中被广泛研究的调控因子,除了在表观遗传和剪接调控等很多方面发挥作用外,Pan等[18]又在ESCC中发现了lncRNA FOXCUT和FOXC1之间的相互关系,预测了lncRNA-mRNA结合新的调控模式。由于FOXCUT位于FOXC1上游启动子区,所以FOXCUT能够竞争启动子抑制RNA聚合酶П,进而抑制FOXC1的表达。另外,由于EMT能够调节VEGF-A参与血管生成,并且FOXC1与EMT和VEGF-A之间的相互作用也已经在其他肿瘤中被证实,所以FOXCUT/FOXC1基因在ESCC肿瘤的血管生成中发挥重要作用。
Cao等[19]也发现当lncRNA ESCCAL-356在ESCC中下调时,其邻近共表达基因LPCAT1在ESCC中表达上调。LPCAT1能够在多种肿瘤组织中表达升高,与肿瘤的增殖、扩散以及恶性程度等显著相关。LPCAT1在乳腺癌中的作用机制可能与乳腺癌组织中脂质表达谱有关[20];在前列腺癌中可能与脂质代谢相关[21]。LPCAT1大多通过脂质的改变发挥作用,在ESCC中,LPCAT1催化溶血磷脂酰胆碱成磷脂酰胆碱来调节磷脂成分,促进癌细胞的增殖扩散。最近,Abdeizaher和Mostafa[22]还揭示了乳腺癌中lncRNA-mRNA的两种不同的共表达模式,可能涉及乳腺癌肿瘤的发生。由此可见,共表达模式在肿瘤的发生发展中起着重要作用。
3 lncRNA在食管鳞癌中的潜在临床应用
ESCC的5年生存率仅为15%~25%,为了提高其5年生存率,及早发现并治疗才是有效的方法。传统的癌症生物标志物,比如糖抗原和癌胚抗原,缺乏敏感性和特异性,不适用于普通人群筛查。所以找到一种有效的生物标志物尤为必要。近年来,由于miRNAs在食管鳞癌中的稳定性和可检测性,已经作为ESCC的诊断和预后的生物标志物被广泛研究。然而,最近人们发现lncRNA在某些癌症中可能和miRNAs发挥相似的生理功能,可以作为一种新的生物标志物。Fu等[23]利用微阵列探针分析了胰腺导管腺癌中lncRNA的表达谱信息,证明了多个lncRNA在胰腺导管腺癌中的潜在作用,有助于胰腺导管腺癌的精确诊断、预后以及治疗评估等。
现有许多研究表明,与肿瘤发生发展有关的lncRNA也与肿瘤的预后有关。Deng等[24]综述lncRNA中的HOTAIR、MALAT1、CCAT2等与乳腺癌、肺癌、结肠癌预后有关的lncRNA同样也与ESCC的预后有关。这些lncRNA在肿瘤组织中较正常组织表达明显增高,肿瘤分化程度较低,进展越迅速,其表达量越高(详见文献[24])。Zhao等[25]的实验研究了lncRNA BC200在ESCC中的表达及其作用,BC200在ESCC组织中表达上调,并且lncRNA BC200表达水平较高的患者生存率更低。这些结果表明,lncRNA可能作为ESCC的诊断预后标志物和治疗靶点。鉴于文献报道的与ESCC预后有关的lncRNA同样在其他肿瘤也存在。因此,新的与ESCC预后有关lncRNA的发现可以在新发现的其他肿瘤相关的lncRNA中寻找,同时寻找ESCC特异的lncRNA将是今后研究的一个热点。
[1]CHEN J, KWONG D L, CAO T, et al. Esophageal squamous cell carcinoma(ESCC): advance in genomics and molecular genetics[J]. Diseases of the Esophagus, 2015, 28(1):84-89.
[2]YANG X, XIE X, XIAO Y F, et al. The emergence of long non-coding RNAs in the tumorigenesis of hepatocellular carcinoma [J]. Cancer Letters, 2015, 360(2):119-124.
[3]HUARTE M. A lncRNA links genomic variation with celiac disease [J]. Science, 2016,352(6281):43-44.
[4]HAN D, WANG M, MA N, et al. Long noncoding RNAs: novel players in colorectal cancer[J]. Cancer Letters, 2015, 361(1):13-21.
[5]FANG X Y, PAN H F, LENG R X, et al. Long noncoding RNAs: novel insights into gastric cancer[J]. Cancer Letters, 2015, 356(2):357-366.
[6]POLLARD K S, SALAMA S R, LAMBERT N, et al. An RNA gene expressed during cortical development evolved rapidly in humans[J]. Nature, 2006, 443(7108):167-172.
[7]MAENNER S, BLAUD M, FOUILLEN L, et al. 2-D structure of the A region of Xist RNA and its implication for PRC2 association[J]. PLoS Bio, 2010, 8(1):e1000276.
[8]WANG P, XUE Y, HAN Y, et al. The STAT3-binding long noncoding RNA lnc-DC controls human dendritic cell differentiation[J]. Science, 2014, 344(6181):310-313.
[9]CASTELLANOS-RUBIO A, FERNANDEZ-JIMENEZ N, KRATCHMAROV R, et al. A long noncoding RNA associated with susceptibility to celiac disease[J]. Science, 2016, 352(6281):91-95.
[10]EDDY S R. Computational analysis of conserved RNA secondary structure in transcriptomes and genomes [J]. Annu Rev Biophys, 2014, 43:433-456.
[11]ZHOU X, REN Y, ZHANG J, et al. HOTAIR is a therapeutic target in glioblastoma[J]. Oncotarget, 2015, 6(10):8353-8365.
[12]BHAN A, MANDAL S S. LncRNA HOTAIR: a master regulator of chromatin dynamics and cancer[J]. Biochim Biophys Acta, 2015, 1856(1):151-164.
[13]GE X S,MA H J,ZHENG X H,et al. HOTAIR,a prognostic factor in esophageal squamous cell carcinoma,inhibits WIF-1 expression and activates Wnt pathway [J]. Cancer Sci, 2013, 104(12):1675-1682.
[14]LI X,WU Z,MEI Q,et al. Long non-coding RNA HOTAIR,a driver of malignancy,predicts negative prognosis and exhibits oncogenic activityinoesophageal squamous cell carcinoma[J]. Br J Cancer, 2013,109(8):2266-2278.
[15]WU Y S, ZHANG L, ZHANG L, et al. Long non-coding RNA HOTAIR promotes tumor cell invasion and metastasis by recruiting EZH2 and repressing E-cadherin in oral squamous cell carcinoma[J]. Int J Oncol, 2015, 46(6):2586-2594.
[16]SUN S, ZHANG Z, SINHA R, et al. SF2/ASF autoregulation involves multiple layers of post-transcriptional and translational control[J]. Nat Struct Mol Biol, 2010, 17(3):306-312.
[17]WANG X, LI M, WANG Z Q, et al. Silencing of long noncoding RNA MALAT1 by miR-101 and miR-217 inhibits proliferation, migration, and invasion of esophageal squamous cell carcinoma cells[J]. J Biol Chem, 2015, 290(7):3925-3935.
[18]PAN F, YAO J, CHEN Y, et al. A novel long non-coding RNA FOXCUT and mRNA FOXC1 pair promote progression and predict poor prognosis in esophageal squamous cell carcinoma.[J]. Int J Clin Exp Pathol, 2014,7(6):2838-2849.
[19]CAO W, WU W, SHI F, et al. Integrated analysis of long noncoding RNA and coding RNA expression in esophageal squamous cell carcinoma[J]. International Journal of Genomics, 2013, 2013:480-534.
[20]ABDELZAHER E, MOSTAFA M F. Lysophosphatidylcholine acyltransferase 1 (LPCAT1) upregulation in breast carcinoma contributes to tumor progression and predicts early tumor recurrence[J]. Tumour Biol, 2015, 36(7): 5473-5483.
[21]GRUPP K, SANADER S, SIRMA H, et al. High lysophosphatidylcholine acyltransferase 1 expression independently predicts high risk for biochemical recurrence in prostate cancers[J]. Mol Oncol, 2013, 7(6): 1001-1011.
[22]ABDELZAHER E, MOSTAFA M F. Lysophosphatidylcholine acyltransferase 1 (LPCAT1) upregulation in breast carcinoma contributes to tumor progression and predicts early tumor recurrence[J]. Tumor Biology, 2015, 36(7): 5473-5483.
[23]FU X L, LIU D J, YAN T T, et al. Analysis of long non-coding RNA expression profiles in pancreatic ductal adenocarcinoma[J]. Scientific Reports, 2016, 6:33535.
[24]DENG H Y, WANG Y C, NI P Z, et al. Long noncoding RNAs are novel potential prognostic biomarkers for esophageal squamous cell carcinoma: an overview[J]. Journal of Thoracic Disease, 2016, 8(8):E653-E659.
[25]ZHAO R H, ZHU C H, LI X K, et al. BC200 LncRNA a potential predictive marker of poor prognosis in esophageal squamous cell carcinoma patients[J]. OncoTargets and Therapy, 2016, 9:2221-2226.
The research development of lncRNA in esophageal squamous cell carcinoma
CHANG Wei-dong, WANG Li, ZHAO Pan-pan, WANG Li-feng, WANG Lin-song
(College of Life Sciences, Henan Normal University, Xinxiang 453007, China)
Q752;Q32;R735
A
2095-1736(2017)05-009-04
2016-08-29;
2016-09-13
河南省教育厅高校重点科研项目(编号:16A180028)
常卫东,硕士研究生,研究方向为生物化学与分子生物学,E-mail:446709355@qq.com
王林嵩,博士,教授,研究方向为生物化学与分子生物学,E-mail: wls@htu.cn
doi∶10.3969/j.issn.2095-1736.2017.05.089
