抗菌药物亚抑菌浓度及其临床意义
2017-10-16王方舟倪文涛崔俊昌
王方舟, 倪文涛, 崔俊昌
·综述·
抗菌药物亚抑菌浓度及其临床意义
王方舟, 倪文涛, 崔俊昌
亚抑菌浓度; 最小选择浓度; 细菌; 耐药
随着抗生素的广泛应用,细菌的耐药性越益严重,而新抗生素的研发速度却在明显下降[1-2],这使得细菌耐药,尤其是多重耐药成为目前威胁人类健康严重的公共卫生问题。为了抑制细菌耐药的产生和发展,制定临床合理的抗生素应用策略,了解细菌耐药产生机制就显得尤为重要。随着人们对细菌耐药产生机制研究的进展,临床抗生素的应用策略也在不断改变,由基于最低抑菌浓度(MIC)的治疗策略,发展为基于耐药突变选择窗(mutant selection window, MSW)理论的治疗策略。近年来,有学者又提出了与细菌耐药产生密切相关的亚抑菌浓度(subinhibitory concentration,Sub-MIC)和最小选择浓度(minimal selective concentration,minSC)概念[3-4],作为对MSW理论的补充,本文就相关研究进展做一综述。
1 Sub-MIC
1.1 Sub-MIC和MIC的概念
Sub-MIC指低于MIC而对细菌有选择作用的抗菌药物浓度,表明药物浓度低于MIC时细菌耐药性仍可增强。Troy将野生型与耐药突变型大肠埃希菌分别标记,以1∶1的比例混合,置于含不同浓度的环丙沙星、链霉素及四环素的培养基中培养,定期进行分类计数。结果显示抗菌药物浓度较低时,野生菌株数量多于突变菌株;但随着抗菌药物浓度增高,突变菌株数量逐渐增加,原因为突变菌株对环境的适应性逐渐增加[5]。突变菌株与野生菌株数量相同时,两者对环境适应性相同,此时抗生素浓度被定义为minSC。实验证实,minSC明显低于与其对应的MIC,minSC最高为MIC的1/4(抗链霉素rpsL105突变),最低仅为1/230(抗环丙沙星gyrASer83Leu突变)[6]。minSC是细菌产生的突变菌株可扩增的最低药物浓度,是Sub-MIC对细菌产生影响的阈值。当抗菌药物浓度低于minSC时,抗菌药物对细菌无选择作用;而高于minSC时,抗菌药物可选择性扩增耐药突变菌株。
1.2 Sub-MIC的理论依据
耐药突变菌需要在菌群中成为优势菌才能使细菌产生耐药性,不同环境中总是适应性强的菌群占据优势。细菌通过自发遗传突变和基因水平转移产生不同的耐药亚群(sub-population),不同亚群对环境有不同的适应性[6]。细菌产生的耐药突变在提高耐药性的同时通常会影响细胞正常功能,一般称之为适应性代价,因此,即使是高耐药的突变菌,也无法在所有环境中都占据优势。如果某种基因型在菌群中所占数量增加,表明此基因型对环境适应性强,反之则表明适应性差。需要注意的是,菌群绝对数量的变化并不能准确反映基因型的适应性,例如菌群数量不断减少,而此基因型所占的比例增加,表明这个基因型在此环境中仍有强适应性[7](图1)。传统MSW理论强调野生菌株对环境的适应性,防突变浓度(MPC),为MSW上限,药物浓度高于MPC时将会同时抑制野生菌株和耐药菌株生长。MIC定义为MSW下限,此时野生菌株生长速度几乎为零,突变菌株被选择性扩增。研究发现突变菌株在低于MIC时生长速度已经高于野生菌株,表明突变菌株在低于MIC环境中适应性强于野生菌株,MIC不适合作为判定药物对细菌是否有选择作用的分界点,应选择耐药菌株与野生菌株的生长速度相同时药物浓度,也就是minSC作为分界点,因为此时耐药菌株与野生菌株适应性相同。
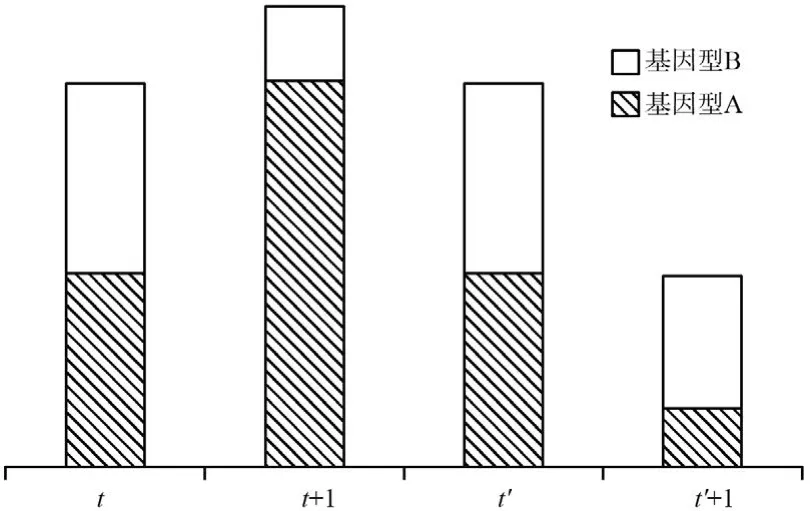
图1 环境适应性
Sub-MIC可增加细菌耐药突变的频率。自发突变是细菌获得耐药性的重要途径,目前人们还无法阻止自发突变产生,但其发生率通常为10-6~10-8[8]。因此需要一定基数的细菌才能产生耐药突变株。Sub-MIC下,氟喹诺酮类、氨基糖苷类及β内酰胺类等药物可通过启动细菌SOS系统、增加氧自由基、干扰DNA表达等多种途径增加细菌自发突变概率,最高可增至自然状态下的15倍[9]。
不同浓度抗生素对细菌会产生不同的选择作用。Sub-MIC下药物对细菌的选择作用需要克服适应性代价才能选择性扩增耐药株,因此更容易富集低适应性代价突变,而不是高耐药性突变菌株。Troy将20株野生型大肠埃希菌分别置于含Sub-MIC抗生素(1/4MIC链霉素,1/10MIC环丙沙星)的培养液中,连续培养并定期测定MIC。20株细菌在400代后对链霉素的MIC为实验开始时的8倍,14株菌600代后MIC增长了16倍。对环丙沙星的实验也有类似的结果[5-6]。这些结果证明Sub-MIC环境下,细菌可产生耐药突变并选择性扩增低适应性代价突变菌株,耐药突变的适应性代价越低,需要产生选择压力的抗菌药物浓度越小,突变株越容易被选择性扩增。现在发现细菌存在代偿性突变机制,细菌通过多次突变,可降低甚至完全消除耐药突变的适应性代价,使得突变菌在竞争中更有优势[10-11]。Sub-MIC药物选择出的低适应性代价突变既可为低耐药性,也可为高耐药性。使得细菌可保留多种不同的耐药突变,为进一步耐药突变的产生作准备。
与Sub-MIC不同,MSW中抗菌药物对细菌具有强选择作用,细菌适应性主要取决于耐药性强弱,药物快速清除敏感菌,并选择性扩增耐药突变菌,在短时间内细菌难以产生高耐药性突变,因此MSW通常选择性富集已存在的高耐药性突变株。
随着抗菌药物在医学、畜牧、种植业的大规模应用,药物以原形或活性代谢产物形式排放,使得环境中存在微量浓度的抗菌药物,极端情况下河水中的环丙沙星浓度可高达31 mg/L,超过抗感染治疗患者的血药浓度(约2~3 mg/L)[12-13]。自然界中大量致病菌与非致病菌长时间暴露于Sub-MIC环境中,增加了耐药突变产生的可能性,使得环境中细菌成为耐药基因的潜在来源。
2 Sub-MIC时耐药产生的机制
Sub-MIC下更容易选择性富集低适应性代价的耐药突变株。Gullberg等[14]和Sun等[15]认为改变底物结构、共同抵抗、改变形态及生物被膜等低适应性代价突变可能为Sub-MIC时耐药产生的主要机制,而产生外排泵、水解酶等高适应性代价机制的可能性较小。Toprak等[16]通过morbidostat设备动态调节氯霉素、多西环素和甲氧苄啶3种药物浓度,保持大肠埃希菌的生长速度衡定为50%最大生长速度。定期测量药物的50%抑制浓度(IC50),结果发现3种药物的IC50均明显升高。氯霉素和多西环素IC50上升曲线平滑,结果显示细菌的mar、acr、cmr等基因存在突变,其中任何一种基因突变均可使IC50增高,产生耐药性对菌群数量要求低,IC50曲线较平滑。甲氧苄啶IC50升高曲线呈现明显阶段性。并且发现几乎所有耐药突变均位于DHFR基因,为g-9a或c-35t(DHFR表达增加相关),且突变按一定顺序产生。因此需要细菌达到一定数量才能产生突变,致使IC50曲线呈阶段性。以上结果表明Sub-MIC时细菌耐药产生受细菌与药物本身种类影响,细菌对不同抗菌药物产生耐药性的途径不同,根本原因为产生的耐药突变不同。
进一步研究发现细菌的耐药性由其耐药基因决定,Chevereau等[17]在研究大肠埃希菌对Sub- MIC呋喃妥因和氯霉素的IC50时发现,呋喃妥因的IC50在前10 d快速升高,而后增速明显降低,基因检测显示细菌在呋喃妥因耐药突变主要位点已产生突变,导致之后仅有少量耐药突变产生。氯霉素的IC50持续升高,结果显示细菌有多个位点可产生耐药突变,各个突变随机产生,在基因位点“消耗”完全之前,细菌耐药性可持续升高。产生耐药突变的位点及途径越多,细菌产生耐药突变的“储备”越多,有可能产生更高的耐药性。见表1。
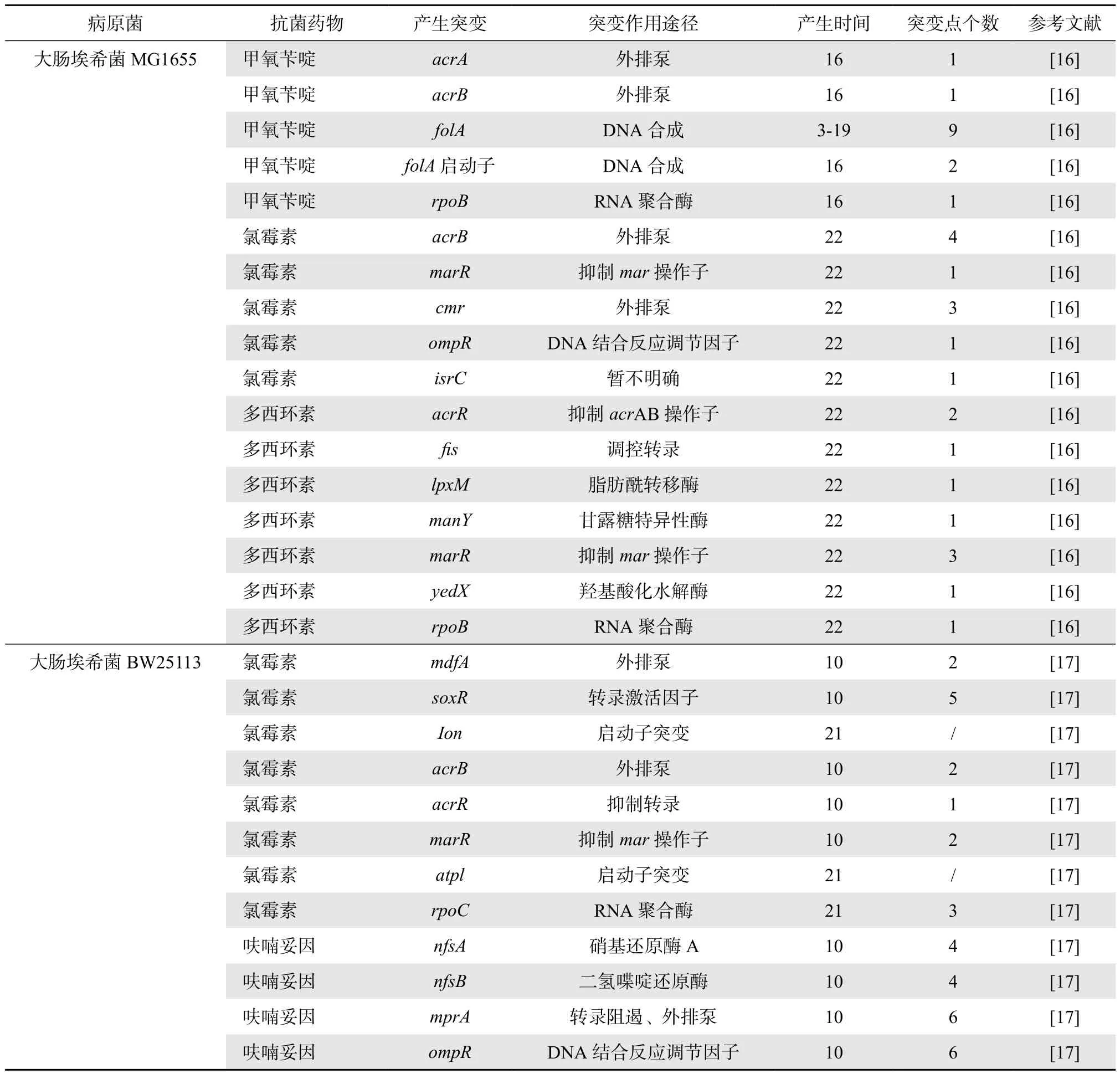
表1 Sub-MIC产生的耐药突变
3 Sub-MIC的临床意义
根据MSW理论,当药物浓度达到MPC以上时,敏感菌株及一步耐药突变菌株的生长菌受到抑制,这时不会有耐药突变菌株被选择性富集,从而也不会产生耐药[18]。但由于药物在人体内分布容积及药物自身不良反应影响,药物浓度往往不能达到MPC以上,此时药物浓度落入MSW中,耐药菌选择性富集,菌群产生耐药,导致治疗失败。药物浓度下降至MIC以下时,细菌便处于Sub-MIC环境,药物从选择高耐药性突变变为选择低耐药性代价突变(图2)。此时细菌既可产生新的耐药突变,也可扩增原有耐药突变菌,为产生更高耐药性菌提供基础。为尽量减少耐药菌的产生和扩增,学者认为可以通过合理应用抗菌药物、联合用药、交替用药等方式减少细菌在Sub-MIC下的暴露时间、减少耐药突变产生,避免细菌产生耐药。
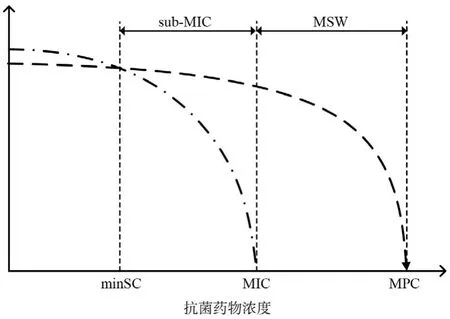
图2 Sub-MIC和MSW
选用在感染部位浓度较高的药物,提高感染部位的药物浓度,尽量减少细菌暴露在Sub-MIC及MSW內的时间,以及处于Sub-MIC下细菌的数量。如利奈唑胺较万古霉素在肺泡上皮衬液及组织液中有较高的浓度[19],治疗肺部感染时可以快速清除细菌,减少暴露于Sub-MIC时间和细菌数量,从而减少耐药突变的产生。
由于药物在体内分布浓度不同、高剂量药物引起的不良反应,以及长期应用抗菌药物导致菌群MIC/MPC升高,细菌难以避免会处于Sub-MIC环境。此时应采用联合用药方式,一方面可以缩小或关闭MSW,减少耐药菌株选择性富集,另一方面可以减少Sub-MIC下新的耐药突变的产生[20]。
实际临床治疗过程中,联合用药治疗效果可能仍不理想,无法清除细菌、减少耐药菌产生[21]。原因可能为药物的相互作用受环境、细菌种类等多种因素影响,治疗过程中细菌的耐药性改变导致药物相互作用改变,随着耐药性增强,药物的协同作用不断减弱。药物对细菌的清除能力下降,菌群数量增加出现更多的耐药突变,导致治疗失败。
4 不足和展望
目前对Sub-MIC与细菌耐药的研究对象大多为大肠埃希菌,耐药产生机制研究主要针对β 内酰胺类、酰氨醇类、氨基糖苷类药物,对当前临床应用药物如甘氨酰环素类、碳青霉烯类药物缺少相关研究。细菌产生耐药性基于抗菌药物种类与细菌自身的基因,不同基因背景可能产生不同的耐药突变。因此现有研究结果不能很好地预测其他细菌及药物的耐药产生机制[22-23]。
细菌除了通过自发突变的方式产生耐药基因之外,水平基因转移也是细菌获得耐药性的重要途径,多重耐药肺炎克雷伯菌可通过获得耐药质粒产生对多种抗生素的耐药性[24]。革兰阴性菌、革兰阳性菌多通过水平基因转移的方式获得β 内酰胺水解酶耐药基因,从而产生对β 内酰胺类药物的耐药性[25],因此仅根据Sub-MIC时耐药产生机制预测β 内酰胺类耐药性并不准确。
Troy认为Sub-MIC下细菌不仅可以产生新的耐药突变,更重要的是已有的耐药突变可以选择性扩增,这与MSW理论中耐药突变菌仅在MSW內富集不同。细菌的自发性突变总在发生,菌群中存在一定量的突变菌株,但如果这些突变菌株不被选择性扩增,成为优势菌,就可以被人体的免疫系统清除[6]。根据Sub-MIC理论,药物对耐药菌株产生选择性扩增作用的最低浓度是minSC,因此也有人将Sub-MIC看作是MSW理论的补充[26]。Sub-MIC耐药机制研究发现不同细菌产生耐药性的速度、耐药性强弱各不相同,这是由细菌产生不同的耐药突变基因决定的。Chevereau等[17]因此认为,Sub-MIC理论可以帮助医师在面对感染时,根据细菌及抗生素种类预测其耐药性变化,采取更有针对性的治疗。
随着细菌耐药问题日趋严重,Sub-MIC理论研究会更加深入,对动物或人体Sub-MIC的研究比体外研究更具实际应用价值,研究结果能更好地帮助解决细菌耐药问题。
[1] PALMER AC, KISHONY R. Understanding, predicting and manipulating the genotypic evolution of antibiotic resistance [J] .Nat Rev Genet, 2013,14(4):243-248.
[2] PENA-MILLER R, LAEHNEMANN D,JANSEN G,et al. When the most potent combination of antibiotics selects for the greatest bacterial load: the smile-frown transition [J].PLoS Biol, 2013,11(4):e1001540.
[3] BOLLENBACH T. Antimicrobial interactions: mechanisms and implications for drug discovery and resistance evolution [J].Curr Opin Microbiol, 2015,27 :1-9.
[4] CHEVEREAU G, BOLLENBACH T. Systematic discovery of drug interaction mechanisms [J]. Mol Syst Biol, 2015,11(4):807.
[5] ANDERSSON DI, HUGHES D.Evolution of antibiotic resistance at non-lethal drug concentrations [J]. Drug Resist Updat, 2012,15(3):162-172.
[6] TROY D, HUIJBEN S,READ AF. Is selection relevant in the evolutionary emergence of drug resistance? [J]. Trends Microbiol, 2015,23(3):126-133.
[7] BELL G. Evolutionary rescue and the limits of adaptation [J].Philos Trans R Soc Lond B Biol Sci, 2013,368(1610):20120080.
[8] SALVERDA ML, DE VISSER JA,BARLOW M. Natural evolution of TEM-1 β-lactamase : experimental reconstruction and clinical relevance [J]. FEMS Microbiol Rev, 2010 ,34(6):1015-1036.
[9] CIRZ RT, CHIN JK,ANDES DR,et al. Inhibition of mutation and combating the evolution of antibiotic resistance [J]. PLoS Biol, 2005,3(6):e176.
[10] ANDERSSON DI, HUGHES D. Gene amplification and adaptive evolution in bacteria [J]. Ann Rev Genet, 2009,43 :167-195.
[11] ANDERSSON DI, HUGHES D. Persistence of antibiotic resistance in bacterial populations [J]. FEMS Microbiol Rev,2011,35(5):901-111.
[12] LARSSON DG, DE PEDRO C,PAXEUS N. Effluent from drug manufactures contains extremely high levels of pharmaceuticals [J]. J Hazard Mater, 2007,148(3):751-755.
[13] TAYLOR NG, VERNER-JEFFREYS DW,BAKER-AUSTIN C. Aquatic systems: maintaining, mixing and mobilising antimicrobial resistance? [J]. Trends Ecol Evol, 2011,26(6):278-284.
[14] GULLBERG E, CAO S,BERG OG,et al. Selection of resistant bacteria at very low antibiotic concentrations [J].PLoS Pathog, 2011,7(7):e1002158.
[15] SUN S, BERG OG,ROTH JR,et al. Contribution of gene amplification to evolution of increased antibiotic resistance inSalmonella typhimurium[J]. Genetics, 2009,182(4):1183-1195.
[16] TOPRAK E, VERES A,MICHEL JB,et al. Evolutionary paths to antibiotic resistance under dynamically sustained drug selection [J]. Nat Genet, 2011,44(1):101-105.
[17] CHEVEREAU G, DRAVECKÁ M,MATUR T,et al.Quantifying the determinants of evolutionary dynamics leading to drug resistance [J]. PLoS Biol, 2015,13(11):e1002299.
[18] DRLICA K. A strategy for fi ghting antibiotic resistance [J]. ASM News,2001,67 :27-33.
[19] MATSUMOTO K, SHIGEMI A,TAKESHITA A,et al.Analysis of thrombocytopenic effects and population pharmacokinetics of linezolid: a dosage strategy according to the trough concentration target and renal function in adult patients [J]. Int J Antimicrob Agents, 2014,44(3):242-247.
[20] MEHTA KC, DARGAD RR,BDRADE DM,et al. Burden of antibiotic resistance in common infectious diseases: role of antibiotic combination therapy [J]. J Clin Diagn Res, 2014,8(6):ME05-8.
[21] SUZUKI S, HORINOUCHI T,FURUSAWA C. Suppression of antibiotic resistance acquisition by combined use of antibiotics[J]. J Biosci Bioeng, 2015,120(4):467-469.
[22] MUNCK C, GUMPERT HK,WALLIN AI,et al. Prediction of resistance development against drug combinations by collateral responses to component drugs [J]. Sci Transl Med, 2014,6(262):262ra156.
[23] LÁZÁR V,PAL SINGH G,SPOHN R,et al. Bacterial evolution of antibiotic hypersensitivity [J]. Mol Syst Biol,2013, 9 :700.
[24] KIM S, LIEBERMAN TD,KISHONY R. Alternating antibiotic treatments constrain evolutionary paths to multidrug resistance[J]. Proc Natl Acad Sci U S A, 2014,111(40):14494-14499.
[25] DAVIES J, DAVIES D. Origins and evolution of antibiotic resistance [J]. Microbiol Mol Biol Rev, 2010,74(3):417-433.
[26] DRLICA K, ZHAO X. Mutant selection window hypothesis updated [J]. Clin Infect Dis,2007,44(5):681-688.
Sub-inhibitory concentration of antimicrobial agents and its clinical implications
WANG Fangzhou, NI Wentao, CUI Junchang. (Department of Respiratory Medicine, Chinese PLA General Hospital, Beijing 100853, China)
R978.1
A
1009-7708 ( 2017 ) 05-0597-05
10.16718/j.1009-7708.2017.05.020
2016-12-08
2017-04-06
三亚市医疗卫生科技创新项目(2016YW35),解放军总医院临床科研扶持基金项目(2014FC-TSYS- 2002)。
中国人民解放军总医院呼吸内科,北京 100853。
王方舟(1993—),男,硕士研究生,主要从事细菌耐药的预防及抗感染药物合理应用。
崔俊昌,E-mail:cuijunchang@163.com。
