石墨烯与抑癌基因p53 DNA片段相互作用的分子模拟与光谱学验证
2017-10-13吴惠丰张明兴李斐曹天贵李雪花赵建民
吴惠丰,张明兴,李斐,曹天贵,李雪花,赵建民
1. 中国科学院烟台海岸带研究所 中国科学院环境过程与生态修复重点实验室,烟台 2640032. 青岛海洋科学与技术国家实验室 海洋渔业科学与食物产出过程功能实验室,青岛 2662373. 中国科学院大学,北京 1000494. 大连理工大学环境学院 工业生态与环境工程教育部重点实验室,大连 116024
石墨烯与抑癌基因p53 DNA片段相互作用的分子模拟与光谱学验证
吴惠丰1,2,*,张明兴1,3,李斐1,曹天贵1,李雪花4,赵建民1
1. 中国科学院烟台海岸带研究所 中国科学院环境过程与生态修复重点实验室,烟台 2640032. 青岛海洋科学与技术国家实验室 海洋渔业科学与食物产出过程功能实验室,青岛 2662373. 中国科学院大学,北京 1000494. 大连理工大学环境学院 工业生态与环境工程教育部重点实验室,大连 116024
石墨烯(graphene, G)及其衍生物由于具有独特的理化性质,被广泛应用于能源、生物医学等领域,但尚缺乏其对生物体和环境潜在危害的研究。采用分子动力学模拟并结合光谱学方法(紫外可见吸收光谱、紫外变温实验及荧光光谱),分析了石墨烯与抑癌基因p53启动子区DNA片段(p53-DNA)间的相互作用,并探讨了相关作用机制。石墨烯的部分芳香环与p53-DNA碱基的芳香环之间存在π-π堆积作用,两者可以通过嵌插作用进行结合,同时还通过沟槽作用进一步结合。光谱实验进一步证实,在石墨烯作用下,p53-DNA的熔点(Tm)值升高,EB-DNA体系发生静态荧光淬灭,说明石墨烯能与p53-DNA结合;同时,p53-DNA与石墨烯结合后在260 nm处的吸光度升高,说明石墨烯对p53-DNA的双螺旋结构具有一定的破坏作用。上述研究结果从分子水平上分析了石墨烯与p53-DNA间的相互作用机制,有助于进一步阐明石墨烯的毒性作用机理。
石墨烯;p53-DNA;相互作用;分子动力学模拟;光谱法
Received4January2017accepted2March2017
Abstract: Graphene and its derivatives have been widely used in the fields ranging from energy to biomedicine because of its peculiar physical and chemical properties. However, limited attention has been paid to potential hazards of graphene to organisms and environments. In this study, the interaction between graphene and the promoter region of p53gene (p53-DNA) was investigated using molecular dynamics simulation (MD) technology and spectroscopic methods based on the combination of UV-vis absorption, DNA melting point test and fluorescent spectra, followed by the illustration of the interaction mechanism. MD results showed that some aromatic moieties of graphene could intercalate into the p53-DNA base pairs, which relied on π-π stacking interaction between aromatic moieties of graphene and p53-DNA base, and the other parts of graphene could further combine with p53-DNA by groove binding. Moreover, spectroscopic tests exhibited that graphene could increase the melting point (Tm) of p53-DNA and lead to static fluorescence quenching of EB-DNA system, suggesting that graphene was indeed able to bind with p53-DNA. Meanwhile, the ascent of absorbance of260nm was found in the p53-DNA as a result of the combination of graphene and p53-DNA, which meant graphene posed some effect on the double helix structure of p53-DNA. In conclusion, the mechanism of interaction between graphene and p53-DNA was investigated at molecular level in this study, which contributed to a further understanding of the toxicological effects of graphene.
Keywords: graphene(G);p53-DNA;binding interaction;molecular dynamics simulations;spectroscopic methods
计算毒理学(computational toxicology)是一门将计算机技术应用于毒理学领域而产生的新兴学科,涉及到计算化学、计算生物学、分子生物学和系统生物学等多个学科,旨在利用计算机模拟技术和数学计算来预测和分析化学污染物的毒副作用及作用机制[1]。由于目前化学品的种类非常多,且数量还在不断增长[2],传统的毒理学试验已经很难满足生态风险评价的需求,因此以计算机模拟为基础的计算毒理学成为对有毒化合物进行筛选和评估的高效工具[3]。基于配体-受体相互作用的分子模拟技术(molecular modeling)是计算毒理学研究中的一种重要手段[4]。而随着计算机技术的快速发展,各种分子模拟技术被迅速应用于研究环境污染物与DNA/蛋白质等生物大分子之间的相互作用[5]。其中,分子对接(molecular docking)和分子动力学模拟(molecular dynamics simulations, MD)技术可以模拟有机污染物与生物分子间的结合模式,有助于深入理解分子间的相互作用机制并揭示污染物的毒性作用机理,因而获得了较为广泛的应用[6]。
p53基因是目前已知的人类肿瘤基因中最主要的肿瘤抑制基因,许多肿瘤的发生都与其突变有关[7]。当某种污染物分子与p53基因相互作用并结合时,DNA的复制或转录就会受到影响,如果这种作用力很强(嵌插或共价结合),则很可能会导致p53基因的突变,从而增加发生癌变的几率[8]。p53基因启动子区域DNA片段(p53-DNA)是研究p53与污染物相互作用模式的常用基因片段[9]。
石墨烯(graphene, G)是一种新型二维碳纳米材料,由于具有独特的热学、光学、电学、力学性质,已经在材料、能源、催化及生物医学等领域广泛应用[10-12]。随着生产工艺的改进,石墨烯及其衍生物的生产成本逐渐降低,其使用领域和生产规模随之不断扩大[13-14],但在生产、使用和废旧材料处理的过程中也会产生更多的含石墨烯衍生物的垃圾,进而在空气、水体、土壤中转移和积累,对生物体和人类产生潜在的威胁[15-16]。已有研究表明石墨烯及其衍生物具有一定的生物毒性,因此石墨烯及其衍生物对生物体和环境的影响受到了越来越多的关注[17-19]。Mu等[20]研究发现,石墨烯横向尺寸的大小对其穿过细胞膜的难易程度有直接影响。Ma等[21]研究也表明,当石墨烯尺寸较大时更容易吸附到细胞表面,而小尺寸的石墨烯则很容易通过胞吞作用或者直接进入细胞内部。因此当生物体或细胞暴露于石墨烯时,较小尺寸的石墨烯很有可能进入细胞内部并与DNA发生相互作用,影响DNA的转录与表达,并可能进一步导致基因突变。所以在分子水平上研究石墨烯与p53-DNA相互作用的机制可以为进一步研究石墨烯的毒理效应提供一定的依据。
光谱学方法(如荧光光谱、紫外可见吸收光谱、圆二色光谱和拉曼光谱等)是研究小分子污染物与DNA分子间相互作用的常用实验方法[22],但每种光谱都存在着一定的局限性,而基于计算机技术的分子模拟方法则可以预测分子间的相互作用模式,分析分子间的相互作用机制,能够弥补光谱学方法的不足。相对有机小分子,石墨烯有较大的尺寸,不利于与DNA的结合,但由于其厚度在纳米尺度并且边缘活性较高,所以很可能会通过边缘部分与DNA结合。本研究使用p53-DNA作为研究对象,通过分子动力学模拟研究了石墨烯与p53-DNA之间的相互作用机制,分析了石墨烯对p53-DNA可能造成的影响,并通过紫外可见吸收光谱、紫外变温实验以及荧光光谱等手段检测了石墨烯与p53-DNA的结合作用,对分子模拟的结果进行了验证。
1 材料与方法(Materials and methods)
1.1 仪器与试剂
仪器:U-3900H型紫外/可见分光光度计(日本Hitachi);LS55型荧光光度计(美国Perkin Elmer);MultiMode 8型原子力显微镜(AFM, 美国Veeco)。
试剂:p53-DNA(中国,上海生工生物技术有限公司);石墨烯(水溶液,浓度为1.0 g·L-1)购自美国Sigma-Aldrich(货号:799092);溴化乙锭(EB,纯度>99%,美国Sigma-Aldrich);磷酸缓冲液(PBS, 0.1 mol·L-1, pH = 7.4)由Na2HPO4和NaH2PO4配制。其他试剂均为国产分析纯,超纯水使用Cascada LS(美国Pall)超纯水机生产。
1.2 分子模拟
通过make-na server网站(http://structure.usc.edu/make-na/server.html)获得p53-DNA的三维结构。该网站内置了Nucleic Acid Builder (NAB)程序,目前广泛应用于核酸(DNA和RNA)的三维结构预测[23-24]。本研究使用Discovery Studio 2.5(简称DS 2.5)软件中的CDOCKER算法进行分子模拟。CDOCKER算法是一种基于格点的分子对接方法,可以通过CHARMm优化力场进行柔性对接,采用高温动力学模拟产生配体分子的空间构象,并使用模拟退火对分子对接结果进行优化,可以精确地计算受体-配体的相互作用能和配体的张力[25-26]。参数设置中设定输出10个平均构象,其他参数不作更改,根据对接能量进行打分和筛选,受体与配体的对接亲和力越大则对接能的负绝对值越大。通过分子对接,可以获得石墨烯与p53-DNA复合物的构象,从而分析石墨烯与p53-DNA间的关键相互作用。
在DS 2.5软件中使用CHARMm力场对石墨烯与p53-DNA复合物体系进行分子动力学模拟(MD)。为了保证对复合物体系进行模拟时有足够的构象变化空间,使用TIP3P水模型作为溶剂,并在溶质外层空间加上厚度为7 Å的水分子层。同时,需要在体系中添加适量的钠离子,用来平衡多余的负电荷,从而保证体系为电中性。在MD模拟之前,先使用最陡下降法对体系进行一次能量优化(设置最大的优化步数为6 000步),以消除高能碰撞的影响。分2步进行MD模拟:首先在设定温度从50 K缓慢升高到300 K的情况下对p53-DNA分子进行20 ps的MD模拟;然后再对整个复合物体系进行500 ps的MD模拟,恒温恒压下进行,不作任何约束条件,每0.5 ps存写一次三维构象。在上述MD模拟过程中,将步长设置为2 fs,静电和范德华等非成键相互作用的截断值半径设为14 Å。
1.3 制备双链p53-DNA
p53-DNA的2条单链序列分别为:5′-CCTCCTCCCCAACTCC-3′,5′-GGAGTTGGGGAGGAGG-3′。使用PBS缓冲液将2条单链互补DNA溶解(5×10-4mol·L-1),于85 ℃保持12 min,之后缓慢冷却至室温,即得5×10-4mol·L-1的双链p53-DNA溶液。检测所得p53-DNA样品在260 nm和280 nm处的紫外吸收,其吸光度的比值A260/A280达到1.84,表明已合成DNA双链,符合实验要求。
1.4 紫外光谱检测
在进行光谱学研究之前,首先使用原子力显微镜(AFM)对石墨烯纳米颗粒进行尺寸表征,测量其厚度和横向尺寸的大小。由于石墨烯没有标准的分子量,无法得知其摩尔浓度,故通过尺寸大小大致估计其分子量,然后使用质量浓度进行实验。
将p53-DNA分别与不同浓度的石墨烯混合,使p53-DNA终浓度为1×10-6mol·L-1,石墨烯终浓度依次为0, 0.1, 0.2, 0.4, 0.6, 0.8, 1.0 mg·L-1,实验前将石墨烯超声震荡30 min以使纳米颗粒均匀分散。静置反应2 h后,检测其紫外可见吸收光谱,参比溶液为PBS缓冲液,扫描范围220~340 nm。
1.5 紫外变温实验
将p53-DNA分别与石墨烯混合,使p53-DNA终浓度为1×10-6mol·L-1,石墨烯终浓度为1.0 mg·L-1,然后程序升温(25℃ ~95 ℃),测定不同温度下纯DNA及混合溶液在260 nm处的吸光度,参比溶液为PBS缓冲液。
1.6 荧光光谱检测
将EB加入p53-DNA溶液,使溶液中EB和DNA的浓度比为6∶1,避光反应30 min,从而得到EB-DNA复合物体系。将EB-DNA体系分别与不同浓度的石墨烯混合,使p53-DNA终浓度为1×10-6mol·L-1,石墨烯终浓度分别为0, 0.1, 0.2, 0.4, 0.6, 0.8, 1.0 mg·L-1,混合前将石墨烯超声震荡30 min。混合均匀后避光反应2 h,然后进行荧光光谱扫描,激发波长设为480 nm,发射波长扫描范围设为500~800 nm。
2 结果(Results)
2.1 分子模拟结果
有机小分子与DNA之间的结合通常为非共价模式,主要包括静电结合、嵌插结合及沟槽结合等3种方式[27]。在这3种方式中,静电结合不具备选择性,但嵌插和沟槽结合却具有选择性,均对DNA分子的构象有一定的影响,且嵌插作用的影响更大[28-29]。分子对接可以获得石墨烯与p53-DNA复合物的空间构象,选择其最佳结合构象,进行了近300 ps的MD研究。图1所示为石墨烯与p53-DNA相互作用的模型图,可以看出,石墨烯的部分芳香环与p53-DNA碱基的芳香环具有很强的π-π堆积作用,能够嵌插到p53-DNA的双螺旋结构中,使石墨烯与p53-DNA牢固结合。同时,石墨烯的其余部分在p53-DNA的沟槽中沿着磷酸骨架伸展,通过沟槽作用增强了两者的结合。图2显示了复合物碳骨架的均方根误差(RMSD)随时间变化的趋势,从中可以看出石墨烯与p53-DNA复合物的RMSD在50 ps后趋于平衡,说明复合物形成后逐渐趋于稳定。

图1 石墨烯与p53-DNA相互作用的分子模拟图注:黄色线表示π-π作用。Fig. 1 The molecular simulation of graphene and p53-DNANote: Yellow lines indicate π-π stacking interaction.
2.2 紫外可见吸收光谱和变温实验
通过AFM扫描结果,可知实验中所使用的石墨烯的厚度约为1 nm,尺寸为10~50 nm。
图3为不同浓度的石墨烯作用前后p53-DNA的紫外可见吸收光谱。从图3可以看出,p53-DNA在260 nm附近出现最大光吸收峰,石墨烯作用后其260 nm处的吸光度升高,且随着石墨烯浓度的增加而不断升高,产生增色效应,最大增色幅度达到11.5%。当DNA分子的双螺旋结构遭到破坏导致氢键断裂形成单链时,其260 nm处的吸光度就会升高[30]。因此,紫外可见吸收光谱测定结果表明,石墨烯能够对p53-DNA的双螺旋结构产生一定的破坏作用。
图4为在1.0 mg·L-1的石墨烯加入前后p53-DNA的紫外变温曲线(熔解曲线)。连续升温可以使DNA的双螺旋结构受到破坏,导致氢键断裂形成单链,从而使DNA溶液在260 nm处的吸光度升高。加热过程中使DNA双螺旋解开的温度范围中点处的温度称为DNA的熔点(Tm),可由熔解曲线计算得出。小分子通过嵌插作用与DNA结合后能够改变其双螺旋结构,最高可使Tm增加5 ℃ ~8 ℃,其他结合方式则不会对Tm产生显著影响[22]。经计算,纯p53-DNA的Tm值为65 ℃,加入1.0 mg·L-1的石墨烯后Tm值升高到66.5 ℃。说明石墨烯的部分结构通过嵌插作用与p53-DNA结合,并改变了p53-DNA的双螺旋结构。
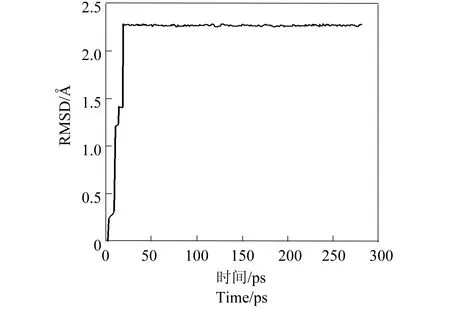
图2 石墨烯与p53-DNA复合物碳骨架RMSD 随时间变化的趋势图Fig. 2 The RMSD changing trend of graphene and p53-DNA complex

图3 石墨烯作用前后p53-DNA的紫外可见吸收光谱注:1~7分别表示石墨烯的不同浓度组0, 0.1, 0.2, 0.4, 0.6, 0.8, 1.0 mg·L-1。Fig. 3 Ultraviolet absorption spectra of p53-DNA before and after graphene treatmentNote: The concentration groups of graphene from 1 to 7 are 0, 0.1, 0.2, 0.4, 0.6, 0.8, 1.0 mg·L-1.

图4 石墨烯加入前后p53-DNA的熔解曲线Fig. 4 Melting curves of p53-DNA in the absence and presence of graphene
2.3 荧光光谱实验结果
经检测,DNA、EB和石墨烯本身的荧光强度均很弱,而EB和DNA结合形成EB-DNA复合体系后则具有较强的荧光强度,因此可通过构建EB-DNA荧光探针体系来分析石墨烯与DNA的相互作用。图5为不同浓度的石墨烯与EB-DNA体系作用前后的荧光光谱图。从图5可以看出,EB-DNA体系荧光光谱的峰位在598 nm左右,并且随着石墨烯浓度的增加,荧光强度逐步减弱,说明石墨烯在一定程度上导致EB-DNA体系的荧光淬灭。当石墨烯浓度最高(1.0 mg·L-1)时,荧光强度下降了10.6%。
根据经典荧光淬灭理论,可通过Stern-Volmer方程求得有机小分子对EB-DNA体系的荧光淬灭常数(Ksv)和淬灭速率常数(Kq)[31]。石墨烯虽然本身尺寸较大,但由于其厚度在纳米尺度并且边缘活性较高,所以也可如有机小分子一样计算其荧光淬灭常数Ksv。经计算,石墨烯对EB-DNA体系的Ksv值为131 L·g-1。根据片层的厚度和尺寸大小可估计出石墨烯的大致分子量,使用摩尔浓度(L·mol-1)换算后其Ksv可超过105数量级。石墨烯对EB-DNA体系的淬灭速率常数Kq为1.31×1010L·g-1·s-1,换算成摩尔浓度表示(L·mol-1·s-1)则高于1013数量级。一般认为,生物大分子发生动态淬灭时其Kq值不会超过2×1010L·mol-1·s-1,实验中测得的Kq值远大于此,因此可认为石墨烯使EB-DNA体系发生荧光淬灭的过程是静态淬灭[32]。

图5 石墨烯作用前后溴化乙锭-DNA(EB-DNA) 体系的荧光光谱图注:1~7表示不同的石墨烯浓度0, 0.1, 0.2, 0.4, 0.6, 0.8, 1.0 mg·L-1。Fig. 5 Fluorescence spectra of ethylene dibromide-DNA (EB-DNA) with grapheneNote: Concentrations of graphene (1-7) is 0, 0.1, 0.2, 0.4, 0.6, 0.8, 1.0 mg·L-1.
此外,可通过Scatchard关系式计算静态淬灭中有机物分子与DNA之间的表观结合常数(Ka)[33]。经计算,石墨烯与p53-DNA之间的表观结合常数Ka为717 L·g-1,用摩尔浓度表示(L·mol-1)则大于105数量级,结合位点数为1.21。
结合紫外可见吸收光谱和紫外变温实验的结果,可推断石墨烯通过以下2种方式导致EB-DNA体系的荧光淬灭:(1) 石墨烯与EB存在着竞争关系,两者会竞争性地通过嵌插作用与p53-DNA结合,而随着石墨烯浓度的不断增加,嵌入p53-DNA双螺旋结构中的EB就会被石墨烯置换出来,使体系中EB-DNA复合体的浓度降低,荧光强度也随之降低;(2) 石墨烯可以通过沟槽作用与p53-DNA结合,从而改变p53-DNA的构象,使EB脱离双螺旋结构。结合分子模拟的结果分析,以上2种方式同时存在,嵌插作用与沟槽结合作用共同导致了石墨烯与p53-DNA的结合。
3 讨论(Discussion)
石墨烯是一种二维碳纳米材料,虽然在厚度上处于纳米级别,但其平面尺寸却相对有机小分子大得多,所以不可能像有机小分子那样完全或大部分嵌入DNA分子中。但由于石墨烯分子很薄且边缘具有很高的活性,所以石墨烯可以通过边缘部分嵌入DNA的双螺旋结构中,从而稳定结合并改变DNA的构象。本研究使用分子动力学模拟的方法分析了石墨烯与p53-DNA间相互作用的机制,发现石墨烯的部分芳香环与p53-DNA碱基的芳香环间具有很强的π-π堆积作用,能够嵌插到p53-DNA的双螺旋结构中,使石墨烯与p53-DNA牢固结合。空间位阻对化合物与DNA的相互作用方式具有很大的影响[34-35],同种化合物经过表面修饰后与DNA的作用方式也会发生变化[36],石墨烯边缘的芳香环结构有利于其与p53-DNA通过嵌插作用结合。同时,由于石墨烯平面尺寸相对有机小分子大得多,它的其余部分可以在p53-DNA的沟槽中沿着磷酸骨架伸展,通过沟槽作用增强两者的结合。有机小分子通常只会以一种方式与DNA结合[37],而石墨烯能同时通过嵌插作用和沟槽作用与p53-DNA结合,展示了其独特的结构和性质。
光谱学实验的结果证实了分子模拟的准确性。在石墨烯作用下,p53-DNA的Tm值升高并且EB-DNA体系发生明显的静态荧光淬灭,说明石墨烯的部分结构能够通过嵌插作用与p53-DNA结合,并改变p53-DNA的双螺旋结构。同时,石墨烯与p53-DNA的结合使p53-DNA在260 nm处的吸光度增加,产生增色效应。有机物分子与DNA结合后,通常都会对DNA的分子构象产生影响[38]。紫外增色效应说明石墨烯在与p53-DNA相结合的过程中改变了其构象,导致其双螺旋结构受到一定的破坏。所以当石墨烯进入细胞后,很可能会对DNA的复制或转录等功能产生影响,甚至导致基因突变。光谱学研究表明,具有平面芳香环结构的分子更容易嵌插到DNA的配对碱基对之间,与DNA的结合作用更强[39]。本研究中石墨烯分子具有众多的芳香环结构,同样显示出与p53-DNA有较强的结合作用。
在研究污染物与生物大分子相互作用时,使用分子动力学模拟的方法进行理论预测,同时通过实验方法加以验证,理论与实验相结合,可以使结果更加准确[40-41]。光谱学方法是研究化合物与DNA分子相互作用的常用实验方法[42],多种光谱学方法的应用可以较为准确地得到化合物与DNA相互作用的信息[43]。本研究通过分子模拟与多种光谱学方法,从理论和实验两方面研究了石墨烯与p53-DNA间的相互作用,获得了基本一致的实验结果。Tian等[44]及Silva等[45]在研究污染物与DNA的相互作用时,也采用了分子模拟与光谱学研究相结合的方法,发现分子模拟所预测的分子结合方式能够很好地解释光谱学实验的结果。
综上,本研究采用分子模拟与光谱学方法共同研究了石墨烯与p53-DNA间的相互作用,两者结果基本一致并相互补充。石墨烯的部分芳香环与p53-DNA碱基的芳香环之间具有很强的π-π堆积作用,主要通过嵌插作用相互结合。同时,石墨烯与p53-DNA之间还存在沟槽作用,增强了两者的结合。石墨烯在与p53-DNA结合后对其双螺旋结构具有一定的破坏作用。本研究从分子水平上阐明了石墨烯与p53-DNA间的相互作用机制,为进一步研究其毒理效应提供了依据。
[1] Thomas R S. Evolution of computational toxicology: From primitive beginnings to sophisticated application [J]. Toxicology Letters, 2016, 258(Supplement): S12
[2] Judson R, Richard A, Dix D J, et al. The toxicity data landscape for environmental chemicals [J]. Environmental Health Perspectives, 2009, 117(5): 685-695
[3] Merlot C. Computational toxicology—A tool for early safety evaluation [J]. Drug Discovery Today, 2010, 15(1-2): 16-22
[4] Li Y L, Wang S J, Wang Q. A molecular dynamics simulation study on enhancement of mechanical and tribological properties of polymer composites by introduction of graphene [J]. Carbon, 2017, 111: 538-545
[5] Li F, Xie Q, Li X H, et al. Hormone activity of hydroxylated polybrominated diphenyl ethers on human thyroid receptor β: In vitro and in silico investigations [J]. Environmental Health Perspectives, 2010, 118(5): 602-606
[6] Mozaffari F. Molecular dynamics simulation study on the structure and the dynamic properties of nano-confined alcohols between graphene surfaces [J]. Fluid Phase Equilibria, 2017, 431: 8-15
[7] Amelio I, Melino G. The p53 family and the hypoxia-inducible factors (HIFs): Determinants of cancer progression [J]. Trends in Biochemical Sciences, 2015, 40(8): 425-434
[8] Laptenko O, Prives C. Transcriptional regulation by p53: One protein, many possibilities [J]. Cell Death and Differentiation, 2006, 13(6): 951-961
[9] 李莉, 鲁嘉, 李卉卉, 等. 光谱联合电泳法研究双酚A与肿瘤相关DNA的相互作用[J]. 化学分析计量, 2014, 23(6): 37-41
Li L, Lu J, Li H H, et al. Studies on the interaction of BPA with tumor related DNA by spectroscopic and electrophoresis methods [J]. Chemical Analysis and Meterage, 2014, 23(6): 37-41 (in Chinese)
[10] Geim A K. Graphene: Status and prospects [J]. Science, 2009, 324: 1530-1534
[11] Wang Y, Li Z, Wang J, et al. Graphene and graphene oxide: Biofunctionalization and applications in biotechnology [J]. Trends in Biotechnology, 2011, 29(5): 205-212
[12] Geim A K, Novoselov K S. The rise of graphene [J]. Nature Materials, 2007, 6: 183-191
[13] Chen Y Y, Mi Y M, Ruan Q C, et al. Progress on preparation of graphene and its application [J]. Bulletin of the Chinese Ceramic Society, 2015, 34(3): 755-763
[14] 徐驰, 朱和国. 石墨烯的制备及其在能源方面的应用研究进展[J]. 材料科学与工程学报, 2016, 34(2): 326-332
Xu C, Zhu H G. Progress in fabrication and application of graphene [J]. Journal of Materials Science and Engineering, 2016, 34(2): 326-332 (in Chinese)
[15] Zhao J, Wang Z, White J C, et al. Graphene in the aquatic environment: Adsorption, dispersion, toxicity and transformation [J]. Environmental Science and Technology, 2014, 48(17): 9995-10009
[16] 田甜, 吕敏, 田旸, 等. 石墨烯的生物安全性研究进展[J]. 科学通报, 2014, 59(20): 1927-1936
Tian T, Lv M, Tian Y, et al. Progress in biological safety of grapheme [J]. Chinese Science Bulletin, 2014, 59(20): 1927-1936 (in Chinese)
[17] Qu G, Liu S, Zhang S, et al. Graphene oxide induces toll-like receptor 4 (TLR4)-dependent necrosis in macrophages [J]. ACS Nano, 2013, 7(7): 5732-5745
[18] Duch M C, Budinger G R S, Liang Y T, et al. Minimizing oxidation and stable nanoscale dispersion improves the biocompatibility of graphene in the lung [J]. Nano Letters, 2011, 11(12): 5201-5207
[19] Zhao S, Wang Q, Zhao Y, et al. Toxicity and translocation of graphene oxide in Arabidopsis thaliana [J]. Environmental Toxicology and Pharmacology, 2015, 39(1): 145-156
[20] Mu Q, Su G, Li L, et al. Size-dependent cell uptake of protein-coated graphene oxide nanosheets [J]. ACS Applied Materials and Interfaces, 2012, 4(4): 2259-2266
[21] Ma J, Liu R, Wang X, et al. Crucial role of lateral size for graphene oxide in activating macrophages and stimulating pro-inflammatory responses in cells and animals [J]. ACS Nano, 2015, 9(10): 10498-10515
[22] Bi S Y, Qiao C Y, Song D Q, et al. Study of interactions of flavonoids with DNA using acridine orange as a fluorescence probe [J]. Sensors and Actuators B: Chemical, 2006, 119(1): 199-208
[23] Ye W, Qin F, Zhang J, et al. Atomistic mechanism of microRNA translation upregulation via molecular dynamics simulations [J]. PLOS ONE, 2012, 7(8): e43788

[25] Pinfield V J, Scott D J. Anomalous small angle X-ray scattering simulations: Proof of concept for distance measurements for nanoparticle-labelled biomacromolecules in solution [J]. PLOS ONE, 2014, 9(4): e95664
[26] Gagnon J K, Law S M, Brooks C L. Flexible cdocker: Development and application of a pseudo-explicit structure-based docking method within charmm [J]. Journal of Computational Chemistry, 2016, 37(8): 753-762
[27] Kumar C V, Asuncion E H. DNA-binding studies and site-selective fluorescence sensitization of an anthryl probe [J]. Journal of the American Chemical Society, 1993, 115(19): 8547-8553
[28] Sarkar D, Das P, Basak S, et al. Binding interaction of cationic phenazinium dyes with calf thymus DNA: A comparative study [J]. The Journal of Physical Chemistry B, 2008, 112(30): 9243-9249
[29] Cai C, Chen X, Ge F. Analysis of interaction between tamoxifen and ctDNA in vitro by multi-spectroscopic methods [J]. Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, 2010, 76(2): 202-206
[30] Oikawa S, Tada-Oikawa S, Kawanishi S. Site-specific DNA damage at the GGG sequence by UVA involves acceleration of telomere shortening [J]. Biochemistry, 2001, 40(15): 4763-4768
[31] Tse W C, Boger D L. A fluorescent intercalator displacement assay for establishing DNA binding selectivity and affinity [J]. Accounts of Chemical Research, 2004, 37(1): 61-69
[32] Zhao N, Wang X, Pan H, et al. Spectroscopic studies on the interaction between tryptophan-erbium(III) complex and herring sperm DNA [J]. Spectrochimica Acta Part A, 2010, 75(5): 1435-1442
[33] Zhuang S, Wang H, Ding K, et al. Interactions of benzotriazole UV stabilizers with human serum albumin: Atomic insights revealed by biosensors, spectroscopies and molecular dynamics simulations [J]. Chemosphere, 2016, 144: 1050-1059
[34] Jalili S, Maddah M, Schofield J. Molecular dynamics simulation and free energy analysis of the interaction of platinum-based anti-cancer drugs with DNA [J]. Journal of Theoretical & Computational Chemistry,2016, 15: 1650054
[36] 倪晗, 易弋, 孙庭广, 等. 用分子模拟的方法研究无乳链球菌GlnR因子与DNA的相互作用[J]. 山东化工,2015, 44(11): 144-148
Ni H, Yi Y, Sun T G, et al. Study on the Streptococcus agalactiae GlnR factor interaction with DNA by molecular modeling methods [J]. Shandong Chemical Industry, 2015, 44(11): 144-148 (in Chinese)
[37] Goswami S, Ray S, Sarkar M. Spectroscopic studies on the interaction of DNA with the copper complexes of NSAIDs lornoxicam and isoxicam [J]. International Journal of Biological Macromolecules, 2016, 93: 47-56
[38] Drsata T, Zgarbova M, Jurecka P, et al. On the use of molecular dynamics simulations for probing allostery through DNA [J]. Biophysical Journal, 2016, 110(4): 874-876
[39] 卜晓阳, 吴敏, 周家宏, 等. 光谱法研究有机农药污染物与DNA的相互作用[J]. 应用化学, 2011, 28(4): 458-463
Bu X Y, Wu M, Zhou J H, et al. Spectrometric study on the interactions between organic pesticide pollutants and DNA [J]. Chinese Journal of Applied Chemistry, 2011, 28(4): 458-463 (in Chinese)
[40] 张瑞芳, 张瑞瑞, 卢宇靖, 等. 苯胺取代白叶藤碱衍生物与双链DNA相互作用的光谱及分子对接研究[J]. 计算机与应用化学, 2013, 30(4): 345-348
Zhang R F, Zhang R R, Lu Y J, et al. Interaction study between aniline-substituted cryptolepine derivatives and double-stranded DNA using spectrometric measurement and docking [J]. Computers and Applied Chemistry, 2013, 30(4): 345-348 (in Chinese)
[41] Xu Z, Liu Y, Zhou S, et al. Analysis of the interaction of Dp44mT with human serum albumin and calf thymus DNA using molecular docking and spectroscopic techniques [J]. International Journal of Molecular Sciences, 2016, 17(11): 1914
[42] Ataci N, Arsu N. Studies of the binding mode of TXNHCH2COOH with calf thymus DNA by spectroscopic methods [J]. Spectrochimica Acta Part A-Molecular and Biomolecular Spectroscopy, 2016, 169: 128-133
[43] Zou N, Wang X, Li G. Spectroscopic and electrochemical studies on the interaction between luteolin and DNA [J]. Journal of Solid State Electrochemistry, 2016, 20(6): 1775-1782
[44] Tian Z, Huang Y, Zhang Y, et al. Spectroscopic and molecular modeling methods to study the interaction between naphthalimide-polyamine conjugates and DNA [J]. Journal of Photochemistry and Photobiology B: Biology, 2016, 158: 1-15
[45] Silva M M, Savariz F C, Silva-Junior E F, et al. Interaction of beta-carbolines with DNA: Spectroscopic studies, correlation with biological activity and molecular docking [J]. Journal of the Brazilian Chemical Society, 2016, 27(9): 1558-1568
◆
StudiesontheInteractionbetweenGrapheneandp53-DNAbyMolecularDynamicsSimulationsandSpectroscopicMethods
Wu Huifeng1,2,*, Zhang Mingxing1,3, Li Fei1, Cao Tiangui1, Li Xuehua4, Zhao Jianmin1
1. Key Laboratory of Coastal Environmental Processes and Ecological Remediation of Chinese Academy of Sciences, Yantai Institute of Coastal Zone Research (YIC), Chinese Academy of Sciences, Yantai264003, China2. Functional Laboratory of Marine Fisheries Science and Food Production Process, Qingdao National Laboratory for Marine Science and Technology, Qingdao266237, China3. University of Chinese Academy of Sciences, Beijing100049, China4. Key Laboratory of Industrial Ecology and Environmental Engineering, School of Environmental Science and Technology, Dalian University of Technology, Dalian116024, China
10.7524/AJE.1673-5897.20170104001
2017-01-04录用日期2017-03-02
1673-5897(2017)3-243-08
X171.5
A
吴惠丰(1977-),男,理学博士,研究员,主要从事生态毒理与计算毒理学方面的研究。
国家自然科学基金(41530642, 21677173);中国科学院青年创新促进会资助(2017255)
吴惠丰(1977-),男,研究员,研究方向为生态毒理学和计算毒理学,E-mail: hfwu@yic.ac.cn
吴惠丰, 张明兴, 李斐, 等. 石墨烯与抑癌基因p53 DNA片段相互作用的分子模拟与光谱学验证[J]. 生态毒理学报,2017, 12(3): 243-250
Wu H F, Zhang M X, Li F, et al. Studies on the interaction between graphene and p53-DNA by molecular dynamics simulations and spectroscopic methods [J]. Asian Journal of Ecotoxicology, 2017, 12(3): 243-250 (in Chinese)
