丹参酮合成相关的候选基因CYP76AK5克隆及生物信息学分析△
2017-09-21浦香东王丽芝
浦香东,王丽芝
(天津中医药大学中药学院,天津 300193)
·基础研究·
丹参酮合成相关的候选基因CYP76AK5克隆及生物信息学分析△
浦香东,王丽芝1*
(天津中医药大学中药学院,天津300193)
目的:基于丹参基因组和转录组筛选、克隆及分析丹参酮合成途径候选的CYP450编码基因。方法:设计引物克隆SmCYP76AK5编码基因,通过MEGA软件构建系统进化树并结合实时定量PCR分析其在根不同组织(周皮、韧皮部、木质部)、根、茎、叶、花中的差异表达;最后利用在线生物信息学工具分析其理化性质,预测其二级结构、保守结构域等。结果:系统进化树分析表明SmCYP76AK5属于CYP71clan;基因差异表达显示SmCYP76AK5在根周皮中表达最高,与丹参酮的合成和积累相一致。结论:预测丹参SmCYP76AK5参与丹参酮的生物合成。
丹参;丹参酮;生物合成;SmCYP76AK5;差异表达
药用植物次生代谢产物是天然药物的主要来源,萜类化合物在植物天然产物中种类最多,如能够有效治疗癌症的紫杉醇和治疗疟疾的青蒿素等。丹参为唇形科鼠尾草属多年生草本植物丹参SalviamiltiorrhizaBunge的干燥根和根茎,在心脑血管疾病诊疗中效果显著,丹参酮类化合物是其重要的活性成分之一[1-5]。以丹参酮为主要成分的复方丹参滴丸于2016年3月16日顺利提前完成美国食品和药品管理局(Food and Drug Administration,FDA)三期临床试验,届时将成为首例获得美国FDA认证的复方中药。由于其重要的药理价值及资源需求,丹参酮生源途径的解析得到国内外的广泛关注。丹参作为药用模式植物,对其丹参酮生物合成途径分子机制的解析具有重要的科学意义和应用价值[6]。
丹参酮上游生物合成途径与其他植物萜类合成途径较一致,具有共同的萜类合成前体,异戊烯基焦磷酸(Isopentenyldiphosphate,IPP),由甲羟戊酸(MVA)和甲基赤藓醇4-磷酸(MEP)途径合成而来[7-8]。MVA途径由6个关键酶(AACT、HMGS、HMGR、MK、PMK、MDC)依次催化乙酰辅酶A(Acetyl-CoA)合成IPP;MEP途径由7个关键酶(DXS、DXR、MCT、CMK、MDS、HDS、HDR)逐步催化3-磷酸甘油醛(Glyceraldehyde-3-phosphate,G3P)和丙酮酸(Pyruvate)合成IPP;萜类前体IPP在异戊烯基焦磷酸异构酶(Isopentenyldiphosphateisomerase,IDI)的作用下可逆催化二甲基丙烯基焦磷酸(Dimethylallyldiphosphate,DMAPP)的合成;然后在牻牛儿基牻牛儿基焦磷酸合成酶(Geranylgeranyldiphosphatesynthase,GGPPS)的催化作用下聚合IPP和DMAPP 形成二萜前体牻牛儿基牻牛儿基焦磷酸(Geranylgeranyldiphosphate,GGPP);最后在关键酶SmCPS和SmKSL的作用下催化GGPP合成次丹参酮二烯(Miltiradiene),其作为丹参酮合成的前体物质,在一系列CYP450s的催化作用下生成丹参酮类化合物[9-12]。
细胞色素P450(CYP450)是一类含亚铁血红素的酶,在植物中CYP超家族基因占蛋白质编码基因的1%,具有广泛的底物选择性,目前植物中已知的底物有数百种,其催化活性多而复杂,如烃基的氧化和过氧化作用[13-15]。丹参酮前体次丹参酮二烯在丹参中通过一系列反应由烯烃前体形成,在不同的CYP450s作用下生成多样的丹参酮类化合物,已知的有:SmCYP76AH1催化次丹参酮二烯合成铁锈醇(Ferruginol);SmCYP76AH3催化铁锈醇合成羟基化铁锈醇(11-hydroxy ferruginol)、柳杉酚(Sugiol)、羟基化柳杉酚(11-hydroxy ferruginol);SmCYP76AK1催化二羟基铁锈醇(11,20-dihydroxy ferruginol)和二羟基柳杉酚(11,20-dihydroxy sugiol)的合成[16-19]。丹参酮的生物合成需要CYP450s参与,本研究基于Xu等筛选的33个丹参根周皮显著高表达的CYP450[12],进一步结合已发表的丹参全基因组及转录组信息,筛选并克隆到可能参与丹参酮合成的SmCYP76AK5,系统分析其进化关系及差异表达,并完成其理化性质、蛋白质二级结构以及三级结构的预测,为下一步研究其功能,实现丹参酮类化合物的体外生产或探索其体内代谢途径奠定基础[20-25]。
1 材料
丹参植物材料(99-3)来自中国医学科学院药用植物研究所。丹参根、茎、叶、花、根周皮、根韧皮部、根木质部均取自中国医学科学院药用植物研究所实验田。用于构建CYP450系统进化树的拟南芥(Arabidopsisthaliana)CYP450氨基酸序列来自于CYP450数据库(http://drnelson.uthsc.edu/Arablinks.html)。
2 方法
2.1丹参酮合成候选CYP450基因的筛选及克隆
基于丹参基因组注释信息,筛选出基因组上注释到的CYP450,统计其在周皮、韧皮部和木质部中的差异表达结果,以log2 ratio≥1(绝对值)、FPKM>10为标准,初步筛选候选的CYP450。分别取丹参根的3个组织(周皮、韧皮部和木质部)以及丹参不同器官(根、茎、叶、花)为材料,采用QIAGEN RNeasy Plus Mini Kit(Cat.74134)试剂盒提取丹参根总RNA,PrimeScriptTMⅡ 1st Strand cDNA Synthesis Kit(Cat.6210A)试剂盒合成一链cDNA。不同组织的RNA样品混合作为模板用Takara Ex Taq DNA 聚合酶对候选CYP450基因进行克隆,引物(5′F-CGCATGCAAGTTTACATTCTTCTC/3′RTTAAAGCTTGATAGGAATAGCCC);反应条件为:94 ℃预变性5 min,然后进行35个循环(94 ℃ 30 s,65℃ 30 s,72℃ 2 min),循环结束后72 ℃充分延伸10 min,10 ℃保温,1%琼脂糖凝胶电泳检测,回收后连接PMD-18T载体测序。
2.2候选SmCYP450基因的系统进化分析
将下载的拟南芥CYP450s、已验证功能的丹参SmCYP76AH1、SmCYP76AH3、SmCYP76AK1、与本研究筛选的CYP450的氨基酸序列进行比对,采用MEGA6 软件构建NJ 系统进化树,bootstrap 选择重复1000次[26]。
2.3候选CYP450基因在丹参不同组织器官的表达分析
采用Primer Premier 6 软件进行qPCR引物设计原则如下:PCR 扩增子区域在100~180 bp;Tm 值在60 ℃左右;GC含量在45%~55%。将扩增子区的序列BLAST 比对到基因组序列,选择特异性高的区段进行PCR 扩增。2%琼脂糖凝胶检测PCR 扩增子大小、引物特异性以及引物二聚体。qPCR引物序列如下(5′F-GCTTGCTCTCCACCAACTC,3′R-GGCTTCTCCGACGTTCATG)。实时定量PCR采用ABI 7500 real-time PCR检测系统,参照Takara公司的SYBR®Premix Ex TaqTMⅡ(TliRNaseH Plus)qPCR试剂(Cat.RR820A)使用说明,选择丹参看家基因SmActin作为内参基因,反应体系:7.5 μL SYBR Premix Ex Taq II(2×)、1.2 μL Primers (F/R) (10 μmol·L-1)、0.3 μL ROX Reference Dye Ⅱ(50×)、2 μL DNA samples、4 μL RNase-free ddH2O,反应条件:预变性95 ℃ 30 s;40 循环:变性95 ℃ 5 s,退火和延伸60 ℃ 34 s;溶解曲线:95 ℃ 15 s,60 ℃ 1 min,95 ℃ 15 s;每组反应设置3个技术重复。采用2-ΔΔCt方法计算基因的相对表达;采用IBM SPSS 20软件的一步法ANOVA 分析多样品间的差异性,P<0.05、P<0.01为差异有统计学意义,采用Pearson’s correction test检测基因共表达。
2.4候选CYP450基因结构分析
基因编码蛋白的理化性质预测采用ExPASy Proteomics Server 提供的在线工具Protparam(http://www.expasy.ch/tools/protparam.html);Predictprotein在线预测蛋白的二级结构(https://www.predictprotein.org);PFAM预测CYP450保守结构域(http://pfam.xfam.org/search/batch)。
3 结果
3.1丹参酮合成候选SmCYP76AK5编码基因的筛选及克隆
丹参基因组共注释到457个CYP450s,根周皮RNA-seq分析显示21% (96/457)的CYP450s 表达水平FPKM > 10[12],本研究筛选出1个与丹参SmCPS1、SmKSL1和SmCYP76AH1共表达(周皮>韧皮部>木质部;log2 ratio≥1;FPKM>10)的CYP450,筛选出的CYP450命名为SmCYP76AK5。比对丹参基因组发现SmCYP76AK5定位在Scaffold12483,基因长度为1503 bp,编码500个氨基酸,由两个外显子组成。根据基因组注释结果设计SmCYP76AK5全长序列的引物进行体外克隆,测序结果显示获得长度为1503 bp的编码基因,与基因组注释完全一致,基因序列上传美国国立生物技术信息中心(NCBI),GenBank登录号KY807047。
3.2候选SmCYP76AK5的系统进化分析
本研究下载来自拟南芥的9个clan的273条CYP450序列,分别为CYP71 clan、CYP72 clan、CYP97 clan、CYP86 clan、CYP85 clan、CYP74 clan、CYP51 clan、CYP711 clan、CYP710 clan,从中筛选出75条拟南芥的CYP450序列与丹参中已被研究的SmCYP76AK1、SmCYP76AH1、SmCYP76AH3以及丹参筛选到的SmCYP76AK5氨基酸序列进行比对后构建系统进化树,结果显示SmCYP76AK5与SmCYP76AK1、SmCYP76AH1、SmCYP76AH3、AtCYP76C1、AtCYP76G1聚为一大枝,属于CYP71clan,与SmCYP76AK1聚在同一小枝属于CYP76AK亚家族,如图1所示。同源性比对分析显示SmCYP76AK5与SmCYP76AK1、SmCYP76AH1、SmCYP76AH3的氨基酸序列同源性分别为69%、48%、48%。

图1 SmCYP76AK5与丹参CYP450s、拟南芥CYP450s的系统进化关系图
3.3SmCYP76AK5基因的差异表达研究
本研究对筛选的SmCYP76AK5在丹参根的3个组织(周皮、韧皮部和木质部)以及丹参不同器官(根、茎、叶、花)中的RNA-seq数据进行差异表达分析,表达量由高到低依次为根周皮、根韧皮部、根、根木质部、茎、叶、花。根周皮的表达量(FPKM)是韧皮部的6.5倍。在叶和花器官中没有检测到SmCYP76AK5的表达,但在根部其表达量明显升高,这与丹参以根或根茎入药相符,见图2A。
根据克隆结果设计qPCR引物,提取丹参根的3个组织(周皮、韧皮部和木质部)以及丹参不同器官(根、茎、叶、花)的RNA,反转为cDNA,利用设计的qPCR引物进行qPCR分析,其结果显示SmCYP76AK5的表达量由高到低依次为周皮、韧皮部、根、木质部、叶、茎、花,与丹参转录组表达趋势基本一致,并且与SmCPS1、SmKSL1呈共表达,见图2B。
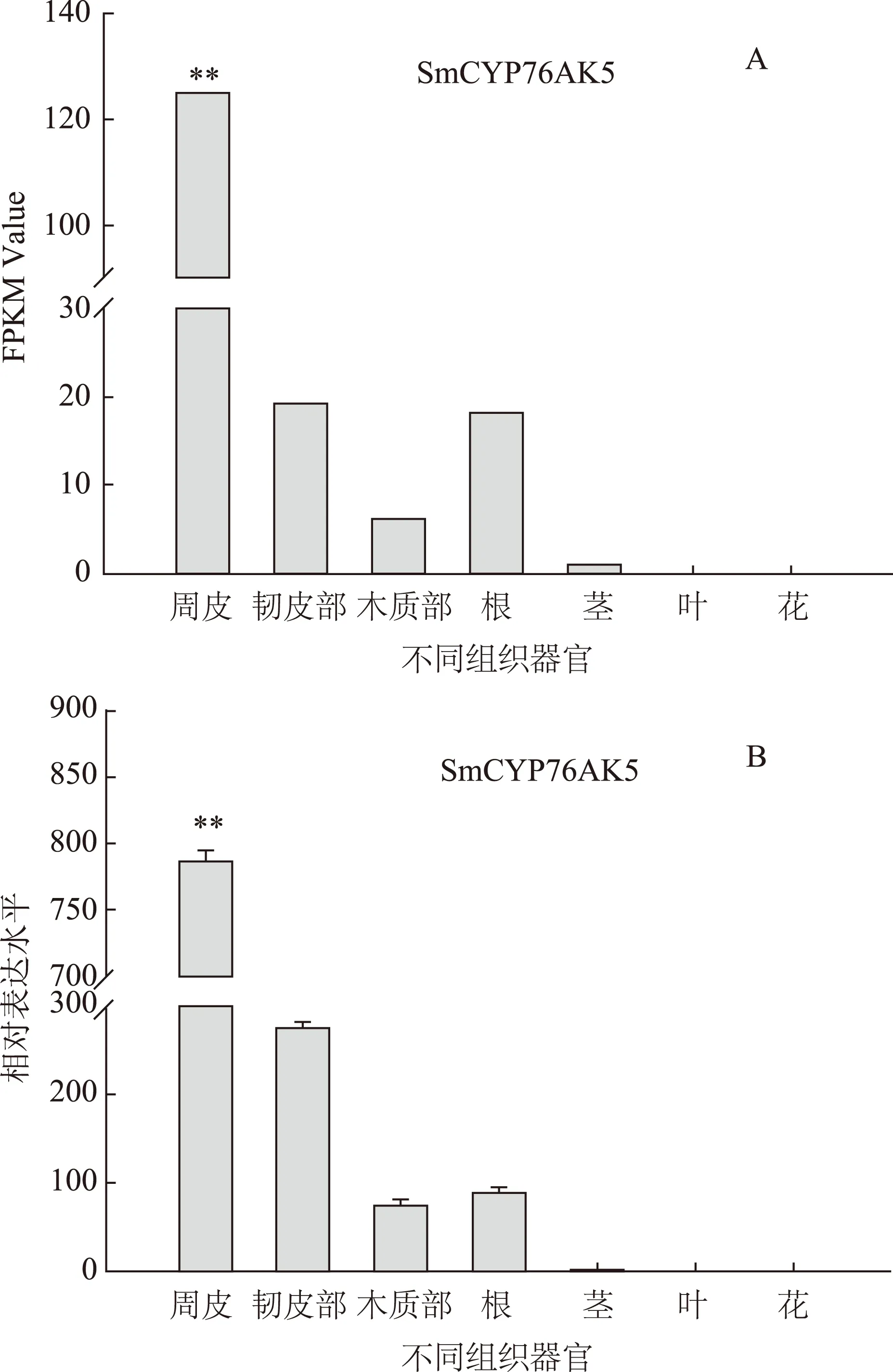
注:A.SmCYP76AK5在不同组织器官的转录组表达差异(FPKM);B.SmCYP76AK5在不同组织器官的qPCR表达分析; **P<0.01。图2 SmCYP76AK5基因在不同组织器官的表达模式
3.4SmCYP76AK5基因的结构特征
SmCYP76AK5基因编码500个氨基酸,利用ExPASy Proteomics Server的在线软件Protparam对SmCYP76AK5氨基酸序列的理化性质进行预测,推测该蛋白的分子式为C2536H4006N700O705S26,分子量是56.4 kD,等电点PI 8.48,带正电残基(Arg+Lys)为57,负电残基(Asp+Glu)为53,该蛋白的不稳定系数为49.80,脂肪系数为93.04,亲水性系数为-0.151。二级结构在线预测结果表明SmCYP76AK5蛋白的二级结构中α-螺旋占50.00%,β-折叠占8.80%,无规卷曲占41.2%。PFAM预测SmCYP76AK5的保守结构域,结果显示该基因编码的蛋白在32-491位点具有CYP450保守结构域(PF00067.20)。
4 讨论
丹参酮作为二萜类化合物,其骨架次丹参酮二烯和铁锈醇的合成已被解析[9],然而到丹参酮的最终合成仍需一系列酶的氧化作用,如氧化酶、脱氢酶等。本研究通过对于丹参基因组以及转录组数据库的分析,验证Xu等对丹参酮合成相关CYP450的鉴定结果,进一步筛选并克隆丹参酮生物合成下游途径的候选基因,为丹参酮生源途径的解析提供参考。
CYP450在修饰萜类骨架产生多种多样萜类化合物中发挥关键作用,这些含血红素的酶通常将氧原子插入C-H键中,使之成为能够被进一步转化的羟基。Boutanaev等报道在真核植物中,参与萜类化合物多样性的CYP450家族成员多属于CYP71 clan[27]。在丹参中,已鉴定SmCYP76AH1、SmCYP76AH3、SmCYP76AK1催化次丹参酮二烯合成铁锈醇、羟基化铁锈醇、柳杉酚、羟基化柳杉酚等丹参酮中间产物[16-17],均归类于CYP71 clan的CYP76亚家族。本研究首次克隆丹参SmCYP76AK5编码基因,与拟南芥CYP450的系统进化分析显示其同属于CYP71 clan的CYP76亚家族。
Xu等证实丹参根周皮是丹参酮合成和积累的主要部位,丹参酮生物合成途径关键酶编码基因的表达在根及根周皮中显著高表达[12]。本研究通过转录组和qPCR揭示SmCYP76AK5编码基因与丹参酮合成途径关键酶编码基因的表达模式相一致,与丹参酮在丹参不同组织器官中的含量呈正相关。基于此,本研究提出SmCYP76AK5催化丹参酮合成的学术假设。
SmCYP76AK5的功能机理仍需要进一步通过体外酶促反应和体内RNAi或过表达研究来证实,本研究为丹参酮合成途径修饰酶编码基因的筛选提供参考,为丹参酮生物合成途径的解析奠定基础。
[1] 国家药典委员会.中华人民共和国药典[S].一部.北京:中国医药科技出版社,2015.
[2] Wang X,Morris-Natschke S L,Lee K H.New developments in the chemistry and biology of the bioactive constituents of Tanshen[J].MedRes Rev,2007,27(1):133-148.
[3] Dong Y,Morris-Natschke S L,Lee K H.Biosynthesis,total syntheses,and antitumor activity of tanshinones and their analogs as potential therapeutic agents[J].Nat Prod Rep,2011,28(3):529-542.
[4] Jackson A J,Hershey D M,Chesnut T,et al.Biochemical characterization of the castor bean ent-kaurene synthase(-like) family supports quantum chemical view of diterpenecyclization[J].Phytochemistry,2014,103:13-21.
[5] Gershenzon J,Dudareva N.The function of terpene natural products in the natural world[J].Nat Chem Biol,2007,3:408-414.
[6] 宋经元,罗红梅,李春芳,等.丹参药用模式植物研究探讨[J].药学学报,2013,48(7):1099-1106.
[7] Ma Y,Yuan L,Wu B,et al.Genome-wide identification and characterization of novel genes involved in terpenoid biosynthesis in Salvia miltiorrhiza[J].J Exp Bot,2012,63:2809-2823.
[8] Cui G,Duan L,Jin B,et al.Functional divergence of diterpene syntheses in the medicinal plant Salvia miltiorrhiza Bunge[J].Plant Physiol,2015,169:1607-1618.
[9] Zhou Y J,Gao W,Rong Q,et al.Modular pathway engineering of diterpenoid synthases and the mevalonic acid pathway for miltiradiene production[J].J Am Chem Soc,2012,134(6):3234-3241.
[10]Peters R J.Two rings in them all:the labdane-related diterpenoids[J].Nat Prod Rep,2010,27:1521-1530.
[11]Kitaoka N,Lu X,Yang B,et al.The application of synthetic biologyto elucidation of plant mono-,sesqui-,and diterpenoid metabolism[J].MolPlant,2015,8:6-16.
[12]Xu Z,Peters R J,Weirather J,et al.Full-length transcriptome sequences and splice variants obtained by a combination of sequencing platforms applied to different root tissues of Salvia miltiorrhiza and tanshinone biosynthesis[J].Plant J,2015,82(6):951-61.
[13]Chen H,Wu B,Nelson D R,et al.Computational identification and systematic classification of novel cytochrome P450 genes in Salvia miltiorrhiza[J].PLoSOne,2014,9:28.
[14]Kaspera R,Croteau R.Cytochrome P450 oxygenases of Taxol biosynthesis[J].Phytochem Rev,2006,5:433-444.
[15]Pateraki I,Heskes A M,Hamberger B.Cytochromes P450 for terpenefunctionalisation and metabolic engineering[J].AdvBiochemEng Biot,2015,148:107-139.
[16]Guo J,Zhou Y J,Hillwig M L,et al.CYP76AH1 catalyzes turnover of miltiradiene in tanshinones biosynthesis and enables heterologous production offerruginol in yeasts[J].ProcNatlAcadSci USA,2013,110:12108-12113.
[17]Guo J,Ma X,Cai Y,Ma Y,et al.Cytochrome P450 promiscuity leads to a bifurcating biosynthetic pathway for tanshinones[J].New Phytol,2016,210(2):525-534.
[18]Zi J,Peters R J.Characterization of CYP76AH4 clarifies phenolic diterpenoid biosynthesis in the Lamiaceae[J].Org Biomol Che,2013,11:7650-7652.
[19]Kai G,Xu H,Zhou C,et al.Metabolic engineering tanshinone biosynthetic pathway in Salvia miltiorrhiza hairy root cultures[J].Metab Eng,2011,13:319-327.
[20]Xu H,Song J,Luo H,et al.Analysis of the Genome Sequence of the Medicinal Plant Salvia miltiorrhiza[J].MolPlant,2016,9(6):949-952.
[21]Luo H,Zhu Y,Song J,et al.Transcriptional data mining of Salvia miltiorrhiza in response to methyl jasmonate to examine the mechanism of bioactive compound biosynthesis and regulation[J].Plant Physiol,2014,152:241-255.
[22]Renault H,Bassard J E,Hamberger B,et al.Cytochrome P450-mediated metabolic engineering:current progress and future challenges[J].CurrOpinPlant Biol,2014,19:27-34.
[23]Wang J W,Wu J Y.Tanshinone biosynthesis in Salvia miltiorrhiza and production in plant tissue cultures[J].ApplMicrobiol B,2010,88:437-449.
[24]李滢,孙超,罗红梅,等.基于高通量测序454 GS FLX的丹参转录组学研究[J].药学学报,2010,45(4):524-529
[25]Xu Z,Ji A,Zhang X,et al.Biosynthesis and Regulation of Active Compounds in Medicinal Model Plant Salvia miltiorrhiza[J].Chinese Herbal Medicines,2016,8(1):3-11.
[26]Castillo D A,Kolesnikova M D,Matsuda S P.An effective strategy for exploring unknown metabolic pathways by genome mining[J].J Am Chem Soc,2013,135(15):5885-94.
[27]Boutanaev A M,Moses T,Zi J,et al.Investigation of terpene diversification across multiple sequenced plant genomes[J].ProcNatlAcadSci USA,2014,E:81-88.
CloningandBioinformaticAnalysisofCYP76AK5RelatedtoTanshinoneBiosynthesisinSalviamiltiorrhiza
PU Xiangdong1,WANG Lizhi1*
(School of Chinese Meteria Medica,Tianjin University of Traditional Chinese Medicine,Tianjin 300193,China)
Objective:The candidate CYP450 gene related to tanshinone biosynthesis was selected and cloned based on genome-wide strategy inSalviamiltiorrhiza.Methods:The specific primers were designed to clone theSmCYP76AK5 gene.The phylogenetic tree was constructed by MEGA software.The different expression ofSmCYP76AK5 in periderm,phloem,xylem,root,stem,leaf and flower was detected by real-time quantitative PCR.The physical and chemical properties,secondary structure,conserved domain were analyzed by a series of bioinformatic tools.Results:The phylogenetic analysis showed that SmCYP76AK5 belongs to CYP71clan.The expression ofSmCYP76AK5 was the highest in periderm,in accordance with the synthesis and accumulation of tanshinone.Conclusion:SmCYP76AK5 was predicted to be involved in tanshinone production inS.miltiorrhiza.
Salviamiltiorrhiza;tanshinone;biosynthesis;SmCYP76AK5;different expression
国家自然科学基金项目(81603221);天津市自然科学基金项目(16JCQNJC09700);天津市高等学校科技发展基金计划项目(20120202/20140209);天津市大学生创新创业训练计划(201610063003)
] 王丽芝,讲师,研究方向:药用植物分子生物学;Email:Lzhwang_2009@163.com
10.13313/j.issn.1673-4890.2017.8.011
2017-03-28)
*[
