北京豆汁微生物群落分析及淀粉絮凝菌分离鉴定
2017-09-18,,
,,
(锦州医科大学食品科学与工程学院,辽宁锦州 121001)
北京豆汁微生物群落分析及淀粉絮凝菌分离鉴定
张莉力,刘黎莹,许云贺*
(锦州医科大学食品科学与工程学院,辽宁锦州 121001)
采集了三个商家的北京豆汁和麻豆腐样本,共12份,通过高通量测序技术分析其中的细菌群落组成及多样性,并利用绿豆汁培养基对优势菌进行分离纯化和鉴定。结果表明,厚壁菌(Firmicutes)、变形杆菌(Proteobacteria)和拟杆菌(Bacteroidetes)是豆汁和麻豆腐的优势菌门。乳酸乳球菌(Lactococcus)、链球菌(Streptococcus)、乳酸杆菌(Lactobacillus)是豆汁中的优势菌属,乳酸乳球菌(Lactococcus)、乳酸杆菌(Lactobacillus)、肺炎克雷伯菌(Klebsiella)和醋酸杆菌(Acetobacter)是麻豆腐中的优势菌属。利用产酸能力和絮凝淀粉能力筛选出两株符合豆汁发酵要求的絮凝产酸菌株D-23和M-10,经16S rRNA 测序鉴定分别为乳酸乳球菌乳酸亚种(Lactococcuslactissubsp.Lactis)和醋酸杆菌(Acetobacterindonesiensis)。这两株菌对北京豆汁工业化纯菌发酵生产具有重要意义。
豆汁,麻豆腐,高通量测序,淀粉絮凝菌,分离鉴定
北京豆汁是具有代表性的老北京小吃之一,其历史悠久,源远流长[1-4]。豆汁是加工绿豆淀粉后的副产物,在绿豆淀粉加工中也被称为酸浆[5]。兑入生豆汁是绿豆淀粉加工的关键工序,生豆汁发挥两方面作用:一方面,生豆汁中的微生物可以作为絮凝剂加速淀粉沉降。生豆汁加入绿豆粉浆中以后,淀粉迅速凝集成絮团并快速沉降,淀粉相对密度最大,占最底层,淀粉上层是含蛋白质、糊精等物质的“麻豆腐”,最上层即为灰绿色的液体,此液体经过6~8 h的发酵变酸后就是生豆汁[6-7],因此兑入生豆汁的另一个作用就是作为发酵剂,使豆汁发酵变酸。
豆汁是自然发酵的产物,产品质量不稳定。已有研究表明,不论是絮凝淀粉活性还是发酵豆汁产酸都是豆汁中的微生物发挥的作用。所以,研究豆汁的微生物区系,筛选出具有絮凝淀粉活性的产酸菌,对于豆汁的工业化发酵具有重要意义[7-10]。发酵产酸是豆汁发酵的主要过程。丁玉振等采用传统平板培养技术分别分析了不同商家和实验室自然发酵豆汁中的主要产酸菌,确定Lactococcuslactis、Lactobacilluscurvatus和Leuconostoccitreum为主要发酵产酸菌[1,11]。传统平板分离技术研究微生物群落具有一定的局限性,为了筛选出可用于豆汁发酵的菌种,需要更加全面探明自然发酵豆汁及麻豆腐微生物区系,本研究利用高通量测序技术,分析不同商家未经煮制的豆汁和麻豆腐的微生物多样性,确定豆汁和麻豆腐的优势菌群,以此为基础结合传统平板分离技术筛选既可以加速淀粉沉降又可以发酵产酸的豆汁发酵菌,为老北京豆汁的工业化生产奠定基础[12-13]。
1 材料与方法
1.1材料与仪器
豆汁 北京不同地点三个商家(DZ、LCQK、LFS)的北京豆汁样本(未煮制)3个和麻豆腐样本3个(每个样本2个重复),豆汁样本标记为D(D-DZ-1、D-DZ-2;D-LCQK-1、D-LCQK-2;D-LFS-1、D-LFS-2),麻豆腐样本标记为M(M-DZ-1、M-DZ-2;M-LCQK-1、M-LCQK-2;M-LFS-1、M-LFS-2),共计12个样本,储存在液氮中,用于DNA 的提取和高通量测序分析;绿豆和绿豆淀粉 市售食用级;蔗糖、葡萄糖、乳糖、酵母膏、磷酸氢二钾、乙酸钠 分析纯;绿豆汁培养基 20 g绿豆放于1 L蒸馏水中,煮沸20 min过滤,加入20 g葡萄糖、2 g乳糖、5 g醋酸钠、5 g酵母提取物、2 g K2HPO4,添加蒸馏水到1 L,121 ℃灭菌15 min;绿豆汁琼脂培养基 绿豆汁培养基1 L、15 g琼脂,121 ℃灭菌15 min。
SW-CJ-2FD型双人单面净化工作台 上海苏净实业有限公司;LRH-350F生化培养箱 上海捷呈实验仪器有限公司;HZQ-F200型振荡培养箱 上海华邻实业有限公司;DNA快速提取试剂盒 天根生化科技(北京)有限公司;TG16K-II台式高速离心机 济南福的机械有限公司;PHS-3B精密pH计 上海雷磁仪器厂;UV754PC紫外分光光度计 上海佑科仪表有限公司;JYL-Y99料理机 九阳股份有限公司;OLYMPUS BX53显微镜 奥林巴斯(中国)有限公司;S900扫描电镜 Hitachi公司。
1.2实验方法
1.2.1 DNA提取与高通量测序 使用DNA提取试剂盒对12个样本的DNA分别进行提取。使用细菌16S V4 rDNA通用引物序列 520F:AYTGGGYD TAAAGNG;802R:TACNVGGGTATCTAATCC[14],扩增16S rDNA V4 可变区。扩增条件:初始变性温度98 ℃、5 min;98 ℃、30 s,50 ℃、30 s,72 ℃、30 s,28个循环;72 ℃、5 min。纯化PCR产物,并利用 TruSeq文库构建试剂盒构建文库。通过Illumina Miseq平台测序。测序得到的原始数据经过质量过滤,利用软件FLASH对通过质量过滤的序列进行连接。运用QIIME进行序列过滤,运用MOTHUR软件中uchime的方法去除嵌合体序列,得到最终用于后续分析的优质序列。16S rDNA V4 可变区扩增和测序工作由上海派森诺生物技术有限公司完成。
1.2.2 操作分类单位(OUTs)聚类 在QIIME 中调用 uCLUST的方法对优质序列按相似度0.97进行聚类,选取每个类最长的序列为代表序列。在 QIIME 中采用 BLAST的方法与序列数据库Greengene进行比对,获得每个 OTUs 分类学信息。根据OTUs列表中的各样本物种丰度情况,应用软件MOTHUR中的Summary single 命令,计算3种常用的生物多样性指数。利用R 软件生成组间OTUs 的维恩图。
1.2.3 菌株的分离 从豆汁和麻豆腐样本中,每个样本取25 mL在30 ℃、150 r/min 孵育5 min,进行系列稀释(10-3~10-8)。每个稀释度取0.1 mL涂布于含有1%碳酸钙的绿豆汁琼脂培养基平板上,30 ℃ 培养 24~48 h[15]。挑取具有碳酸钙溶解圈的菌落进行纯化,直至获得纯菌落为止。将分离到的菌株接入到绿豆汁培养基中,30 ℃培养24 h,以絮凝能力为指标,筛选出对淀粉有絮凝活性的菌株。
1.2.4 絮凝率(FR)和pH测定 将分离筛选到的菌株接入绿豆汁培养基中,在30 ℃培养24 h,经两次扩培,测定发酵液的pH和絮凝率。絮凝率测定方法:80 mL 蒸馏水中加入0.4 g绿豆淀粉,再加入2 mL菌株发酵液,在容器中搅拌2 min 后,静置5 min。对照组使用蒸馏水替代发酵液。通过测量上清液吸光度的变化,计算絮凝率,公式如下:
式中:A为550 nm对照组光密度值,B为550 nm样本光密度值。
1.2.5 16S rDNA扩增与测序 取筛选菌株发酵液5 mL,4000×g离心10 min,弃上清,使用DNA提取试剂盒提取DNA。使用引物27F(AGAGTTTGA TCCTGGCTCAG)和1492R(CTACGGCTACCTTGTT ACGA)扩增菌株的16S rDNA,扩增条件为 95 ℃、5 min后进入35个扩增循环(95 ℃、30 s,58 ℃、30 s,72 ℃、90 s),最后72 ℃延伸7 min。纯化PCR 产物,进行测序,测序工作由上海派森诺生物技术有限公司完成。测得的序列与GenBank 序列进行比对(BLASTn)[16-18]。
1.2.6 细菌黏附淀粉颗粒的扫描电镜观察 将分离到的菌株(D-23和M-10)分别接入绿豆汁培养基中,在30 ℃培养24 h后为分离菌株培养液。取绿豆淀粉2.5 g 加水100 mL搅拌均匀,加入分离菌株培养液10 mL,搅拌均匀,沉淀5 min后去上清,收集底部结合细菌的淀粉颗粒。结合细菌的淀粉颗粒使用3%戊二醛溶液固定,置于离子溅射仪的样本仓中,喷金后,取出,在扫描电镜观察室进行观察[19]。
1.2.7 统计分析 数据统计采用SPSS 19.0 进行ANOVA单因素方差分析及LSD多重检验(p<0.05),数值以均值±标准误表示[20]。
2 结果与分析
2.1 DNA提取与高通量测序
使用DNA提取试剂盒对样本的DNA进行提取后,应用细菌16S V4 rDNA通用引物进行PCR扩增,所有样本都能扩增出有效条带,满足后续Illumina Miseq平台测序要求。高通量测序建库过程中的PCR扩增会产生嵌合体序列,测序过程中会产生点突变等测序错误,为了保证分析结果的准确性,需要对有效序列进行进一步过滤和去除嵌合体处理,得到最终用于后续分析的优质序列。测序得到每个样本的有效序列数和优质序列数见表1。
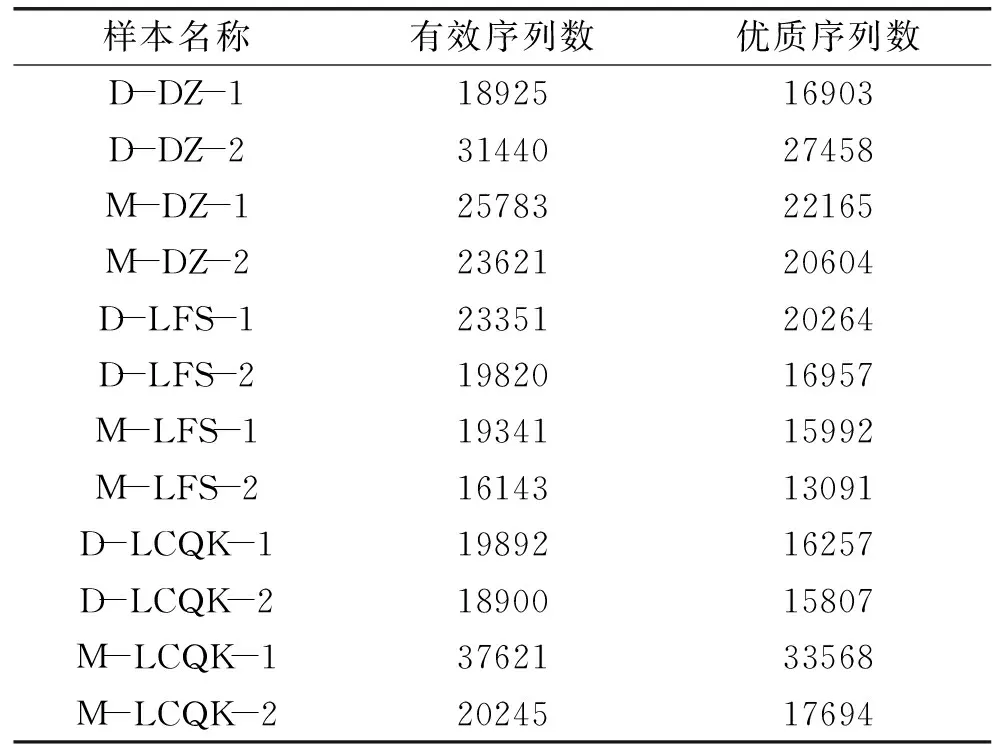
表1 高通量测序获得样本序列数 Table 1 Number of samples sequence by high-throughput sequencing
2.2 OUTs聚类及注释
通过对样本V4 可变区进行PCR扩增,使用Illumina Miseq平台测序,各组获得的OTUs数量见图1。D组(D-DZ、D-LFS、D-LCQK)共获得637个OTUs,其中D-DZ 组428个,D-LFS组449个,D-LCQK组391个(图1a)。M组(M-DZ;M-LFS;M-LCQK)共获得656个OTUs,其中M-DZ组450个,M-LFS组406个,M-LCQK组327个(图1b)。豆汁组中共有OTUs个数为218个,而在麻豆腐组中共有OTUs个数为147个。从OUT总数(637和656)来看,豆汁和麻豆腐差不多,但是从不同商家共有的OUTs数量(218和147)来看,豆汁中多于麻豆腐。

图1 各组间OTUs分布Fig. 1 Analysis of shared OTUs of different groups
菌群Alpha多样性是指一个特定区域或生态系统内的多样性,多样性指数是反映丰富度和多样性的综合指标。Chao和ACE指数是计算菌群丰度的指数,Chao或ACE指数越大,说明群落丰富度越高;Shannon指数是计算菌群多样性的指数,Shannon值越大,说明群落多样性越高。从表2可以看出,豆汁样本间多样性指数差异不显著(p>0.05),说明豆汁中细菌的丰富度和多样性在不同商家之间变化不大。麻豆腐是生产绿豆淀粉的副产物,位于豆汁的下层,其中含有蛋白质等适于微生物生长丰富的营养物质,在麻豆腐中存在大量具有絮凝淀粉能力的微生物。从表3可以看出,不同商家之间麻豆腐中的细菌多样性差异显著(p<0.05),即M-LCQK组的Shannon指数显著低于(p<0.05)M-DZ和M-LFS组。

表2 豆汁中微生物多样性指数Table 2 Microbiota diversity index of Douzhir
注:同列肩标相同小写字母表示差异不显著(p>0.05)。

表3 麻豆腐微生物多样性指数Table 3 Microbiota diversity index of Ma tofu
注:同列肩标不同小写字母表示差异显著(p<0.05);表4同。
2.3豆汁和麻豆腐中微生物组成分布
12个样本中共检测到6个门,三个商家的豆汁微生物区系相近,Firmicutes是最丰富的菌门(67%以上),其次是Proteobacteria和Bacteroidetes(图2)。 Firmicutes和Proteobacteria菌门在D-DZ、D-LFS和D-LCQK组中分别占有97.95%、97.79%和96.28%。在M-DZ和 M-LFS组中 Firmicutes是最丰富的菌门,但是在M-LCQK 组中Proteobacteria 是最丰富的菌门。由此可以看出,除了M-LCQK组外,所有的豆汁样本及M-DZ和M-LFS组都是Firmicutes占绝对优势,其次为Proteobacteria(图2)。在三个商家的豆汁和麻豆腐中,Firmicutes和Proteobacteria两个门的微生物之和占94%以上。

图2 豆汁和麻豆腐中微生物在门水平的组成Fig.2 Distribution of Douzhir and Ma tofu microbiota composition at phylum level注:a. D-DZ;b. D-LFS;c. D-LCQK;d. M-DZ;e. M-LFS;f. M-LCQK;图3同。
豆汁在属水平上的分布见图3。三个不同商家来源的豆汁中优势菌属均为Lactococcus、Streptococcus、Klebsiella和Lactobacillus,所有豆汁样本中Lactococcus含量均最高,从37.6%至54.53%。
麻豆腐样本在属水平上的分布见图3。M-LFS和M-LCQK中均含有Acetobacter,且占绝对优势,M-LCQK中含量达80.67%,M-LFS为31.09%。M-DZ中,Lactococcus为优势菌属,三个不同商家来源的麻豆腐中都含有Lactobacillus,且含量均在10%以上。
在M-LFS和M-LCQK样本中占绝对优势的Acetobacter在豆汁样本中含量不高。麻豆腐的主要成分是蛋白质,绿豆淀粉乳加入生豆汁后pH下降,一些蛋白质沉降下来,位于淀粉上层,豆汁下层;而豆汁的主要成分是一些可溶性的物质,二者的营养成分的差异可能是豆汁与麻豆腐菌群差异的主要原因[2]。
陈宇翔等[3]采用传统平板培养技术分析了北京豆汁的优势菌群,分离出36株乳酸菌,确定豆汁主要是乳酸菌发酵的产物,Lactococcuslactis和Lactobacilluscurvatus为主要发酵乳酸菌。丁玉振等[1]在实验室进行了豆汁自然发酵实验,认为细菌产酸是绿豆乳自然酸化的主要原因,主要产酸微生物为Lactococcuslactis和Leuconostoccitreum。高通量测序技术与传统培养技术对豆汁的微生物菌群的检测结果存在一定的差异,平板培养技术未分离检测到Klebsiella、Streptococcus和Acetobacter等菌属,这可能是由于传统技术受所选择培养基和培养温度等条件的限制,并不适合所有微生物的生长。其次,豆汁本身就是自然发酵的产物,其菌群受到自然条件和操作条件的影响,不同批次、不同商家和不同季节的豆汁其菌群存在差异[12-13,21]。
2.4絮凝淀粉菌株的分离和鉴定
利用含有碳酸钙的绿豆汁培养基平板,从样本中分离出56株有碳酸钙溶解圈的产酸菌株,之后以是否对淀粉具有絮凝能力为指标,初步筛选出8株对淀粉有絮凝活性的菌株。以絮凝率和pH为指标复筛发酵液pH在4.0以下且絮凝率高的菌株。从表4中可以看出,菌株D-23和M-10的絮凝率大于50%,显著高于其他菌株(p<0.05),而且发酵液pH在4以下。
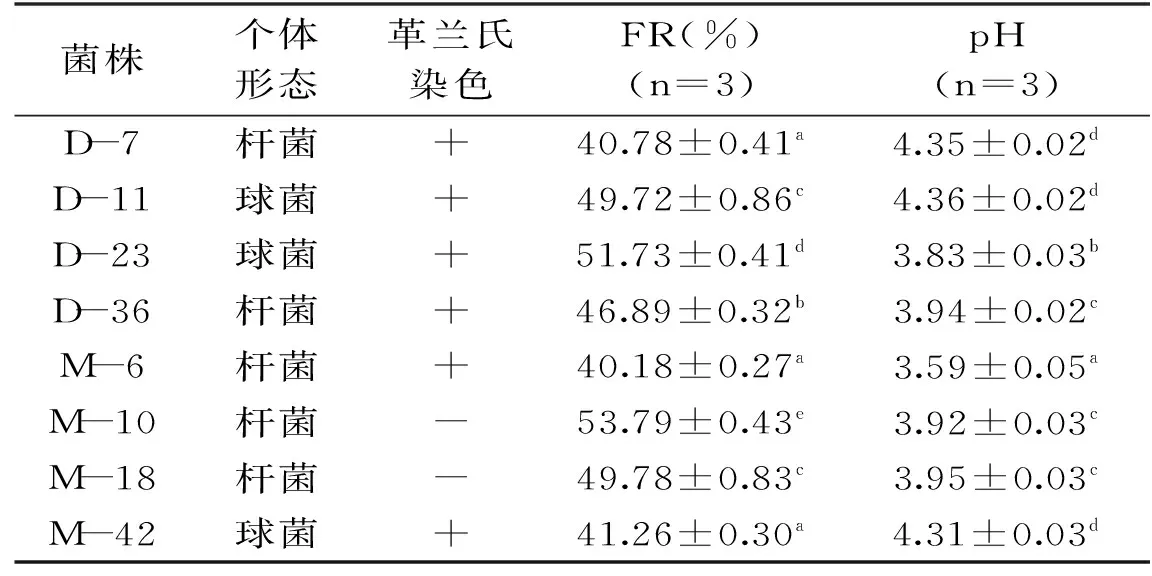
表4 初筛菌株的絮凝率和pHTable 4 FR and pH of primary screening strains
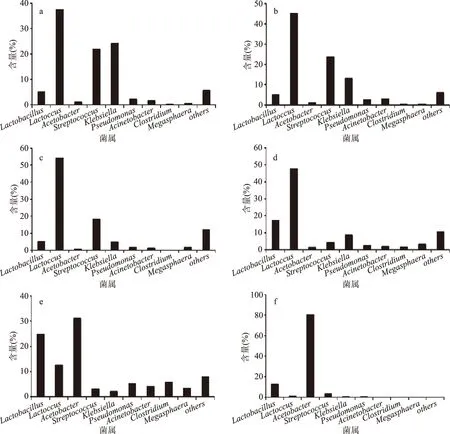
图3 豆汁和麻豆腐中微生物在属水平的组成Fig.3 Distribution of Douzhir and Ma tofu microbiota composition at genera level
16S rRNA测序结果比对表明 D-23 菌株为Lactococcuslactissubsp.lactis,M-10菌株为Acetobacterindonesiensis。D-23菌株是从豆汁样本中分离筛选到的,从图3可以看出,三个不同商家来源的豆汁中优势菌属均为Lactococcus。M-10菌株是从麻豆腐样本中分离筛选到的,从图3可以看出,Acetobacter是麻豆腐样本中M-LFS和M-LCQK组的优势菌属。可见,本研究筛选到的两株菌D-23和M-10都来自于样本中的优势菌属。
2.5菌株黏附淀粉颗粒的显微分析
在光学显微镜下观察到加入筛选菌株发酵液前后绿豆淀粉的分布状态:图4(a)为加入筛选菌株发酵液前,淀粉颗粒在显微镜下均匀分布;图4(b、c)为加入筛选菌株D-23和M-10发酵液后,众多淀粉颗粒凝聚成大的絮凝体。通过扫描电镜可以观察到菌体细胞结合到淀粉颗粒表面(图5),将众多淀粉颗粒粘结在一起,使淀粉颗粒凝集成大的絮凝体,正是淀粉颗粒变大后重力增加,从而加速了淀粉的沉降。研究表明,Lactococcuslactis在用于绿豆淀粉加工的酸浆(豆汁在绿豆淀粉加工中也被称为酸浆)中也被分离出来过,且被证明是该菌体而不是菌体产的代谢产物具有加速绿豆淀粉沉降的作用[6-7]。对甘薯酸浆中的微生物絮凝性研究也表明,絮凝活性物质分布于菌体上[22-23]。Acetobacterindonesiensis是本研究首次分离出来被证明具有絮凝淀粉活性的产酸菌。

图4 淀粉颗粒凝集的光学显微镜图 Fig.4 Optical micrograph of starch granule aggregation

图5 菌株D-23和M-10黏附淀粉颗粒扫描电镜图 Fig.5 Scanning electron micrograph of starch granules with attached D-23 and M-10 cells
本研究所筛选的2株有淀粉结合活性的产酸菌株可以作为开发豆汁发酵剂的菌种,为北京豆汁的工业化生产奠定了基础。
3 结论
Firmicutes、Proteobacteria和Bacteroidetes是豆汁和麻豆腐的优势菌门,Firmicutes和Proteobacteria两个门的微生物之和占94%以上。Lactococcus、Streptococcus、Klebsiella和Lactobacillus是豆汁中的优势菌属,Lactococcus、Lactobacillus和Acetobacter是麻豆腐中的优势菌属。
利用平板分离技术从豆汁和麻豆腐的优势菌中分离筛选出2株具有絮凝淀粉活性的产酸菌(Lactococcuslactissubsp.Lactis和Acetobacterindonesiensis),2株菌对淀粉的絮凝率均在50%以上,发酵液的pH在4.0以下。本研究为北京豆汁工业化纯菌发酵生产奠定了基础。
[1]丁玉振,张绍英,梁尽祎,等. 北京传统小吃“豆汁”制作工艺研究[J]. 食品科学,2010,31(2):280-283.
[2]苗志伟,刘玉平,黄明泉,等. 豆汁熬制过程中挥发性香成分的变化[J]. 中国食品学报,2013,13(2):199-204.
[3]陈宇翔,陈历俊,姜铁民. 北京豆汁优势菌群的探究及其发酵性能测试[J]. 食品科技,2013(8):67-70.
[4]Wu H,Rui X,Li W,et al. Mung bean(Vigna radiata)as probiotic food through fermentation with Lactobacillus plantarum B1-6[J]. Lwt-food Science and Technology,2015,63(1):445-451.
[5]Deng F M,Mu T H,Zhang M,et al. Composition,structure,and physicochemical properties of sweet potato starches isolated by sour liquid processing and centrifugation[J]. Starch-Stärke,2013,65(1-2):162-171.
[6]Liu W,Shen Q. Structure analysis of mung bean starch from sour liquid processing and centrifugation[J]. Journal of Food Engineering,2007,79(4):1310-1314.
[7]Liu W J,Shen Q. Studies on the physicochemical properties of mung bean starch from sour liquid processing and centrifugation[J]. Journal of Food Engineering,2007,79(1):358-363.
[8]Cocolin L,Bisson L F,Mills D A. Direct profiling of the dynamics in wine fermentation[J]. FEMS Microbiol Lett,2000, 189(1):81-87.
[9]Li Z,Liu W,Shen Q,et al. Properties and qualities of vermicelli made from sour liquid processing and centrifugation starch[J]. Journal of Food Engineering,2008,86(2):162-166.
[10]Chang Y H,Lin C L,Chen J C. Characteristics of mung bean starch isolated by using lactic acid fermentation solution as the steeping liquor[J]. Food Chemistry,2006,99(4):794-802.
[11]Ding Y Z,Zhang S Y,Peng L,et al. Microbiological and biochemical changes during processing of the traditional Chinese food douzhi[J]. Food Control,2009,20(12):1086-1091.
[12]Hamady M,Knight R. Microbial community profiling for human microbiome projects:Tools,techniques,and challenges[J].Genome Research,2009,19(7):1141-1152.
[13]Escobar-Zepeda A,Sanchez-Flores A,Quirasco B M. Metagenomic analysis of a Mexican ripened cheese reveals a unique complex microbiota[J]. Food Microbiology,2016,57:116-127.
[14]Blanton L V,Charbonneau M R,Salih T,et al. Gut bacteria that prevent growth impairments transmitted by microbiota from malnourished children[J]. Science,2016,351(6275):aad3311-aad3311.
[15]Ryan S M,Fitzgerald G F,Van S D. Screening for and identification of starch-,amylopectin-,and pullulan-degrading activities in bifidobacterial strains[J]. Applied & Environmental Microbiology,2006,72(8):5289-5296.
[16]Altschul S F,Madden T L,Schäffer A A,et al. Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J]. Nucleic Acids Research,1997,25(17):3389-3402.
[17]Gevers D,Cohan F M,Lawrence J G,et al. Opinion:Re-evaluating prokaryotic species[J]. Nature Reviews Microbiology,2005,3(9):733-739.
[18]Guo X H,Kim J M,Nam H M,et al. Screening lactic acid bacteria from swine origins for multistrain probiotics based oninvitrofunctional properties[J]. Anaerobe,2010,16(4):321.
[19]O’Riordan K,Muljadi N,Conway P. Characterization of factors affecting attachment of Bifidobacterium species to amylomaize starch granules[J]. Journal of Applied Microbiology,2001,90(5):749.
[20]Xu Y,Yang H,Zhang L,et al. High-throughput sequencing technology to reveal the composition and function of cecal microbiota in Dagu chicken[J]. Bmc Microbiology,2016,16(1):259.
[21]Riesenfeld C S,Schloss P D,Handelsman J. Metagenomics:Genomic Analysis of Microbial Communities[J]. Annual Review of Genetics,2004,38(1):525.
[22]张莉力,许云贺,李新华. 对甘薯淀粉具有絮凝活性的乳酸菌的分离鉴定及其特性研究[J]. 食品科学,2010,31(7):228-231.
[23]张莉力,许云贺,李新华. 甘薯酸浆中微生物絮凝性研究[J]. 食品工业科技,2010,31(6):172-175.
AnalysisofBeijingDouzhirmicrobiotaandisolationandidentificationofadvantagebacteriumcapableofflocculatingstarch
ZHANGLi-li,LIULi-ying,XUYun-he*
(College of Food Science and Engineering,Jinzhou Medical University,Jinzhou 121001,China)
12 samples of Douzhir(D groups)and Ma tofu(M groups)were collected from three manufacturers. High-throughput sequencing was used to analyze composition and diversity of bacterial community. Based on this method,dominant bacteria were screened and identified using mung bean juice as medium. In both Douzhir and Ma tofu,dominant bacteria belonged to Firmicutes,Proteobacteria and Bacteroidetes. In Douzhir of three manufacturers,predominant genera wereLactococcus,Streptococcus,KlebsiellaandLactobacillus. In Ma tofu of three manufacturers,predominant genera wereLactococcus,LactobacillusandAcetobacter. After sequencing of 16S rRNA,they belong toLactococcuslactissubsp.LactisandAcetobacterindonesiensisrespectively. These two strains had important significance for industrial fermentation production of Beijing Douzhir.
Douzhir;Ma tofu;high-throughput sequencing;starch-flocculating strains;isolation and identification
2017-02-28
张莉力(1977-),女,博士,副教授,主要从事食品微生物方面的研究,E-mail:13634967549@163.com。
*通讯作者:许云贺(1978-),男,博士,副教授,主要从事畜牧微生物应用方面的研究,E-mail:sn_97@126.com。
国家自然科学基金项目(31301499);辽宁省自然科学基金项目(2014022052,2014022046)。
TS201.3
:A
:1002-0306(2017)16-0136-06
10.13386/j.issn1002-0306.2017.16.026
