基于DNA条形码技术的储粮害虫碎片鉴定研究
2017-09-15江亚杰汪中明贺培欢李福君
江亚杰 汪中明 张 涛 贺培欢 李福君 曹 阳 伍 祎
(国家粮食局科学研究院1,北京 100037) (河南工业大学粮油食品学院2,郑州 450001)
基于DNA条形码技术的储粮害虫碎片鉴定研究
江亚杰1,2汪中明1张 涛1贺培欢1李福君1曹 阳1伍 祎1
(国家粮食局科学研究院1,北京 100037) (河南工业大学粮油食品学院2,郑州 450001)
储粮害虫(螨)个体微小,种类繁多,与人类生活紧密相关,具有重要经济意义,储粮害虫快速鉴定是进行储粮害虫综合防治的前提和基础。传统的形态鉴定技术难以实现粮食及食品中害虫的非成虫态和碎片的准确鉴定,DNA条形码是近年来出现的物种分子鉴定技术,能够实现对昆虫非成熟虫态及碎片的快速鉴定。本研究利用DNA条形码技术和联合开发的中国储粮害虫DNA条形码鉴定系统(GPDBIS),针对某食品有限公司送检的储粮害虫碎片样品进行了序列测定、比对和分析,最终实现碎片的快速准确鉴定。结果显示,在所检测的7个害虫碎片样品中,有4头为锈赤扁谷盗CryptolestesferrugineusStephens,3头为赤拟谷盗TriboliumcastaneumHerbst,DNA条形码技术是储粮害虫快速准确鉴定的有效手段。
储粮害虫 害虫碎片 DNA条形码 GPDBIS系统 分子鉴定
储粮害虫是一类与人类生活紧密相关,具有重要经济意义的害虫。粮食储藏期间,环境适宜、食源丰富,导致储量害虫频繁发生,严重影响粮食的数量和质量、种子的发芽率、粮油产品的营养价值,同时储粮害虫的代谢物和虫尸是成品粮的主要卫生污染物[1-2],为微生物在粮油产品上滋生提供有利条件,甚至导致人类过敏反应。
储粮昆虫种类调查及其快速鉴定是进行储粮害虫综合防治的前提和基础,截止2000年,全世界已记载的仓储昆虫和螨类约500种,我国有383种,其中仓储昆虫242种,螨类141种[3]。储粮昆虫种类多,个体微小,传统的储粮昆虫(包括螨类)鉴定工作主要依靠形态学特征,区分不同昆虫种类之间的形态学差别往往非常细微和复杂,需要严格的专业知识学习和培训,且很难对未成熟虫态(卵、幼虫及蛹)、残损样品以及形态相似种类进行快速鉴定。利用现代分子生物学技术进行储粮昆虫鉴定,不受样品个体发育状态和环境条件的影响,对非成虫态昆虫及形态特征不完整的样品,可直接从DNA水平上得到准确、可靠的鉴定。
近年来,随着DNA检测和分析技术的快速发展,各种分子生物技术手段被广泛应用于仓储害虫的种类鉴定。在仓储害虫中,已成功运用随机扩增多态性(Random Amplified Polymorphic DNA,RAPD)[4-5]、限制性片段长度多态性(Restriction Fragment Length Polymorphism,RFLP)[6-7]和基于PCR特异引物技术实现了物种的快速鉴定[8-10]。通过测定某个或某几个基因的序列来进行种类鉴定和区分的DNA条形码(DNA barcode)技术也得到了迅速的发展和使用[11]。分类学家 Hebert 等[12-13]首次提出“生物条形码”的概念,认为线粒体COI基因一段约700 bp的片段可作为动物 DNA 条形码,这一技术可以让非专业人员在很短时间内准确、经济地对目标物种进行鉴定。
基于线粒体上“生物条形码”COI基因,国家粮食局科学研究院和中国农业大学生物入侵检疫实验室联合测试了我国常见储粮虫螨条形码序列,并且设计开发了中国储粮害虫DNA条形码鉴定系统GPDBIS(the Grain Pests DNA Barcode Identification System)。该系统涵盖了我国15个省市34个粮库粮堆中42种储粮虫螨的COI条形码序列,其中鞘翅目17种,啮虫目10种,螨类9种,鳞翅目3种,半翅目2种,膜翅目1种。GPDBIS是为我国储粮害虫(螨)DNA条形码的获得、分析及鉴定提供帮助的信息平台。通过整合生物分子、形态、区域分布及防治的数据,为我国储粮害虫的综合防治提供信息支持。
本研究基于GPDBIS系统,应用DNA条形码分子鉴定技术,对2015年某食品有限公司送检的害虫碎片样品进行mtDNA COI基因片段的序列检测,序列比对和系统发育分析,实现对储粮害虫碎片的快速、准确鉴定。
1 材料与方法
1.1 试验材料
储粮害虫碎片来自于某食品有限公司方便面包装盒上,7个碎片,收集后液浸于100%的乙醇中并于-20 ℃保存。
1.2 试验仪器
Leica M数字显微镜:Leica公司;MicroCL 17台式离心机、VXE380超低温冰箱:Thermo scientific公司;Veriti TM 96-well Thermal CyclerPCR仪:Applied Biosystems公司;Gel Logic 212 PRO凝胶成像系统:Kodak公司;DYCP-31D水平式电泳槽、DYY-7C稳压稳流电泳仪:北京六一仪器厂;UV-Vis spectrophotometer Q5000微量核酸浓度检测仪:Quawell公司。
1.3 试验方法
1.3.1 DNA提取
单一碎片样品基因组DNA的提取选用天根生化科技(北京)有限公司商业化试剂盒“微量基因组DNA提取试剂盒”,提取过程参照试剂盒的使用说明。提取完成后取1 μL DNA模板溶液,用微量核酸浓度检测仪检测DNA的浓度和纯度,剩余基因组DNA放置于-20 ℃保持备用。
1.3.2 PCR扩增及序列测定
扩增的目的片段是mtDNA中COI基因,约650 bp的一段序列,扩增引物参考Folmer所使用的LCO-1490和HCO-2198[14],LCO-1490 5′-GGTCAACAAATCATAAAGATATTG-3′,HCO-2198 5′-TAAACTTCAGGGTGACCAAAAAATCA-3′,由北京奥科生物科技有限公司合成。PCR反应的总体积为52 μL,具体包含:6.0 μL 10×buffer缓冲液,3.0 μL MgCl2,2.0 μL dNTPS, 0.4 μLTaqDNA聚合酶,上下游引物各3.0 μL(10 μmol/L), ddH2O 32.6 μL,DNA模板2.0 μL。反应条件为94 ℃加热3 min,然后进行39次PCR循环:94 ℃变性1 min,50 ℃退火1 min,72 ℃延伸1 min,最后72 ℃延伸10 min,PCR产物保存于4 ℃。取4 μL PCR产物,加入1 μL 6×Loading buffer,混匀,在1%的琼脂糖凝胶上进行电泳检测(1×TAE缓冲液,电压100 V,30 min),采用marker D2000作为分子质量的标准对照,EB染色15 min后在紫外分光光度计下观察PCR的扩增结果。为保证获取序列的准确性,以同一碎片的DNA为模板,进行3次PCR扩增,将获取的PCR反应产物委托生工生物工程(上海)股份有限公司进行纯化并测序。测序反应在ABI-3730测序仪上进行双向测定。
1.4 数据分析
将获得的序列利用软件Chromas 1.45[15]检测序列峰图,利用DNAMAN 5.2(Lynnon BioSoft)进行序列双向比对拼接,获取拼接序列后去除两端引物序列,得到单条准确序列。然后在GPDBIS数据库中的“序列比对和鉴定”功能进行相似性比对,以确定所扩增的序列的种类,然后查看鉴定结果以及相似性数据,最后利用MEGA 5.0[16]软件采用最大似然法(ML)、邻接法(NJ)和简约法(MP)构建所检测的7个碎片样品与GPDBIS数据库中相关储粮害虫种的系统发育树,以核实确定鉴定的结果,明确待鉴定种类在主要储粮害虫中的系统关系。
2 结果与分析
2.1 形态观察结果
通过数字显微镜观察,待鉴定样品缺乏主要的形态鉴定特征,初步确定7片送检样品为鞘翅目碎片,可能为赤拟谷盗/杂拟谷盗/锈赤扁谷盗/长角扁谷盗/土耳其扁谷盗等(图1),通过形态鉴定无法鉴定到种的水平。

注:a 待测碎片,b 赤拟谷盗,c 锈赤扁谷盗。图1 待测样品形态鉴定
2.2 PCR扩增及序列测定结果
PCR产物经凝胶电泳检测后含有一段700 bp左右片段,结果如图2所示,其中M泳道为Marker,1~7泳道为7个储粮害虫碎片的扩增产物,第8泳道为阴性对照。条形码基因扩增结果条带清晰,可作为测序和后续研究。获得的序列经过正反向拼接、同物种序列比对和人工校正,最终得到了长度为650 bp的高质量COI序列7条。
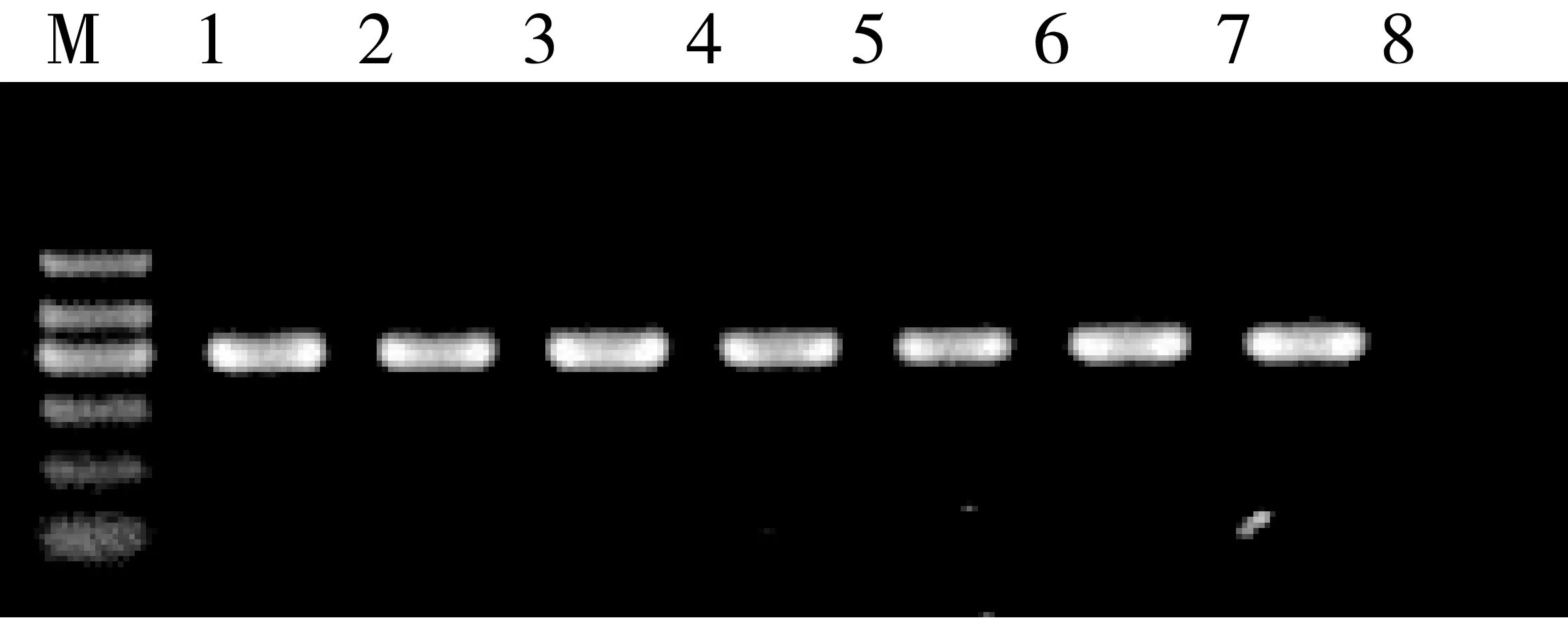
注:M为D2000 Marker,1~7为7个储粮害虫碎片,8为阴性对照。图2 储粮害虫碎片COI基因扩增结果
2.3 相似性比对鉴定结果
经GPDBIS的分子鉴定结果为:7个碎片中包括了2种鞘翅目害虫,其中4个为锈赤扁谷盗,3个为赤拟谷盗。锈赤扁谷盗有4头,对应上述PCR扩增结果的第1、3、4、7泳道,其中1、4、7序列完全相同,序列单倍型命名为CF-Hap1;泳道3的锈赤扁谷盗序列命名为CF-Hap2、CF-Hap1与CF-Hap2存在5个碱基的变异(表1)。这2条序列与GPDBIS中收录的锈赤扁谷盗比对均能找到相似性为100%的序列,其中与CF-Hap1完全一致的锈赤扁谷盗序列包括广东、广西、重庆,与CF-Hap2完全一致的序列来自广东、海南。赤拟谷盗为3头,对应上述PCR扩增结果的第2、5、6泳道,其中2、6序列完全相同,序列命名为TC-Hap1;泳道5的序列命名为TC-Hap2,TC-Hap1与TC-Hap2存在9个碱基变异(表1)。这2条序列与GPDBIS中收录的赤拟谷盗比对,结果显示广东、广西与TC-Hap1完全一致,而广东、四川成都的种群与TC-Hap2完全一致。进一步将获得的2个种4条单倍型序列与GenBank上的数据比对分析,表明待鉴定的种分别为锈赤扁谷盗和赤拟谷盗。

表1 同一物种不同单倍型间的碱基差异
2.4 系统发育树
利用MEGA 5.0软件,分别采用最大似然法、邻接法和简约法构建各条序列与GPDBIS数据库中主要鞘翅目害虫序列之间的系统发育树,3种方法构建的系统树基本相似,以邻接法构建的结果进行分析(图3)。分属各物种的个体的聚集趋势十分显著,待鉴定的4条序列中,2条与锈赤扁谷盗聚为一个类群,2条与赤拟谷盗聚为一个类群,这表明同一物种的个体可以和其他物种的个体很好地区分开,系统发育树的构建更明了的展现了待鉴定害虫碎片的种类,同时可直观看出各个害虫种类在系统树上的关系,如4种扁谷盗聚类在一个大的分支,米象和玉米象,赤尼谷盗与杂尼谷盗等近似种分别聚在同一个大的分支上,且每一个种有其独立的小分支。进一步说明运用该方法和GPDBIS系统能准确鉴定储粮害虫碎片。
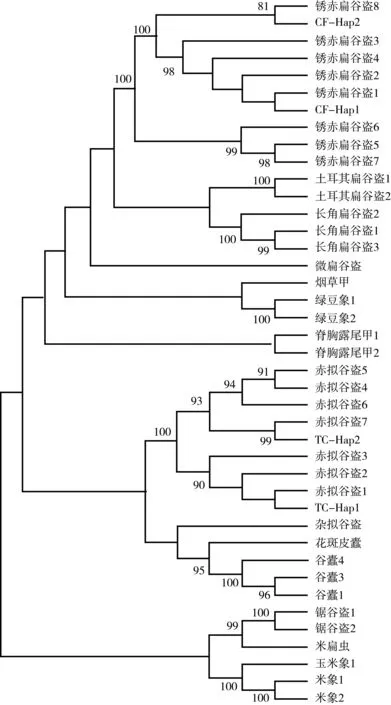
图3 NJ法构建未知种及其相关种系统发育树
3 结论
通过进行线粒体DNA上COI基因700 bp序列的测定和基于GPDBIS系统的比较分析,送检的7个害虫碎片样本得到了成功的鉴定和区分,其中锈赤扁谷盗4头,赤拟谷盗3头。本研究得到的序列可以作为DNA 条形码序列补充进GPDBIS系统,该系统可以应用于原粮、食品加工厂以及一线口岸的进出口粮食及食品实际检疫工作中的害虫鉴定,这种方法的推广在保证物种鉴定准确性的同时,将大大降低非成虫态及虫尸碎片在实际鉴定工作中的难度,提高鉴定效率。同时,本研究进一步验证了650 bpCOI基因片段序列可以对储粮害虫达到种水平的区分,从而实现有关物种的快速、准确鉴定。
GPDBIS系统作为我国储粮虫螨的分子鉴定系统需要逐步的完善和改进。首先,虫螨的种类需要进一步丰富,目前该系统基础数据库仅包括42种储粮虫螨,虽然由于储粮设施和技术的改进,主要储粮害虫种类有所下降,但是我国建国以来进行了6次仓储害虫调查,每次的种类均在百种以上,因此要达到全面的应用需要不断丰富和增加虫螨种类。其次,GPDBIS系统在功能上需要完善,系统目前仅包括单一的基因mtDNA COI片段,以后将逐步补充其他基因片段,以针对不同对象,选取合适的基因进行鉴定、地理种群差异分析等。
各种DNA分子标记已经被广泛应用于亲子鉴定和个体识别领域[17],在储粮害虫分子鉴定领域也有了长足进展[18]。某些物种在长期的进化中,由于外界因素(温度、湿度、光照以及地形地貌等)产生了地理隔离,形成了不同的地理种群品系,这种变异在形态上很难区分,但是会在基因水平上表现出来,通过分子技术手段得到直观的体现。随着分子技术的发展,除了对物种的准确快速鉴定外,未来将逐步实现不同区域、不同品系的鉴定和区分,这对追溯食品害虫来源,保障食品安全将起到重要作用。
[1]Trematerra P,Stejskal V,Hubert J. The monitoring of semolina contamination by insect fragments using the light filth method in an Italian mill[J]. Food Control,2011,22(7): 1021-1026
[2]Wu Y,Li F,Li Z,et al. Microsatellite markers forCryptolestesferrugineus(Coleoptera: Laemophloeidae) and otherCryptolestesspecies[J]. Bulletin of Entomological Research, 2016,106(2):154-160
[3]李隆术,赵志模. 我国仓储昆虫研究和防治的回顾与展望[J]. 昆虫知识,2000,37(2):84-88 Li Longshu,Zhao Zhimo.Review and prospect on the research and control of storage insects in China [J]. Entomological Knowledge,2000,37(2):84-88
[4]Cenis J L,Beitia F. Application of the technique RAPD-PCR to the identification of insects[J]. Investigation Agraria Production by Protection Vegetales,1994,9(2):289-297
[5]Brown R J,Malcolm C A,Mason P L,et al. Genetic differentiation between and within strains of the saw-toothed grain beetle,OryzaephilussurinamensisatRAPD loci[J]. Insect Biology,1997,6(3):285-289
[6]Hidayat P,Phillips T W,Ffrench-Constant R H. Molecular and morphological characters discriminateSitophilusoryzaeandS.zeamais(Coleoptera:Curculionidae) and confirm reproductive isolation[J]. Annals of the Entomological Society of America,1996,89(5):645-652

[8]Mohammad A,Francisco M O,George P O,et al. PCR and isothermal-based molecular identification of the stored-product psocid pestLepinotusreticulatus(Psocoptera:Trogiidae)[J]. Journal of Stored Products Research,2012,49:184-188

[10]林阳武. 应用PCR方法鉴定菜豆象、四纹豆象和绿豆象的研究[D]. 福州:福建农林大学,2008 Lin Yangwu. Detection ofAcanthoscelidesobtectus(Say)、Callosobruchusmaculatus(Fabricius) andCallosobruchuschinensis(Linnaeus) by PCR [D]. Fuzhou: Fujian Agriculture and Forestry University,2008
[11]杨倩倩,李志红,伍祎,等.线粒体COⅠ基因在昆虫DNA条形码中的研究与应用[J]. 应用昆虫学报,2012,49(6):1687-1695 Yang Qianqian,Li Zhihong,Wu Yi,et al. Advance and application of mtDNA COI barcodes on insects[J]. Chinese Journal of Applied Entomology,2012,49(6):1687-1695
[12]Hebert P D N,Cywinska A,Ball S L,et al. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society of London Series B-Biological Sciences,2003a,270(1512):313-321
[13]Hebert P D N,Gregory T R. The promise of DNA barcoding for taxonomy[J]. Systematic Biology,2005,54(5):852-859
[14]Folmer O,Black M,Hoeb W,et al. DNA primers for amplification of mitochondrial cytochrome C oxidase subunit I from diverse metazoan invertebrate[J]. Molecular Marine Biology & Biotechnology,1994,3(5):294-299
[15]Goodstadt L,Ponting C P. CHROMA: consensus-based colouring of multiple alignments for publication[J]. Bioinformatics,2001,17(9):845-846
[16]Tamura K,Peterson D,Peterson N,et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution,2011,28(10):2731-2739[17]李孟华,王海生,赵书红,等. DNA分子标记在动物个体识别与亲权鉴定方面的应用[J].生物技术通报,2001(5):4-7 Li Menghua,Wang Haisheng,Zhao Shuhong,et al. The application of DNA molecular markers on animal individualIdentification and paternity tests [J]. Biotechnology Bulletin,2001(5):4-7
[18]伍祎,李志红,李燕羽,等. DNA分子遗传标记技术在仓储害虫中的研究与应用[J]. 中国粮油学报,2015,30(10):140-146 Wu Yi,Li Zhihong,Li Yanyu,et al. The research and application of DNA molecular genetic markers in stored-product pests [J]. Journal of the Chinese Cereals and Oils Association, 2015, 30(10): 140-146.
Molecular Identification of Stored Grain Pest Fragments by DNA Barcoding
Jiang Yajie1,2Wang Zhongming1Zhang Tao1He Peihuan1Li Fujun1Cao Yang1Wu Yi1
(Academy of State Administration of Grain1, Beijing 100037) (College of Grain, Henan University of Technology2, Zhengzhou 450001)
Stored grain pest (mite) was small individuals with various species, which was related to human lifeclosely and has important economic significance. Rapid identification of stored grain pests was the prerequisite and foundation of integrated pest management. It was difficult to identify immatures and fragments of insect in grain and food by morphological characteristics. Emerging as a promising molecular technology of species identification, DNA barcodes could realize rapid identification of the immatures and fragments. In this study, in order to identify pest fragments samples collected from a food Co., Ltd., based on the DNA barcoding and GPDBIS system, a series of studies, including sequencing, alignment and analysis, were conducted. The results showed that four of the seven insect fragments specimens wereCryptolestesferrugineusStephens, and three of them wereTriboliumcastaneumHerbst.DNA barcode could be used to identify the species of stored-grain insects/mites rapidly and accurately.
stored grain pest, Insect fragments, GPDBIS, DNA barcode, molecular identification
国家重点研发计划(2016YFD0401004),粮食公益性行业科研专项(201513002)
2016-06-20
江亚杰,男,1991年出生,硕士,农业昆虫与害虫防治
伍祎,女,1980年出生,副研究员,植物保护 曹阳,男,1958年出生,研究员,粮油储藏
Q-1
:A
:1003-0174(2017)08-0131-05
