环境因子对太湖底泥细菌群落结构的影响
2017-09-11朱艳霞邱业先
钱 玮,朱艳霞,邱业先
(苏州科技大学 化学生物与材料工程学院,江苏 苏州215009)
环境因子对太湖底泥细菌群落结构的影响
钱 玮,朱艳霞,邱业先*
(苏州科技大学 化学生物与材料工程学院,江苏 苏州215009)
以太湖底泥为研究对象,测定样品中的总氮(TN)、总磷(TP)、总有机碳(TOC),采用变性梯度凝胶电泳(DGGE)研究底泥细菌多样性。此外,还采用冗余分析(RDA)的方法研究了底泥中一些环境因子对微生物群落结构的影响。结果表明:TN、TP、TOC变化能解释大部分细菌群落结构的差异(54.27%),其中TN对细菌群落结构的影响最大,其次是TP,再次是TOC。该研究对太湖地区微生物资源的调查及环境监测具有重要意义。
太湖底泥;变性梯度凝胶电泳;冗余分析
太湖是中国第二大淡水湖,位于长江三角洲地区腹地,总面积2 425 km2,平均水深2.1 m,贮水量44.4亿m3。太湖流域是我国经济最发达、最具活力的地区之一。然而,随着流域内工业化、城市化进程的加快和农业生产的发展,大量未处理达标的工业、农业和生活污水排入,太湖水质不断恶化,富营养化程度加剧。2000年太湖水质平均为Ⅳ类,其中1/3湖区为Ⅴ类或劣Ⅴ类水体,约70%的水体处于富营养状态[1]。底泥是湖泊生态系统的重要组成部分,其中栖息着大量的能为水体自净提供驱动力的微生物,它们是底泥生物活性的源泉。微生物可以通过同化、异化等生化活动以及改变环境条件来影响底泥中的营养物质的分布、转化,而水质变化如污染和富营养化反过来也会引起底泥中微生物群落的改变。底泥微生物群落变化的研究有助于从侧面了解上覆水水质、推测其污染及演化历史;底泥中微生物物种、基因等资源的鉴定和获得有助于丰富当前对微生物资源的认识。因此,湖泊底泥中微生物的群落结构及作用成为新兴的研究热点之一[2]。
20世纪90年代以来,微生物生态学已经从宏观水平发展到微观和分子水平,很多新兴的分子生物学技术如分子杂交、克隆文库、荧光原位杂交、限制性片段长度多态性分析、变性梯度凝胶电泳(DGGE)等技术都被应用在基因水平上估计微生物物种的个体丰度、均度,寻找物种的变异情况以及探寻群落中微生物间的相互关系,从而更加客观地认识环境中微生物真实的生态状况。DGGE相比其他技术具有快速(少于一天)、全面(能检测丰度大于1%的微生物)等优势,能够比较真实反映环境中微生物的群落结构,非常适用于湖泊底泥微生物群落多样性分析研究[3]。笔者以典型的富营养化湖泊——太湖中不同区域底泥作为研究对象,首先,分析底泥中营养物质的分布特征;然后,采用宏基因组学策略和PCR、DGGE、克隆测序等分子生态学方法,对太湖底泥中的微生物多样性进行研究,比较分析不同采样点微生物群落结构特征和差异。笔者旨在探究太湖底泥微生物的群落结构,同时也为以微生物为基础的环境生态监测方法的建立提供理论依据[4]。
1 材料与方法
1.1 实验材料
1.1.1 样品采集
底泥样品于2012年5月29日在西太湖的梅梁湖和竺山湖区域采集,共设有5个采样点,分别如图1所示。1号点位于梅梁湖湖心;2号点位于分水口,主要污染源是上游分水、漕桥等处的农村面源污染;3号点位于浯溪口,主要污染源是浯溪、殷村港等处的工业废水排放;4号点大平场位于连接竺山湖和梅梁湖的马山水道西侧;5号点位于梅梁湖湾。用沉积物柱状采样器采集约50 cm的柱状泥样,每个采样点设置3个重复,取上层10 cm混匀后分成2份,置于冷藏箱中带回实验室-80℃保存,1份用于测定理化指标,1份用于微生物多样性分析[5]。

图1 采样点分布示意图
1.1.2 试剂
PCR扩增试剂盒、DNA胶回收试剂盒、PCR引物、溶菌酶、水饱和酚等均购自上海生工生物工程有限公司;氯仿、异戊醇、异丙醇、乙酸钠等购自上海国药集团化学试剂有限公司。
1.1.3 仪器
高速冷冻离心机(GL-20G-Ⅱ,上海安亭),小型台式高速离心机(5417R,德国Eppendorf),凝胶成像分析仪(Universal Hood II,美国 Bio-Rad),PCR 扩增仪(5332,德国 Eppendorf),恒温水浴锅(HH·S21-4-S,上海精宏),高压蒸汽灭菌锅(YXQ-LS-50SII,上海博讯),DCode 突变检测系统(Bio-Rad)。
1.2 实验方法
1.2.1 底泥理化性质测定
底泥样品的理化性质参照《土壤农化分析》,含水率采用烘干恒重法测量,总有机质含量采用灼烧法测量,总氮采用半微量凯氏定氮法测量,总磷采用HClO4-H2SO4消解钼锑抗比色法测定[6]。
1.2.2 底泥DNA提取
底泥总DNA提取参照朱艳霞等[7]的方法,略有改动。称取3 g底泥样品于10 mL离心管中,加入6 mL DNA提取液,涡旋混匀5 min。加入溶菌酶至终浓度为2 mg·mL-1,摇床上37℃,225 r·min-1振荡1 h。加入0.1倍体积20%SDS,混匀后65℃水浴2 h,期间每15 min轻轻颠倒混匀。室温8 000 r·min-1离心10 min收集上清,上清中加0.5倍体积的40%PEG-8000混匀,4℃沉淀过夜。4℃ 12 000 r·min-1离心20 min,收集沉淀。 沉淀用 600 μL TE 溶解,加入等体积的酚-氯仿-异戊醇(25∶24∶1),4 ℃ 12 000 r·min-1离心 10 min 收集上清。 加入等体积的氯仿-异戊醇(24∶1),4℃ 12 000 r·min-1离心15 min,收集上清。 加入0.1倍体积的5 mol·L-1的乙酸钠和0.6倍体积-20℃预冷的异丙醇室温沉淀4 h,4℃ 12 000 r·min-1离心15 min弃上清。沉淀用-20℃预冷的70%乙醇2次洗涤,沉淀晾干后加200 μL TE缓冲液溶解,收集于1.5 mL离心管中。
1.2.3PCR-DGGE
PCR引物为
GC-341F(5′-CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGGCCTACGGGAGGCAGCAG-3′)下划线为 GC 夹子,518R (5′-ATTACCGCGGCTGCTGG-3′)[8]。PCR 反应为 20 μL 体系,10 μL 2×premix,上下游引物各1μL,1μL模板DNA,7 μL ddH2O。PCR 反应条件为 94℃预变性3 min,94℃变性 1 min,56℃退火30 s,72℃延伸30 s,重复35个循环,最后72℃延伸5 min。
DGGE 使用 DCode突变检测系统,参考 Zhou等[9]的方法,使用 8%聚丙烯酰胺(Acr∶Bis=37.5∶1),变形梯度范围为40%~60%,电泳条件为80 V,12 h。然后银染,扫描成像。
1.2.4 数据分析
DGGE图谱分析采用Quantity One 4.62软件,经过去除背景、泳道识别、条带识别和配对得到数字化的图谱,分别计算样本的物种丰富度(S),香农多样性指数(H)和Pielou均匀度指数(J),然后计算不同泳道的Dice相似性系数,用非加权组平均法(Unweighted Pair Grouping Method with Arithmetical Averages,UPGMA)进行了聚类分析(Cluster analysis)并构建聚类图。理化因子等数据分析采用SPSS15.0软件,样本均值的显著性分析采用单因素方差分析(ANOVA),并进行Tukey验后检验。冗余分析(RDA)采用R语言3.2 vegan软件包[10]。
2 结果与分析
2.1 底泥理化性质
底泥的理化性质见表1,5个采样点底泥中总氮含量没有显著差异,而含水率、有机质、总磷含量则有显著差异。 从有机质含量来看,分水口(2号)底泥中含量最高(P<0.01),为(5.76±0.08)%,梅梁湖湾(5 号)底泥中有机质最少。就总磷含量来看,分水口(2号)和浯溪口(3号)底泥中含量最高,梅梁湖心(1号)和大平场(4号)底泥含量次之,梅梁湖湾(5号)底泥含量最低(P<0.01)。

表1 底泥理化性质分析
2.2 DGGE图谱分析
底泥样品总DNA经PCR-DGGE后得到的指纹图谱如图2所示,5个采样点间的细菌群落结构有比较大的差异,采用Quantity One软件对图谱进行数字化识别,采用非加权组平均法(UPGMA)进行聚类分析(图3),发现梅梁湖心(1号)与其他样品的差异比较大,单独聚成一支,分水口(2号)、浯溪口(3号)底泥中细菌组成比较相近,而大平场(4号)与梅梁湖湾(5号)相似度较高。
根据公式计算出各个样品的基因型丰富度(Genotypic Richness,S)、香农多样性指数(Shannon-Wiener diversity index,H)和 Pielou 均匀度指数(Pielou evenness index,J)(见表 2),结果显示5 号样品具有最高的丰富度(34)、多样性(3.44)和均匀度(0.98),而浯溪口(3号)各项指标均为最低。
2.3 理化因子与群落结构相关性分析

图2 底泥DNA细菌V3区DGGE图谱
根据DGGE图谱得到的条带组成矩阵 (共57个条带),选取5个样品中平均丰度前20的条带,以及与条带组成相关性较高的3个环境变量(总磷、总氮、总有机质),各组变量经标准化后应用软件包对条带组成和环境因子进行冗余分析(RDA)(图4)。总体来看,第一轴(RDA1)能解释底泥细菌群落结构差异的32.80%,第二轴(RDA2)能解释21.47%的差异,总体能解释样本间差异的54.27%。总氮和总有机质与第一轴的夹角较小,说明这2个变量是各个样品细菌群落结构在第一轴上的排布的主要影响因素。总磷含量的不同是第二轴差异的主要因素。环境因子中总氮的箭头最长,说明总氮对各样本细菌组成的影响最大,其次是总磷,然后是总有机质[11]。
根据采样点在RDA排序图的中分布来看,分水口(2号)和浯溪口(3号)细菌群落结构比较接近,这与聚类分析结果一致,而且在环境变量箭头上的排序靠前,说明高负荷的碳、氮、磷等富营养元素污染是此类样品的细菌群落结构形成的动因。
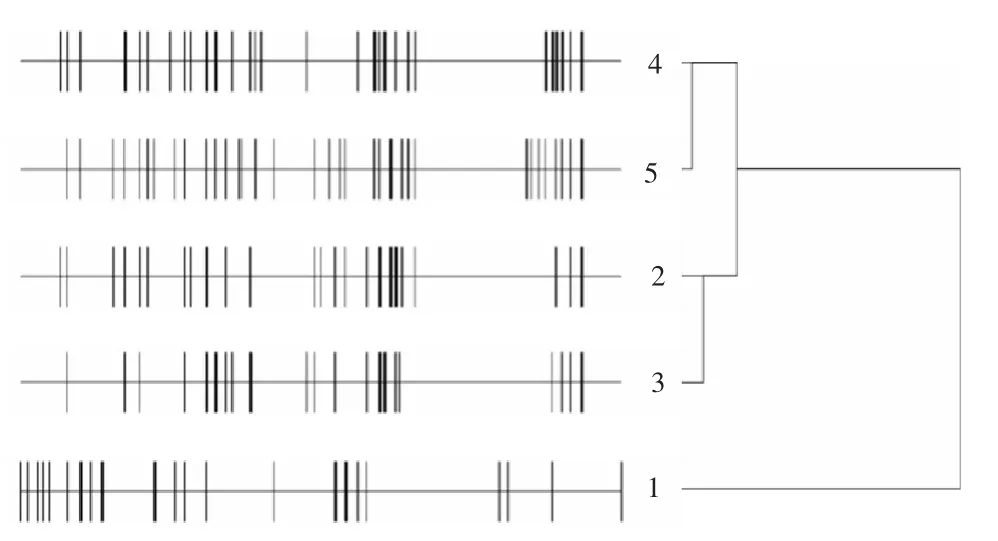
图3 DGGE条带识别和聚类分析

表2 样品的多样性指数
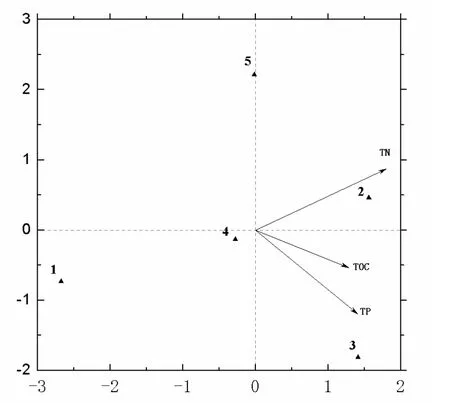
图4 细菌群落结构与环境因子的冗余分析
3 结语
采用PCR-DGGE方法对太湖底泥中细菌群落结构进行研究,发现不同位置底泥中的细菌组成有较大差别,结合底泥理化性质进行RDA分析,发现底泥中总氮、总磷和总有机质含量的变化能解释大部分群落结构的差异。分水口(2号)和浯溪口(3号)底泥的细菌组成是由高浓度的碳、氮、磷污染造成的,其中总氮对细菌群落结构的影响最大。后续研究可以扩大样本量,通过相关性分析找出高度富营养化底泥中的特征微生物,作为指示微生物应用到环境监测和预警中。
[1]秦伯强.长江中下游浅水湖泊富营养化发生机制与控制途径初探[J].湖泊科学,2002,14(2):193-202.
[2]屈建航,李宝珍,袁红莉.沉积物中微生物资源的研究方法及其进展[J].生态学报,2007,27(6):2636-2641.
[3]TIJDENS M,HOOGVELD H L,KAMST-VAN AGTERVELD M P,et al.Population dynamics and diversity of viruses,bacteria and phytoplankton in a shallow eutrophic lake[J].Microbial ecology,2008,56(1):29-42.
[4]王晓丹,李艳红.分子生物学方法在水体微生物生态研究中的应用[J].微生物学通报,2007,34(4):777-781.
[5]LING N,DENG K,SONG Y,et al.Variation of rhizosphere bacterial community in watermelon continuous mono-cropping soil by long-term application of a novel bioorganic fertilizer[J].Microbiological Research,2014,169(7/8):570-578.
[6]史瑞和,鲍士旦,秦怀英.土壤农化分析[M].北京:中国农业出版社,2000:29-82.
[7]朱艳霞,邱业先,钱玮.一种适用于太湖底泥基因组 DNA的提取方法[J].江苏农业科学,2012,40(7):31-33.
[8]MUYZER G,DE WAAL E C,UITTERLINDEN A G.Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J].Applied and Environmental Microbiology,1993,59(3):695-700.
[9]ZHOU X,GAO D,LIU J,et al.Changes in rhizosphere soil microbial communities in a continuously monocropped cucumber (Cucumis sativus L)system[J].Eur J Soil Biol,2014,60(1):1-8.
[10]OKSANEN J,KINDT R,LEGENDRE P,et al.The vegan package[J].Community Ecology Package,2007,10(1):2008.
[11]白晓旭,史荣久,尤业明,等.河南宝天曼不同林龄与林型森林土壤的细菌群落结构与多样性[J].应用生态学报,2015,26(8):2273-2281.
Influence of environmental factors on bacterial community structures in sediments of Lake Tai
QIAN Wei,ZHU Yanxia, QIU Yexian*
(School of Chemistry,Biology and Material Engineering,SUST,Suzhou 215009,China)
Denatured gradient gel electrophoresis (DGGE) was used to study the diversity of bacteria in sediments of Lake Tai.Total nitrogen (TN),total phosphorus (TP) and total organic Carbon (TOC) were quantified.Then,Redundancy Analysis (RDA) was carried out to explore the influence of environmental factors on bacterial community structures in sediments of Lake Tai.Results show that the gradient of TN,TP and TOC can explain 54.27%variations of bacterial community structures.TN was the most important impact factor,followed by TP and TOC.This study has provided valuable data for microbial resources investigation and environmental monitoring of Lake Tai.
sediments of Lake Tai;denatured gradient gel electrophoresis(DGGE);Redundancy Analysis(RDA)
Q33
A
2096-3289(2017)03-0050-05
责任编辑:李文杰
2015-12-22
苏州科技学院科研基金资助项目(XKQ201313)
钱 玮(1983-),男,江苏泰州人,实验师,硕士,研究方向:环境微生物学。
*通信作者:邱业先(1954-),男,博士,教授,博士生导师,E-mail:qyx542@163.com。
