D N A甲基化对抑郁症患者抗抑郁药物反应影响的研究进展☆
2017-09-11王培培易正辉吕钦谕张翠珍蔡卫民
王培培易正辉吕钦谕张翠珍蔡卫民
·综 述·
D N A甲基化对抑郁症患者抗抑郁药物反应影响的研究进展☆
王培培*易正辉△吕钦谕△张翠珍*蔡卫民*
表观遗传学 DNA甲基化 抑郁症 抗抑郁药
抗抑郁药物治疗抑郁症 (major depressive disorder,MDD)即使在足量足疗程的方案下,临床上仍有50%~60%的患者无法获得完全缓解[1]。药物反应受许多因素影响,至今临床仍无确切抗抑郁药物反应预测指标,治疗药物的选择及治疗方案无确切依据可寻。以往许多学者认为基因多态性是导致抗抑郁药物治疗个体差异的重要原因,多项研究发现,基因突变与药物反应密切相关[2-3]。但研究结果尚不明确统一,且不能完全解释抗抑郁药物反应个体间的差异。近年来,表观遗传学尤其是DNA甲基化作为可能影响药物反应的因素越来越受到各界关注,多项研究发现DNA甲基化在个体间的差异与药物疗效和不良反应在个体间的差异有一定相关性。本文将对相关基因DNA甲基化与抗抑郁药物反应关联性研究进行回顾探讨,进而提出DNA甲基化可能成为早期预测药物反应的一个潜在生物标志物。
1 DNA甲基化
表观遗传学(epigenetics)是研究不涉及DNA序列改变的可遗传的基因结构修饰对基因表达的改变,其中DNA甲基化是研究最多的表观遗传调节方式。DNA甲基化主要发生富含CpG双核苷酸的DNA片段即CpG岛,通过DNA甲基化转移酶(DNA methyltransferases,DNMT)修饰调控。DNMT家族主要有DNMT1、DNMT2、DNMT3a和DNMT3b等4种亚型,DNMT1主要负责维持DNA复制过程甲基化水平,DNMT3a和DNMT3b结合形成复合体负责基因的起始甲基化,而DNMT2作用微弱[4]。基因启动子区甲基化可影响转录激活因子与其识别序列的结合而直接抑制基因表达,或通过结合甲基化结合蛋白 (methy CpG binding domain protein,MBD)募集抑制因子抑制基因转录,导致基因表达沉默[5]。见图1。
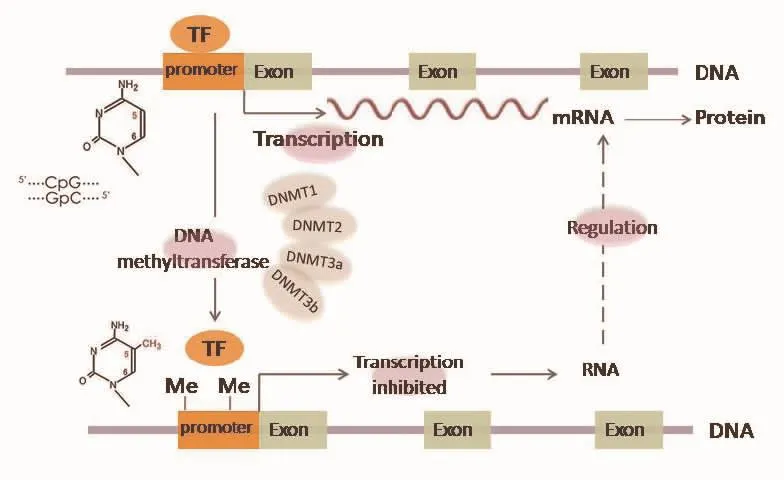
图1 DNA甲基化调控示意图
DNA甲基化在基因表达过程中相对稳定遗传,但长期压力、生活事件、早年创伤、药物干预等具有逆转环境因素所诱导表观遗传改变的潜能。实验发现,长期慢性刺激引发抑郁样症状同时会引起促肾上腺皮质激素释放激素(corticotropin releasing hormone,CRH)表达上调,激活肾上腺轴活动,而基因启动子区DNA甲基化降低;抑郁模型小鼠经丙咪嗪药物治疗后CRH mRNA表达降低,DNA甲基化水平升高[6]。DNA甲基化在组织间差异较大,大脑内羟甲基胞嘧啶水平约为胚胎干细胞的10倍,尤其是位于神经突触内的基因对精神疾病表观遗传研究至关重要。但文献报道,外周组织DNA甲基化状态接近脑组织表达,可一定程度反映脑组织DNA甲基化水平[7-8]。LOWE等[9]研究发现,颊粘膜细胞可能比外周血更能代替大脑甲基化水平,可能是因其细胞异质性低,与脑组织从同一胚层发育分化。近年来,脑脊液在肿瘤和阿尔茨海默病表观遗传研究应用越来越多,研究者认为脑脊液更接近大脑组织,可以更真实反映脑组织表达情况[10]。未来研究精神疾病应该重点考虑表观调控修饰在脑组织和其他组织细胞异质性。
2 DNA甲基化与抗抑郁药物反应
抗抑郁药物反应影响因素众多,除了个体遗传学差异,DNA甲基化研究逐渐成为寻找疗效预测标记物研究的新热点。但是,因当前研究样本量有限,且种族、研究及基因位点的不同,甲基化状态影响药物反应的研究结果存在较大争议。目前大部分临床研究集中在基线期 (用药前)DNA甲基化研究,而非药物干预对功能性基因位点的甲基化水平逆转。文献检索发现,DNA甲基化对抗抑郁药物干预影响的研究也集中于基因 SLC6A4、HTR1B、MAOA、BDNF、IL-11[11-18](见表1)。
2.1 5-羟色胺转运体(5-HTT)5-羟色胺转运体(serotonin transporter,5-HTT,编码基因SLC6A4)在抑郁症发病分子生物学机制和抗抑郁药物干预过程中均有重要作用。表观遗传研究发现[19-20],位于SLC6A4转录控制区的CpG岛DNA甲基化水平与LPR基因型相互作用可以影响5-HTT表达水平,并且SLC6A4启动子区DNA甲基化化与中风后抑郁和抑郁症状严重程度密切相关。PHILIBERT等[21]研究发现SLC6A4启动子区CpG岛基因位点甲基化参与基因活性调控,SLC6A4 DNA甲基化是5-HTT功能的重要调节方式。
2013年,KANG等[11]开展了一项关于抑郁症患者SLC6A4基因DNA甲基化状态与早期创伤、临床特征和治疗效果关系的研究,结果发现,抑郁症患者SLC6A4启动子区高甲基化与其童年期创伤、抑郁家族史、主观压力高、严重抑郁症状等相关, 与药物治疗反应无关。2014年,DOMSCHKE等[12]报道SLC6A4外显子1A转录区DNA甲基化对抗抑郁药物反应的影响。该研究纳入94例高加索抑郁症患者,予以艾司西酞普兰抗抑郁药物治疗并随访6周,结果显示,SLC6A4平均甲基化水平低者预示药物疗效较差,尤其在CpG 2位点具有显著相关性[12]。研究者们推测SLC6A4转录区DNA低甲基化可能通过增加5-HTT表达而减少脑内5-羟色胺含量,从而导致较差的药物疗效;此外,研究发现5-HTTLPR/rs25531单倍型对SLC6A4甲基化状态及治疗效果均无影响。为了探索预测抑郁症诊断与抗抑郁药物反应标志物,OKADA等[13]在50例日本抑郁症患者和50例健康对照中进行一项药物干预研究。结果发现,入组时患者组与对照组SLC6A4启动子区DNA甲基化水平无明显差异,而用药后患者组几个位点DNA甲基化水平显著变化,且入组时甲基化水平高的抑郁症患者表现出更好的治疗效果[17]。另外与DOMSCHKE等[12]研究结果相同,OKADA等[13]也未发现SLC6A4片段长度多态性5-HTTLPR对其基因甲基化水平有影响。
虽然上述研究都对SLC6A4 DNA甲基化水平进行了探索,但纳入的位点数量、位置不尽相同,结果有一定差异,DOMSCHKE等[12]证明低甲基化预示较差的治疗效果,而KANG等[11]发现高甲基化与HAMD评分提高有一定相关趋势。
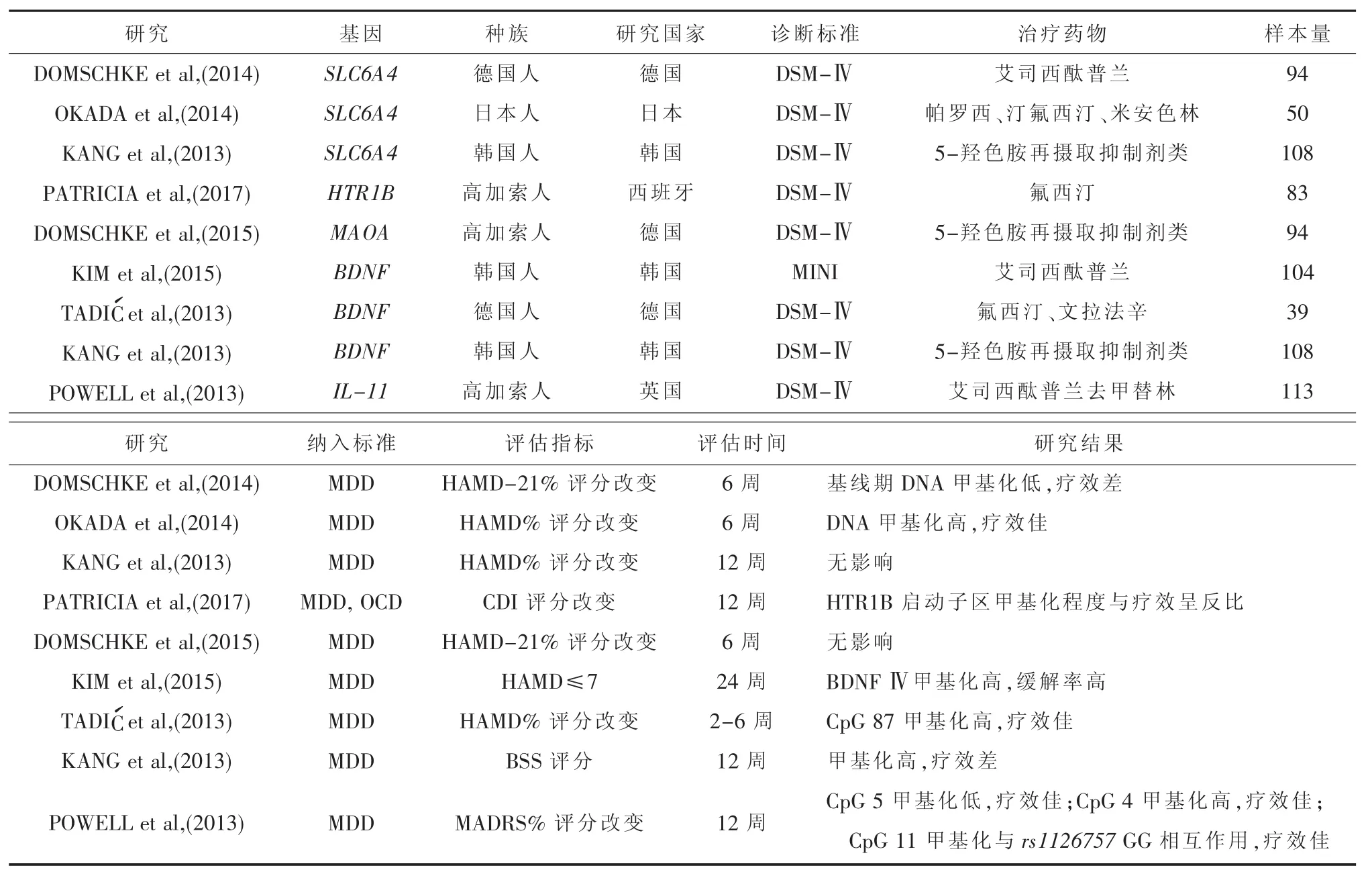
表1 D N A甲基化相关研究文献基本信息一览表
2.2 5-羟色胺受体1B(5-HTR1B)5-羟色胺受体能控制进入突触间隙5-羟色胺(5-hydroxytryptamine,5-HT)的合成和释放,动物研究证实,抗抑郁药物可减少动物脑内5-HT受体密度,且受体结合能力与抑郁状态变化有关[22],因此推测影响5-HT受体表达的基因多态性和DNA甲基化可能同抗抑郁药物疗效相关。2016年GASSò 等[23]研究纳入83例青少年抑郁症患者,给予氟西汀抗抑郁药物治疗并随访12周,儿童抑郁量表 (children's depression inventory,CDI)评估疗效,检测外周血HTR1B DNA甲基化水平及基因型,综合分析结果发现,rs9361233和rs9361235基因多态性与用药后疗效密切相关,杂合子携带者用药后CDI评分较高,抗抑郁疗效较差;而HTR1B启动子DNA甲基化水平与患者用药后临床改善呈负相关。基因甲基化导致基因表达沉默,而HTR1B启动子低甲基化可能通过增强基因转录活性,使5-HT水平降低,从而提高抗抑郁药物治疗反应。
2.3 单胺氧化酶A(MAOA)单胺氧化酶(monoamine oxidase A,MAOA)主要参与5-羟色胺、肾上腺素、去甲肾上腺素等单胺递质的氧化降解过程,抑郁症自杀患者下丘脑区MAOA活性增加,单胺氧化酶抑制剂就是通过抑制MAOA活性、减少中枢单胺递质分解以提高突触间隙单胺递质浓度来达到抗抑郁作用的目的。文献报道,MAOA基因突变可能与抑郁症发病风险相关[24],但是对药物干预是否有影响仍不清楚。表观修饰可以通过影响基因转录水平从而影响基因的表达,SHUMAY等[25]研究证实MAOA基因启动子区DNA甲基化水平影响脑内MAOA酶活性及表达水平。2014年DOMSCHKE等[14]首次报道MAOA外显子调控区DNA甲基化对抗抑郁药物反应的影响。结果显示,在女性亚组中,CpG低甲基化与疗效欠佳呈现一定相关性,但经过多重检验校正后相关性无统计学意义[14]。
2.4 脑源性生长因子(BDNF)脑源性生长因子BDNF可以促进神经元细胞的生长、增殖、分化等,并参与神经突触可塑性和认知功能调控[26]。研究表明,外周血BDNF表达降低与抑郁症发病密切相关,而有效的抗抑郁药物治疗可以提高外周血BDNF表达水平[27]。表观遗传修饰参与BDNF表达调控。研究发现,BDNF甲基化水平增高可以影响其外周血表达水平,且高甲基化可能与抑郁症高发病风险及药物反应有关[28]。
2013年,TADIò等[15]研究发现,BDNF启动子区CpG-87位点未甲基化预示治疗效果欠佳,且药物治疗后血浆BDNF水平降低,而高甲基化携带患者疗效较好并伴随血浆BDNF水平升高。抑郁症是引起自杀的重要精神因素,KANG等[26]研究表观调控与抑郁症患者自杀行为及药物治疗后自杀观念改变的关系,结果发现,BDNF高甲基化与自杀史及药物治疗后自杀观念缓解差显著相关。许多躯体疾病会合并抑郁症,抑郁症状会严重影响躯体疾病进展。KIM等[17]通过研究BDNF DNA甲基化对急性冠脉综合征合并抑郁症患者在使用抗抑郁药治疗24周后疗效影响发现,BDNF高甲基化组表现出更好的治疗效果。综上研究,BDNF甲基化可能成为预测药物反应的生物标记物,尚待更多研究进行深入探索。
2.5 白介素-11(interleukin-11,IL-11)
免疫功能激活是抑郁症发病机制的重要补充,细胞因子参与神经递质系统、神经内分泌及神经可塑性等多个系统。IL-11是一个多功能的细胞因子,近来多项研究发现IL-11表达与抑郁症及药物干预密切相关。POWELL等[29]研究显示抑郁症患者抗抑郁药物治疗后有效组较无效组IL-11表达水平降低,IL-11基因多态性rs1126757与艾司西酞普兰疗效有关。在此研究基础上,2013年POWELL等[18]进一步研究了表观遗传修饰对抗抑郁药物反应的预测作用。该研究纳入抑郁症药物基因组学研究项目(GENDEP)中的113例抑郁症患者,予以艾司西酞普兰或去甲替林药物治疗,分析外周血IL-11 DNA甲基化水平与治疗效果的关系,结果发现IL-11低甲基化状态与较好的治疗效果显著相关[18]。研究者们提出该研究中可能存在一些不足,尤其是甲基化存在组织特异性,外周血甲基化水平不能完全代替脑部甲基化水平,但IL-11的表观修饰在药物干预中的影响仍值得进一步研究关注。
3 结论
目前,表观遗传相关的DNA甲基化对药物干预影响的研究较少,还处于起步阶段。但已有的研究结果显示,DNA甲基化可能成为预测抗抑郁药物反应的重要生物标志物,值得引起重视。多项研究由于样本量、研究对象种族、疗效评价标准、目的基因位点差异等因素使得研究结果无法获得统一,因此需要大量长期随机对照研究来补充验证。未来,表观遗传在个体化治疗中的研究和应用将为精神疾病治疗提供更多的理论依据。
[1]FAVA M.Diagnosis and definition of treatment-resistant depression[J].Biol Psychiatry,2003,53(8):649-659.
[2]PORCELLI S,FABBRE C,SERRETTI A.Meta-analysis of serotonin transporter gene promoter polymorphism(5-HTTLPR) association with antidepressant efficacy [J].Eur Neuropsychopharmacol,2012,22(4):239-258.
[3]KARLSSON L,GREEN H,ZACKRISSON AL,et al.ABCB1 gene polymorphisms are associated with fatal intoxications involving venlafaxine but not citalopram[J].Int J Legal Med,2013, 127(3):579-586.
[4]WEBER M,SCHUBELER D.Genomic patterns of DNA methylation:targets and function of an epigenetic mark[J].Curr OpinCell Biol,2007,19(3):273-280.
[5]VIALOU V,FENG J,ALFRED J,et al.Epigenetic Mechanisms of Depression and Antidepressants[J].Annu Rev Pharmacol Toxicol,2013,53:59-87.
[6]ELLIOTT E,EZRA-NEVO G,REGEV L,et al.Resilience to social stress coincides with functional DNA methylation of the Crf gene in adult mice[J].Nat Neurosci,2010,13(11):1351-1353.
[7]BYUN H,D.Siegmund K,Pan F,et al.Epigenetic profiling of somatic tissues from human autopsy specimens identifies tissueand individual specific DNA methylation patterns[J].Hum Mol Genet,2009,18(24):4808-4817.
[9]LOWE R,GEMMA C,BEYAN H,et al.Buccals are likely to be a more informative surrogate tissue than blood for epigenomewide association studies[J].Epigenetics,2013,8(4):445-454.
[10]BEKRIS L,LUTZ F,MONTINE T,et al.MicroRNA in Alzheimer’s disease:an exploratory study in brain,cerebrospinal fluid and plasma[J].Biomarkers,2013,18(5):455-466.
[11]KANG H,KIM J,STEWART R,et al.Association of SLC6A4 methylation with early adversity,characteristics and outcomes in depression[J].Prog Neuro-Psychopharmacology Biol Psychiatry, 2013,44:23-28.
[12]DOMSCHKE K,TIDOW N,SCHWARTE K,et al.Serotonin transporter gene hypomethylation predicts impaired antidepressant treatment response[J].Int J Neuropsychopharmacol,2014,17 (8):1167-1176.
[13]OKADA S,MORINOBU S,FUCHIKAMI M,et al.The potential of SLC6A4 gene methylation analysis for the diagnosis and treatment of major depression[J].J Psychiatr Res,2014,53:47-53.
[14]DOMSCHKE K,TIDOW N,SCHWARTE K,et al.Pharmacoepigenetics of depression:no major influence of MAO-A DNA methylation on treatment response[J].J Neural Transm,2015,122 (1):99-108.
[16]KANG H,KIM J,LEE J,et al.BDNF promoter methylation and suicidal behavior in depressive patients[J].J Affect Disord,2013, 151(2):679-685.
[17]KIM J,STEWART R,KANG H,et al.BDNF methylation and depressive disorder in acute coronary syndrome:The K-DEPACS and EsDEPACS studies[J].Psychoneuroendocrinology,2015,62: 159-165.
[18]POWELL TR,SMITH RG,HACHINGER S,et al.DNA methylation in interleukin-11 predicts clinical response to antidepressants in GENDEP[J].Transl Psychiatry,2013,3(9):e300.
[19]PHILIBERT R,MADAN A,ANDERSEN A,et al.Serotonin Transporter mRNA Levels are Associated With the Methylation of an Upstream CpG Island[J].Am J Med Genet B Neuropsychiatr Genet,2007,144B(1):101-105.
[20]JINYING Z,GOLDBERG J,BREMNER J,et al.Association Between Promoter Methylation of Serotonin Transporter Gene and Depressive Symptoms:A Monozygotic Twin Study[J].Psychosom Med,2013,75(6):doi:10.1097.
[21]PHILIBERT RA,SANDHU H,HOLLENBECK N,et al.The relationship of 5HTT(SLC6A4)methylation and genotype on mRNA expression and liability to major depression and alcohol dependence in subjects from the Iowa Adoption studies[J].Am J Med Genet B Neuropsychiatr Genet,2008,147(5):543-549.
[22]LI YAN,RAABY KF,SANCHEZ C,et al.Serotonergic receptor mechanisms underlying antidepressant-like action in the progesterone withdrawal model of hormonally induced depression in rats[J].Behav Brain Res,2013,256:520-528.
[24]ZICHAO L,LIANG H,XIONGJIAN L,et al.MAOAVariants and Genetic Susceptibility to Major Psychiatric Disorders[J]. Mol Neurobiol,2016,53(7):4319-4327.
[25]SHUMAY E,LOGAN J,VOLKOW N,et al.Evidence that the methylation state of the monoamine oxidase A (MAOA)gene predicts brain activity of MAOA enzyme in healthy men[J].Epigenetics,2012,7(10):1151-1160.
[26]AUTRY AE,MONTEGGIA LM.Brain-derived neurotrophic factor and neuropsychiatric disorders[J].Pharmacol Rev,2012, 64:238-258.
[27]BOCCHIO-CHIAVETTO L,BAGNARDI V,ZANARDINI R,et al.Serum and plasma BDNF levels in major depression:a replication study and meta-analyses[J].World J Biol Psychiatry, 2010,11(6):763-773.
[28]MARTINOWICH K,HATTORI D,WU H,et al.DNA methylation related chromatin remodeling in activity-dependent BDNF gene regulation[J].Science,2003,302(5644):890-893.
[29]POWELL TR,SCHALKWYK LC,HEFFERNAN AL,et al.Tumor necrosis factor and its targets in the inflammatory cytokine pathway are identified as putative transcriptomic biomarkers for escitalopram response[J].Eur Neuropsychopharmacol,2013,23(9): 1105-1114.
R749 (
2017-04-14)
A (责任编辑:肖雅妮)
10.3969/j.issn.1002-0152.2017.07.015
☆国家自然科学基金项目(编号:81671326)
* 复旦大学药学院临床药学(上海 201203)
△上海交通大学医学院附属精神卫生中心
