甘蓝型油菜株高QTL定位及主效QTL区间候选基因预测
2017-09-01姜成红耿鑫鑫魏文辉姜慧芳
姜成红,耿鑫鑫,魏文辉,姜慧芳
(中国农业科学院 油料作物研究所/农业部油料作物生物学与遗传育种重点实验室,湖北 武汉 430062)
甘蓝型油菜株高QTL定位及主效QTL区间候选基因预测
姜成红,耿鑫鑫,魏文辉*,姜慧芳*
(中国农业科学院 油料作物研究所/农业部油料作物生物学与遗传育种重点实验室,湖北 武汉 430062)
为了研究油菜株高的遗传基础,以2个甘蓝型油菜株系DH-7-9(矮杆)× DH-G-42(高杆)杂交后代连续自交的重组自交系群体(190个家系)为材料,在西宁和武汉2种环境下进行株高性状鉴定,结果显示,该重组自交系群体的株高表现连续变异并且符合正态分布。利用前期构建的遗传连锁图,结合2种环境下株高性状鉴定数据,采用WinQTLcart 2.5 软件复合区间作图法(CIM)进行QTL定位和效应估计,结果表明,在2种环境下共检测到11个与株高性状相关的QTL,单个QTL可解释的表型变异为1.17%~10.45%。在A10连锁群上,主效QTL(qPH-X-A10或qPH-W-A10)在两环境下可重复检测到,可解释10.24%~10.45%的表型变异。将156个拟南芥株高基因与该主效QTL置信区间对应的油菜基因组上的723个基因进行同源比较分析,在主效QTL区域内预测到2个株高候选基因BnaA10g07740D和BnaA10g12020D,其对应的拟南芥同源基因分别为ATGA20ox2、GA5/ATGA20ox1和STA1,均与拟南芥株高相关。
甘蓝型油菜; 株高; QTL定位; 候选基因
油菜(Brassicanapus)是世界范围内广泛种植的主要油料作物,也是我国唯一的冬季油料作物,我国油菜栽培面积和总产量均居世界第1位[1]。株高是油菜产量相关性状之一,影响其株型、种植密度和抗倒伏系数,直接或间接影响油菜的产量和品质[2]。适宜的株高对油菜抗倒性、产量提高及机械收割均有重要意义[3]。油菜株高表现出连续变异的特点,为多基因控制的数量性状,且受环境影响较大[4]。
近年来,利用QTL作图和图位克隆方法,一些影响作物株高的基因已被克隆。Ghd7是一个控制水稻穗粒数、抽穗期和株高的多效性基因,对光周期敏感,编码产物为具有CCT结构域的锌指转录因子。长日照条件下,Ghd7基因表达增强,引起穗粒数增加、抽穗期延迟及株高增加[5]。Ghd8是一个与Ghd7类似的同时控制水稻穗粒数、抽穗期和株高的基因,编码CCAAT盒结合蛋白转录因子的HAP3亚基。在长日照条件下,Ghd8能显著延迟抽穗期,同时增加株高和每穗粒数[6]。油菜株高的QTL定位研究相对滞后,但也取得了一些进展[4,7-9]。易斌等[4]检测到3个与油菜株高相关的QTL,分别定位于第4、第9和第10连锁群上,解释了9.42%~17.58%的表型变异。张倩[7]在LG1和LG2连锁群上检测到4个与油菜株高相关的QTL,可解释8.54%~17.04%的表型变异。Zhang等[8]在第4和第21连锁群上检测到3个与油菜株高性状相关的QTL,分别解释28.60%、23.85%和11.11%的表型变异。张凤启等[9]利用SNP芯片在单一环境和多环境下共检测到27个油菜株高QTL,位于8个连锁群上,单个QTL可解释的表型变异为0.70%~26.10%。尽管已知控制油菜株高的相关QTL分布在一些具体的连锁群上[8-18],但QTL克隆及QTL区段内候选基因预测的相关报道较少。鉴于此,以甘蓝型油菜重组自交系群体为材料,对油菜株高进行QTL定位,根据主效QTL置信区间对应的甘蓝型油菜基因组序列,分析该区间的全部基因,并与Shi等[15]报道的拟南芥156个株高基因进行同源性比对,筛选候选基因,以期为后续分子标记辅助育种及相关基因克隆奠定基础。
1 材料和方法
1.1 材料
以甘蓝型油菜株系矮杆小粒材料DH-7-9为母本及高杆大粒材料DH-G-42为父本,构建重组自交系群体,该群体包含190个株系。DH-7-9和DH-G-42均通过小孢子培养技术获得[19]。
1.2 田间试验与株高性状考察
将重组自交系群体于2013年9月种植于中国农业科学院油料作物研究所武汉市武昌区试验田(F8),2014年5月种植于青海省西宁市青海大学油菜繁育中心试验田(F9)。所有田间试验均按株系分区种植,3个重复,每小区3行,行距40 cm、株距20 cm。按常规生产方式进行田间管理。收获时从每小区内随机选取10株,用于株高性状的鉴定。
1.3 株高的QTL定位
利用本实验室前期通过F2代群体构建的遗传连锁图谱进行QTL定位[20]。该连锁图覆盖20个连锁群,包含228个标记,图谱总长1 546.6 cM。采用WinQTLcart 2.5软件复合区间作图(CIM)法[21-22]定位该重组自交系群体的株高QTL。选用1 cM的步长,在α=0.05的水平上,利用Permutation检验法重复检验1 000次,LOD阈值设定为2.5,确定QTL在染色体上的位置及数目。QTL命名参照McCouch等[23]的规则,用SPSS 13.0软件统计分析表型数据。
1.4 候选基因的筛选
下载NCBI上156个与拟南芥株高相关的基因序列[15],然后与检测到的主效QTL置信区间对应的甘蓝型油菜基因组序列[24]进行BlastN比对,筛选候选基因。
2 结果与分析
2.1 甘蓝型油菜双亲及重组自交系群体株高的表型变异
试验所选用的甘蓝型油菜矮杆株系DH-7-9和高杆株系DH-G-42在株高上存在极显著差异,在武汉和西宁2种环境中,DH-G-42的株高均极显著高于DH-7-9 (图1)。统计分析表明,株高在重组自交系群体中呈现超双亲分离和连续性分布(表1,图2),说明株高是由多个基因控制的数量性状,适合于QTL分析。
2.2 甘蓝型油菜重组自交系群体的株高QTL定位
利用WinQTLcart 2.5软件对2种环境下重组自交系群体的株高进行QTL分析。在西宁环境下,共检测到5个与株高相关的QTL,分别位于A02、A07、A09、A10和C03连锁群上,单个QTL贡献率为1.17%~10.24%,其中1个QTL表现为正向加性效应,其他均表现为负向加性效应(表2)。在武汉环境下,共检测到6个与株高性状相关的QTL,分别位于A07、A08、A10、C01、C04和C07连锁群上,单个QTL贡献率为1.18%~10.45%,其中2个QTL表现为正向加性效应, 其他均表现为负向加性效应(表2)。位于A10连锁群上的qPH-X-A10和qPH-W-A10遗传贡献率较大,分别为10.24%和10.45%,应为主效QTL。qPH-X-A10定位于分子标记MR156与Na10-E08之间,qPH-W-A10定位于分子标记Na10-E08与Ra2-E03之间,距Na10-E08较近,遗传距离为0.046 cM(表2,图3)。通过置信区间对比发现,qPH-X-A10的置信区间位于qPH-W-A10置信区间之内,加性效应方向一致且均为正值(表2,图3,图4),说明此QTL位点具有正向加性效应和增效作用,且增效等位基因来自母本DH-7-9。因此推测,qPH-X-A10与qPH-W-A10为2个环境下重复检测到的相同QTL。A07连锁群上的qPH-X-A07和qPH-W-A07置信区间部分重合,加性效应方向一致但均为负值(表2),说明增效基因来自父本DH-G-42。因此,qPH-X-A07和qPH-W-A07是不同环境下检测到的相同QTL,但是这2个QTL贡献率都较小,分别为1.53%和3.91%。

不同大写字母表示同一环境下亲本间差异达极显著水平 (P<0.01)图1 在武汉和西宁环境下2个亲本的株高
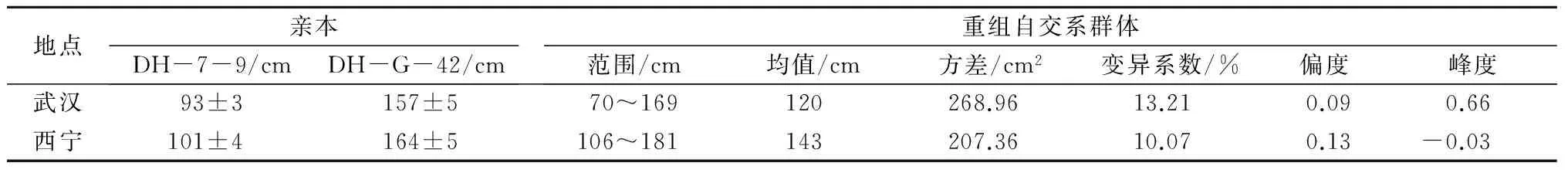
表1 在武汉与西宁环境下亲本及重组自交系群体的株高及统计分析

图2 重组自交系群体株高在武汉和西宁环境下的频次分布

表2 西宁和武汉环境下检测出的株高QTL

黑色为qPH-X-A10置信区间位置,白色为qPH-W-A10置信区间位置 图3 A10连锁群上主效QTL位置
2.3 甘蓝型油菜株高主效QTL区间候选基因筛选
将上述主效QTL置信区间(物理距离约4.8 Mb)与甘蓝型油菜基因组序列[24]比对,得到723个注释基因。将这些基因与拟南芥株高基因进行同源比对,发现有2个株高相关基因位于主效QTL置信区间内,分别为BnaA10g07740D和BnaA10g12020D。其中, 候选基因BnaA10g07740D在拟南芥中有2个同源基因,为ATGA20ox2和GA5/ATGA20ox1(表3)。根据拟南芥的基因功能注释,ATGA20ox2和GA5/ATGA20ox1均参与赤霉素的生物合成[25-26],赤霉素可以促进细胞的伸长从而引起植物株高增加[27]。候选基因BnaA10g12020D与拟南芥基因STA1同源,拟南芥STA1基因突变体Starik中非血红素蛋白含量明显高于野生型植株,STA1的突变导致植株的矮化[28]。

实线代表qPH-X-A10,虚线代表qPH-W-A10 图4 A10连锁群上主效QTL峰
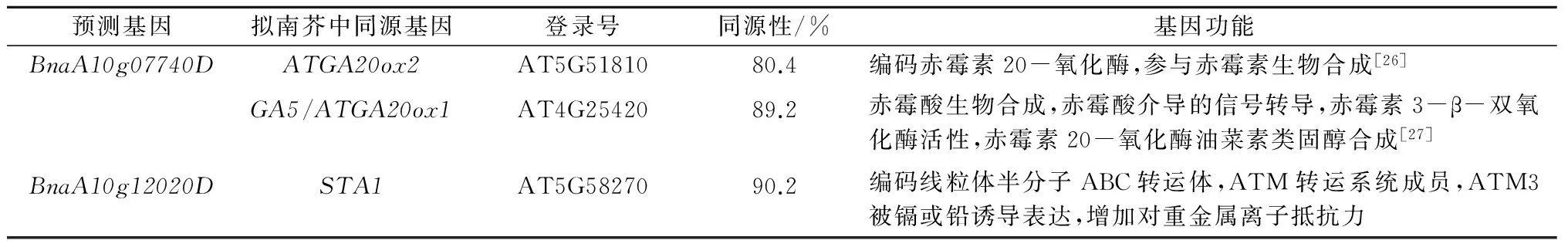
表3 甘蓝型油菜株高主效QTL置信区间候选基因与拟南芥相关基因的比对
3 结论与讨论
株高作为油菜重要的农艺性状之一,对油菜产量的潜在影响较大,合理的株高对油菜育种非常重要,因此,定位与株高相关的QTL对发掘株高基因和培育高产油菜品种具有重要潜在意义。本研究所选用的2个亲本材料是经过小孢子培养途径获得的纯系,且株高存在显著差异,其重组自交系群体株高呈现连续分布,因此,这2个亲本材料适合用于株高遗传研究。本研究发现,位于A10和A07连锁群上的株高QTL在不同环境下可重复检测到,为稳定表达的QTL,而其余QTL只能在1种环境下检测到,说明株高相关基因受环境影响较大,不同环境诱导了不同的株高基因表达。
前人研究表明,油菜株高QTL主要分布在A02、A03、A07、A08、C02、C03、C06和C07连锁群[8-18],其中主效QTLph13b[16]、ph12.3和ph2.3[17]分别位于A02、C02和C03连锁群。A10连锁群上也曾有相关QTL的报道,Basunanda等[14]和Udall等[17]分别利用DH系群体在A10连锁群上定位了株高QTL,但其贡献率均较小。张凤启等[9]在A10连锁群上定位的株高QTLqPH10-1(贡献率9.23%)与本研究的QTL贡献率接近,但是通过物理距离对比发现,其QTL峰值对应的标记与本研究中的QTL峰值对应的标记相距较远。因此,本研究在A10连锁群上检测到的QTL可能为一个新的QTL。
一般认为,贡献率在10%以上的QTL为主效QTL,LOD值越大,QTL的准确率越高。本研究在2种环境下共定位到11个QTL,除2个QTL贡献率大于10%以外,其他QTL贡献率均较小,其中有4个QTL贡献率小于2%,说明控制油菜株高性状的遗传基础比较复杂。单个环境下检测到的QTL分布在不同连锁群上,未见同一连锁群出现多个QTL的现象。本研究将主效QTL定位在A10连锁群上,但A10连锁群上标记较少,标记之间遗传距离较大,增加了后续目的基因克隆的难度。因此,开发亲本间新的多态性分子标记,增加遗传图谱标记密度,是进一步精细定位目标QTL及发现新的主效QTL的有效途径。
通过在QTL置信区间进行候选基因预测,可以在一定程度上验证QTL定位的准确性。本研究在A10连锁群上的主效QTL置信区间预测到2个候选基因,有3个拟南芥基因与其同源,同源性分别为80.4%、89.2%、90.2%,同源性较高。根据拟南芥基因功能注释,这3个同源基因均与株高发育相关,推测A10连锁群上的主效QTL极可能是控制株高的遗传位点。后续将利用株高主效QTL附近的分子标记逐步构建目的基因的近等基因系,通过精细定位和图位克隆方法最终获得目的基因。
[1] James C.Global review of commercialized transgenic crops[J].Curr Sci,2003,84:303-309.
[2] Hedden P.The genes of the green revolution[J].Trends Genetics,2003,19(1):5-9.
[3] 张芳.我国油菜品种审定管理与育种趋势研究[D].北京:中国农业科学院,2012.
[4] 易斌,陈伟,马朝芝,等.甘蓝型油菜产量及相关性状的QTL分析[J].作物学报,2006,32(5):676-682
[5] Xue W,Xing Y,Weng X,etal.Natural variation inGhd7 is an important regulator of heading date and yield potential in rice[J].Nature Genetics,2008,40(6):761-767.
[6] Yan W H,Wang P,Chen H X,etal.A major QTL,Ghd8,plays pleiotropic roles in regulating grain productivity,plant height,and heading date in rice[J].Molecular Plant,2011,4(2):319-330.
[7] 张倩.甘蓝型油菜主要株型性状的遗传分析和QTL初步定位[D].重庆:西南大学,2013.
[8] Zhang S F,Fu T D,Zhu J C,etal.QTL mapping and epistasis analysis for plant height and height to the first branch in rapeseed(BrassicanapusL.)[J].Biotechnology(Gene Cloning Functional Analysis),2007,2:232-235.
[9] 张凤启,刘越英,程晓辉,等.利用高密度SNP标记定位甘蓝型油菜株高QTL[J].中国油料作物学报,2014,36(6):695-700.
[10] Zhao Z K,Wu L K,Nian F Z.Dissecting quantitative trait loci for boron efficiency across multiple environments inBrassicanapus[J].PLoS One,2012,7(9):e45215.
[11] Shi T X,Li R Y,Zhao Z K,etal.QTL for yield traits and their association with functional genes in response to phosphorus deficiency inBrassicanapus[J].PLoS One,2013,8(1):e54559.
[12] Ding G D,Zhao Z K,Liao Y,etal.Quantitative trait loci for seed yield and yield-related traits,and their responses to reduced phosphorus supply inBrassicanapus[J].Annals of Botany,2012,109(4):747-759.
[13] Shi J,Li R,Zou J,etal.A dynamic and complex network regulates the heterosis of yield-correlated traits in rapeseed(BrassicanapusL.)[J].PLoS One,2011,6(7):e21645.
[14] Basunanda P,Radoev M,Ecke W,etal.Comparative mapping of quantitative trait loci involved in heterosis for seedling and yield traits in oilseed rape(BrassicanapusL.)[J].Theor Appl Genet,2010,120(2):271-281.
[15] Shi J Q,Li R Y,Qiu D,etal.Unraveling the complex trait of crop yield with quantitative trait loci mapping inBrassicanapus[J].Genetics,2009,182(3):851-861.
[16] Mei D S,Wang H,Hu Q,etal.QTL analysis on plant height and flowering time inBrassicanapus[J].Plant Breeding,2009,128(5):458-465.
[17] Udall J A,Quijada P A,Lambert B,etal.Quantitative trait analysis of seed yield and other complex traits in hybrid spring rapeseed(BrassicanapusL.):2.Identification of alleles from unadapted germplasm[J].Theor Appl Genet,2006,113(4):597-609.
[18] Würschum T,Liu W,Maurer H P,etal.Dissecting the genetic architecture of agronomic traits in multiple segregating populations in rapeseed(BrassicanapusL.)[J].Theor Appl Genet,2012,124(1):153-161.
[19] 介智靖,闫晓红,方小平,等.甘蓝型油菜DH植株创建及其倍性的流式鉴定[J].中国油料作物学报,2010,32(3):349-353.
[20] 朱恒星,闫晓红,方小平,等.甘蓝型油菜千粒重性状的QTL初步定位研究[J].植物遗传资源学报,2012,13(5):843-850.
[21] Zeng Z B.Theoretical basis for separation of multiple linked gene effects in mapping quantitative trait loci[J].Proc Natl Acad Sci USA,1993,90(23):10972-10976.
[22] Zeng Z B.Precision mapping of quantitative trait loci[J].Genetics,1994,136(4):1457-1468.
[23] McCouch S R,Cho Y G,Yano M,etal.Report on QTL nomenclature[J].Rice Genet Newsl,1997,14:11-13.
[24] Chalhoub B,Denoeud F,Liu S,etal.Early allopolyploid evolution in the post-NeolithicBrassicanapusoilseed genome[J].Science,2014,345:950-953.
[25] Hedden P,Phillips A L.Gibberellin metabolism:New insights revealed by the genes[J].Trends in Plant Science,2000,5(12):523-530.
[26] Xu Y L,Li L,Wu K,etal.TheGA5 locus ofArabidopsisthalianaencodes a multifunctional gibberellin 20-oxidase:Molecular cloning and functional expression[J].Proc Natl Acad Sci USA,1995,92(14):6640-6644 .
[27] Ogawa M,Hanada A,Yamauchi Y,etal.Gibberellin biosynthesis and response duringArabidopsisseed germination[J].Plant Cell,2003,15(7):1591-1604.
[28] Kushnir S,Babiychuk E,Storozhenko S,etal.A mutation of the mitochondrial ABC transporter Sta1 leads to dwarfism and chlorosis in theArabidopsismutant starik[J].The Plant Cell,2001,13(1):89-100.
QTL Mapping of Plant Height and Prediction of Candidate Genes in Major QTL Interval ofBrassicanapus
JIANG Chenghong,GENG Xinxin,WEI Wenhui*,JIANG Huifang*
(Oil Crops Research Institute,Chinese Academy of Agricultural Sciences/Key Laboratory of Biology and Genetic Improvement of Oil Crops,Ministry of Agriculture,Wuhan 430062,China)
For studying the genetic basis controlling the plant height of rapeseed(Brassicanapus),a recombinant inbred line population(190 lines) derived from a cross between a dwarfB.napusline DH-7-9 and a tallB.napusline DH-G-42 constructed by selfing for successive generations was used as material.This population was grown in Xining and Wuhan.The plant heights of individual lines were measured.The QTLs of plant height were detected using the genetic map constructed before by the complex interval graph method(CIM) of WinQTLcart 2.5 software under two environments.The results showed that a total of 11 putative QTLs for plant height were detected under two environments,each of them explained 1.17%—10.45% of phenotypic variation.In linkage group A10,one major QTL(qPH-X-A10 orqPH-W-A10) was identified under two environments repeatedly,explaining 10.24%—10.45% of the phenotypic variation.We collected 156 genes associated with the plant height ofArabidopsisthalianaand then searched the genomic region corresponding to the major QTL confidence interval to screen the possible candidate genes inB.napus.Two candidate genes,BnaA10g07740DandBnaA10g12020D,associated with plant height were founded.They had three orthologous genesATGA20ox2,GA5/ATGA20ox1 andSTA1,all involving in plant height differentiation ofA.thaliana.
Brassicanapus; plant height; QTL mapping; candidate genes
2017-02-19
国家自然科学基金项目(31371664);武汉市科技攻关计划项目(2013020501010174);中央级公益性科研院所基本科研业务费专项资金项目(1610172012001)
姜成红(1990-),男,湖北红安人,在读硕士研究生,研究方向:作物遗传学。E-mail:jiangchenghong1234@163.com
*通讯作者:魏文辉(1971-),男,湖北红安人,教授,博士,主要从事作物基因组学与分子育种研究。 E-mail:whwei88@hotmail.com 姜慧芳(1963-),女,湖北新洲人,研究员,博士,主要从事花生种质资源研究。E-mail:peanut@oilcrops.cn
S565.4
A
1004-3268(2017)08-0027-05
