大肠杆菌和肺炎克雷伯氏菌的共培养研究
2017-08-02陈柳霓李笑寒田平芳北京化工大学生命科学与技术学院北京100029
陈柳霓,李笑寒,田平芳(北京化工大学生命科学与技术学院,北京100029)
大肠杆菌和肺炎克雷伯氏菌的共培养研究
陈柳霓,李笑寒,田平芳
(北京化工大学生命科学与技术学院,北京100029)
为研究大肠杆菌(Escherichia coli)和肺炎克雷伯氏菌(Klebsiella pneumoniae)混菌体系,构建了基于抗生素筛选的2株重组菌。将携带卡那霉素抗性基因的载体pET-28a转化到肺炎克雷伯氏菌中,获得重组菌K.pneumoniae(pET-28a);将携带氯霉素抗性基因cm的重组载体pET-cm(卡那霉素和氯霉素双抗性载体)转化到大肠杆菌中,获得重组菌E.coli(pET-cm),在卡那霉素抗性培养基中单独培养及混合培养上述两株重组菌。结果发现:单独培养条件下,E.coli与K.pneumoniae的相对菌体密度为57.87%;混合培养条件下,E.coli与K.pneumoniae的相对菌体密度为1.94%;E.coli和K.pneumoniae相对于各自单独培养的存活率分别为2.57%和76.60%。上述结果表明,K.pneumoniae为混菌体系的优势菌,可强烈抑制E.coli的生长。
大肠杆菌;肺炎克雷伯氏菌;混合培养;相对密度;氯霉素;卡那霉素
微生物合成目标产物通常涉及多酶催化反应。一般多在单菌中超表达多个酶基因,其载体构建难度大且菌体代谢负荷重。相比之下,混菌体系可将代谢途径分摊给不同的菌,即一种菌的产物为另一种菌的底物,避免了单菌承载太长的代谢途径[1-2]。近年来,混菌体系已用于化工原料生产等方面[1-2]。例如,在酵母和梭状芽孢杆菌混菌体系中,梭状芽孢杆菌将纤维素转化为单糖,酵母将单糖转化为乙醇[3]。将达到相对稳态的混菌体系称为顶级群落(climax community),其种间的代谢流分配趋于稳定[4]。
通过遗传改造实现菌间互利共生是构建人工混菌体系的目标。E.coli遗传背景清晰,作为宿主菌已广泛用于化学品生产[5-6]。肺炎克雷伯氏菌(Klebsiella pneumoniae)虽非模式生物,但能天然合成一系列大宗重要化学品,包括3-羟基丙酸[7]、1,3-丙二醇[8]及2,3-丁二醇[9],因此成为当前工业微生物的研究热点。
本文中,笔者首先构建携带不同抗性的重组大肠杆菌(E.coli)和肺炎克雷伯氏菌(K.pneumoniae),然后在碳源充足情况下进行单独培养和混合培养,考察两种菌之间的关系,以期为建立混菌体系奠定基础。
1 材料与方法
1.1 材料
E.coli BL21和K.pneumoniae DSM 2026保存于笔者所在实验室。限制性内切酶、T4 DNA连接酶、质粒提取和PCR产物回收试剂盒,博迈德生物技术有限公司。DNA测序由北京六合华大基因科技股份有限公司完成。
LB液体培养基(g/L):酵母粉5,NaCl 10,蛋白胨10;pH 7.0。
LB固体培养基:向LB液体培养基中加入1.5 g/L琼脂。
卡那霉素抗性培养基:向LB液体培养基中加入50μg/mL卡那霉素。
氯霉素抗性培养基:向LB液体培养基中加入170μg/mL氯霉素。
1.2 方法
总体思路如图1所示。首先构建两株重组菌,在试管中单独培养和混合培养大肠杆菌与肺炎克雷伯氏菌,按相等菌量(以OD600计算)涂布于氯霉素抗性和卡那霉素抗性LB平板,统计菌落数。在液体培养基中进行单独培养和混合培养24 h,每3 h用紫外分光光度计测定菌体量,绘制生长曲线。
1.2.1 重组菌的构建
1)氯霉素抗性基因cm的克隆。根据GenBank公布的氯霉素抗性基因cm的序列设计引物,以实验室保藏cm质粒为模板,将带有氯霉素启动子等元件的cm表达框完整地克隆出来。上游引物引入酶切位点SacⅠ,下游引物引入SalⅠ。上游引物: 5'-TCCGAGCTCCAATAAACCGGTAAACCAG-3';下游引物:5'-GACGTCGACAGCTGATAGAAACAGAAGC-3'(下划线序列为酶切位点)。PCR反应体系: ddH2O 8.8μL,模板0.4μL,上游引物0.4μL,下游引物0.4μL,2×PCR mix 10μL;总体积20μL。PCR反应参数:94℃变性3 min;94℃1 min,55℃40 s,72℃1 min,共30个循环;72℃10 min;即得到cm基因表达框,16℃保存。

图1 实验流程示意Fig.1 Schematic diagram of this study
2)载体pET-cm的构建与转化。基因cm和载体pET-28a经过双酶切后连接,转化E.coli BL21,用菌落PCR及质粒双酶切鉴定。将空质粒pET-28a电击转化K.pneumoniae DSM 2026感受态细胞,获得携带卡那霉素抗性的肺炎克雷伯氏菌K. pneumoniae(pET-28a)。由此获得同时携带氯霉素和卡那霉素抗性的重组菌E.coli(pET-cm)和单独携带卡那霉素抗性的肺炎克雷伯氏菌K.pneumoniae (pET-28a)。
1.2.2 重组菌的培养
菌种复苏培养于15 mm×150 mm试管,内装4 mL LB培养基,接种量1%(体积分数),在37℃、200 r/min条件下培养。将两种重组菌按1%接种量单独培养和混合培养于100 mL LB液体培养基中,在37℃、200 r/min条件下培养24 h,每隔3 h测OD600。
将重组大肠杆菌和肺炎克雷伯氏菌进行单独培养和混合培养,按1%的接种量接种于4 mL LB液体培养基中,在37℃、200 r/min条件下培养11 h,测OD600。根据计算结果,用相等菌量菌涂布平板,过夜倒置培养于37℃恒温培养箱中,统计菌落数。1.2.3菌体生物量及菌落数分析测定
采用比浊法测定菌体生物量。将过夜培养的原菌液稀释10倍,用分光光度计测OD600。据此计算单独培养和混合培养涂布时所需的菌体量。
涂布时,将原菌液分别稀释106、107、108倍,培养15 h后统计菌落数。单独培养中,大肠杆菌涂于卡那霉素抗性平板,计数;肺炎克雷伯氏菌涂于卡那霉素抗性平板,计数。混合菌以等菌体量分别涂于氯霉素抗性平板和卡那霉素抗性平板,计数。
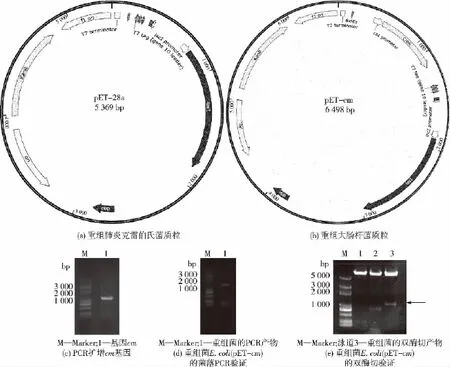
图2 重组质粒示意图与基因克隆电泳图Fig.2 Schematic diagram of vector construction and validation by PCR and electrophoresis
2 结果与讨论
2.1 重组菌E.coli(pET-cm)和K.pneumoniae
(pET-28a)的构建
载体构建及cm基因的PCR扩增验证结果见图2。由图2可知:PCR扩增能够得到cm基因条带,为1 100 bp(图2(c)),说明测序表明其序列正确。将重组质粒pET-cm转化E.coli BL21,挑取单菌落进行PCR,得到约1 400 bp的条带(图2(d))(引物为cm基因的上游引物和pET-28a的通用下游引物),提取质粒进行双酶切验证(图2(e)泳道3),表明重组质粒pET-cm已转化至E.coli BL21。将空质粒pET-28a电击转化K.pneumoniae,复苏后涂布于卡那霉素抗性平板,培养24 h后挑取单菌落,利用pET-28a质粒的通用引物进行菌落PCR鉴定,表明pET-28a已转化至K.pneumoniae。
2.2 生物量测定
将重组菌单独及混合培养于含抗生素的100 mL LB液体培养基中,在37℃、200 r/min条件下培养,24 h后测定生物量,结果见图3。由图3可知,K.p (kana)和E.coli(kana)分别为重组肺炎克雷伯氏菌和大肠杆菌在卡那霉素抗性培养基生长良好。在相同培养条件下,肺炎克雷伯氏菌的生长明显快于大肠杆菌。但在混合培养条件下,这两种的生物量均低于单独培养K.pneumoniae的生物量,但都高于单独培养E.coli的生物量,表明两菌存在生长竞争。
2.3 单独培养和混合培养的菌落数
将两株重组菌分别单独培养与混合培养于4 mL卡那霉素抗性培养基中,在37℃、200 r/min条件下培养11 h后测OD600,据此计算涂布所需的菌量,结果见表1。由表1可知:由于重组大肠杆菌中的质粒负担要大于肺炎克雷伯氏菌,因此大肠杆菌的生长慢于肺炎克雷伯氏菌。即使排除质粒干扰,大肠杆菌自身的生长也慢于肺炎克雷伯氏菌。
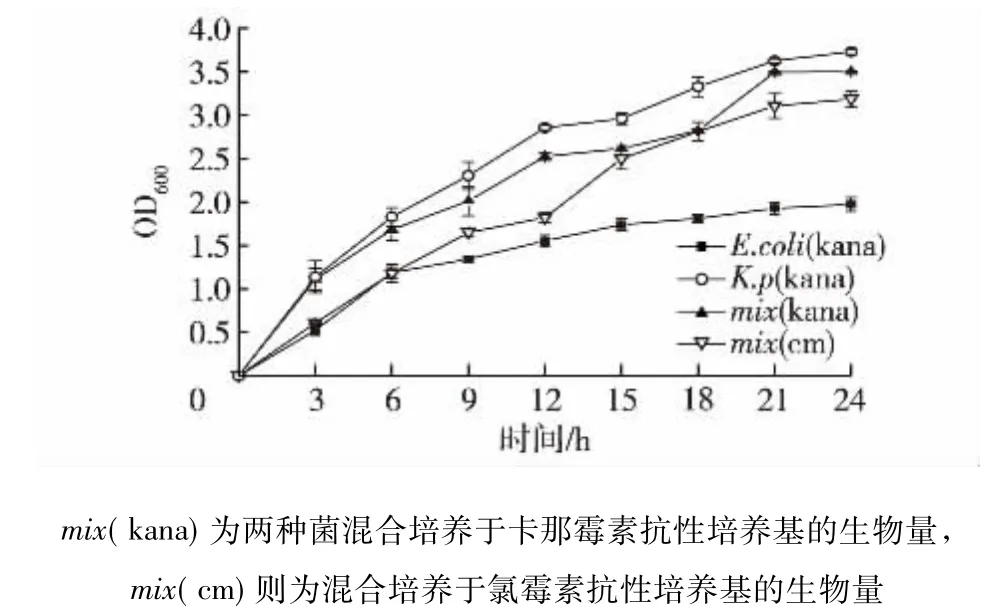
图3 重组菌的生物量Fig.3 Shake flask cultivation of the recombinant strains
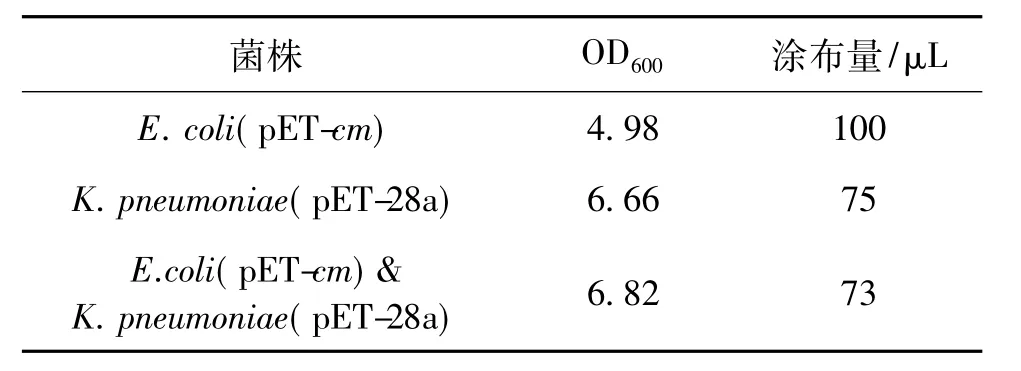
表1 涂布过程参数Table 1 Parameters for bacterial inoculation
因此,考察单独培养与混合培养的生长菌活数,以确定不同抗生素对单独培养和混合培养的抑菌效果。按不同稀释倍数涂布后,于37℃恒温倒置培养15 h,统计菌落数,结果见表2。

表2 单独培养与混合培养的菌落数Table 2 The colony number in separate culture and mixed culture
由表1与表2可知:在单独培养的条件下,K.pneumoniae(pET-28a)的单菌落数明显多于E.coli (pET-cm),在一定程度上说明,K.pneumoniae的生长速度快于E.coli,两种菌虽然具有一定同源性,推测其代谢途径与代谢能力各不相同,导致其自身生长中存在较大差异。
之后计算两种菌混合培养相对于单独培养的存活率,结果见表3。
由表3可知:在梯度稀释106倍时,E.coli(pET-cm)的存活率明显低于K.pneumoniae(pET-28a)。同源性较高的两种菌在混合培养中存活率表现出了明显的差异性,由此可以推测,大肠杆菌和肺炎克雷伯氏菌在培养中存在相互抑制或者相互竞争的关系。在现实工业生产中,若想实现这两种微生物的混合培养,势必还要进行相应研究。后续将探究重组菌的相对菌体密度。

表3 混合培养中重组菌存活率Table 3 Survival rate of recombinant strains in mixed culture
以肺炎克雷伯氏菌为100%,考察大肠杆菌单独培养和混合培养对于肺炎克雷伯氏菌的相对菌体密度的影响,结果见表4。
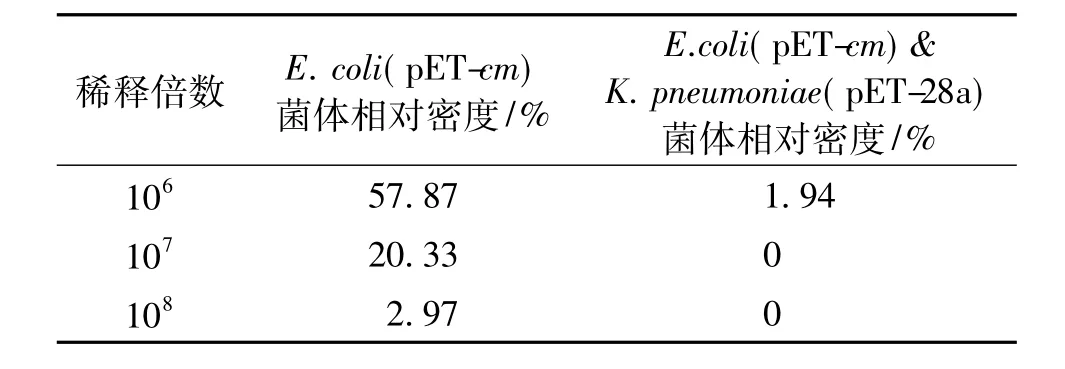
表4 重组菌的相对菌体密度Table 4 Relative density of recombinant strains
由表4可知:在单独培养条件下,大肠杆菌的生长速度慢于肺炎克雷伯氏菌,推测是由于不同菌之间代谢的差异性导致;在混合培养时,大肠杆菌的生长情况证明了两种菌在生长中存在相互抑制关系,其中抑制关系使得大肠杆菌生长速度变慢,使肺炎克雷伯氏菌为混菌体系中的优势菌。在之后的研究中可利用这种天然的生长优势来达到混合培养过程中对于两种菌生物量的调控,也可对两种菌进行代谢流的探究与改造,以期实现两种菌的互利共生。
重复进行试验,测定不同抗性条件下的单独培养及混合培养条件下的菌落数量,结果见图4。
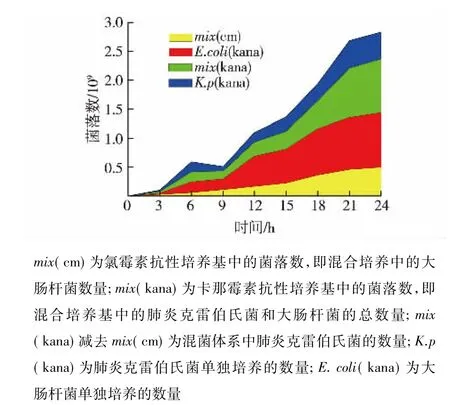
图4 重组菌的菌落数Fig.4 Colony numbers of the recombinant strains
由图4可知:就单独培养而言,肺炎克雷伯氏菌具有生长优势,生物量明显要大于大肠杆菌,生长速度快,说明肺炎克雷伯氏菌具有工业应用的潜力。但由于对其研究有限,要想成为工业化生产的宿主菌,还需要经过后续一系列研究。在混合培养中,这两种菌的生长均低于单独培养,证明两种菌的生长均受到抑制,表明存在竞争关系,如何改造或者利用这种关系有待进一步研究。
虽然肺炎克雷伯氏菌具有荚膜毒性,但其代谢甘油能力很强[10],生长速度快,从生物量来看非常适合用作表达宿主。实验表明,肺炎克雷伯氏菌的生长明显快于大肠杆菌。由于肺炎克雷伯氏菌能自身产生辅酶B12[11],无需在培养基中额外添加B12,因此,它比大肠杆菌具有明显成本优势。
后期研究可致力混菌培养。混菌培养可发挥不同菌的优势,但需进行相对密度控制策略:①针对混菌体系代谢途径的特点,借助动态控制的代谢模拟手段,如(COBRA工具箱)[12],模拟菌种代谢流平衡体系,实现菌体相对密度的动态优化;②重构两菌的群体感应(quorum sensing),实现两菌生长速度的相互制约[13-14];③构建两菌内部的基因回路,包括双稳态开关[15]和基于共有代谢物的生物量控制装置[16],从而动态调控两菌的代谢流量分配。因为合成生物学不仅能实现单菌基因表达的自动化和智能化,而且有望改变不同菌的种间关系(interspecific relationship)。因此,有望将大肠杆菌和肺炎克雷伯氏菌之间的抑制关系改为互利共生,从而发挥各自优势,拓宽发酵底物和产物的范围。
3 结论
1)克隆了E.coli的cm抗性基因,构建了携带不同抗性的重组大肠杆菌E.coli(pET-cm)和肺炎克雷伯氏菌K.pneumoniae(pET-28a);
2)研究了两种菌的存活率和相对菌落数,结果表明,混菌体系中大肠杆菌比肺炎克雷伯氏菌受抑制程度更大,肺炎克雷伯氏菌为混菌体系的优势菌。该研究也为食品微生物防控提供了依据。
[1]CONTAG P R.Organism co-culture in the production of biofuels: US,008986962B2[P].2015-03-24.
[2]ZHOU K,QIAO K J,EDGAR S,et al.Distributing a metabolic pathway among a microbial consortium enhances production ofnatural products[J].Nat Biotechnol,2015,33(4):377-383.
[3]ZUROFF T R,XIQUES S B,CURTIS W R.Consortia-mediated bioprocessing of cellulose to ethanol with a symbiotic Clostridium phytofermentans yeast co-culture[J].Biotechnol Biofuels,2013,6 (1):59.
[4]MACLEAN R C,FUENTES-HERNANDEZ A,GREIG D,et al.A mixture of"cheats"and"co-operators"can enable maximal group benefit[J].PLoS Biol,2010,8(9):e1000486.
[5]GRILLO M A,COLOMBATTO S.S-adenosylmethionine and its products[J].Amino Acids,2008,34(2):187-193.
[6]HE W,FU L,LI G,et al.Production of chondroitin in metabolically engineered E.coli[J].Metab Eng,2015,27: 92-100.
[7]ASHOK S,SANKARANARAYANAN M,KO Y,et al.Production of3-hydroxypropionic acid from glycerolby recombinant Klebsiella pneumoniaΔdhaTΔyqhD which can produce vitamin B12naturally[J].Biotechnol Bioeng,2013,110(2):511-524.
[8]HUANG Y,LI Z,SHIMIZU K,etal.Simultaneous production of3-hydroxypropionic acid and 1,3-propanediol from glycerol by a recombinant strain of Klebsiella pneumoniae[J].Bioresour Technol,2012,103(1):351-359.
[9]PETROV K,PETROVA P.High production of 2,3-butanediol from glycerol by Klebsiella pneumonia G31[J].Appl Microbiol Biotechnol,2009,84(4):659-665.
[10]FENG X,DING Y,XIAN M,et al.Production of optically pure D-lactate from glycerol by engineered Klebsiella pneumoniae strain[J].Bioresour Technol,2014,172:269-275.
[11]FORAGE R G,FOSTER M A.Glycerol fermentation in Klebsiella pneumoniae:functions of the coenzyme B12-dependent glycerol and diol dehydratases[J].J Bacteriol,1982,149(2):413-419.
[12]SCHELLENBERGER J,QUE R,FLEMING R M T,et al.Quantitative prediction of cellular metabolism with constraintbased models:the COBRA Toolbox v2.0[J].Nat Protoc,2011,6 (9):1290-1307.
[13]YOU L,COX R S III,WEISS R,et al.Programmed population control by cell-cell communication and regulated killing[J].Nature,2004,428:868-871.
[14]AN J H,GOO E,KIM H,et al.Bacterial quorum sensing and metabolic slowing in a cooperative population[J].Proc Natl Acad Sci USA,2014,111(41):14912-14917.
[15]GARDNER T S,CANTOR C R,COLLINS J J.Construction of a genetic toggle switch in Escherichia coli[J].Nature,2000,403: 339-342.
[16]MINTY J J,SINGER M E,SCHOLZ S A,et al.Design and characterization of synthetic fungal-bacterial consortia for direct production of isobutanol from cellulosic biomass[J].Proc Natl Acad Sci USA,2013,110(36):14592-14597.
(责任编辑荀志金)
Co-culture of Escherichiacoli and Klebsiella pneumoniae
CHEN Liuni,LI Xiaohan,TIAN Pingfang
(College of Life Science and Technology,Beijing University of Chemical Technology,Beijing 100029,China)
To study the co-culture of Escherichia coli and Klebsiella pneumoniae,we constructed distinguishable recombinant strains by recruiting distinct resistance genes.Briefly,the vector pET-28a harboring kanamycin resistance gene(Kanr)was transformed into K.pneumoniae,leading to recombinant strain K.pneumoniae(pET-28a).The vector pET-cm harboring both Kanrand chloramphenicol resistance gene(cm)was transformed into E.coli,resulting in recombinant strain E.coli(pET-cm).The two recombinant strains were subjected to monoculture and co-culture.Under monoculture conditions,the relative density between E.coli(pET-cm)and K.pneumoniae(pET-28a)was 57.87%,whereas it was only 1.94%under co-culture conditions.More interestingly,the relative survival rates of E.coli and K.pneumoniae under co-culture conditions were 2.57%and 76.60%,respectively,compared with that of monoculture conditions.Overallthese results indicate that K.pneumoniae(pET-28a)was the predominant species and strongly inhibited the growth of E.coli(pET-cm).
Escherichia coli;Klebsiella pneumoniae;co-culture;relative density;choramphenicol;kanamycin
Q81
A
1672-3678(2017)04-0034-06
10.3969/j.issn.1672-3678.2017.04.006
2017-06-09
国家自然科学基金(21276014、21476011);国家高技术研究发展计划(863计划)(2015AA021003)
陈柳霓(1992—),女,重庆涪陵人,研究方向:微生物代谢工程;田平芳(联系人),教授,E-mail:tianpf@mail.buct.edu.cn
