野生大豆对中国大豆育成品种遗传贡献的分子印证
2017-07-18吕祝章
吕祝章
(日照职业技术学院,山东 日照 276826)
野生大豆对中国大豆育成品种遗传贡献的分子印证
吕祝章
(日照职业技术学院,山东 日照 276826)
采用SSR标记鉴定野生大豆对10个大豆育成品种的遗传贡献.结果表明:野生大豆含有较多的特有等位变异,利用率为17.27%;回交能降低野生大豆特有等位变异利用率;野生大豆在16个位点上与粒重、荚粒数、高硬脂酸含量、抗胞囊线虫等有关的19个特有等位变异易被育成品种遗传利用;10个大豆育成品种在13个位点上产生了18个新的等位变异.利用野生大豆改良和创新大豆有较大空间.不同的杂交组合方式对育成品种的遗传贡献有明显差异.野生大豆的小粒、多荚、高硬脂酸含量以及抗胞囊线虫等优良性状基因易被育成品种选择利用.
野生大豆; 育成品种; SSR; 等位变异; 遗传贡献
中国是大豆的起源地,除新疆、青海、海南外均有野生大豆.如此广泛的地理分布和巨大的环境差异,生长出适应不同生态条件的野生大豆类型.我国现收集保存了6 672份野生大豆资源,占世界总数的90%以上,其中蕴藏着高蛋白[1,2]、高亚油酸[2,3]、高亚麻酸和低植酸[4]、胰蛋白酶抑制剂缺失基因或无脂氧酶[5]、抗逆性强、多花多荚、丰产性好[6,7]等一大批特有的优异基因型[8-12].
我国利用野生大豆培育出200余份各具特性的新种质,其中审定品种达20余个[13-17].杨光宇等[14]利用野生大豆小粒、高蛋白质、抗逆性强等特性育出高蛋白、耐干旱大豆品种吉育59号,高产品种吉林66号和出口专用小粒黄豆系列品种吉林小粒1-8号(吉林小粒1号1995年获国家发明四等奖).姚振纯等[15]选育出蛋白质含量48%以上的龙品8807(被评为“九五”国家科技攻关项目重大科技成果一级优异种质、2002年获黑龙江省科技进步二等奖)及审定熟期最早的特用小粒大豆品种龙小粒豆1号(2003年获黑龙江省科技进步二等奖).李福山等[16]选育出高产、耐盐大豆新品种中野1号、中野2号.这些都足以证明野生大豆中的有益性状基因在栽培大豆的改良中起到了重要作用.
关于野生大豆×栽培大豆后代品系农艺品质性状表现及遗传变异分析已有不少报道[18-27].但运用SSR技术进行有关野生大豆对生产上大面积推广应用的育成品种的遗传贡献方面的研究还鲜少报道.
本试验采用SSR标记技术对利用野生大豆育成的10个大豆品种及其17个亲本材料进行分析,目的是鉴定野生大豆DNA片段被大豆育成品种的利用情况,分析野生大豆导入片段与其育成品种农艺性状及栽培特性形成的关系,发掘出一批农艺品质性状优异位点、等位变异及携带优异等位变异的野生大豆载体材料,为大豆分子标记辅助育种等提供参考信息.
1 材料与方法
1.1 试验材料
选用了10个具有野生血缘且在生产上大面积推广应用的大豆品种及其17个亲本,亲本中的黑龙江小粒豆、察隅1号未被搜集到,各品种的杂交组合方式见表1.
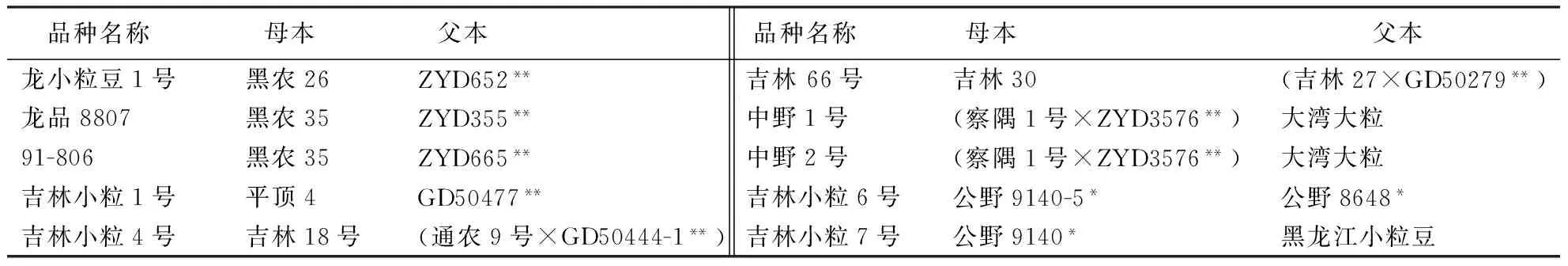
表1 利用野生大豆育成的大豆推广品种系谱1)
1)*和**分别代表半野生和野生大豆.
1.2 试验方法
1.2.1 SSR分析 每份材料从入库的500 g纯净种子中随机取出100粒种子磨成豆粉,用SDS法[28]提取DNA.PCR反应体系为20 μL,其中含有40 ng基因组DNA模板、1×PCR缓冲液、1.25 mmol·L-1MgCl2、0.2 mmol·L-1dNTP、0.2 μmol·L-1SSR引物和1 UTagDNA聚合酶.选用分布于全基因组的88对SSR引物进行分析.反应在PE-9600型号的PCR扩增仪上进行.反应程序为95 ℃预变性4 min;95 ℃变性30 s,47 ℃退火30 s,72 ℃延伸30 s,运行35个循环;72 ℃延伸10 min,4 ℃保存.扩增产物用6%聚丙烯酰胺、8 mol·L-1脲素制成的测序胶分离,银染技术检测.
1.2.2 数据统计分析 每个SSR位点,根据条带的有无记录等位变异数目.
2 结果与分析
2.1 大豆育成品种与亲本间的遗传关系
在20个连锁群上选取88个SSR位点进行分析,9个野生及半野生大豆亲本共检测到339个等位变异,与8个栽培亲本相比较,共拥有110个特有等位变异,其中Satt591、Satt304、Satt294、Satt180、Satt291、Sat_252、Satt184、Satt014、Satt012、Satt192、Sat_219、Satt239、Satt232、Satt446、Sat_099、Satt387等16个位点上的19个特有等位变异被8个大豆育成品种所利用,特有等位变异利用率达17.27%;野生亲本在Satt461、Sat_252、Satt294、Satt192、Sct_189和Satt596等6个位点上分布的特有等位变异数较多,都在4个以上;野生亲本在Satt005、Sat_219、Satt239、Sct_189等位点上拥有特有等位变异相对集中,都有6个野生大豆在上述4个位点上拥有特有等位变异;野生亲本ZYD355、ZYD3576、GD50444-1、GD50477和ZYD652分别含有58、27、26、19和17个特有等位变异,其中GD50477和ZYD355特有等位变异利用率较高,分别达31.58%和17.24%,ZYD652的利用率为0,公野8646和公野9140等半野生大豆材料含有的特有等位变异数较少,可能与种间杂种材料的选择与驯化有关;龙品8807和吉林小粒1号利用其野生亲本特有等位变异数较多,分别遗传利用了ZYD355亲本7个位点上的10个和GD50477亲本6个位点上的6个特有等位变异(表2、表3).8个栽培大豆亲本共检测到259个等位变异,与9个野生及半野生大豆亲本相比较,有30个特有等位变异,其中10个特有等位变异被其育成品种利用,利用率为33.33%.10个大豆育成品种共检测到264个等位变异,其中包括在13个位点上新产生的18个特有等位变异.

表2 野生大豆特有等位变异分布及利用情况
续表2

序号连锁群位点野生大豆特有等位变异数拥有特有等位变异野生大豆野生大豆特有等位变异利用数野生大豆特有等位变异利用品种及野生亲本41ISat_2191ZYD355、ZYD665、GD50477、ZYD652、GD50444-1、公野86481龙品8807(ZYD355)42ISatt2393ZYD355(2)、GD50477、ZYD652、GD50444-1、公野8648、ZYD35761吉林小粒1号(GD50477)43ISatt2923ZYD355、ZYD665、GD50444-1、ZYD3576(2)44ISct_1894ZYD355(4)、ZYD665、GD50477、ZYD652、GD50444-1、ZYD357645JSatt5964ZYD355(4)、ZYD665、GD50444-1、GD50279、ZYD357646JSatt2443ZYD355(2)、ZYD665、GD50444-147KSatt1671公野864848KSatt5591GD50444-149LSatt2322ZYD355、ZYD6521龙品8807(ZYD355)50LSatt4461ZYD3551龙品8807(ZYD355)51LSatt1823GD50477、GD50444-1、GD50279、ZYD3576(2)52LSatt4621ZYD355、GD50279、ZYD357653LSatt4811GD5047754LSat_0992ZYD355(2)2龙品8807(ZYD355)55LSatt3731GD50477、GD50444-1、GD5027956MSatt5902ZYD355(2)、ZYD652、GD50279、ZYD357657MSatt5401ZYD355、ZYD65258MSatt3061GD50444-159NSatt3871GD50477、ZYD6521吉林小粒1号(GD50477)60OSatt4871ZYD665、GD50477、GD5027961OSatt2431ZYD355总计11019
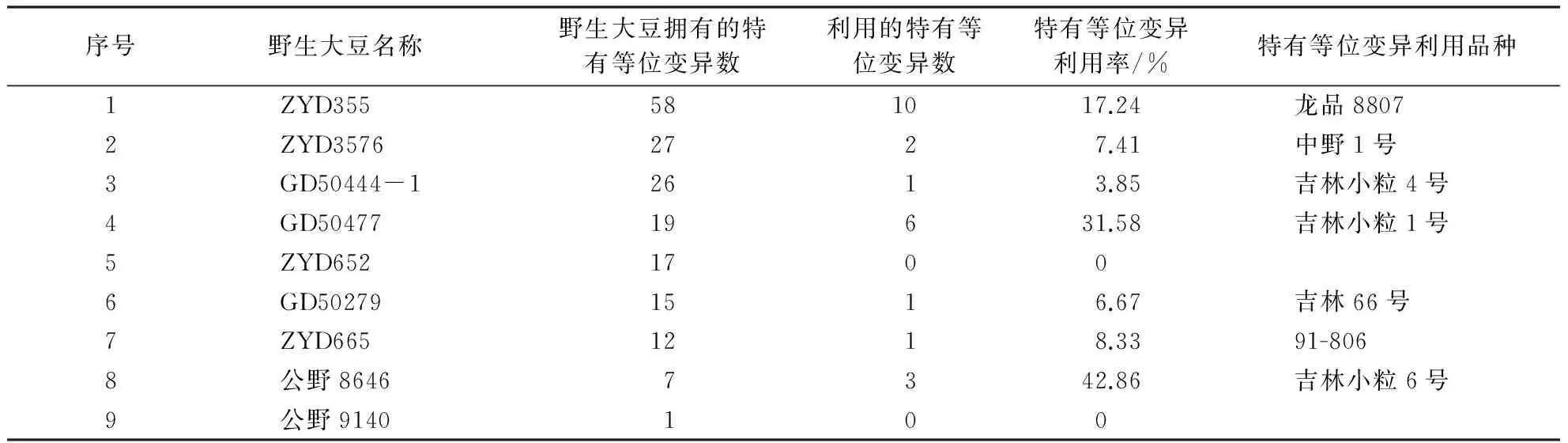
表3 各野生大豆拥有特有等位变异数及利用情况
2.2 同组合中野生大豆对其育成品种的遗传贡献
以组合为单位,对每个组合中野生亲本相对于本组合中的栽培亲本所拥有的“特有”等位变异情况进行分析表明:野生和半野生大豆亲本在60个位点上有着不同程度的“特有”等位变异,这些“特有”等位变异也不同程度地被其育成品种所遗传利用.育成品种利用其野生亲本的区段情况见表4,从表中可以看出,野生大豆M连锁群Satt210位点上的“特有”等位变异最易被其育成品种遗传利用,有6个品种遗传利用了其野生亲本该位点上的等位变异;其次是位于B2连锁群上的Satt556位点,有5个品种分别遗传利用了其野生亲本该位点上的“特有”等位变异;再次是位于A1连锁群上的Satt591、B1连锁群上的Satt453、D1a连锁群上的Satt184、F连锁群上的Satt114、L连锁群上的Satt182、N连锁群上的Satt530等6个位点,有4个品种分别遗传利用了其野生亲本该位点上的等位变异.

表4 同组合中野生大豆对其育成品种的遗传贡献情况1)
续表4

序号位点连锁群利用同组合中野生大豆“特有”等位变异品种53Satt540M龙品8807、91-80654Satt175M中野2号55Satt210M龙小粒豆1号(2)、龙品8807、中野1号、中野2号、吉林小粒6号(公野8648)、吉林小粒6号(公野9140)、吉林小粒7号56Satt530N吉林小粒4号、吉林小粒6号(公野8648)、吉林小粒6号(公野9140)、吉林小粒7号、龙小粒豆1号57Satt387*N吉林小粒1号58Satt487O吉林小粒6号(公野8648)、吉林小粒7号59Satt345O吉林小粒1号、吉林小粒7号60Satt243O吉林小粒6号(公野8648)、吉林小粒6号(公野9140)、吉林小粒7号
1)*表示9个野生及半野生大豆共有的特有等位变异被育成品种利用的位点.
表4、表5列出了大豆育成品种利用其同组合中野生亲本“特有”等位变异情况,利用其野生亲本“特有”等位变异数最多的品种是吉林小粒1号,其利用了野生大豆GD50477在Satt197、Satt415、Satt453、Satt556、Satt180、Satt291、Satt286、Sat_312、Sat_252、Satt184、Satt014、Satt002(2个)、Satt226、Satt309、Satt279、Satt302、Satt239、Satt596、Sat_099、Satt387、Satt345等21个位点上的22个特有等位变异,占GD50477特有等位变异总数的35.48%;吉林小粒4号和吉林66号利用其野生亲本“特有”等位变异数最少,特有等位变异利用率也最低,分别为6.98%和7.32%.吉林小粒6号利用了公野8648在Satt591、Satt236、Satt556、Satt184、Satt461、Satt012、Satt192、Satt302、Satt571(2个)、Satt210、Satt530、Satt487、Satt243等13个位点上的14个特有等位变异,占公野8648“特有”等位变异总数的45.16%;利用了公野9140-5在Satt591、Satt174、Satt453、Satt202、Satt179、Satt157、Satt005、Satt002、Satt461、Satt114、Satt335、Satt182、Satt210、Satt530、Satt243等15个位点上的15个特有等位变异,占公野9140-5“特有”等位变异总数的48.39%,特有等位变异利用率最高.

表5 育成品种利用其野生亲本“特有”等位变异情况
2.3 野生大豆对拓宽大豆育成品种遗传基础的组配贡献方式
表5数据显示:野生及半野生大豆其“特有”等位变异数被其育成品种利用的比例范围为6.98%~48.39%,其中通过回交方式育成的吉林小粒4号、吉林66号、中野2号、中野1号等4个品种利用其野生亲本“特有”等位变异的利用率分别为6.98%、7.32%、17.07%和17.14%,“特有”等位变异平均利用率12.13%;吉林小粒4号和吉林66号是采用栽培大豆做母本进行回交转育而成,“特有”等位变异平均利用率为7.15%,而中野1号和中野2号是采用栽培大豆做父本进行回交转育而成,“特有”等位变异平均利用率为17.11%,从此可以看出,回交方式的不同决定了野生亲本的“特有”等位变异利用率存在着较大的差别.通过单交方式育成的龙品8807、91-806、吉林小粒1号、龙小粒豆1号等4个品种利用其野生亲本“特有”等位变异利用率为23.29%~35.48%,平均利用率为29.07%;而利用具有野生大豆血缘的种间杂种材料育成的吉林小粒6号平均利用率高达46.78%.
3 结论
研究结果表明:野生大豆共有的分布在16个位点上与产量和粒重有关的Satt304[29]、Satt294[30]、Satt291[30]、Satt184[31]、Satt014[32]、Sat_099[29,33]、Satt210[34-35],与产量和株高有关的Sat_252[34,36]、Satt387[37-38],与荚粒数有关的Sat_219[34]、Satt239[25,26,32,34,39],与脂肪和高硬脂酸含量有关的Satt180[36]、Satt591[40],与抗胞囊线虫有关的Satt012[41-43]、Satt453[44]和与倒伏性有关的Satt232[45]等位点上的特有等位变异被育成品种利用.由此可见,野生大豆的小粒、多荚、高硬脂酸含量以及抗胞囊线虫等优良性状基因易被育成品种选择利用.
与栽培大豆相比,野生大豆含有较多的等位变异数和特有等位变异数,但其特有等位变异的整体利用率仅为17.27%,说明在杂交育种中利用野生大豆作为亲本进行大豆改良和创新仍有较大的空间.
野生大豆、种间杂交形成的半野生大豆及其杂交组合方式的不同对育成品种的遗传贡献有明显差异,研究结果表明:回交方式能降低野生大豆的特有等位变异利用,使品种更趋向栽培选择利用的目标性状,并能便捷有效地缩短育种时间.种间杂种材料的等位变异更易被其育成品种利用,在育种实践中可利用优异的种间杂种材料做亲本以有效提高其优良基因及性状的选择效率,对改良大豆的品质与抗性性状更具有实际意义.
10个大豆育成品种在13个位点上产生了18个新的等位变异,与Sjakste et al[46]在研究7个大麦品种与其父母本的遗传物质传递时发现有13.93位点存在与父母本均不相同的等位变异情况相同.表明野生和栽培大豆的种间杂交并不单纯是遗传物质的简单组合,更重要的是它可以通过基因的重组创造出新的优良基因型种质.
[1] 李福山.中国野生大豆资源的地理分布及生态分化研究[J].中国农业科学,1993,26(2):47-55.
[2] 徐豹.中国野生大豆(G.soja)研究十年[J].吉林农业科学,1989(1):5-13.
[3] 王连铮,吴和礼,姚振纯,等.黑龙江省野生大豆的考察和研究[J].植物研究,1983(3):116-130.
[4] RABOY V, DICKINSON D B. Phytic acid level in seeds ofGlycinemax×G.Sojaas influenced by phosphorus status[J]. Crop Science, 1993,33(6):1 300-1 305.
[5] 吴燕,赵秋,刘澍才.辽宁省野生大豆种质资源及其创新利用价值[J].杂粮作物,2004,24(3):182-183.
[6] 林红.黑龙江省野生大豆优异资源鉴定与筛选[J].作物品种资源,1999(2):41-42.
[7] 林红,齐宁,李向华,等.黑龙江省野生大豆资源考察研究[J].中国油料作物报,2006,28(4):427-430.
[8] HAJIKA M, TAKAHASHI M, SAKAI S, et al. A new genotype of 7S globulin (.BETA.-conglycinin) detected in wild soybean (GlycinesojaSieb.etZucc.)[J]. Japanese Journal of Breeding, 1996,46(4):385-386.
[9] HAJIKA M, TAKAHASHI M, SAKAI S, et al. Dominant inheritance of a trait lacking β-conglycinin detected in a wild soybean Line[J]. Breeding Sci, 1998,48(4):383-386.
[10] 盖钧镒,许东河,高忠,等.中国栽培大豆和野生大豆不同生态类型群体间遗传演化关系的研究[J].作物学报,2000,26(5):513-520.
[11] 李向华,田子罡,李福山.新考察收集野生大豆与已保存野生大豆的遗传多样性比较[J].植物遗传资源学,2003,4(4):345-349.
[12] 田清震,盖钧镒,喻德跃,等.我国野生大豆与栽培大豆AFLP指纹图谱研究[J].中国农业科学,2001,34(5):480-485.
[13] 杨光宇,王洋,马晓萍,等.小粒大豆新品种吉林小粒6号选育报告[J].中国农业信息,2008(4):19.
[14] 杨光宇,王洋,马晓萍,等.野生大豆种质资源评价与利用研究进展[J].吉林农业科学,2005,30(2):61-63.
[15] 姚振纯,林红,来永才.蛋白质与脂肪总含量66.16%大豆种间杂交新种质的选育[J].作物品种资源,1999(3):6-7.
[16] 王克晶,李福山.野生大豆种质杂交导人的育种效果[J].植物遗传资源科学,2000,1(3):34-38.
[17] 杨明亮,张东梅,常玉森,等.特用大豆优质种质资源利用与创新[J].黑龙江农业科学,2016(8):15-18.
[18] 赵洪锟,王玉民,李启云,等.中国不同纬度野生大豆和栽培大豆SSR分析[J].大豆科学,2001,20(3):172-176.
[19] 裴颜龙,王岚,葛松,等.野生大豆遗传多样性研究Ⅰ. 4个天然居群等位酶水平的分析[J].大豆科学,1996,15(4):302-309.
[20] 周晓馥,庄炳昌,王玉民,等.利用RAPD与SSR技术进行野生大豆种群内分化的研究[J].中国生态农业学报,2002,10(4):6-9.
[21] 许东河,高忠,盖钧镒,等.中国野生大豆与栽培大豆等位酶、RFLP和DRPD标记的遗传多样性与演化趋势分析[J].中国农业科学,1999,32(6):16-22.
[22] 郑翠明,常汝镇,邱丽娟,等.利用RAPD技术分析野生大豆×栽培大豆后代品系遗传组成[J].大豆科学,2000,19(2):97-104.
[23] 丁艳来,赵团结,盖钧镒.中国野生大豆的遗传多样性和生态特异性分析[J].生物多样性,2008,16(2):133-142.
[24] 范虎,文自翔,王春娥,等.中国野生大豆群体农艺加工性状与SSR关联分析和特异材料的遗传构成[J].作物学报,2013,39(5):775-788.
[25] 文自翔,赵团结,郑永战,等.中国栽培和野生大豆农艺及品质性状与 SSR标记的关联分析Ⅱ.优异等位变异的发掘[J].作物学报,2008,34(8):1 339-1 349.
[26] 文自翔,赵团结,郑永战,等.中国栽培和野生大豆农艺品质性状与SSR标记的关联分析Ⅰ.群体结构及关联标记[J].作物学报,2008,34(7):1 169-1 178.
[27] 范虎,赵团结,丁艳来,等.中国野生大豆群体特征和地理分化的遗传分析[J].中国农业科学,2012,45(3):414-425.
[28] 关荣霞,常汝镇,邱丽娟.用于SSR分析的大豆DNA的快速提取[J].大豆科学,2003,21(1):73-74.
[29] ORF J H, CHASE K, JARVIK T, et al. Genetics of soybean agronomic traits Ⅰ. Comparison of three related recombinant inbred populations[J]. Crop Sci, 1999,39(6):1 642-1 651.
[30] YUAN J, NJITI V N, MEKSEM K, et al. Quantitative trait loci in two soybean recombinant inbred line populations segregating for yield and disease resistance[J]. Crop Sci, 2002,42:271-277.
[31] PANTHEE D R, PANTALONE V R, WEST D R, et al. Quantitative trait loci for seed protein and oil concentration and seed size in soybean[J]. Crop Sci, 2005,45:2 015-2 022.
[32] CHAPMAN A, PANTALONE V R, USTUN A, et al. Quantitative trait loci for agronomic and seed quality traits in an F2 and F4:6 soybean population[J]. Euphytica, 2003,129(3):387-393.
[33] ADLER F R, CHASE K, LARK K, et al. Genetics of soybean agronomic traits: Ⅱ. Interactions between yield quantitative trait loci in soybean[J]. Crop Sci, 1999,39(6):1 652-1 657.
[34] 张军,赵团结,盖钧锰.大豆育成品种农艺性状QTL与SSR标记的关联分析.作物学报,2008,34(12):2 059-2 069.
[35] CSANADI G, VOLLMANN J, STIFT G, et al. Seed quality QTLs identified in a molecular map of early maturing soybean[J]. Theoretical and Applied Genetics, 2001,103(6):912-919.
[36] 郑永战,盖钧镒,卢为国,等.大豆脂肪及脂肪酸组分含量的QTL定位[J].作物学报,2006,32(12):1 823-1 830.
[37] BAIANU I C, DIERS B W, FEHR W R, et al. Putative alleles for increased yield from soybean plant introduction[J]. Crop Sci, 2004,44:784-791.
[38] 杨胜先,牛远,李梦,等.栽培大豆农艺性状的关联分析及优异等位变异挖掘[J].中国农业科学,2014,47(20):3 941-3 952.
[39] LI Y H, SMULDERS M J M, CHANG R Z, et al. Genetic diversity and association mapping in a collection of selected Chinese soybean accessions based on SSR marker analysis[J]. Conservation Genetics, 2011(12):1 145-1 157.
[40] HYTEN D L, PANTALONE V R, SAMS C E, et al. Seed quality QTL in a prominent soybean population[J]. Theor Appl Genet, 2004,109(3):552-561.
[41] 高利芳,郭勇,郝再彬,等.大豆株高QTL的“整合”及Overview分析[J].遗传,2013,35(2):215-224.
[42] 张闻博,蒋洪蔚,李灿东,等.基于元分析的大豆胞囊线虫抗性QTL的整合[J].中国油料作物学报,2010,32(1):104-109.
[43] CONCIBIDO V C, DIERS B W, ARELLI P R. A decade of QTL mapping for cyst nematode resistance in soybean[J]. Crop Sci, 2004,44(4):1 121-1 131.
[44] GUO B, SLEPER D A, ARELLI P R, et al. Identification of QTLs associated with resistance to soybean cyst nematode races 2, 3 and 5 in soybean PI 90763[J]. Theor Appl Genet, 2005,111(5):965-971.
[45] SPECHT J E, CHASE K, MACRANDE M, et al. Soybean response to water: A QTL analysis of drought tolerance[J]. Crop Sci, 2001,41:493-509.
[46] SJAKSTE T G, RASHAL I, RODER M S. Ineritance of microsatellite alleles in pedigrees of Latvian barley varieties and related European ancestors[J]. Theor Appl Genet, 2003,106(3):539-549.
(责任编辑:吴显达)
Molecular confirmation of genetic contribution of wild soybean to Chinese soybean cultivars
LÜ Zhuzhang
(Rizhao Polytechnic College, Rizhao, Shandong 276826, China)
SSR markers were used to identify the genetic contribution of wild soybean to 10 soybean cultivars. The results show that wild soybean contains a few specific alleles, with 17.27% utilization rate. Backcross can reduce the utilization rate of specific alleles of wild soybean, and 19 specific alleles, associated with pod size and number, stearic acid level and anti SCN, which located in 16 loci, can be easily utilized by genetic methods. Ten soybean cultivars produced 18 new alleles from 13 loci. In conclusion, improvement and innovation of soybean is promising based on wild soybean. Genetic contribution of different hybrids upon bred cultivars varied significantly. Desirable traits, including small grain, multi pod, high stearic acid level and resistance to cyst nematode are accessible to breeding and hybridization.
wild soybean; cultivar; SSR; allelic variation; genetic contribution
2016-12-02
2017-02-22
国家自然科学基金(30490250);国家自然科学基金(30671310)
吕祝章(1964-),男,教授,博士.研究方向:油料作物遗传育种及分子生物学.Email:ytzhuzhang@163.com.
S565.1
A
1671-5470(2017)04-0379-08
10.13323/j.cnki.j.fafu(nat.sci.).2017.04.003
