基于RNA-seq数据大规模挖掘蜜蜂球囊菌的SSR分子标记
2017-07-18李汶东熊翠玲王鸿权侯志贤童新宇付中民郑燕珍陈大福
李汶东, 熊翠玲, 王鸿权, 侯志贤, 童新宇, 张 璐, 付中民, 郑燕珍, 陈大福, 郭 睿
(福建农林大学蜂学学院,福建 福州 350002)
基于RNA-seq数据大规模挖掘蜜蜂球囊菌的SSR分子标记
李汶东, 熊翠玲, 王鸿权, 侯志贤, 童新宇, 张 璐, 付中民, 郑燕珍, 陈大福, 郭 睿
(福建农林大学蜂学学院,福建 福州 350002)
基于已获得的球囊菌转录组数据预测微卫星标记(SSR),并进行SSR位点的信息分析和SSR引物的挖掘.利用软件MISA对球囊菌转录组中42610条unigenes进行搜索,共预测出7968个SSRs,分布于5233条unigenes中,其中最主要的重复类型为三核苷酸重复(53.15%),其次为二核苷酸重复(32.32%)和四核苷酸重复(8.46%).二核苷酸重复中的基序主要是AG/CT(占总量的15.8%).针对所有的SSR位点,利用Primer Premier 5软件设计出6956对SSR特异性引物,随机选取20对引物对两个不同来源的球囊菌样品进行SSR位点扩增,共有6对引物成功扩增出符合预期的目的片段.研究结果表明,利用球囊菌转录组数据开发SSR引物是可行的.
蜜蜂球囊菌; 微卫星标记; 转录组; RNA-seq
微卫星(simple sequence repeat, SSR)是以1~6个核苷酸碱基为重复单元(motif)组成的简单串联重复序列.与同类分子标记技术相比,SSR具有多态性高、呈共显性遗传、重复性好、实验操作容易等特点[1],在动植物及微生物等学科领域得到了广泛应用,但是SSR的特异性一定程度上限制了其引物的通用性[2].对于基因序列未知的物种而言,SSR的开发较为困难.传统SSR开发以基因文库构建法(包括SSR富集文库)为主[3-4],不仅试验过程繁杂,而且效率低下.SSR还可以利用公共基因数据库(NCBI, EMBL, DDBJ)中的共享序列来发掘,然而对于模式生物或新物种,有限的基因序列信息严重制约SSR的开发.2005年以来,第二代高通量测序技术发展迅猛,为规模化遗传变异检测[5]和标记位点开发[6-8]带来了新机遇.如何利用高通量测序数据高效、快速地挖掘SSR位点,是当前分子遗传学领域研究的热点之一.
蜜蜂球囊菌(Ascosphaeraapis)简称球囊菌,是一种特异性侵染蜜蜂幼虫的致死性真菌病原.白垩病的发病时期一般为春季和初夏,虽然不对蜂群造成毁灭性打击,但却能造成成年蜜蜂数量及蜂群群势的大幅下降,从而严重影响蜂蜜等产品的产量[9-10],据报道,白垩病可造成蜂蜜产量下降5%~37%[11].目前尚无抗真菌药物被批准用于养蜂生产,一些替代的防治方法,如饲养管理[12-13]等,虽取得一定效果,但总体并不理想.已鉴定的球囊菌SSRs极少,严重阻碍球囊菌的分子遗传学研究.前期研究中,本课题组已成功组装并注释了球囊菌的参考转录组(未发表数据),可为球囊菌的分子研究提供依据,也为球囊菌的SSR位点挖掘提供宝贵资源.本研究基于已获得的球囊菌转录组数据,利用MISA软件进行大规模的SSR位点预测,并设计特异性SSR引物对不同来源的球囊菌样本进行SSR位点扩增.
1 材料与方法
1.1 球囊菌转录组数据
球囊菌转录组数据委托广州基迪奥生物科技有限公司完成,测序平台为Illumina HiSeq 2500,采用双端(PE125)测序,RNA-seq数据共组装得到42610条unigenes.前期获得的球囊菌转录组数据已上传美国国家生物技术信息中心(NCBI)SRA数据库(SRA464366).
1.2 供试球囊菌样品
本研究中用于验证SSR引物的球囊菌样品来源于福建福州和浙江苍山的白垩病幼虫尸体.从上述虫尸上刮取部分菌丝和孢子,放入研钵,加入适量无菌水充分研磨,将研磨液转移至干净的EP管,利用真菌基因组DNA提取试剂盒(TIANamp Genomic,中国)抽提总DNA,作为PCR模板,-20 ℃保存备用.
1.3 SSR鉴定
利用软件MISA(http://pgrc.ipk-gatersleben.de/misa/)搜索unigenes的微卫星标记,按照以下标准从unigenes中查找SSR位点:二核苷酸重复≥6次,三核苷酸重复≥5次,四核苷酸重复≥5次,五核苷酸重复≥5次和六核苷酸重复≥5次.
1.4 SSR引物设计与验证
根据MISA的输出结果,利用Primer Premier 5(PREMIER Biosofe Int., Palo Alto, CA)对每个含有16 bp碱基重复的SSR设计引物.目标扩增片段设置为必须包含SSR起始-3 bp,终止+6 bp,扩增片段80~300 bp.引物长度设置为18~25 bp,最适长度为22 bp,引物最大允许有1个不能识别的碱基;引物的退火温度(Tm)设置为55~65 ℃,最适Tm为58 ℃,上下游引物间的Tm差异最大允许3 ℃,引物末端稳定性最大为250.引物由上海生工生物工程有限公司合成.为了验证所设计的引物能否稳定扩增,从批量设计出的引物中随机挑选20对引物进行验证.PCR反应体系为20 μL:上述DNA模板1 μL,Mixture 10 μL,上下游引物(10 μmol·L-1)各1 μL,无菌水补足20 μL.反应程序:94 ℃预变性5 min;94 ℃变性50 s,55 ℃退火30 s,72 ℃延伸30 s,共33个循环;72 ℃延伸10 min,4 ℃保存.PCR产物用4%琼脂糖凝胶电泳检测.
2 结果与分析
2.1 球囊菌转录组中SSR位点的分布特点
利用软件MISA对球囊菌转录组42 610条unigenes的数据进行搜索,共找到7 968个SSR位点,分布于5 233条unigenes,发生频率(含有SSR的unigene数量与总unigene数量之比)为12.28%.含1个以上SSR位点的unigene序列有1 615条,以复合型形式存在的SSR序列数目1 012条,SSR的分布频率(SSR个数与总unigene数量比)为18.70%.这些SSR基序包含1~5 bp的串联重复序列.在球囊菌转录组数据中,SSR基序的重复类型占SSR总数最多的是三核苷酸重复,达53.15%;其次是二核苷酸重复与四核苷酸重复,分别占32.32%和8.46%;五核苷酸和六核苷酸重复的含量相对较少,分别占SSR总数的3.31%和2.76%(表1).球囊菌转录组SSR重复单元的重复次数分布在4~30次之间.二核苷酸重复次数介于6~30次,三核苷酸重复次数介于5~30次,四、五和六核苷酸重复次数最多分布在4次.其中重复次数最多为5次,占30.40%.

表1 基于重复单元数目中SSRs在蜜蜂球囊菌转录组中的出现频率
根据不同SSR基序出现频率分析(图1),二核苷酸重复中主要是AG/CT基序,占总量的15.8%,其它基序百分比超过2%的有AC/GT、AT/AT、AAC/GTT、AAG/CTT、AAT/ATT、ACC/GGT、ACG/CGT、AGC/CTG、AGG/CCT、ATC/ATG和CCG/CGG,其比例分别为4.1%、11.9%、5.1%、7.3%、3.3%、4.6%、5.6%、11.6%、6.4%、6.1%和2.3%,三核苷酸中ACT/AGT基序最少,占SSR总数的0.8%.
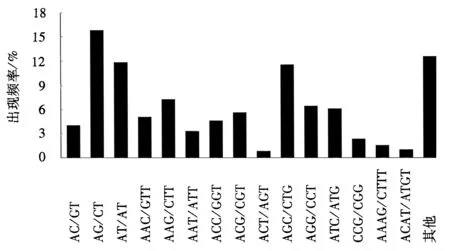
图1 基于基序类型中SSRs在蜜蜂球囊菌转录组中的出现频率
2.2 球囊菌SSR引物设计
基于筛选的SSRs,运用Primer Premier 5软件进行引物的批量设计,按照已设置好的参数,共对6956个unigenes设计出15963对SSR引物.随机挑选其中的20对引物对来源于福建福州与浙江苍山的白垩病幼虫样本DNA进行SSR位点扩增(表2),电泳结果显示共有6对引物可成功扩增,且扩增片段符合预期(图2),有效扩增率达到30%,说明基于RNA-seq数据开发SSR引物的方法可行,这些SSR位点多态性的丰富度有待于进一步的试验评估.

表2 SSR引物的特征
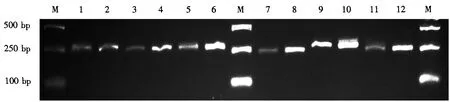
M:DNA marker;泳道1、3、5、7、9、11:来源于福建福州的球囊菌样品;泳道2、4、6、8、10、12:来源于浙江苍山的球囊菌样品;泳道1和2使用的是SSR21引物;泳道3和4使用的是SSR22引物;泳道5和6使用的是SSR35引物;泳道7和8使用的是SSR36引物;泳道9和10使用的是SSR37引物;泳道11和12使用的是SSR38引物.
3 讨论
此前,本课题组利用RNA-seq技术对球囊菌进行深度测序,成功组装并注释其参考转录组.在此基础上,本研究利用生物信息学方法从球囊菌的转录组数据中预测出多达7968个SSR位点,这些SSR位点种类丰富,以三核苷酸重复类型为主,其次为二核苷酸重复.目前,有关球囊菌的微卫星标记的研究报道几乎没有,其它近缘真菌微卫星标记的研究报道也较少.Qu et at[14]基于糙皮侧耳(Pleurotusostreatus)基因组预测了2114个SSRs并对其进行了功能富集,发现外显子中的三核苷酸重复类型SSR最为丰富,与本研究结果一致.Catalina et al[15]基于单轴霉(Plasmoparaobducens)的基因组开发了37个SSR分子标记.Liu et al[16]基于金针菇(Flammulinavelutipes)的基因组预测出1321个SSRs,从中挑取115个SSRs对124个F.velutipes品种进行PCR扩增,有109个可以扩增出目的片段,其中有25个SSR位点具有多态性.
在一些昆虫如黄粉虫(Tenebriomolitor)[17]、扶桑绵粉蚧(Phenacoccussolenopsis)[18]和沙葱萤叶甲(Galerucadaurica)[19]中,基于转录组数据挖掘SSR位点的研究结果显示,单核苷酸重复是最主要的重复类型,其次为三核苷酸重复.在本课题组的另一研究中,基于中华蜜蜂(Apisceranacerana)幼虫肠道转录组数据开发SSR分子标记的研究结果显示,SSR位点种类以二核苷酸重复类型为主,其次为三核苷酸重复.在表达序列标签(ESTs)中,多数情况下单核苷酸重复最多,其次为三核苷酸重复.对于昆虫,三核苷酸重复是EST-SSR中优势的核心重复类型,因为三核苷酸核心基元在编码区较其它重复基元类型更加稳定,极少产生编码框滑动突变现象[20].动物和植物的转录组或基因组中,二碱基重复的SSR中GC/CG是非常稀有的重复基元.本研究发现球囊菌转录组中二核苷酸重复的SSR虽然占比达到32.32%,但未发现GC/CG重复单元存在,这与动植物转录组或基因组中的情况相似.
本研究中,为了验证MISA软件预测出的球囊菌的SSR引物,随机挑选20对SSR引物,对国内两个不同来源的球囊菌样品SSR位点进行PCR扩增,结果显示有6对引物可有效扩增出SSR位点,有效扩增率达30%,说明基于RNA-seq数据预测出的SSR位点只有部分真实存在,然而,通过此法开发出的可用于多态性分析的SSR位点数目仍非常可观.本研究只针对两个来源的球囊菌样本进行验证,样本数量较少,下一步将继续扩大采样区域并采集更多的球囊菌样本,以获得不同地域的球囊菌在遗传分类学上的依据.本研究采用的是琼脂糖凝胶电泳法,虽然此法较聚丙烯酰胺凝胶电泳(PAGE)法分辨率低,但研究结果表明部分SSR位点真实存在,这些SSR位点多态性的确定有待于进一步研究.未来的工作重点是进一步利用已得到的球囊菌转录组数据挖掘更多的SSR位点,并采用分辨率更高的PAGE法和荧光标记的毛细管电泳法对已初步鉴定的SSR位点进行多态性验证.
开发SSR分子标记的传统试验方法繁琐且效率低下,以RNA-seq为代表的二代测序技术解决了传统微卫星标记开发的瓶颈问题,使微卫星标记的大规模开发成为现实.球囊菌是非模式生物,其基因组序列信息早在2006年就已公布[21],但其基因功能注释信息直到2016年才公布.本研究说明基于高通量测序数据开发非模式生物的SSR分子标记是一种快速、高效的途径.目前,以单分子测序技术为代表的三代测序技术逐渐兴起,有望使SSR分子标记开发及功能基因定位变得更为方便和快捷.
[1] JARNE P, LAGODA P J L. Microsatellites, from molecules to populations and back[J]. Trends in Ecology and Evolution, 1996,11(10):424-429.
[2] GLENN T C, SCHABLE N A. Isolating microsatellite DNA loci[J]. Methods in Enzymology, 2005,395:202-222.
[3] HAMILTON M B, PINCUS E L, DI-FIORE A, et al. Universal linker and ligation procedures for construction of genomic DNA libraries enriched for microsatellites[J]. Biotechniques, 1999,27(3):500-507.
[4] ZANE L, BARGELLONI L, PATARNELLO T. Strategies for microsatellite isolation: A review[J]. Molecular Ecology, 2002,11(1):1-16.
[5] O′ NEILL E M, SCHWARTZ R, BULLOCK C T, et al. Parallel tagged amplicon sequencing reveals major lineages and phylogenetic structure in the North American tiger salamander (Ambystomatigrinum) species complex[J]. Molecular Ecology, 2013,22(1):111-129.
[6] DAVEY J W, HOHENLOHE P A, ETTER P D, et al. Genome-wide genetic marker discovery and genotyping using next-generation sequencing[J]. Nature Reviews Genetics, 2011,12(7):499-510.
[7] YU J N, WON C, JUN J, et al. Fast and cost-effective mining of microsatellite markers using NGS technology: an example of a Korean water deerHydropotesinermisargyropus[J]. PLoS One, 2011,6(11):e26933.
[8] PANDEY G, MISRA G, KUMARI K, et al. Genome-wide development and use of microsatellite markers for large-scale genotyping applications in foxtail millet [Setariaitalica(L.)][J]. DNA Research, 2013,20(2):197-207.
[9] Bailey L, Ball B V. Honey Bee Pathology[M]. San Diego: Academic Press Inc, 1991:13.
[10] WOOD M. Microbes help bees battle chalkbrood[J]. Agricultural Research Washington D.C, 1998,46(8):16-17.
[11] ZAGHLOUL O A, MOURAD A K, EL K M B, et al. Assessment of losses in honey yield due to the chalkbrood disease, with reference to the determination of its economic injury levels in Egypt[J]. Communications in Agricultural & Applied Biological Sciences, 2005,70(4):703-714.
[12] ANDERSON D L, GIACON H, GIBSON N. Detection and thermal destruction of the chalkbrood fungus (Ascosphaeraapis) in honey[J]. Journal of Apicultural Research, 1997,36(3-4):163-168.
[13] BAGGIO A, GALLINA A, DAINESE N, et al. Gamma radiation: a sanitating treatment of AFB-contaminated beekeeping equipment[J]. Apiacta, 2005,40:22-27.
[14] QU J, HUANG C, ZHANG J. Genome-wide functional analysis of SSR for an edible mushroomPleurotusostreatus[J]. Gene, 2016, 575(2): 524-530.
[15] CATALINA S S, YAZMIN R, DANIEL V, et al. Polymorphic SSR markers forPlasmoparaobducens(Peronosporaceae), the newly emergent downy mildew pathogen of impatiens (Balsaminaceae)[J]. Applications in Plant Sciences, 2015,3(11):1500073.
[16] LIU X B, FENG B, LI J, et al. Genetic diversity and breeding history of Winter Mushroom (Flammulinavelutipes) in China uncovered by genomic SSR markers[J]. Gene, 2016,591:227-235.
[17] 朱家颖,吴国星,杨斌.基于转录组数据高通量发掘黄粉甲微卫星引物[J].昆虫学报,2013,56(7):724-728.
[18] 罗梅,张鹤,宾淑英,等.基于转录组数据高通量发掘扶桑绵粉蚧微卫星引物[J].昆虫学报,2014,57(4):395-400.
[19] 张鹏飞,周晓榕,庞保平,等.基于转录组数据高通量发掘沙葱萤叶甲微卫星引物[J].应用昆虫学报,2016,53(5):1 058-1 064.
[20] WANG B, EKBLOM R, CASTOE T A, et al. Transcriptome sequencing of black grouse (Tetraotetrix) for immune gene discovery and microsatellite development[J]. Open Biology, 2012,2(4):120 054.
[21] QIN X, EVANS J D, ARONSTEIN K A, et al. Genome sequences of the honey bee pathogensPaenibacilluslarvae, andAscosphaeraapis[J]. Insect Biochemistry and Molecular biology, 2006,15(5):715-718.
(责任编辑:吴显达)
Large scale development of SSR molecular markers ofAscosphaeraapisbased on RNA-seq data
LI Wendong, XIONG Cuiling, WANG Hongquan, HOU Zhixian, TONG Xinyu, ZHANG Lu,FU Zhongmin, ZHENG Yanzhen, CHEN Dafu, GUO Rui
(College of Bee Science, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China)
To illustrate the molecular genetics and functional genomics ofAscosphaeraapis, simple sequence markers (SSRs) were predicted based on previous transcriptome data ofA.apis, and SSR loci were analyzed and the specific SSR primers were developed. Firstly, 42610 unigenes assembled from RNA-seq data were interpreted by MISA software, and 7968 SSR loci were detected, they were distributed in 5233 unigenes. Among these SSR loci, tri-nucleotide repeats were the dominant repeat type, accounting for 53.15% of the total, which was followed by di-nucleotide repeats (32.32%) and tetra-nucleotide repeats (8.46%). The AG/CT motif was the majority (15.8%) in di-nucleotide repeats. In total, 6956 pairs of primers were designed from all SSRs. Among them, 20 SSR primer pairs were randomly selected and used to amplify the SSR loci inA.apissamples from 2 regions in China. It turned out that 6 pairs of primers were able to amplify the target fragments with expected sizes, indicating that large-scale development ofA.apisSSR primers on the basis of transcriptome data is feasible.
Ascosphaeraapis; microsatellite; transcriptome, RNA-seq
2017-01-03
2017-02-20
现代农业产业技术体系建设专项资金(CARS-45-KXJ7);福建农林大学科技发展资金(KF2015123);国家自然科学基金(30800806).
李汶东(1996-),男.研究方向:蜜蜂分子生物学.Email:wendongli22@163.com.通讯作者郭睿(1987-),男,博士,讲师.研究方向:蜜蜂分子生物学.Email:ruiguo@fafu.edu.cn.
S895.1+3
A
1671-5470(2017)04-0434-05
10.13323/j.cnki.j.fafu(nat.sci.).2017.04.013
