福建柏优树子代ISSR遗传多样性与指纹图谱
2017-07-18陈剑成沈少炎何天友荣俊冬郑郁善
万 娟, 罗 睿, 陈剑成, 沈少炎, 何天友, 荣俊冬, 郑郁善,
(1.福建农林大学林学院;2.福建农林大学园林学院,福建 福州 350002)
福建柏优树子代ISSR遗传多样性与指纹图谱
万 娟1, 罗 睿1, 陈剑成1, 沈少炎1, 何天友2, 荣俊冬1, 郑郁善1,2
(1.福建农林大学林学院;2.福建农林大学园林学院,福建 福州 350002)
采用ISSR分子标记技术对24个福建柏家系的遗传多样性进行分析.结果表明:16条引物共扩增出215个位点和190个多态性位点,多态位点百分率达86.74%,表现出较好的多态性,福建柏各家系间存在较大的遗传变异,且ISSR标记能有效揭示家系间的多态性;家系间遗传相似系数为0.58~0.87,遗传距离为0.13~0.50,表现出丰富的遗传多样性;以遗传距离L1为界,将24个福建柏优树子代划分为3类,不存在明显的地域性规律;仅用UBC841就能完全鉴别出所有待测家系.表明建立的福建柏DNA指纹图谱ISSR 分子标记技术可有效地用于福建柏家系的资源评价和指纹图谱构建.
福建柏; ISSR分子标记; 遗传多样性; 指纹图谱; 聚类分析
福建柏(Fokieniahodginsii)又名建柏、滇柏等,属柏科福建柏属,是中国特有的单属种植物,已被列为国家二级保护植物[1-3].福建柏不仅具有良好的生态效应,更因其具有良好的木材物理力学性质,在木材加工中得到广泛应用,是我国珍贵的用材树种[4-8].同时福建柏在药用[5]和园林观赏[6]方面都存在一定的研究价值.国内对福建柏的研究较多,但大多集中于福建柏的种实表型变异、苗期选择和子代林测定等方面[7-8],但植物表型以及生理生化性状受环境条件影响显著,难以真实反映植物的遗传变异状况.而采用分子标记手段可以更加准确地对植物遗传多样性进行研究.
遗传多样性水平可以反映出植物在其生长环境中基因的丰富程度,也决定其对于环境变化的适应能力,使用合适的分子标记方法对研究植物种质资源的遗传多样性及亲缘关系具有重要意义[9].因ISSR分子标记方法克服了RAPD、AFLP和SSR分子标记技术的局限性,并表现出更高的多态性、重复性和可靠性,已被广泛应用于植物研究[10-13],但在柏科植物中应用研究报道不多.叶睿超等[14]利用ISSR分子标记技术标记了65个垂枝香柏的个体,并分析其遗传多样性;齐明[15]为了鉴定杉木与侧柏远交杂种的真实性,采用ISSR分子标记技术对其杂交后代进行鉴定,并检测到了多条父系特征谱带;李单琦等[16]对观赏性福建柏的遗传多样性进行ISSR分析;张金玉等[17]利用ISSR分子标记研究了115个无性系福建柏之间的亲缘关系.有关福建柏优树子代实生苗的分子标记研究目前尚未见报道.本试验利用ISSR分子标记技术对24个福建柏家系的实生苗进行遗传多样性分析,构建DNA指纹图谱,以期为福建柏资源的合理开发利用与福建柏种质资源的良种选育工作提供依据.
1 材料与方法
1.1 供试材料
24个福建柏优树子代材料来源于广西、福建等4个省11个地区(表1),2013年10—11月从各地林场预选出的优树上采回球果,经过发芽处理后,于2015年2月下旬在福建安溪白濑国有林场苗圃地中育苗[18];2015年12月下旬在每个福建柏家系中选取3~5株长势适中的植株,低温保存后速带回实验室液氮冷冻,并于-80 ℃冰箱中保存,以便进行DNA提取.
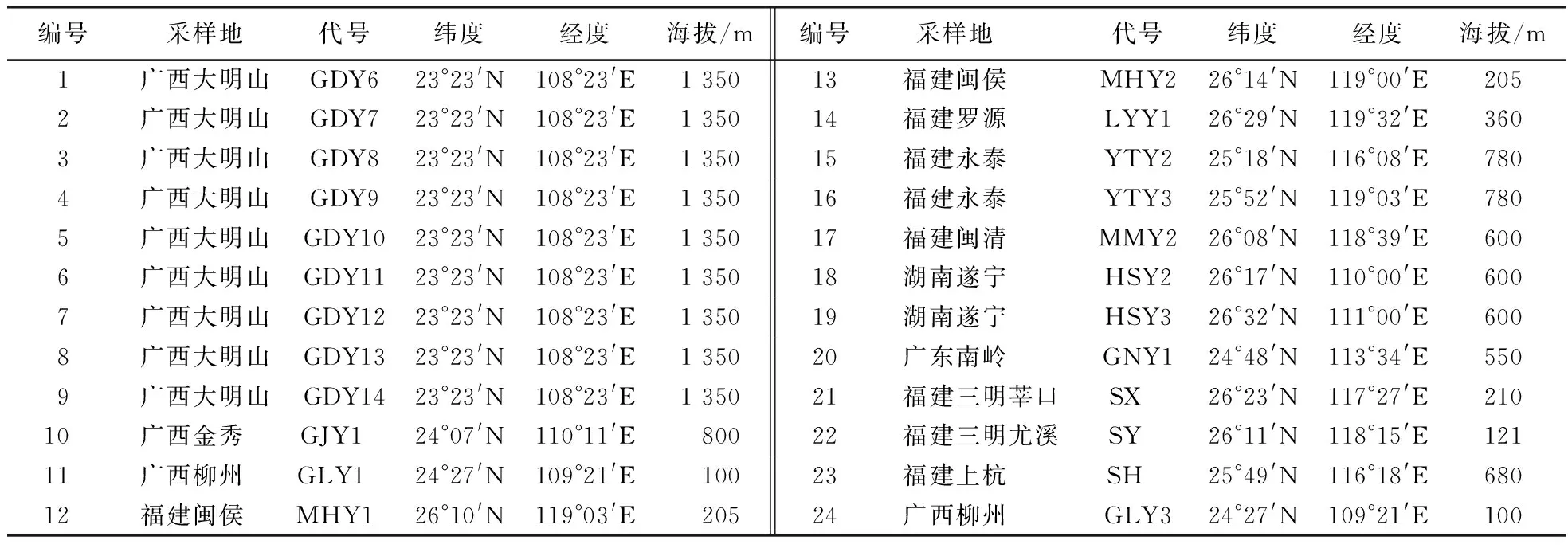
表1 地理气象因子
1.2 试验方法
1.2.1 DNA提取 参照文献[19]中基因组DNA提取方法.取0.2 g福建柏叶片,加入适量PVP,于液氮中研磨成粉,转入已于65 ℃水浴锅中预热过的650 μL的2×CTAB提取缓冲液(其中已添加β-巯基乙醇);65 ℃水浴,每隔5~10 min振荡摇匀1次,45 min后取出于室温放置5 min;按1∶1比例加入氯仿、异戊醇(24∶1)混合液,关紧盖子,翻转混匀至混合液呈乳液状,15 000g离心15 min;取上清液转入新的离心管中并重复上一步骤;取上清液转入新的离心管中,加入0.8~1倍体积的异丙醇(-20 ℃)与1/10倍体积的NaAc,充分混匀后-20 ℃静置1 h;去废液,用75%(体积分数)乙醇漂洗沉淀2~3次;干燥,DNA沉淀;加入100~200 μL TE缓冲液充分溶解沉淀(4 ℃);加入适量RNase A充分混匀后分装保存(4 ℃),并取3 μL样品于1.0%(质量分数)琼脂糖凝胶上电泳检测.
1.2.2 ISSR-PCR反应体系的建立 采用李单琦等[6,16]建立的PCR反应体系与扩增程序,对24个福建柏优树子代的DNA进行扩增,配制1.5 g·mL-1的琼脂糖凝胶,并加入适量Gold View核酸染料检测扩增产物,通过全自动数码凝胶成像分析仪观察扩增结果并拍照.
1.3 ISSR扩增引物的筛选
从哥伦比亚大学公布的100条通用引物中筛选出条带清晰、不模糊、不弥散、质量好的条带引物,对24个福建柏优树子代的DNA模板进行扩增,并检验其有效性和反应体系.
1.4 数据处理
对各家系的DNA模板进行ISSR-PCR扩增后,观察电泳图上是否有条带.ISSR分子标记扩增谱带多为显性表现,用“1”和“0”分别表示“有”和“无”,记录清晰、重复性好的ISSR扩增条带;对每一引物扩增结果构建二元数据矩阵,统计总位点数和多态位点数.采用POPGENE 32和DPS 7.05软件对此数据矩阵进行分析,根据遗传距离采用非加权配对算数平均法进行聚类分析,绘制树状图.
2 结果与分析
2.1 DNA检测结果
DNA电泳检测结果表明,基本无拖带或拖带不明显(图1).采用紫外分光光度计(德国赛默飞NANODROP 2000C)检测到的所有DNA的光密度(D260 nm和D280 nm)均为1.8~2.0,说明提取的DNA纯度较高,符合要求.

图1 24份福建柏样本DNA电泳图
2.2 不同引物的ISSR标记多态性
筛选出的16条扩增效果较好的引物见表2.多态位点百分率(PPB)表示遗传变异水平,反映被测植物的环境适应能力,PPB值高表明该植物的环境适应性强,多态性丰富[20].从表2可看出:16条引物在所有供试样品DNA中共扩增出215个位点、190个多态性位点,PPB为88.37% ;每个引物平均扩增13.44个位点,多态位点有11.88个,各引物的PPB均为57%~100%.引物扩增结果如图2、3所示,扩增的DNA片段分子质量为250~2 000 bp.结果表明福建柏各家系间有较大的遗传变异,且ISSR标记能有效揭示材料间的多态性.

表2 福建柏ISSR分析引物
2.3 各福建柏优树子代遗传相似系数
Nei相似系数是用来比较群体或个体间相似程度的度量参数,平均相似系数越高,说明相似程度越大,遗传背景一致性越强[21].本试验利用POPGENE 32对24份福建柏优树子代的供试材料进行遗传相似系数分析(表3),结果表明24个福建柏家系之间的相似系数为0.58~0.87,平均值为0.72.其中来自广西大明山的GDY7与GDY8的相似度最高,相似系数为0.87,亲缘关系最近;来自广西大明山的GDY9与YTY2的相似度最低,相似系数为0.58,亲缘关系最远.表明24份福建柏优树子代的相似系数均在0.58以上,且不存在明显的地域趋势.

图2 引物UBC841的福建柏扩增图谱
图3 引物UBC855的福建柏扩增图谱

ID2345678910111213141516171819202122232410.840.800.730.790.730.760.730.740.680.770.770.760.730.690.750.800.740.680.730.720.730.750.7820.870.710.750.760.780.730.790.650.810.720.800.780.690.730.800.750.690.750.750.790.790.8130.700.740.720.700.730.760.670.790.740.790.740.680.750.790.690.670.730.690.790.750.7740.670.690.650.650.710.710.710.710.710.630.580.660.710.620.690.640.660.680.700.6850.800.840.730.780.620.800.750.730.740.740.750.780.750.670.800.700.780.770.8060.820.730.810.640.790.740.740.740.730.760.740.690.680.760.750.790.800.7870.730.780.640.800.730.740.720.780.740.730.790.640.790.760.770.770.8080.800.660.770.730.690.740.760.750.770.750.630.770.720.750.750.8090.670.790.780.780.780.710.790.810.730.700.780.740.790.800.78100.650.650.690.610.630.660.680.660.700.650.680.700.660.66110.790.780.800.730.740.800.750.710.810.730.820.840.84120.810.780.690.820.830.690.730.750.740.810.780.78130.790.770.800.820.740.690.770.730.770.760.79140.740.790.810.720.680.760.700.780.790.79150.750.740.760.600.750.710.730.740.75160.840.720.700.790.730.760.770.79170.760.770.800.770.810.820.80180.640.770.750.730.760.80190.710.670.730.730.70200.770.800.850.81210.750.770.75220.840.82230.85
2.4 聚类分析
基于DPS 7.05和Nei遗传距离采用UPGMA法对24个福建柏家系进行聚类分析,绘制聚类分支树状图(图4).结果(图4)表明,利用ISSR标记能将24个福建柏家系完全区分开.以L1为界可将供试材料分为3类:第1类包含了21个家系;第2类包含来自广西的GDY9和GJY1;来自湖南绥宁的HSY2单独聚为一类.来自广西金秀的子代材料与来自广西大明山的GDY9同聚为一类;来自湖南绥宁的HSY2单独聚为一类,而与之来自同一地区的HSY3却与其余20个家系聚为一类.说明聚类受材料地理来源的影响较小.

图4 24个福建柏优树子代UPGMA聚类图
2.5 指纹图谱的构建
由表4可知16条引物对24个福建柏家系的鉴别情况,其中,仅引物UBC841就能将所有被测家系鉴别出来,表现出良好的鉴别能力;其他引物通过不同组合也可将所有被测家系鉴别出来,例如引物UBC840、UBC811分别与UBC836(UBC873)和UBC808(UBC855)组合.

表4 各引物的鉴别样本
3 小结与讨论
本研究结果表明福建柏优树子代存在较为丰富的遗传多样性,这与李单琦等[16,17]对福建柏种群的研究结果相似.福建柏属于我国特有的濒危单属种植物,主要分布于我国长江以南地区,少量分布于越南北部地区,分布区较小.一般认为物种遗传多样性与其地理分布区域呈显著正相关,许多我国所特有的濒危植物遗传多样性都较低[22].福建柏作为华夏植物区系的孑遗种,可能受第四纪冰川时期的影响较弱,在中国南部地区得以存留,幸存个体可能保留了其先祖丰富的遗传基础[23].
对福建柏聚类的分析结果表明其受地理因子影响较小,这可能是由于试验采用的是福建柏1年实生苗作为试验对象,幼龄实生苗遗传不稳定,或者受到某些试验尚未考虑到的因素的影响较大.
本研究所构建的指纹图谱中引物UBC841能将24个福建柏优树子代全部鉴定出来,鉴定能力最强;而其他引物如UBC840、UBC811需要分别与UBC836(UBC873)和UBC808(UBC855)组合,鉴别能力次之.李单琦等[16]所构建的指纹图谱需要2个或2个以上的引物组合才能将参试样品全部鉴定.说明目前仅利用ISSR分子标记技术就可以绘制福建柏的指纹图谱,为福建柏种质资源的识别与鉴定提供较为全面的分子生物学信息[24].
[1] 中国树木志编辑委员会.中国树木志[M]. 北京:中国林业出版社,1983:344-345.
[2] 傅立国.中国植物红皮书——稀有和濒危植物(第1卷)[M].北京:科学出版社,1991:125-126.
[3] 高兆蔚.珍贵用材树种——福建柏[J].福建林业科技,1994,21(2):62- 66.
[4] 陈祖松.福建柏人工林木材物理力学性质的试验研究[J]. 福建林学院学报,1999,19(3):223-226.
[5] WU X D, HE J, LI X Y, et al. Diterpenoids from the twigs and leaves ofFokieniahodginsii.[J]. Journal of Natural Products, 2013,76(6):1 032-1 038.
[6] 李单琦.福建柏遗传多样性研究与景观型福建柏良种选择[D].福州:福建农林大学,2015.
[7] 何开跃,李晓储,黄利斌,等.福建柏抗寒生理指标变化研究(英文)[J].南京林业大学学报(自然科学版),2002(5):10-14.
[8] 郑仁华,苏顺德,赵青毅,等.福建柏种源生长性状遗传变异及种源选择[J].福建林学院学报,2014,34(3):249-254.
[9] 许先松,刘志钦,林晓丹,等.基于形态及SRAP标记的辣椒资源遗传多样性及亲缘关系比较[J].福建农林大学学报(自然科学版),2011,40(1):48-53.
[10] ZIETKIEWIEZ E, RAFSLSKI A, LABUDA D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplifi cation[J]. Genomics, 1994(20):176-183.
[11] 赵谦,杜虹,庄东红.ISSR分子标记及其在植物研究中的应用[J].分子植物育种,2007(S1):123-129.
[12] 朱岩芳,祝水金,李永平,等.ISSR分子标记技术在植物种质资源研究中的应用[J].种子,2010(2):55-59.
[13] 朱岩芳.作物品种分子标记鉴定及指纹图谱构建研究[D].杭州:浙江大学,2013.
[14] 叶睿超,陈照,董艳芳,等.冶勒自然保护区垂枝香柏的ISSR遗传多样性研究[J].四川大学学报(自然科学版),2009(4):1 207-1 212.
[15] 齐明.运用ISSR分子标记鉴定杉木×侧柏远交杂种[J].浙江林学院学报,2008,(5):666-669.
[16] 李单琦,谢德金,王汉琪,等.福建柏遗传多样性ISSR分析[J].中南林业科技大学学报,2016,36(5):63-67.
[17] 张金玉.福建柏无性系亲缘关系的分子标记鉴定[D].福州:福建师范大学,2014.
[18] 陈澜.不同种源福建柏苗期性状差异性研究及优良种源选择[D].福州:福建农林大学,2015.
[19] DOYLE J. DNA protocols for plants-CTAB total DNA isolation[C]∥HEWITT G M, JOHNSTON A. Molecular techniques in taxonomy. Germany, Berlin: Springer, 1991.
[20] 黄树军,陈礼光,肖永太,等.大明竹属遗传多样性ISSR分析及DNA指纹图谱研究[J].生态学报,2013,33(24):7 863-7 871.
[21] NEI M. Estimation of average heterozygosity and genetic distance from a small number of individual[J]. Genetics, 1978(89):583-590.
[22] XIE G W, WANG D L, YUAN Y M, et al. Population genetic structure ofMonimopetalumchinense(Celastraceae), an endangered endemic species of eastern China[J]. Ann Bot, 2005,95(5):773-777.
[23] XUE D W, GE X J, HAO G, et al. High genetic diversity in a rare, narrowly endemic primrose species:Primulainterjacensby ISSR analysis[J]. Acta Botanica Sinica, 2004,46(10):1 163-1 169.
[24] 巫桂芬,徐鲜均,徐建堂,等.利用SRAP、ISSR、SSR标记绘制黄麻基因源分子指纹图谱[J].作物学报,2015,41(3):367-377.
(责任编辑:叶济蓉)
Genetic diversity and fingerprint ofFokieniahodginsiiby ISSR
WAN Juan1, LUO Rui1, CHEN Jiancheng1, SHEN Shaoyan1, HE Tianyou2, RONG Jundong1, ZHENG Yushan1,2
(1.College of Forestry; 2.College of Landscape Architecture, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China)
The genetic diversity of 24Fokieniahodginsiiwas analyzed by ISSR markers. The results showed that 16 ISSR primers produced 215 loci among 24 families ofF.hodginsii, of which 190 were polymorphic. Polymorphism of primers is satisfactory, with percentage of polymorphic loci being 86.74%, indicating great genetic variation among the families. The genetic similarity coefficient among families ranged between 0.58 and 0.87, and genetic distance ranged between 0.13 and 0.50, confirming abundant genetic diversity among the families. The 24 families were divided into 3 groups at the genetic distance of L1, but without significant regional regularity. And DNA fingerprints can be identified by UBC841. In conclusion, ISSR molecular markers could be effectively used in genetic diversity and fingerprint analysis forF.hodginsii.
Fokieniahodginsii; ISSR mark; genetic diversity; fingerprint; cluster analysis
2017-01-03
2017-05-15
福建省林业厅资助项目(闽林科[2013]1号).
万娟(1992-),女,硕士研究生.研究方向:自然地理.Email:745065754@qq.com.通讯作者郑郁善(1960-),男,教授,博士生导师.研究方向:园林植物应用、园林规划设计及森林培育学.Email:zys1960@ 163.com.
S791.43
A
1671-5470(2017)04-0404-06
10.13323/j.cnki.j.fafu(nat.sci.).2017.04.008
