鲤饲料转化率性状的关联分析及优异等位变异挖掘
2017-07-10张晓峰李超鲁翠云曹顶臣孙效文
张晓峰,李超,鲁翠云,曹顶臣,孙效文
(中国水产科学研究院黑龙江水产研究所淡水鱼类育种国家地方联合工程实验室,黑龙江 哈尔滨 150070)
鲤饲料转化率性状的关联分析及优异等位变异挖掘
张晓峰,李超,鲁翠云,曹顶臣,孙效文
(中国水产科学研究院黑龙江水产研究所淡水鱼类育种国家地方联合工程实验室,黑龙江 哈尔滨 150070)
饲料转化率是鲤养殖重要的亟待改良的经济性状之一。本研究利用分布于鲤全基因组的1 536个SNP标记,分析68尾全同胞家系镜鲤Cyprinuscarpio的基因分型,采用TASSEL软件的GLM和MLM模型检测与饲料转化率性状紧密关联的标记位点,发掘相关位点内的优异等位变异。本研究共获得17个与饲料转化率性状显著关联(P<0.05)的SNP标记,贡献率在6.4%~16.6%之间,分布于鲤基因组第2、6、11、16、20、32、33、41,和42染色体以及Scaffold1032、1078和2323上,注释了一批饲料转化率相关的候选基因。从显著关联的标记位点内发掘了16种优异等位变异,其平均表型效应值较群体均值高6.2%,较非优势等位变异高12.5%。其中,SNP0919、SNP0167和SNP1016 3个标记的优异等位变异增减效差异最大,暗示这些标记对性状的选择效果明显。本研究发掘的饲料转化率相关的标记及优异等位变异可为鲤饲料转化率性状的分子标记辅助选择及分子设计育种提供依据。
鲤;饲料转化率性状;SNP;关联分析;优异等位变异
饲料约占我国养殖业成本的70%,是影响养殖效益的关键因素[1-3]在养殖业中占有举足轻重的地位。饲料转化率(Feed conversion efficiency,FCE)性状是十分复杂的数量性状,受多种因素影响,如生长环境、饲料配方、饲养设备、健康状况、营养状况等,但同等条件下品种或品系在饲料转化率中起决定性作用[4-6]。随着全球对食物需求的增长及可用于农业生产的土地资源的萎缩,可持续性地生产出人们消费需要的产品及最大限度发挥生产能力,培育饲料转化效率最佳的养殖动物是科学工作者面临的巨大挑战[7,8]。
将分子标记辅助选择用于育种实践,增强对数量性状的遗传操控能力和数量性状优良基因型的选择效率,是提高育种效果的有效途径。近年来,随着现代分子生物技术的快速发展,分子标记种类、检测手段以及分析方法日趋完善,基于SNP标记的全基因组关联分析所发掘的标记解析率高,在MAS中可提高选择的目的性和准确性,提高育种效率,已成功应用于农作物[9-11]、畜禽[12-14]等多种生物重要经济性状的遗传研究中。
饲料转化率性状也是水产业中最重要的经济性状之一,饲料费用约占总产值的65%~75%[15,16],但由于水产动物个体饲料转化率性状难于测量,很多水产养殖种类都没有开展性状分子育种工作,仅在鲤中有少量报道[17-19]。鲤是世界和我国主要水产养殖种之一,是人类重要的动物蛋白源。据统计,鲤养殖成本中饲料成本接近70%[20],因此,迫切需要利用现代分子育种技术改良现有鲤品种的饲料转化率,提高饲料的利用效率。饲料转化率的选择需要做个体饲料转化率测定,在育种中操作繁琐,如能找到与饲料转化率高度相关的分子标记,使选择相对简单。在此基础上,选择正向效应较大的等位基因,在育种中有目的地聚合或替代,以达到提高育种效率的目的。
本研究以镜鲤Cyprinuscarpio心家系为试验群体,利用SNP标记进行饲料转化率性状的全基因组关联分析,试图从全基因组范围内发掘与饲料转化率相关的标记,挖掘鲤饲料转化率的优异等位基因,为鲤分子辅助选择奠定基础。
1 材料与方法
1.1 试验家系及表型测定
试验家系选用黑龙江水产研究所试验站的镜鲤群体一龄个体,父母本为国家鉴定为良种德国镜鲤选育系的后代。随机选取68尾,初始体质量为(58.1±14.9)g,单尾饲养于水族箱中,每尾鱼单独配置饲料盒,每日定时饱食投食3次,记录未被摄食的饵料量。连续饲养3个月,每月定期测定体质量,记录饲料投喂量及体质量变化,试验结束鱼平均体质量达(322.1±73.0)g。饲料转化率计算:
饲料转化率(FCE)=(W-W0)/Id×100%,其中W0为初始体质量(g),W为终末体质量(g),Id为测定期摄入的干物质总量(g)。
第一、二、三月龄的饲料转化率分别用FCE1、FCE2和FCE3表示,用FCEa表示3个月总平均饲料转化率。
试验结束时,将试验鱼麻醉,抽取尾部静脉血液1mL,放入盛有抗凝剂的2mL离心管中迅速摇匀,放置-4℃备用。采用QIAampDNA Blood MidiKit血液提取试剂盒,参照标准流程提取血液样本的DNA。
1.2 GoldenGate芯片设计及SNP基因分型
本试验SNP标记序列来自镜鲤高通量转录组测序,选择均匀分布于鲤全基因组1 536个SNP标记合成寡核苷分析芯片。芯片制作及芯片杂交实验,委托上海博豪生物芯片有限公司完成[21]。分型数据由基因分型软件自动读取和分析,手工剔除错误分型标记。
1.3 性状和标记的关联分析及优异等位变异发掘
(1)用SPSS11.5软件计算性状的最大值(maximum,Max)、最小值(minimum,Min)、平均值(mean)、标准差(standard deviation,SD)和变异系数(coefficientofvariation,CV)。利用SPSS 11.5的GLM对数据进行方差分析。
(2)利用TASSEL5.2.1软件[22](http://www.maizegenetics.net/)的一般线性模型(general linearmodel,GLM)和混合线性模型(mixed linearmodel,MLM)进行标记和性状之间的关联分析。参考Machay等[23]的方法,用随机分布于鲤全基因组的383个SNP标记评估群体的遗传结构。采用STRUCTURE软件[24]的混合模型来确定群体结构,获得混合线性模型分析所需的Q值。将群体结构分析所得的Q值和亲缘关系Kinship作为协变量,进行MLM关联分析。选取两种模型共同关联到的显著(P<0.05)标记位点,并计算这些标记位点对表型变异的贡献率(R2)。
(3)参考文自翔等[25]描述的方法,对饲料转化率性状显著关联的标记位点进一步发掘优异等位基因,计算各等位变异的表型效应值。对关联分析中检测到的每个标记,按照基因型对表型进行分组。采用Dunnett-t检验比较不同基因型间性状平均值与对照的差异显著性,这里的表型效应值为优异等位基因的性状平均值与对照的差值。公式为:

式中,ɑi代表第i个等位变异的表型效应值,xij为携带第i个等位变异在第j个个体中表型值,ni为具有第i等位变异的个体数,Nk为携带无效等位变异(某一位点中,若其中一个等位变异被视为“有效”,则其他等位变异相应地被视为“无效”)的第k个个体的表型值,nk为具有无效等位变异的个体数。若ɑi>0,则认为该等位变异为增效等位变异,反之为减效等位变异[25]。
增效(减效)等位变异平均效应(Average positive(negative)allele effectof locus,AAE)和增减百分比的计算方法为:AAE=Σɑc/nc,其中ɑc为某关联位点内第c个增效(减效)等位变异表型效应值,nc为位点内增效(减效)等位变异数。增效(减效)等位变异表型效应的增效(减效)百分比为:
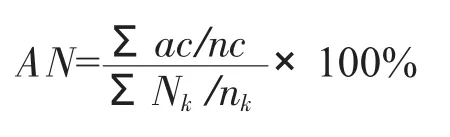
2 结果与分析
2.1 饲料转化率性状表型变异
由表1可知,不同月龄镜鲤的饲料转化率性状表现出丰富的表型变异。试验群体3个月龄个体间饲料转化率的变异幅度分别为0.38~0.99、0.39~0.93及0.26~0.90,其中第三月龄的变异幅度较大,三月平均为0.41~0.88。不同月龄的饲料转化率性状变异系数为13%~26%,其中第三月龄变异最大(26%),其他月龄及总平均变异系数基本一致。
2.2 鲤饲料转化率性状的关联分析
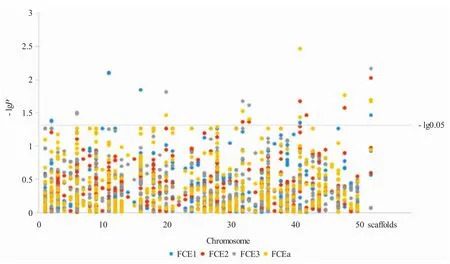
图1 镜鲤FCE的关联分析曼哈顿图Fig.1 M anhattan plot for association analysis of four FCE traits in m irror carp
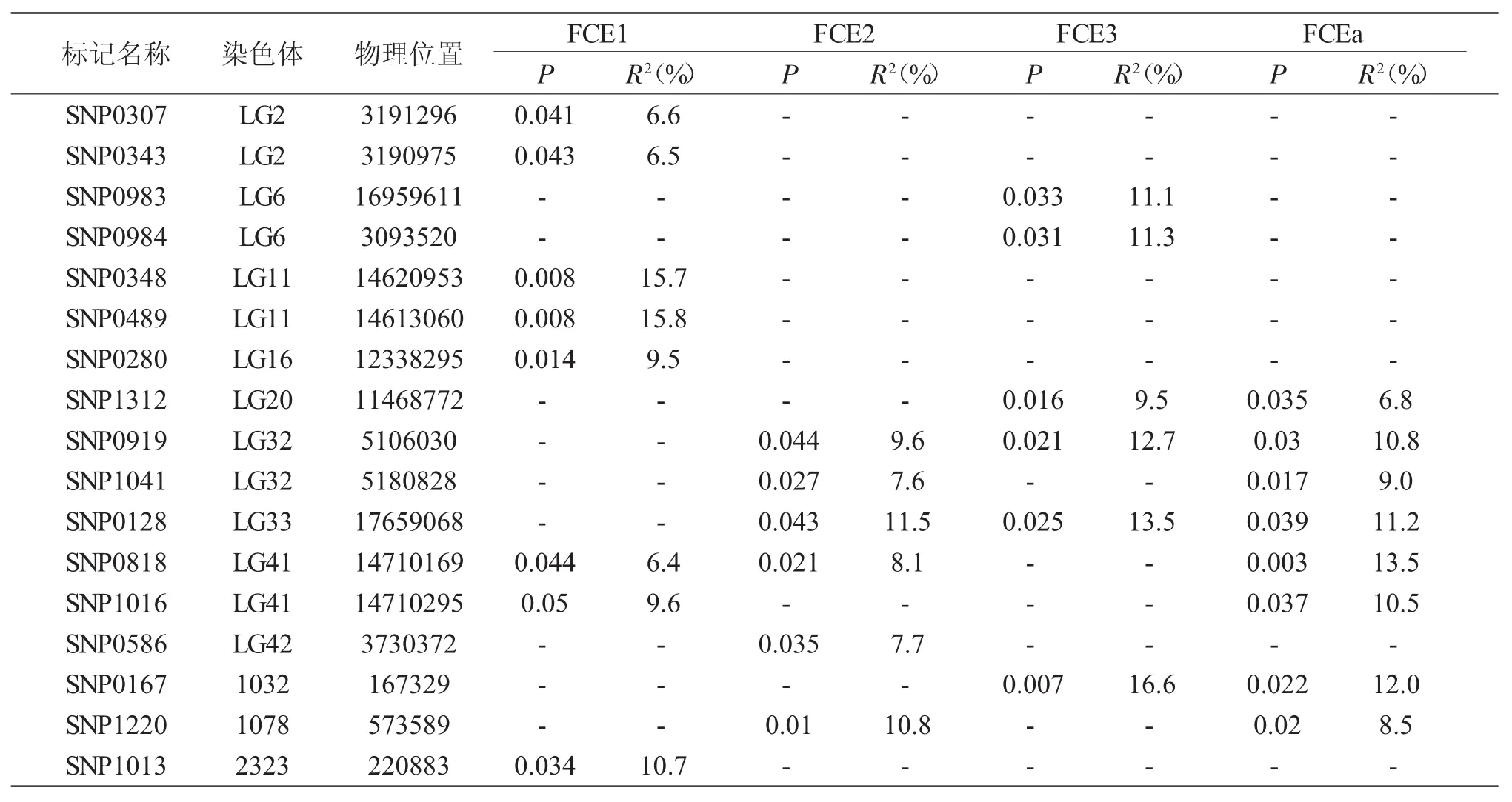
表2 两种线性模型检测到的与镜鲤食物转化率相关的标记位点(P<0.05)及其对表型贡献值Tab.2 M arker lociassociated w ith feed conversion efficiency in m irror carp(P<0.05)detected by two linear models and their phenotypic contributions
采用TASSEL软件GLM模型和MLM模型程序,将SNP标记基因分型结果与3个月份下的表型数据进行关联分析。两种模型共检测到17个标记位点与饲料转化率性状显著相关(P<0.05),分别位于第 2、6、11、16、20、32、33、41和 42染色体及scafford1032、1078和2323上,单个关联位点表型变异贡献率范围为6.4%~16.6%(图1,表2)。其中与FCE1显著关联的分子标记有8个,分别位于染色体2、11、16、41,和scafford2323上,各标记位点对表型贡献为6.4%~15.8%,其中第11染色体上的两个标记(SNP0348和SNP0489)对表型的贡献较大(15.7%和15.8%)。与FCE2关联的分子标记有6个,分别位于染色体32、33、41、42,和scafford1078上,各标记位点对表型的贡献为67.6%~11.5%,其中位于第33染色体上的SNP0128对表型的贡献最大(11.5%)。与FCE3关联的分子标记有6个,分别位于染色体6、20、32、33,和scafford1032上,各标记位点对表型的贡献为9.5%~16.6%,其中位于scafford1032上的 SNP0167对表型的贡献最大(16.6%)。与FCEa关联的位点有8个,分别位于染色体20、32、33、41、42、scafford1032,和scafford1078上,各标记位点对表型的贡献为6.8%~13.5%,其中5个标记对表型的贡献达到10%以上,分别为SNP0818、SNP0167、SNP0128、SNP0919,和SNP1016,其中SNP 0818对表型贡献最大,达到13.5%。
本研究没有检测到与4个性状都关联的共同分子标记位点。与两个性状相关的分子标记位点有5个,其中SNP1016标记与FCE1和FCEa相关;SNP1041和SNP1220与FCE2和FCEa显著相关;SNP1213和SNP0167与FCE3和FCEa显著相关。与 3个性状相关的标记位点有 3个,分别为SNP0919、SNP0128和SNP0818。其中SNP0818标记与 FCE1、FCE2及 FCE3显著相关;SNP0919和SNP0128两个位点对FCE2、FCE3及总平均显著相关。其余9个标记仅与一个饲料转化率性状相关,说明饲料转化率性状既存在相对稳定效应的基因,不同月龄对其影响也较大。
2.3 关联位点优异等位变异鉴定
在关联分析中,找到与性状关联位点只是第一步,还需进一步明确各显著关联位点中等位变异的差异,发掘出优异等位变异。本研究利用SPSS软件,采用Dunnett-t检验比较不同等位基因性状平均值与对照的差异显著性,将表现差异显著的等位基因作为优异等位基因,表型效应值为优异等位基因的性状平均值与对照的差值。对本研究鉴定的与镜鲤饲料转化率相关的17个位点,同一位点等位变异间表型效应差异明显,除了标记SNP307标记外,其余16个标记在各等位变异均存在显著差异(表3)。表3还列出了各关联位点增效/减效表型效应值及增/减效等位变异平均效应的较平均值增减的百分比。在FCE1关联的7个位点中,SNP 1013位点增效等位变异平均效应最高(+4.7%),减效平均效应值为-8.0%,如果把增效等位变异的基因型定义为优势基因型,则减效等位变异定义为非优势等位基因型,则优势等位变异和非优势等位变异之前平均变异达到12.7%,说明优势基因型对性状的贡献非常显著。同理,SNP0348、SNP0489和SNP1016等3个标记的这种差异也达到10%以上,暗示优势基因型选择效应显著。与FCE2关联的6个位点中,SNP0128位点增效平均效应最高(+11.2%),增/减效等位基因间差异为11.9%,SNP1220和SNP0919标记优势基因型和非优势基因型之间效应差异也达到10%以上,其中SNP0919增/减效应差异高达17.2%。与FCE3关联的6个位点中,增效变异较前两个月龄明显增大,其中增效等位变异平均效应最高的SNP0167位点,达到13.4%,其次是SNP0128(11.2%)。同时,这些位点的增/减效差异也较大,SNP0167位点的增/减效达到22.9%。与FCEa关联的7个位点中,SNP0167、SNP0919和SNP1016三个位点增/减效差异也分别达到10%以上。
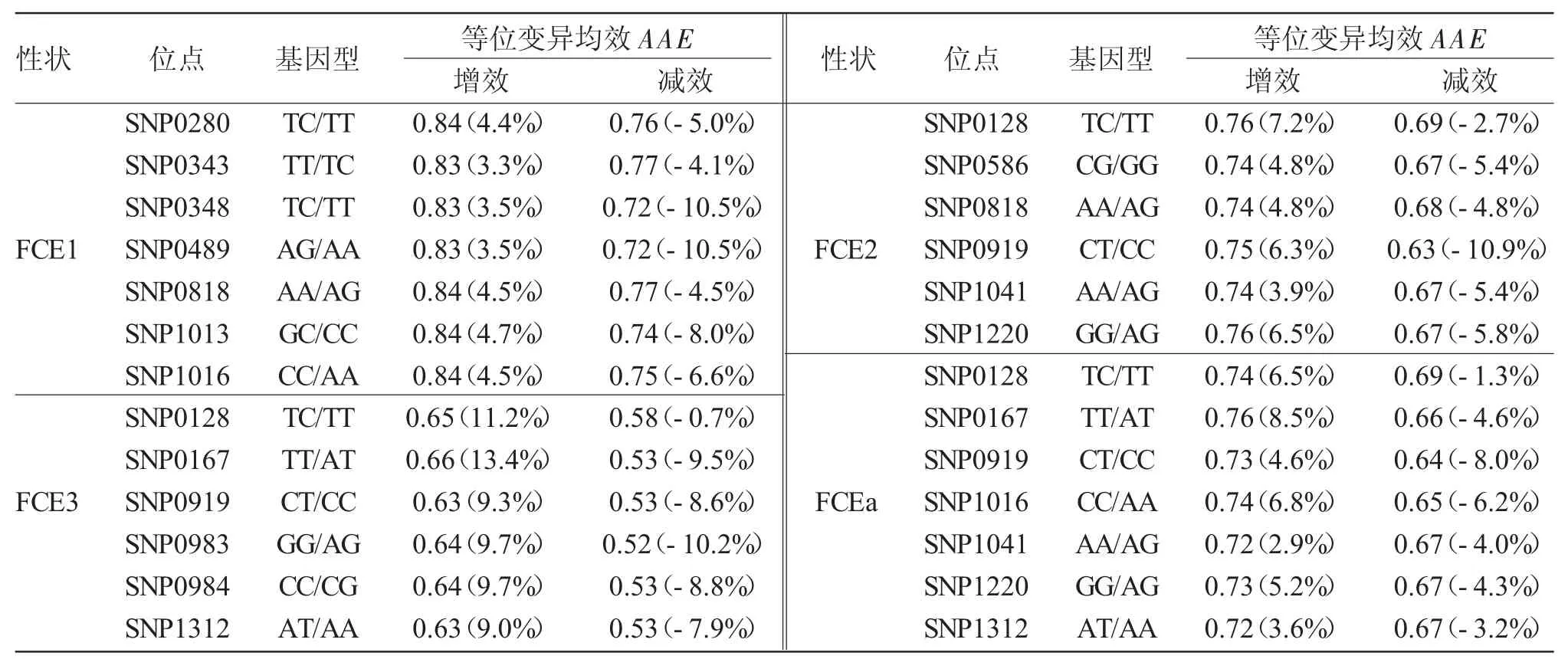
表3 与镜鲤食物转化率性状关联的SNP位点增效(减效)优异等位变异的表型效应Tab.3 Phenotypic effects of elite alleles at locistrongly associated w ith feed conversion efficiency traits in m irror carp
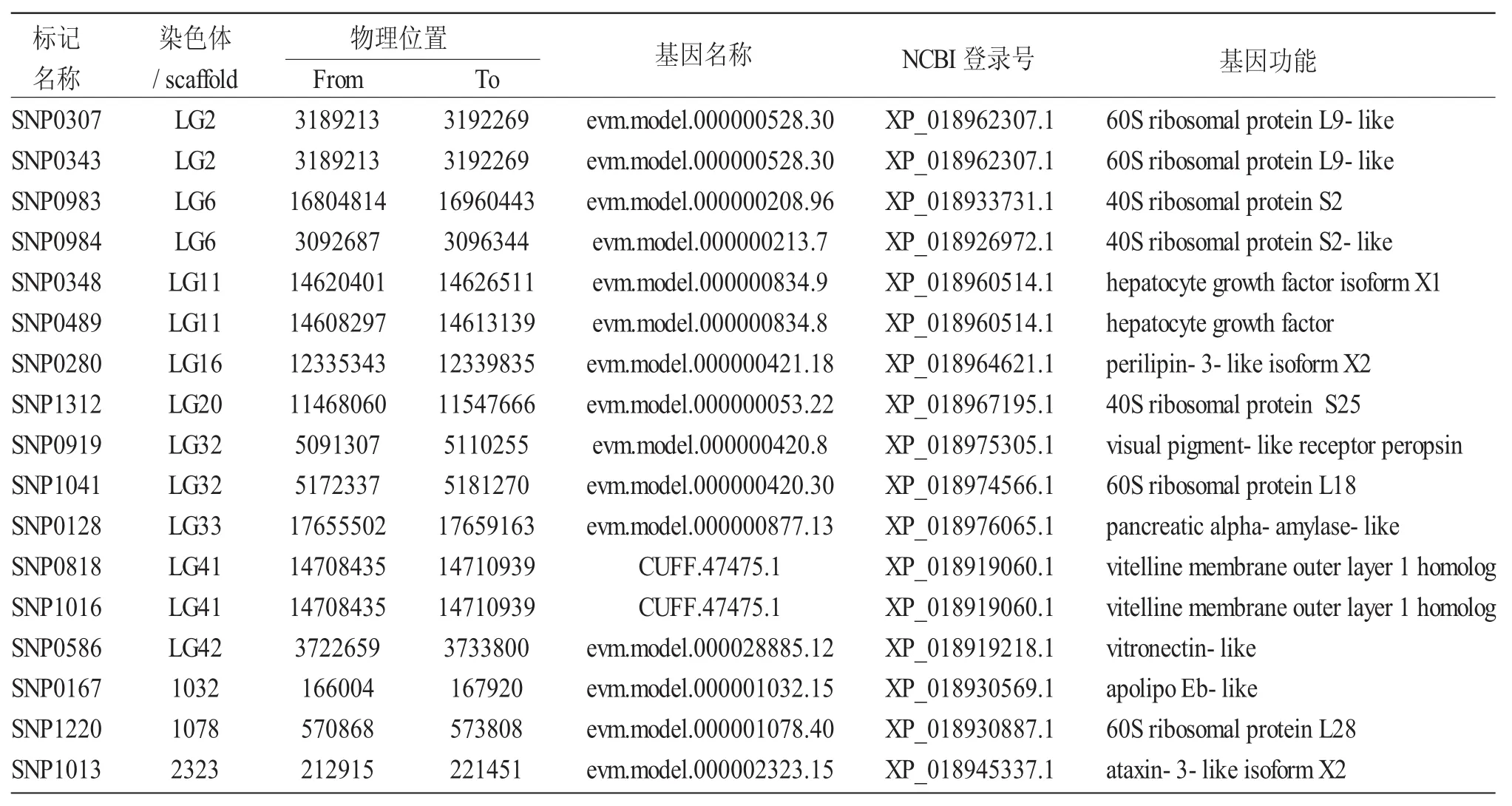
表4 食物转化率性状关联标记对应的候选基因Tab.4 Candidate genes associated w ith feed conversion efficiency related SNPs refined to them irror carp genome
纵观四个性状,优异等位变异的表型效应较群体均值平均高6.2%,较非优势基因型平均高12.5%。其中,SNP0919标记的优势基因型CT对FCE2、FCE3和FCEa三个性状的增减效差异分别为17.2%、17.9%和12.6%;SNP0167标记的优势基因型TT对FCE3和FCEa的增减效差异分别为22.9%和13.0%;SNP1016标记的优势基因型CC对FCE1和FCEa两个性状的增减效差异分别为11.1%和13.0%;暗示这些标记对性状的选择效果明显。
2.4 候选基因分析
对17个与饲料转化率显著关联的SNP标记位点与鲤全基因组物理图谱进行了扫描分析,获得17个候选基因(表4)。对这些基因进行注释,发现这些基因分别为:核糖体蛋白基因家族(60S ribosomal protein L9-like,60S ribosomal protein L28,60S ribosomal protein L28,40S ribosomal protein S2,40S ribosomal protein S2-like及40S ribosomal protein S25)、肝细胞生长因子及其异构体(hepatocyte growth factor和hepatocyte growth factor isoform X1)、脂滴包被蛋白(perilipin-3-like isoform X2)、视觉受体样蛋白(visualpigment-like receptor peropsin)、胰腺α-淀粉酶(pancreatic alpha-amylase-like)和卵黄膜外层1同源基因(vitellinemembrane outer layer 1 homolog)、玻连蛋白(vitronectin-like)、载脂蛋白(apolipo Eb-like)及共济失调蛋白(ataxin-3-like isoform X2)等。这些功能基因的鉴定将为鲤饲料转化率性状的分子机理研究提供信息。
3 讨论
利用分子标记辅助选择技术提高育种效率、降低育种成本是近30年来农作物、畜禽及水产遗传育种研究的主要内容[10-14]。找到重要经济性状相关的标记或基因,并据此进行遗传改良,将分子标记辅助选择用于育种实践,增强对数量性状的遗传操控能力,提高育种中对数量性状优良基因型选择效率。
连锁分析和关联分析是现代分子育种中常用的解析复杂性状的方法,但二者有所不同。对一些具有较小效应的基因,关联分析的发现能力比连锁分析高得多[26,27]。本研究采用一般线性模型和混合线性模型,对性状进行关联分析,可有效消除伪关联,增加关联结果的可信度[28]。共获得了17个不同阶段鲤饲料转化率相关的SNP标记,其中有两个标记(SNP0818和SNP1041)与前人的研究结果一致[19],绝大多数是检测到的新标记。贡献率大于10%的标记占总数的50%以上,其中最大的达16.6%,说明这些标记对饲料转化率性状具有重要的作用。在不同月龄镜鲤中检测出的位点不完全一致可能是不同生长阶段影响的结果,这是多基因控制的数量性状的突出特点。有些关联位点在两个以上月龄镜鲤中才能检测到,可视为相对稳定的位点。如SNP0919、SNP0167和SNP1016等三个位点在两个以上不同月龄的饲料转化率都显著相关,说明这些标记位点效应稳定,比仅在某一特定月龄下表达的位点更有育种价值。
饲料转化率性状是一个复杂的数量性状,受多种因素影响,但品种或品系在饲料转化率方面起决定性的作用,有些品种/品系将饲料转化为肌肉的能力比其他遗传品种/品系更高效。同一品种的不同品系采用不同的育种目标或育种计划育成的两个品系,可能具有显著不同的饲料转化率,这在不同畜禽育种中已得到证实[12-14,29]。本研究结果表明:鲤家系内个体间饲料转化率性状存在明显差异,不同月龄的鲤家系内饲料转化率性状存在15%~26%的变异。前期对该群体遗传结构分析的研究也表明,该群体具有丰富的遗传变异[30],说明可通过家系选育获得更高饲料转化率的品系或品种可能性。不同基因型的个体饲料转化率性状上差异明显。本研究中鲤家系优势基因型较群体平均值高6.2%,较非优势基因型更高,高达12.5%。预计用此来培育新品种可减少5%以上的饲料量,每km2效益提高20%,每km2增加纯利6 000元以上。按我国鲤养殖面积约13万km2计算[31],每年节约的饲料成本相当可观。
由于本研究的SNP标记来自转录组测序,每个标记位点均位于鲤基因的编码区内,因此找到了与性状相关的标记也就找到了与性状相关的候选基因,17个饲料转化率相关的标记对应17个候选基因。其中,酶胰腺α-淀粉酶是最重要的水解碳水化合物的酶,参与碳水化合物的消化和吸收[32]。肝细胞生长因子及其异构体能促进多种类型的细胞分化、增殖,是一种多功能的细胞因子,对许多组织细胞的增生修复有重要作用[33]。脂滴包被蛋白在脂肪分解调控中起到“分子开关”的作用,决定了机体脂肪的沉降量[34]。核糖体蛋白基因家族除了负责细胞内蛋白质的合成以外,还与细胞的生长、分化、胚胎发育等有关[35,36]。这些基因参与鱼体内食物转化的具体过程还需进一步研究验证。
利用关联分析发掘优异基因是目前动植物分子育种研究的重要方法之一。本研究解析了与鲤饲料转化率稳定关联的位点内等位变异的表型效应,在性状的表型与基因型之间用特定的等位变异建立起联系,发掘了饲料转化率关联的优异等位变异。在鲤育种工作中,可以利用携带这些优异等位变异的材料做亲本,并借助分子标记辅助选择将多个目标基因导入或聚合,提高育种选择效率。本研究只选择了1 536个标记,只是粗略意义上的全基因组关联分析,已检出的关联位点还需得到紧密相邻的其他位点关联性的支持,以提高可信度。
[1] 詹志春.饲料原料的全数据分析与养殖业的可持续发展[J].养殖与饲料,2016(5):7.
[2] 宋明.浅谈如何节约饲料成本[J].当代畜禽养殖业,2016(5):15.
[3] 张长春.猪饲料成本的影响因素及控制措施[J].现代畜牧科技,2016,9(21):58.
[4] Skinner-Noble D,Jones R and Teeter R.Components of feed efficiency in broiler breeding stock:is improved feed conversion associated with increased docility and lethargy in broilers[J].Poultry Science,2003,82(4):532-537.
[5] Hafla A N,Carstens G E,Forbes TD,et al.Relationships between postweaning residual feed intake in heifers and forage use,body composition,feeding behavior,physical activity,and heart rate ofpregnantbeef females[J].Journal ofAnimalScience,2013,91(11):5353-5365.
[6] Woyengo T A,Beltranena E and Zijlstra R T.Controlling feed costby including alternative ingredients into pig diets: a review[J].JournalofAnimalScience,2014,92(4):1293.
[7] HoqueM A,KadowakiT,Shibata T,etal.Genetic parameters formeasuresof residual feed intake and growth traits in seven generationsofDuroc pigs[J].Livestock Science,2009, 121(1):45-49.
[8] 夏俊花.改善饲料转化率是提高养猪生产成本效益的最大机会[J].期国外畜牧学(猪与禽),2015,35(9):1-3.
[9] Sivakumar S,Susanne D,Marta L,etal.Genome-wide association study for grain yield and related traits in an elite springwheatpopulation grown in temperate irrigated environments[J].Theoretical and Applied Genetics,2015,18(2):353-363.
[10] KenjiY,EijiY,Koichiro E,etal.Genome-wide association study usingwhole-genome sequencing rapidly identifies new genes influencing agronomic traits in rice[J]. Nature Genetics,2016,48(8):927-934.
[11] Cui Z,Luo J,QiC,etal.Genome-wide association study(GWAS)reveals the genetic architecture of four husk traits inmaize[J].BMCGenomics,2016,17(1):946.
[12] Sherman E L,Nkrumah JD,Moore S S,et al.Whole genome SNP associationswith feed intake and feed efficiency in beefcattle[J].JournalofAnimalScience,2010, 88(1):16-22.
[13] Jiao S,Maltecca C,Gray K A,etal.Feed intake,average daily gain,feed efficiency,and real-time ultrasound traits in Duroc pigs:II.Genome-wide association[J].Journalof AnimalScience,2014,2(7):2846-2860.
[14] Jingwei Y,Kehua W,Guoqiang Y,et al.Genome-wide association studies for feed intake and efficiency in two laying periods of chickens[J].Genet Selection Evolution, 2015,47(1):82.
[15] Gjedrem T and Baransk M.Selective Breeding in Aquaculture.An Introduction[M].Springer,New York,2009.
[16] Marineharvest.TheMarineharvestSalmon Industry Handbook[M].Marineharvest,Bergen,2012:41.
[17] 李鸥,曹顶臣,张研,等.利用EST-SSR分子标记研究鲤的饲料转化率性状[J].水产学报,2009,33(4):624-631.
[18] 张丽博,张晓峰,曹顶臣,等.利用SSR及EST标记对鲤鱼饲料转化率的QTL分析[J].农业生物技术学报, 2010,18(5):963-967.
[19] 王宣朋,张晓峰,李文升,等.鲤饲料转化率性状的QTL定位及遗传效应分析[J].水生生物学报,2012(2): 177-196.
[20] 李同国.我国2016年鲤鱼养殖渔情分析专题报告[R].中国水产频道/水产前沿.
[21] Fan JB,Gunderson K L,Bibikova M,etal.Illumina universalbeadarrays[J].Methods in Enzymology,2006,410: 57-73.
[22] Bradbury P J,Zhang ZW,Kroon D E,et al.TASSEL:Software for associationmapping of complex traits in diverse samples[J].Bioinformatics,2007,23:2633-2635.
[23] Mackay Iand PowellW.Methods for linkage disequilibriummapping in crops[J].Trends in Plant Science,2007, 12(2):57-63.
[24] EvannoG,RegnautSand Goudet J.Detecting the number of clusters of individualsusing the software STRUCTURE: a simulation on study[J].Molecular Ecology,2005,14(8): 2611-2620.
[25] 文自翔,赵团结,郑永战,等.中国栽培和野生大豆农艺及品质性状与SSR标记的关联分析:II.优异等位变异的发掘[J].作物学报,2008(34):1339-1349.
[26] Lander E S.Genetic dissection of complex traits science[J].Nature Genetics,1996,12(4):356-358.
[27] Pritchard JK,Stephens M and Donnelly P.Inference of population structure usingmultilocus genotype data[J]. Genetics,2000,155(2):945-959.
[28] 杨胜先,牛远,李梦,等.栽培大豆农艺性状的关联分析及优异等位变异挖掘[J].中国农业科学,2014,4(20): 3941-3952.
[29] Schumann Band Zijlstra R.Improving feed efficiency[J]. Proceedings of the London Swine Conference,2014: 131-133.
[30] 张晓峰,郑先虎,匡友谊,等.利用GoldenGate分析的高通量SNP芯片对鲤鱼基因进行分型[J].水产学杂志, 2016,29(6):19-25.
[31] 农业部渔业渔政管理局.中国渔业统计年鉴[M].北京:中国农业出版社,2016:55-62.
[32] Leloup V M,Colonna P and Marchis-Mouren G.Mechanism of the adsorption of pancreatic alpha-amylase onto starch crystallites[J].Carbohydrate Research,1992,232(2):367-374.
[33] Miyazawa K,Hirothito T,Nake D,etal.Molecular cloning and sequence analysis of cDNA for human hepatocyte growth factor[J].Biochemical&Biophysical Research Communications,1989,163(2):967.
[34] Londos C,Brasaemle D L,Schultz C J,etal.On the controlof lipolysis in adipocytes[J].Annals of the New York Academy ofSciences,1999,892(1):155-168.
[35] MaheshwariY,RaoM,Sykes D E,etal.Changes in ribosomalprotein and ribosomalRNA synthesis during rat intestinaldifferentiation[J].CellGrowth&Differentiation the Molecular Biology Journalof the American Association for CancerResearch,1993,4(9):745-752.
[36] Lai K,Amsterdam A,Farrington S,etal.Many ribosomal protein mutations are associated with growth impairment and tumor predisposition in zebrafish[J].Developmental Dynamicsan Official Publication of the American Association ofAnatomists,2009,238(1):76-85.
Association Analysis of Feed Conversion Efficiency and Exp loration of Elite Alleles in Common Carp
ZHANG Xiao-feng,LIChao,LU Cui-yun,CAODing-chen,SUN Xiao-wen
(Nationaland LocalUnited Engineering Laboratory for Freshwater Fish Breeding,Heilongjiang River FisheriesResearch Institute, Chinese Academy of Fishery Sciences,Harbin 150070,China)
Feed conversion efficiency(FCE)is one of the importanteconomic traits to be improved.In this study,a total of 1 536 SNPswere used to genotype a full-sib panelof 68 individualsofm irror carp(Cyprinuscarpio)based on the Illum ina GoldenGate assay.The association between SNPs andFCEphenotypic datawas analyzed by general and m ixed linearmodels(GLM and MLM)methods in software of TASSEL,and the significantly associated lociofFCEwere analyzed forexploring the elite alleles.A totalof 17 SNPswere found to be significantly associated w ithFCEata threshold ofP<0.05 in nine chromosomes(2,6,11,16,20,32,33,41 and 42)and three Scaffolds(1032,1078 and 2323).TheFCEassociated locihad contribution varying from 6.4%to 16.6%,and the phenotypic allele effectwasestimated and then the average positiveand negative allele effectofa locuswas calculated over the phenotypic effects of allalleles.A totalof 16 elite alleleswere screened outamong the 17 lociw ith repeated associations,w ith 6.2%higher average phenotypic allele effect than that in the population mean and 12.5%higher than the negative ones.Three loci such as SNP0919,SNP0167 and SNP1016 w ith themaximaldifferences in average phenotypic allele effect revealed the obvious effecton the selection ofFCEtrait.Meanwhile,17 candidate geneswere scanned and annotated.The lociand elite alleles ofFCEidentified w ill provide important foundation forsubsequentstudies includingmolecularmarkerassisted selection andmolecularbreeding by design.
common carp;feed conversion efficiency;singlenucleotide polymorphism(SNP);association analysis;elite allele
S917
A
1005-3832(2017)03-0011-08
2017-02-06
中国水产科学研究院基本业务费重点项目(2016ZD0301);农业部"948"计划项目(2016-X15).
张晓峰(1973-),博士,副研究员,从事水产基因组研究.E-mail:zhangxiaofeng@hrfri.ac.cn
孙效文(1955-),研究员,博士生导师,从事水产分子育种研究.E-mail:sunxw2002@163.com
