猪流行性腹泻病毒山西分离株全基因组序列分析
2017-06-26刘文俊樊振华王娟萍姚敬明韩一超薛翼鹏米瑞娟李红丽
刘文俊,樊振华,王娟萍,姚敬明,吴 忻,孟 帆,韩一超,薛翼鹏,米瑞娟,李红丽,赵 岳
(山西省农业科学院畜牧兽医研究所,山西太原 030032)
猪流行性腹泻病毒山西分离株全基因组序列分析
刘文俊,樊振华*,王娟萍,姚敬明,吴 忻,孟 帆,韩一超,薛翼鹏,米瑞娟,李红丽,赵 岳
(山西省农业科学院畜牧兽医研究所,山西太原 030032)
用RT-PCR扩增分离的一株PEDV CH-SXCH11-2015的全基因组序列,测序拼接后,进行了序列分析。CH-SXCH11-2015全长28 038 bp比CV777长5 bp,与BJ-2011-1同源性最高,为98.9%;与LZC、SM98同源性最低,为96.6%。S基因全长均为4 161 bp,突变主要发生在S1区,同源性分析表明,CH-SXCH11-2015核苷酸同源性与BJ-2011-1和CH/ZJJS-Z/2012的最高为98.9%,与SM98同源性最低为93.6%,与CV777、CV777vaccine同源性分别为94.2%和93.8%。CH-SXCH11-2015 S基因氨基酸同源性与CH/ZJJS-Z/2012的最高为98.3%,与CV777vaccine、SQ2012同源性最低为91.8%,与CV777的同源性为92.9%。ORF3基因全长均为675 bp,无核苷酸插入和缺失,同源性分析表明,CH-SXCH11-2015的ORF3基因与CH/HBQX/10核苷酸同源性最高,为99.3%,与CV777 truncated核苷酸同源性最低,为90.6%。CH-SXCH11-2015的ORF3基因推导的氨基酸与(CH/HBQX/10、CH/S、CH/SHH/06、 CH/TJ-2012、CH/ZKXH/11、CPF299、GZGY-10、PFF188、PFF285)同源性最高,为98.7%;与CV777 truncated毒株最低,为88%。CH-SXCH11-2015的ORF3基因与CV777、attenuated DR13的核苷酸同源性分别为96.6%和97.6%,氨基酸同源性分别为95.1%和97.1%。遗传进化分析表明,CH-SXCH11-2015全基因组、S基因、ORF3基因与2010年以后我国分离的毒株亲缘关系较近,与CV777、2个疫苗株(attenuated DR13、CV777vaccine)的亲缘关系较远。
猪流行性腹泻病毒;全基因组序列;序列分析
猪流行性腹泻(Porcine epidemic diarrhea,PED)是由猪流行性腹泻病毒(Porcine epidemic diarrhea virus,PEDV)引起的猪的一种急性、高度传染性肠道传染病,临床上以呕吐、水样腹泻和脱水死亡为特征[1]。该病1978年首次报道于比利时和英国[2-3],1984年宣化等证实该病在我国存在[4]。2010年10月,中国南方暴发PED[5],给养猪业造成巨大损失。2014年冬季至2015年春季,山西部分规模化猪场暴发哺乳仔猪腹泻病,临床主要表现为哺乳仔猪水样腹泻、严重脱水和呕吐,发病率和病死率高达90%~100%,给养猪业造成巨大的经济损失。为了查清山西猪流行性腹泻的流行情况,课题组对山西11个地市的30个规模化猪场的仔猪腹泻发病情况进行了流行病学调研,并对采集的253份病料进行猪流行性腹泻病毒、猪传染性胃肠炎病毒(TGEV)检测,PEDV阳性率为73.26%,TGEV阳性率为7.36%。表明山西猪群发生的腹泻,主要是由猪流行性腹泻病毒引起。课题组从阳性病料中分离出一株PEDV,命名为CH-SXCH11-2015。本研究通过对基因组序列的分析,为PED防控提供理论依据和参考。
1 材料与方法
1.1 材料
1.1.1 毒株 2015年从山西某种猪场采集的疑似PED病料中分离出一株PEDV,命名为CH-SXCH11-2015。
1.1.2 主要试剂 RNAiso Plus、PrimeScriptTMⅡ 1st Strand cDNA Synthesis Kit、PremixTaqTM、DNA Marker DL2000、pMD18-T Vector购自宝生物工程(大连)有限公司;DNA胶回收试剂盒、质粒提取试剂盒购自上海生工生物工程技术服务有限公司;E.coliDH5α菌株购自天根生物科技(北京)有限公司;乙醇、异丙醇、氯仿等试剂均为分析纯。
1.2 方法
1.2.1 引物的设计与合成 根据GenBank中收录的PEDV CV777、CH/FJND-3/2011全基因组序列,设计了31对引物,引物序列见表1。引物由上海生工生物工程技术服务有限公司合成。
1.2.2 病毒总RNA的提取 按RNAiso Plus试剂盒操作说明书,从细胞培养液中提取病毒基因组RNA,用适量的DEPC水溶解,置-20℃保存,备用。
1.2.3 PEDV全基因组的扩增、克隆和测序 以提取的RNA为模板制备cDNA,参照Prime ScriptTMⅡ 1st Strand cDNA Synthesis Kit操作说明书进行。在PCR管中依次加入,RNase free H2O 7 μL、RNA 1 μL 、dNTP 1 μL,下游引物1 μL;混匀后,65℃保温5 min后,冰上迅速冷却;在上述的PCR管加入5×Prime Script Ⅱ buffer 4 μL、RNase Inhibitor 0.5 μL、 PrimeScript Ⅱ RTase 1 μL、 RNase free dH2O 4.5 μL。缓慢混匀,30℃、10 min,42℃、60 min,95℃保温5 min,冰上放置。PCR反应体系:PremixTaq25 μL,上下游引物各1 μL,cDNA模板2 μL,灭菌蒸馏水21 μL,混匀后进行扩增。扩增反应条件:94℃ 3 min;94℃ 30 s,55℃ 45 s,72℃ 80 s,35个循环;再72℃延伸10 min。取10 μL PCR扩增产物进行15 g/L琼脂糖凝胶电泳,观察结果。将PCR产物纯化后与pMD18-T载体连接,转化DH5a感受态细胞,经鉴定后将阳性菌液送上海生工生物工程技术服务有限公司进行测序。
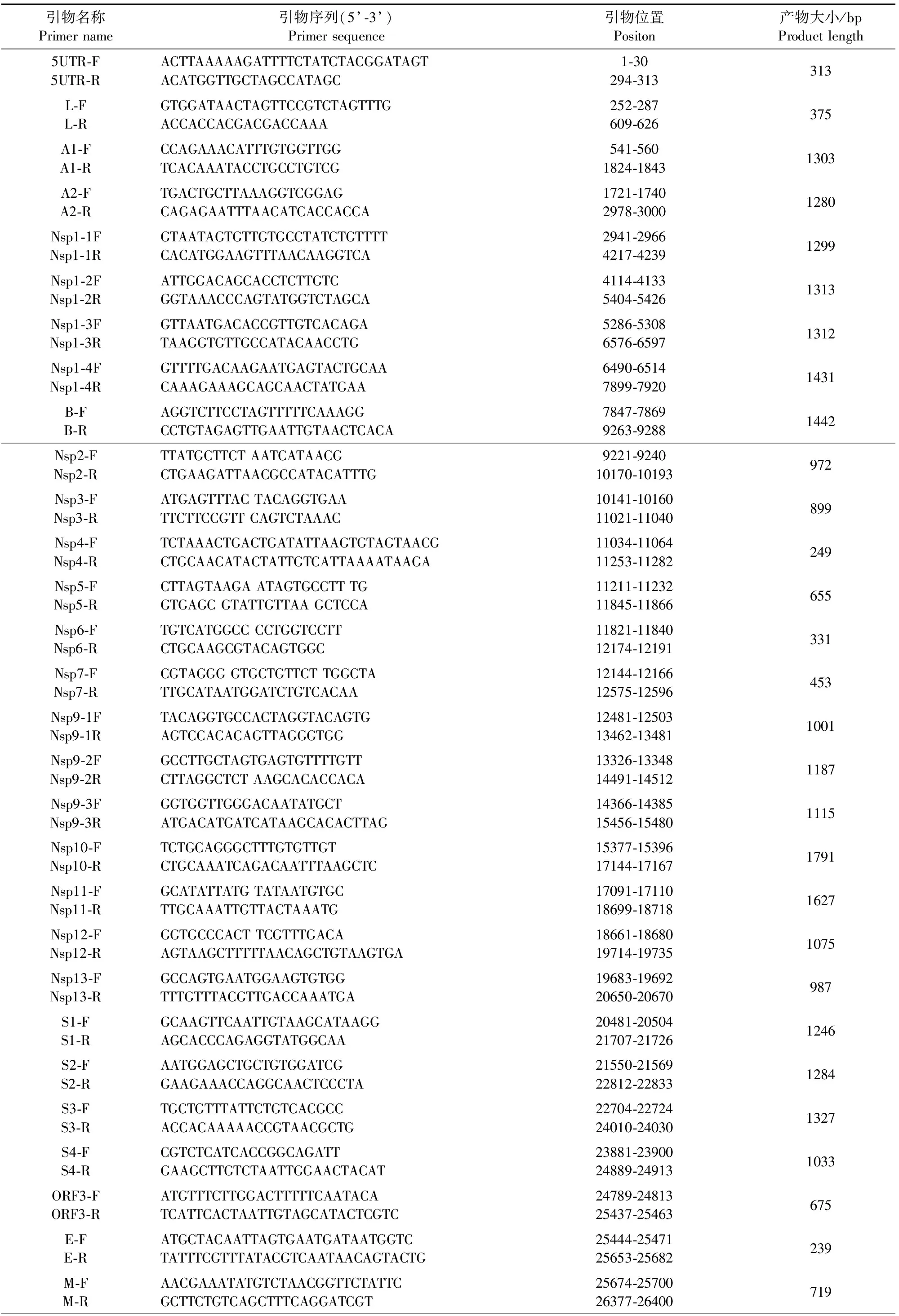
表1 扩增PEDV全基因序列的引物
1.2.4 PEDV全基因组、S和ORF3基因的序列比较分析 参考CV777毒株的全基因组序列,用DNAMAN将测得的序列进行拼接。用MegAlign分析CH-SXCH11-2015与GenBank中收录的国内外PEDV参考毒株的全基因组序列和S、ORF3基因同源性和变异情况,并用MEGA5.0分析CH-SXCH11-2015与参考毒株之间遗传进化关系。
2 结果
2.1 全基因组序列序列分析
用DNAMAN将测得的序列进行拼接,结果显示,CH-SXCH11-2015毒株全长28038bp,基因结构为5’UTR-ORF1-S-ORF3-E-M-N-3’UTR,符合PEDV基因组的特征。同源性分析表明,CH-SXCH11-2015与BJ-2011-1同源性最高,为98.9%;与LZC、SM98同源性最低,为96.6%。用MEGA5.0分析CH-SXCH11-2015毒株与GenBank中收录的其他PEDV毒株的遗传进化关系,结果见图1。由图1可见,PEDV毒株可分为2个群,G1包括CV777、我国2010年以前分离的的毒株(LZC、CH/S)和3个韩国毒株(SM98、attenuated DR13、virulent DR13)。CH-SXCH11-2015与5个我国2010年及以后分离的毒株(CH/FJND-3/2011、CH/HNLH/2015、SD2014、CHYJ130330、BJ-2011-1)组成G2群。结果表明CH-SXCH11-2015与我国2010以后分离的毒株亲缘关系较近,与CV777、我国早期分离的的毒株(LZC、CH/S)和3个韩国毒株(SM98、attenuated DR13、virulent DR13)亲缘关系较远。
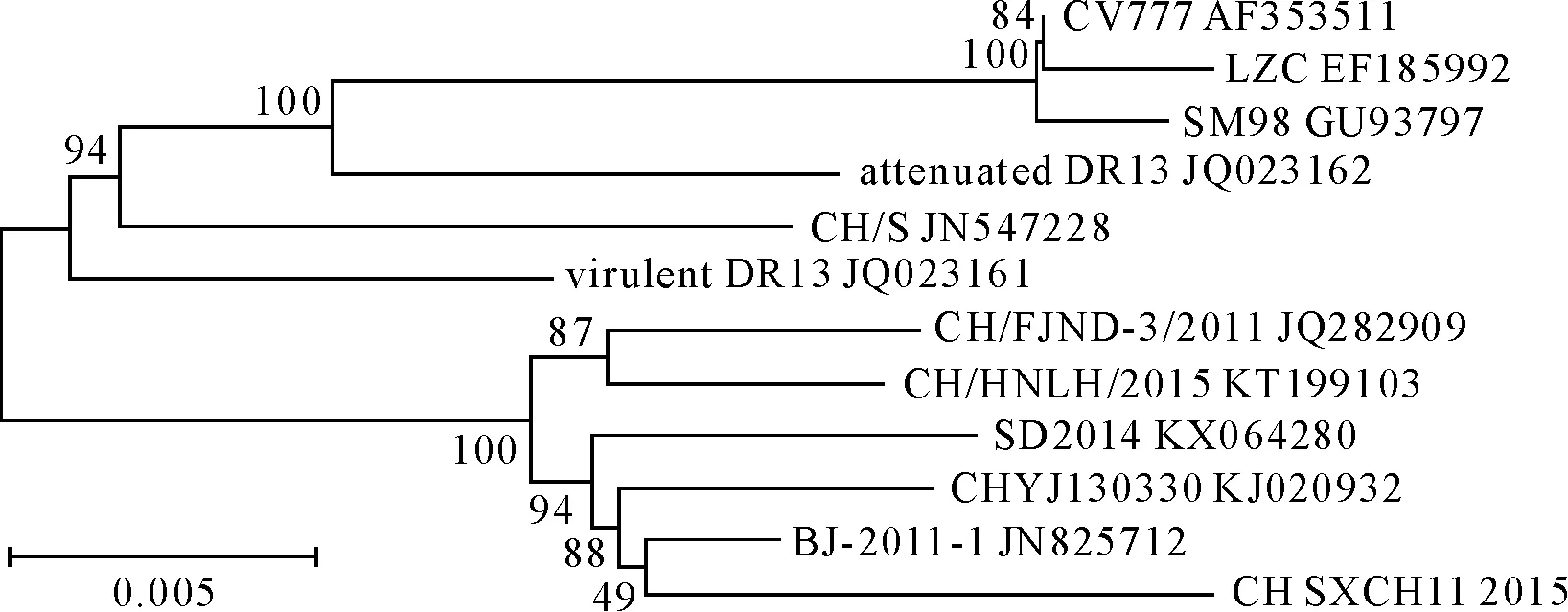
图1 基因PEDV全基因组序列构建的遗传进化分析
2.2 S基因序列分析
2.2.1 S基因序列变化 CH-SXCH11-2015株S基因全长均为4161bp,与CV777比较,既存在碱基位点突变,又存在碱基的插入和缺失现象。在170~171之间插入AAACCAGGGTGT12个碱基,在401~402之间插入TAA,在467-474位之间缺失TGGAAA。共有251个碱基发生突变,其中S1区有183个碱基发生突变。CH-SXCH11-2015株S基因推导的氨基酸与CV777比较,在58~59位之间插入QGVN,在135-136位之间插入N,在153-156位之间缺失DG,共有97个氨基酸发生突变,其中在S1区发生突变的氨基酸有72个,占发生突变的氨基酸比例为74.23%。
2.2.2 S基因同源性分析 CH-SXCH11-2015株S基因核苷酸同源性与BJ-2011-1和CH/ZJJS-Z/2012的最高为98.9%,与SM98同源性最低为93.6%,与CV777、CV777vaccine同源性分别为94.2%、93.8%。CH-SXCH11-2015株S基因氨基酸同源性与CH/ZJJS-Z/2012的最高为98.3%,与CV777vaccine、SQ2012同源性最低为91.8%,与CV777的同源性为92.9%。
2.2.3 S基因的遗传进化分析 CH-SXCH11-2015株S基因与GenBank已收录的PEDV的23株进行比对,构建进化树,见图2。由图2可知,PEDV毒株可分为2个群(G1和G2)。G1群又可分为2个亚群(G1-1和G1-2),G1-1包括,4株2010年以前分离的中国株(LJB/03、LZC、JS-2004-2、CH/S、)、2014年分离的SQ2014、4株韩国毒株(attenuated DR13、virulent DR13、KPEDV-9、SM98)、1株日本毒株83P-5、 CV777vaccine、CV777。CH-SXCH11-2015与9个2010年以后我国分离的毒株(CH/HNQX-3/14、CH/FHND-3/2011、CHYJ130330、BJ-2011-1、CH/ZJJS-Z/2012、FJ-PT/2013、SH1404、CH/HNLH/2015、SD2014)组成G1-2群。1个韩国毒株Chinju99和1个日本毒株KH组成G2群。结果表明,CH-SXCH11-2015与2010年以后我国流行毒株(除SQ2014)的亲缘关系较近,与2010年以前我国流行毒株、2个日本株、5个韩国株、2个疫苗株(attenuated DR13、CV777vaccine)、CV777的亲缘关系较远。
2.3 ORF3基因的序列分析
2.3.1 ORF3基因序列变化 CH-SXCH11-2015毒株的ORF3基因全长均为675 bp,与CV777 ORF3基因比较相比,无核苷酸插入和缺失,有23个相同的核苷酸突变,分别为:62、 162、 360、 393位核苷酸由T→C,54、160、235、301位核苷酸由G→A,63、237 、243 264、274、369、439、450位核苷酸由C→T,238、546位核苷酸由T→G,438位核苷酸由T→A,497位核苷酸由A→G, 616位核苷酸由A→T,630位核苷酸由A→C,631位核苷酸由T→A。CH-SXCH11-2015毒株的ORF3基因推导的氨基酸与CV777相比,第21位氨基酸由V→A,第54、79位由V→I,第80位F→V,第92由L→F,第101由A→T,第165由N→S,第182由H→Q。CH-SXCH11-2015毒株的ORF3基因与attenuated DR13比较,在244-245位之间多了51个核苷酸,推导的氨基酸序列比attenuated DR13在81-82位氨基酸之间多了17个氨基酸。
2.3.2 ORF3基因同源性分析 CH-SXCH11-2015与CH/HBQX/10核苷酸同源性最高,为99.3%,与CV777 truncated核苷酸同源性最低,为90.6%。CH-SXCH11-2015的ORF3基因推导的氨基酸与(CH/HBQX/10、CH/S、CH/SHH/06、CH/TJ-2012、CH/ZKXH/11、CPF299、GZGY-10、PFF188、PFF285)同源性最高,为98.7%;与CV777 truncated毒株最低,为88%。CH-SXCH11-2015的ORF3基因与CV777、attenuated DR13的核苷酸同源性分别为96.6%和97.6%,氨基酸同源性分别为95.1%和97.1%。
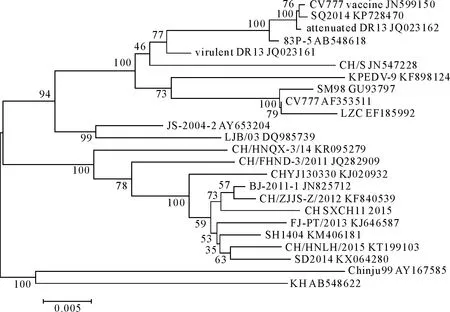
图2 基因PEDV S基因序列构建的遗传进化树

图3 基因PEDV ORF3基因组序列构建的遗传进化树
2.3.3 ORF3基因的遗传进化分析 CH-SXCH11-2015的ORF3基因与GenBank已收录的PEDV的ORF3基因序列比对,利用MEGA5.0建进化树(图3)。由图3可知,PEDV毒株可分为2大群(G1和G2),其中G1群又可分为3个亚群(G1-1、G1-2和G1-3)。CH-SXCH11-2015与11个我国2010年以来分离的毒株(CH/HNLH/2015、GZGY-01、BJ-2011-1、CH/HBQX/10、CH/HNQX-3/14、CH/ZJHZHY-6/2013、SH5、CH/TY/12、CH/TJ-2012、CH/ZKXH/11、CH/FJND-3/2011)和2009分离的CH/JL/09组成G1-1。G1-2群包括1个我国2010年以前我国分离的毒株(CH/SHH/06)、1个我国2010年以后我国分离的毒株(SD2014)和3个韩国毒株(PFF188、PFF285、CPF299)。G1-3群包括我国早期分离的毒株CH/S、3个韩国毒株(Chinju99、virulent DR13、attenuated DR13)和CV777 truncated。CV777、Brl/87、LZC组成G2群。遗传进化关系:CH-SXCH11-2015毒株与11个我国2010年以来分离的毒株(CH/HNLH/2015、GZGY-01、BJ-2011-1、CH/HBQX/10、CH/HNQX-3/14、CH/ZJHZHY-6/2013、SH5、CH/TY/12、CH/TJ-2012、CH/ZKXH/11、CH/FJND-3/2011)的亲缘关系较近,与CV777、LZC、Brl/87、2个弱毒株(attenuated DR13和CV777 truncated)亲缘关系较远。
3 讨论
用DNA Man将测得的序列进行拼接,结果显示,CH-SXCH11-2015毒株全长28 038 bp,基因结构为5’UTR-ORF1-S-ORF3-E-M-N-3’UTR,符合PEDV基因组的特征。CH-SXCH11-2015比CV777多5个碱基,是由于CH-SXCH11-2015的5UTR比CV777少了4个碱基,S基因比CV777多了9个碱基。全基因组序列分析表明,CH-SXCH11-2015与2010年以后分离的毒株位于同一亚群,亲缘关系较近,同源性为98.3%~98.9%。2010年以前分离的2个毒株(CH/S、LZC),3个韩国毒株(SM98、virulent DR13、 attenuated DR13)和CV777位于另一亚群,与CH-SXCH11-2015的亲缘关系较远,同源性为96.6%~97.3%。
CH-SXCH11-2015的S基因全长均为4 161 bp, 与CV777比较,共有251个碱基发生突变,其中S1区有183个碱基发生突变。氨基酸序列比较,共有97个氨基酸发生突变,其中在S1区发生突变的氨基酸有72个,占发生突变的氨基酸比例为74.23%,表明突变主要发生在S1区,这一结果与郑逢梅[6]、刘艳成[7]等的结果一致。遗传进化树分析表明,CH-SXCH11-2015与2010年以后我国流行毒株的亲缘关系较近,与2010年以前我国流行毒株、疫苗株的亲缘关系较远。
CH-SXCH11-2015的ORF3基因与GenBank已收录PEDV进行对比分析,CH-SXCH11-2015与11个2010年以来我国各地方分离的毒株和2009年我国分离的CH/JL/09毒株亲缘关系较近,与CV777、LZC、Brl/87、2个弱毒株(attenuated DR13和CV777 truncated)亲缘关系较远。Park[8]等研究发现,经过细胞适应的毒株比亲本毒株和野生毒株的ORF3基因有51个核苷酸的缺失。CH-SXCH11-2015与attenuated DR13比较,在244~245之间多了51个核苷酸,表明CH-SXCH11-2015为野毒株。CH-SXCH11-2015 的ORF3与CV777基因相比,无核苷酸插入和缺失,有多处核苷酸发生变异,与董彦鹏[9]、顾超逸[10]、黄冬妮[11]等分离的毒株既有相同的变异位点,又有不同的变异位点,可能是由于受到各个地方当地的环境、免疫压力或药物等因素的影响产生不同变异,变异的多样性,增加了PED的防控难度。
本研究对2015年从山西发病猪群中分离的PEDV毒株CH-SXCH11-2015进行了遗传变异分析,结果表明,CH-SXCH11-2015全基因与弱毒株attenuated DR13亲缘关系较远,CH-SXCH11-2015的S基因和ORF3基因与弱毒株attenuated DR13和CV777亲缘关系较远。因此,PEDV发生变异,现在的疫苗不能提供很好的保护,可能是PED流行的主要原因。现在急需从变异毒株中筛选新的疫苗毒株,用来防控PED的流行。
[1] 斯特劳 B E.猪病学[M].9版,赵德明,张仲秋,沈建忠,主译,北京:中国农业大学出版社,2008:399-403.
[2] Chasey D,Cartwright S F.Virus-like particles associated with porcine epidemic diarrhea [J].Res Vet Sci,1978,25(2):255-256.
[3] Pensaert M B,Debouck P.A new coronavirus-like particles associated with diarrhea in swine[J].Arch Virol,1978,58 (3):243-247.
[4] 宣 化,邢德坤,王殿瀛,等.应用猪胎肠单层细胞培养猪流行性腹泻病毒的研究[J].中国人民解放军兽医大学学报,1984,4(3):202-208.
[5] Sun R Q,Cai R J,Chen Y Q,et al.Outbreak of porcine epidemic diarrhea in sucking piglets,China[J].Emerg Infect Dis,2012,18(1):161-163.
[6] 郑逢梅,霍金耀,赵军,等.2010-2012年华中地区猪流行性腹泻病毒分子特征和遗传进化分析[J]病毒学报,2013,29(2):197-205.
[7] 刘艳成.内蒙古猪病毒性腹泻流行病学调查及猪流行性腹泻病毒M、S基因序列分析[D].内蒙古呼和浩特市:内蒙古农业大学,2015.
[8] Park S J,Kim H K,Song D S,et al.Complete genome sequences of a Korean virulent porcine epidemic diarrhea virus and its attenuated counterpart [J].J Virol,2012,86(10):59-64.
[9] 董彦鹏,白 娟,范宝超,等.猪流行性腹泻病毒RT-PCR检测与ORF3基因分子流行病学调查[J].中国预防兽医学报,2013,35(7):555-558.
[10] 顾超逸,孙 裴,施雷.安徽省猪流行性腹泻病毒的ORF3基因序列分析[J].西北农林科技大学学报(自然科学版),2015,43(2):1-6.
[11] 黄冬妮,黄宗良,马春全,等.广东省猪流行性腹泻病毒ORF3和M基因的克隆与序列分析[J].黑龙江畜牧兽医(科技版),2015(3):136-139.
Analysis of Complete Genome Sequence of Porcine Epidemic Diarrhea Virus CH-SXCH11-2015
LIU Wen-jun,FAN Zhen-hua,WANG Juan-ping,YAO Jing-ming,WU Xin,MENG Fan,HAN Yi-chao,XUE Yi-peng,MI Rui-juan,LI Hong-li,ZHAO Yue
(InstitutionofAnimalHusbandryandVeterinary,ShanxiAcademyofAgriculturalSciences,Taiyuan030032,China)
Through the RT-PCR method,the complete genome sequence of CH-SXCH11-2015 was amplified,and the sequence data were assembled and analyzed by using Lasergene software (DNAstar Inc.,USA).The complete genome sequence of PEDV CH-SXCH11-2015 is 28 038 nucleotides (nt) in length,and has 5 bp longer than that of CV777.The highest sequence homology of the complete genome sequence of CH-SXCH11-2015 with that of BJ-2011-1 was 97.6%,and the lowest sequence homology of CH-SXCH11-2015 with LZC and SM98 was 96.6%.The length of S gene of CH-SXCH11-2015 was 4161 nucleotides,and was 9 bp longer than that of CV777 strain.The mutations of the S gene of CH-SXCH11-2015 occurred mainly in the S1 region.The CH-SXCH11-2015 had the highest nucleotide homology of 98.9% with BJ-2011-1 and CH/ZJJS-Z/2012,had the lowest nucleotide homology of 93.6% with SM98,and had the nucleotide homologies of 94.2%,93.8% with CV777,vaccine strain-CV777,respectively.The amino acid homology of S gene of CH-SXCH11-2015 was highest of 98.3% with CH/ZJJS-Z/2012,and the lowest homology of 91.8%with CV777vaccine and SQ2012,and the homology was 92.9% with CV777.The ORF3 gene of CH-SXCH11-2015 was 675bp in length,and no nucleotide insertion and deletion.The ORF3 gene of CH-SXCH11-2015 had the highest nucleotide homology of 99.3% with CH/HBQX/10,had the lowest nucleotide homology of 93.6% with CV777 truncated,and had the nucleotide homologies of 96.6% and 95.1% with CV777 and attenuated DR13,respectively.The amino acid homology of ORF3 gene of CH-SXCH11-2015 was highest of 98.7% with CH/HBQX/10,CH/S,CH/SHH/06,CH/TJ-2012,CH/ZKXH/11,CPF299,GZGY-10,PFF188,PFF285,and the lowest homology of 88%withCV777 truncated,and the homologies were 97.6% and 97.1% with CV777 and attenuated DR13.Phylogenetic analysis showed that complete genome sequence,S gene,ORF3 gene of CH-SXCH11-2015 have close relationship with the strains isolated in China after 2010,and have genetically quite distant(far genetic relationship) from CV777,and 2 different strains of vaccine strains (attenuated DR13,CV777vaccine).
Porcine epidemic diarrhea virus; complete genome sequence; sequence analysis
2016-09-28
山西省科技攻关项目(20140311020-2-A);山西省科技攻关项目(20150311018-3)
刘文俊(1979-),男,山西河津人,助理研究员,硕士,主要从事动物疫病分子生物学研究。*通讯作者
S852.65
A
1007-5038(2017)06-0006-07
