银杏MYB转录因子的鉴定及特征分析
2017-06-15石晓雯孙岩丁娜孙惠英王婷贾小云李润植
石晓雯,孙岩,丁娜,孙惠英,王婷,贾小云*,李润植*
(1.山西农业大学,山西 太谷 030801; 2.山西省农科院果树研究所,山西 太谷 030800)
银杏MYB转录因子的鉴定及特征分析
石晓雯1,孙岩1,丁娜1,孙惠英2,王婷1,贾小云1*,李润植1*
(1.山西农业大学,山西 太谷 030801; 2.山西省农科院果树研究所,山西 太谷 030800)
[目的]MYB蛋白是真核生物中一类重要的转录因子,在植物形态建成、生长发育、初生和次级代谢产物合成等生命过程中起重要调控作用。银杏作为植物中的“活化石”,其提取物中含有的活性成分次生代谢物类黄酮等对人体有益。在类黄酮的生物合成中,MYB转录因子扮演着重要角色。本研究从转录组水平详细鉴定和分析了银杏GbMYB转录因子,为今后GbMYB的功能研究奠定基础,也为其他物种MYB转录因子的分析提供方法。[方法]本文首先利用BlASTp和PlantTFbcat等生物信息学工具对银杏的MYB蛋白序列进行筛选和鉴定;其次运用MEME和MEGA 7.0软件分别对GbMYB蛋白进行基序和系统进化分析;最后使用MeV对GbMYB在银杏各组织中的表达水平进行聚类分析。[结果]从41151个银杏蛋白中共鉴定出60个MYB转录因子,根据MYB保守域的特点将其分为R1-MYB、R2R3-MYB和R1R2R3-MYB三大类。系统进化树分析发现60个GbMYB可分为16个亚家族且具有相似功能的蛋白序列通常聚在一个家族。在银杏GbMYB蛋白中共检测到21种不同的保守基序。表达谱数据分析表明,一些GbMYB基因的表达具有组织特异性。[结论]本研究利用生物信息学方法,在银杏转录组水平上共鉴定出参与调控银杏各个组织发育的60个GbMYB转录因子;结合拟南芥AtMYB家族分类法将GbMYB分为16个亚家族;保守基序分析显示GbMYB在功能上具有多样性。研究结果为全面解析银杏MYB基因结构与生物学功能提供了新信息,也为进一步研究银杏GbMYB转录因子在次生代谢产物中的调控功能奠定了基础。
MYB转录因子; 银杏; 类黄酮; 转录组
转录因子又称反式作用因子,是与真核生物基因的顺式作用元件结合并调控下游基因表达的一类蛋白分子。植物中常见的转录因子有:NAC、AP2、HD-ZIP、b-ZIP、APF、WRKY、APF、MYB等。MYB是植物中种类最多且功能最重要的转录因子家族之一。MYB家族成员的典型特征是有一段由50~53个氨基酸构成的保守性极强的DNA结合域(即MYB domain,用R表示)。在该结构域中每隔18或19个氨基酸有1个保守的色氨酸(W),参与疏水核心的组成,对维持MYB转录因子构象的稳定起主导作用。依据MYB蛋白含有的结构域数量不同将其分为4类,即R1R2R3-MYB、R2R3-MYB、R1-MYB及4R-MYB[1]。尽管不同的MYB蛋白在结构和序列上存在着相似性和保守性,但功能各异。这种差异会表现在不同物种之间或在同一个体的不同组织器官之间。在植物中发现的第一个MYB转录因子是玉米穗基因C1编码的参与花青素代谢合成的c-myb-like[2]。随着MYB转录因子在不同植物中被陆续发现,对其功能的研究也日益增多。现发现MYB类转录因子对植物的整个生命过程都具有重要的意义,包括细胞形态建成[3]、生长发育[4]、对生物和非生物胁迫的应答[5,6]以及植物初生和次生代谢[7]等。
尤其MYB转录因子参与调控次级代谢产物类黄酮的生物合成在其他植物中多次被报道。Qi[8]和Yuan[9]等人将黄芩MYB转录因子SbMYB2,SbMYB7和SbMYB8的过表达载体转入烟草后发现:参与类黄酮生物合成相关基因的表达量增加,植物的抗氧化性明显提高。Xu[10]等人发现银杏GbMYBF2转录因子负调控类黄酮的合成。Yan[11]和PerezDiaz[12]等研究发现,GmMYB100和VvMYB4-like基因分别负调控大豆和葡萄中黄酮类化合物的合成。在苹果中,Espley[13]等发现MdMYB1和MdMYB2与果皮中花青素的合成有关,而MdMYB10调控苹果果实中花青素的合成。
银杏(GinkgobilobaL)属于古老的孑遗植物,已有2.7亿年的生活史,是非开花植物中唯一存活的代表,素有“活化石”的美称[10],其典型的扇形叶性状已保留了数百万年,这种情况在其他幸存的植物物种中很少存在,因此银杏在植物进化史中拥有非常独特的地位。银杏生命力极强,对真菌、虫害和病毒等具有较强的抗性。在草本植物中,银杏叶提取液中含有6%的萜内酯类化合物和24%的类黄酮化合物[14],在全球极为畅销,具有很高的药用价值。类黄酮有很高的抗氧化能力,对心血管疾病和癌症有防治作用。植物中类黄酮来源于苯丙酸合成途径的一个分支,其合成途径主要受一些酶基因和转录因子的调控。因此,弄清银杏类黄酮分子代谢途径,发现并鉴定类黄酮代谢过程中起调控作用的转录因子进而揭示其转录调控机制,有利于对药用植物的进一步开发和利用[15]。
本研究通过对药用植物数据库中银杏的蛋白序列进行生物信息学分析,共鉴定出60个银杏GbMYB转录因子,并依据其所含有的MYB domain 数目将其分为3类。通过对银杏MYB转录因子蛋白序列进行基序、聚类分析、系统进化以及表达分析,为今后进一步解析银杏MYB转录因子的结构和功能奠定基础,也为其他物种的MYB转录因子的研究提供方法。
1 材料与方法
1.1 蛋白序列的收集与整理
银杏(GinkgobilobaL.)和拟南芥(Arabidopsisthaliana)的蛋白序列分别从药用植物数据库(Medicinal Plant Genomics Resource,http://medicinalplant genomics.msu.edu/)和拟南芥信息资源数据库(The Arabidopsis Information Resource,http://www.arabidopsis.org/)中下载。将获得的蛋白序列用Notepad++软件进行编辑。若某蛋白有不同的序列,选择最长的氨基酸序列。并将蛋白序列的编号进行了简化处理,如>gba_locus_10001简记为>gba10001。然后将编辑好的蛋白序列拷贝到Excel文件中,作为后续分析的蛋白序列数据库。
1.2 银杏GbMYB转录因子的筛选、鉴定和验证
将NCBI数据库中已知的银杏MYB蛋白进行序列比对,得到一段由104个氨基酸组成的高度保守的GbMYB片段。用该片段对上述编辑好的银杏蛋白数据库中的序列进行BLASTP比对,初步确定MYB转录因子的数目。为保证获得的MYB转录因子的准确性,将上述所鉴定的蛋白序列提交至CDD (http://www. ncbi.nlm.nih.gov/cdd/)数据库进行鉴定。最后,将含有MYB功能域的序列在PlantTFbcat (http://plantgrn.noble.org/ PlantTFcat/)中进行比对分析,进一步验证其是否属于MYB类转录因子。
1.3 银杏GbMYB转录因子的分类与结构域基序分析
对验证正确的MYB蛋白序列进行SMART (http://smart.embl-heidelberg.de/) 批量搜索[16],根据所含有的DNA-binding domain的数量对其进行分类。利用MEGA 7.0对银杏中数量最多的R2R3-MYB类蛋白序列进行比对,并保存比对结果。然后将得到的比对结果删除不保守区域后,利用weblogo(http://weblogo.berkeley.edu/logo.cgi)对R2R3-MYB的DNA domain进行结构域特征分析[17]。
1.4 银杏MYB转录因子保守基序组成
GbMYB蛋白基序用MEME 4.8.0 (http://meme.nbcr.net/meme/intro.html)在线软件进行分析[18]。具体参数为:基序位点数量1~50;基序重复次数为any;每个基序的长度为6~70个氨基酸残基;预测基序的数量最多为21个。
1.5 银杏R2R3-MYB家族进化分析
为进一步研究银杏MYB 转录因子的功能,运用MEGA7.0软件采用邻近法(Neighbor-Joining)构建包含125个拟南芥MYB转录因子及55个银杏MYB转录因子(其中有五条序列由于同源性相差太大,剔除)的系统发育树。其中Model设置为p-distance, Bootstrap值设为3 000,其余参数设为默认。
1.6 银杏GbMYB表达分析
银杏转录组测序的表达数据从MPGR数据库下载。GbMYB基因在根、熟果、幼果、叶和茎中表达数据系统聚类及其表达模式分析选用MeV4.9.0软件,聚类结果中红色和绿色分别表示与对照相比表达上调和下调的GbMYB基因。
2 结果与分析
2.1 银杏MYB转录因子的鉴定
为鉴定银杏GbMYB转录因子,首先从药用植物数据库中检索到41 511个单拷贝蛋白序列。然后用已知的银杏MYB蛋白与前面得到的序列进行比对,经BLASTP鉴定得到191条含有MYB结构域的蛋白序列。最后,去除短序列及冗余转录本序列后,剩余蛋白序列进一步经NCBI在线保守域分析和PlantTFbcat转录因子软件鉴定,最终得到银杏MYB蛋白序列共60条。
2.2 银杏MYB蛋白序列的分类与结构域特征分析
对得到的60条银杏MYB蛋白序列进行批量SMART搜索后按照所含有的 DNA binding domain的数量进行分类。含有1个DNA binding domain的MYB为R1-MYB,同理含有2个 DNA binding domain的MYB 为R2R3-MYB 类转录因子,含有3个DNA binding domain 的MYB为R1R2R3-MYB 类转录因子,如图1。共鉴定出 R1-MYB 12个,R2R3-MYB 47个,R1R2R3-MYB 1个,而没有发现包含4R-MYB类的转录因子。
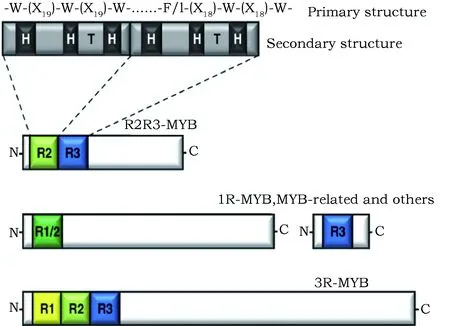
图1 银杏MYB 转录因子分类示意图Fig.1 Classification of GbMYB transcription factors W: 色氨酸残基;X: 其它氨基酸残基;F: 苯丙氨酸残基; I: 异亮氨酸残基。W: Tryptophane; X: Other amino acid; F: Phenylalanine; I: Isoleucine.
利用weblogo在线分析软件对银杏中数量最多的R2R3-MYB类转录因子的DNA结合结构域进行特征分析。氨基酸出现频率越大在图中对应位点的字母长度越高,表明氨基酸残基越保守(图2)。在长度约为51个氨基酸残基的R2结构域中含有3个高度保守的色氨酸残基(分别位于第4位、24位和44位),且每个色氨酸残基间隔18~19个氨基酸(图2)。在R3结构域中第一个色氨酸残基往往会被苯丙氨酸(F)所替代(图2中第57位),第2个和第3个色氨酸残基仍具有高度保守性(图中第76位和95位)。这些都与其他物种已鉴定出的MYB结构域的特点一致[19]。除了高度保守的色氨酸残基外,R2和R3结构域中也存在大量其他的保守氨基酸残基,如在结构域起始位置的赖氨酸(K)、精氨酸(R)、甘氨酸(G)以及在R2结构域中的GKSCRL序列和R3结构中存在LPGRTDN序列。这些保守氨基酸残基可能与色氨酸残基共同维持MYB转录因子形成的螺旋-转角-螺旋(HTH)结构的稳定,进而保证能与DNA分子大沟结合行使其功能。
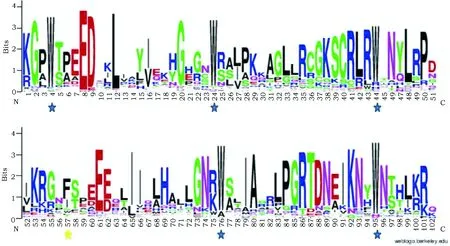
图2 银杏R2R3-MYB转录因子DNA结合结构域特征示意图Fig.2 Structural characteristics of R2R3-MYB DNA binding domain of Ginkgo biloba
2.3 银杏MYB转录因子的聚类分析
为鉴定GbMYB的功能,采用Neighbor-Joining法分析55个银杏MYB蛋白和125个拟南芥MYB蛋白序列之间的系统发育关系。依据Dubos[20]对拟南芥MYB蛋白的分类方法,将银杏MYB蛋白序列分为16个亚家族G1-G16(图3)。在这16个亚家族中G8亚家族含有的成员数目最多,共有8个GbMYB,而G7亚家族仅含有一个GbMYB。本研究发现银杏中的G8亚族与拟南芥S7亚族可聚成一类,Stracke[21]的研究表明拟南芥S7亚族中的成员(AtMYB11、AtMYB12和AtMYB111)可参与调控黄酮类化合物的合成;Huang[22]等人通过实验证实传统中药材淫羊藿中与AtMYB111聚在一类的EsMYBF1同样能调控类黄酮的合成。同样在Nakatsuka[23]等人的研究中也发现与拟南芥S7亚族成员有较高亲缘性的GtMYBP3、GtMYBP4参与调控龙胆花中黄酮类化合物的生物合成。因此,我们推测银杏G8亚族与拟南芥S7亚族可能具有相同的功能。银杏G1、G4、G3亚家族与拟南芥中发育相关的MYB转录因子[24~26]聚在一起,推测这几类GbMYB可能与银杏的细胞发育相关。G15和G12亚族可能与银杏的非生物胁迫有关,因为与G15聚在一起的AtMYB60和AtMYB96通过ABA信号途径调控气孔移动分别响应干旱和病害胁迫[27~29],而与G12聚成一族的AtMYB15参与ABA信号转导途径响应冷害胁迫[30]。
2.4 银杏GbMYB基序组成分析
MYB转录因子含有很多的保守序列,如图2中R2和R3结构域中保守的氨基酸残基,这些保守序列可能与DNA分子的某些部位相结合,在表达调控过程中起重要作用。为识别银杏GbMYB家族中潜在的保守基序,我们通过MEME在线分析软件对GbMYB蛋白序列进行了基序组成分析,如图4中所示:共鉴定出21个保守基序(表1)。Motif 1是MYB蛋白N端结构域,几乎存在于所有的银杏GbMYB中(图4)。相对N端来说,C端的Motif的易变性较高,每条序列不尽相同,大约有18到250个核苷酸的差异,这些特异性的基序预示着不同亚族可能具有不同的功能[31]。位于同一进化分支的GbMYB蛋白含有相同的保守基序种类且位置大体一致,推测这些转录因子的功能可能相似。不同进化分支中GbMYB蛋白保守基序种类和位置差异较大。gba196、gba57103、gba3518没有与其他的任何GbMYB蛋白序列聚到一起,他们的基序组成也与其他分支的蛋白序列存在较大差异(图4)。银杏GbMYB基序组成分析结果也表明了同一亚族的蛋白序列在结构上是保守的。
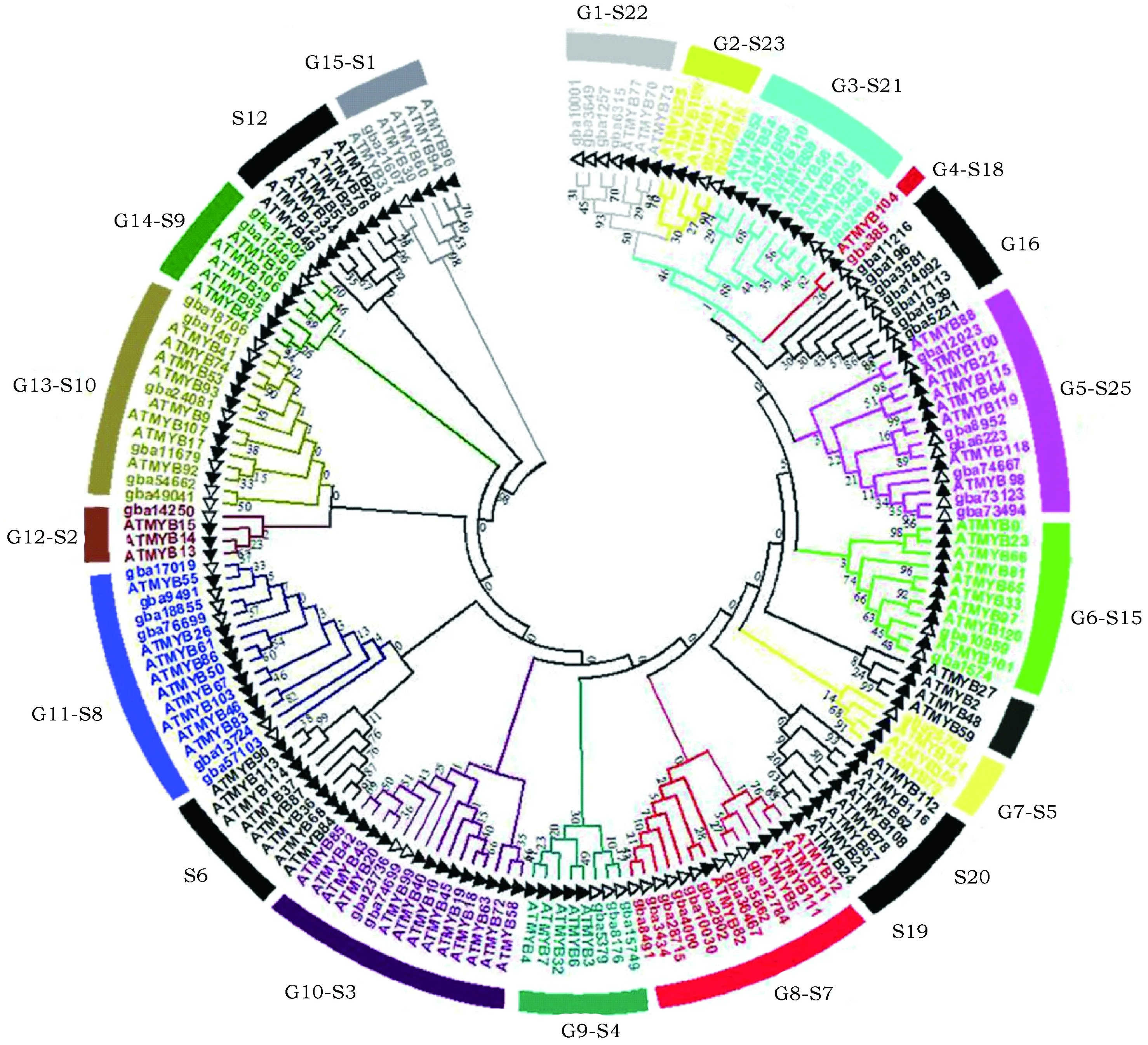
图3 银杏和拟南芥MYB蛋白聚类分析Fig.3 Cluster analysis of MYB protein from Ginkgo biloba and Arabidopsis thaliana
2.5GbMYB基因的表达分析
为进一步了解银杏GbMYB转录因子的生物学功能,我们使用Mev软件对GbMYB在根、茎、叶、幼果、熟果中的表达进行了聚类分析并绘制热图。如图5所示,有16个GbMYB在叶片组织中的表达量较高,7个GbMYB在茎中的表达量较高,11个GbMYB在根中表达较高,3个GbMYB基因在根和茎中都有表达,11个GbMYB在熟果中没有检测到表达量。以上不同GbMYB基因在不同组织中的差异表达情况表明GbMYB转录因子可能参与调控银杏各个组织的发育。
3 讨论
MYB蛋白是植物中数量最多的一类转录因子家族,现已在多种植物中被鉴定出来,如:拟南芥、水稻、玉米和大豆[32]。通常,MYB转录因子依据所含的DNA结合结构域的个数可被分为R1-MYB、R2R3-MYB、R1R2R3-MYB和4R-MYB四类。而在植物中最常见的是R2R3-MYB类转录因子。转录水平的调控是目前研究最多的,也是生物体最重要的调控方式。本研究首次在转录组水平上利用PlantTFbcat和BLASTp在线软件对银杏MYB转录因子进行了鉴定和筛选。我们共鉴定出60个MYB转录因子,其中1R-MYB类成员的数目占20%,R2R3-MYB类占78%,而R1R2R3-MYB类只含有1个,而没有4R-MYB类转录因子,这种分布比例与拟南芥、小麦和玉米等基本相似[33]。但是从总数上来说本研究鉴定的GbMYB蛋白相对较少,可能是因为本次分析是在银杏的转录组水平,可用的基因组信息量较少,所以导致一些GbMYB未被鉴定。对47条银杏R2R3-MYB类转录因子的保守域基序分析表明其含有MYB家族典型的DNA binding domain。
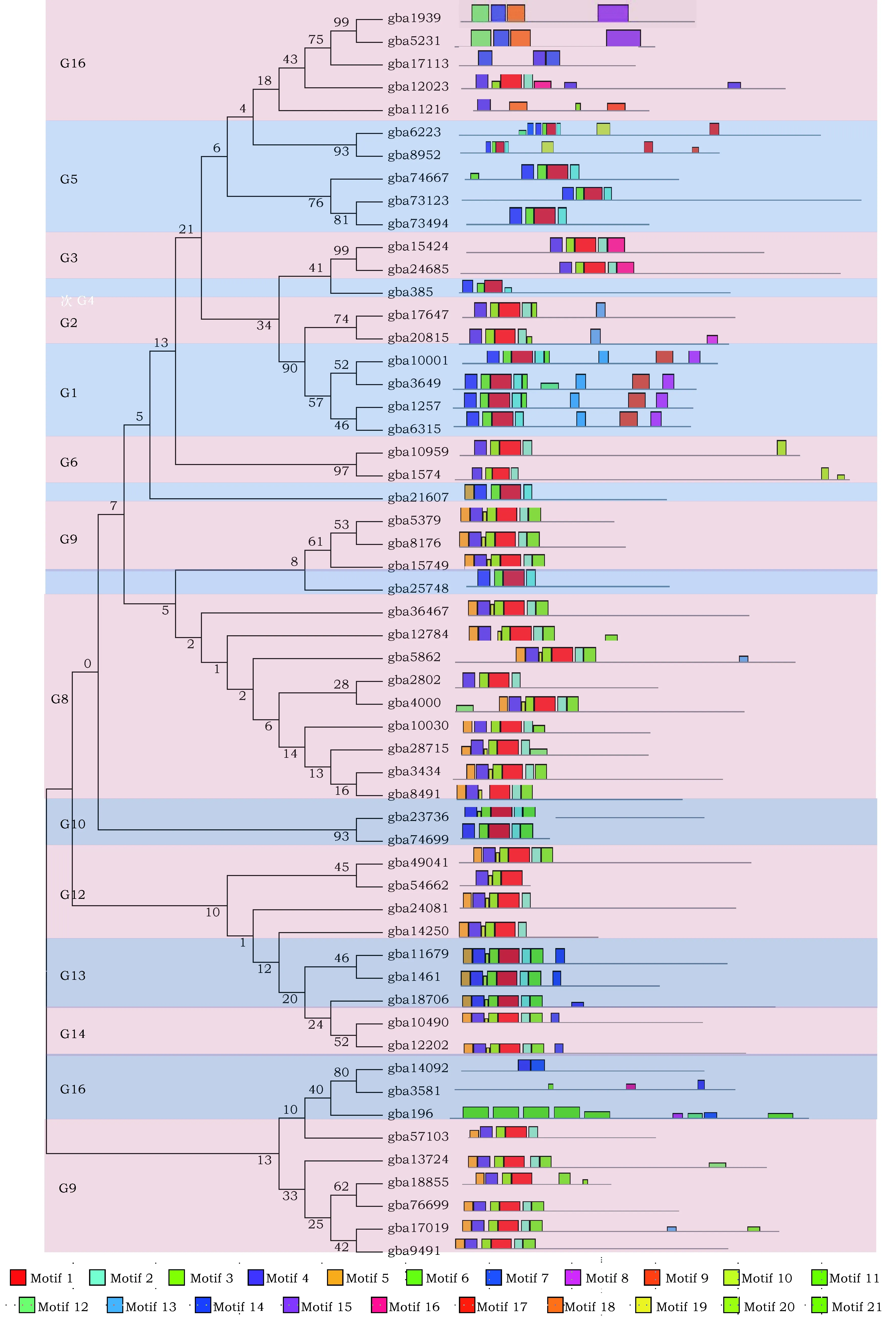
图4 银杏GbMYB蛋白序列的MEME保守基序及进化分析Fig.4 Phylogenetic analyses of conserved motifs of GbMYB
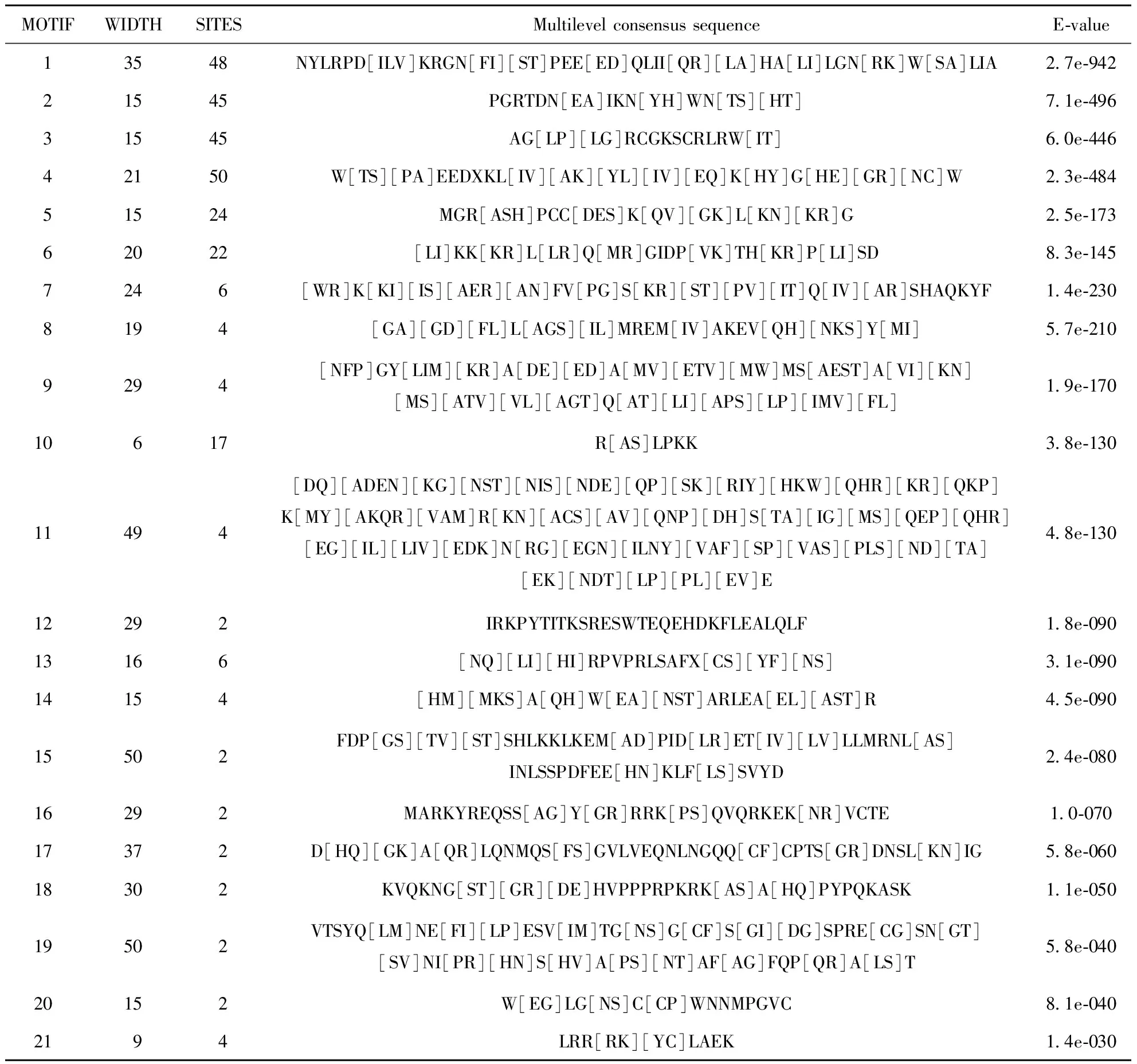
MOTIFWIDTHSITESMultilevelconsensussequenceE⁃value13548NYLRPD[ILV]KRGN[FI][ST]PEE[ED]QLII[QR][LA]HA[LI]LGN[RK]W[SA]LIA27e⁃94221545PGRTDN[EA]IKN[YH]WN[TS][HT]71e⁃49631545AG[LP][LG]RCGKSCRLRW[IT]60e⁃44642150W[TS][PA]EEDXKL[IV][AK][YL][IV][EQ]K[HY]G[HE][GR][NC]W23e⁃48451524MGR[ASH]PCC[DES]K[QV][GK]L[KN][KR]G25e⁃17362022[LI]KK[KR]L[LR]Q[MR]GIDP[VK]TH[KR]P[LI]SD83e⁃1457246[WR]K[KI][IS][AER][AN]FV[PG]S[KR][ST][PV][IT]Q[IV][AR]SHAQKYF14e⁃2308194[GA][GD][FL]L[AGS][IL]MREM[IV]AKEV[QH][NKS]Y[MI]57e⁃2109294[NFP]GY[LIM][KR]A[DE][ED]A[MV][ETV][MW]MS[AEST]A[VI][KN][MS][ATV][VL][AGT]Q[AT][LI][APS][LP][IMV][FL]19e⁃17010617R[AS]LPKK38e⁃13011494[DQ][ADEN][KG][NST][NIS][NDE][QP][SK][RIY][HKW][QHR][KR][QKP]K[MY][AKQR][VAM]R[KN][ACS][AV][QNP][DH]S[TA][IG][MS][QEP][QHR][EG][IL][LIV][EDK]N[RG][EGN][ILNY][VAF][SP][VAS][PLS][ND][TA][EK][NDT][LP][PL][EV]E48e⁃13012292IRKPYTITKSRESWTEQEHDKFLEALQLF18e⁃09013166[NQ][LI][HI]RPVPRLSAFX[CS][YF][NS]31e⁃09014154[HM][MKS]A[QH]W[EA][NST]ARLEA[EL][AST]R45e⁃09015502FDP[GS][TV][ST]SHLKKLKEM[AD]PID[LR]ET[IV][LV]LLMRNL[AS]INLSSPDFEE[HN]KLF[LS]SVYD24e⁃08016292MARKYREQSS[AG]Y[GR]RRK[PS]QVQRKEK[NR]VCTE10⁃07017372D[HQ][GK]A[QR]LQNMQS[FS]GVLVEQNLNGQQ[CF]CPTS[GR]DNSL[KN]IG58e⁃06018302KVQKNG[ST][GR][DE]HVPPPRPKRK[AS]A[HQ]PYPQKASK11e⁃05019502VTSYQ[LM]NE[FI][LP]ESV[IM]TG[NS]G[CF]S[GI][DG]SPRE[CG]SN[GT][SV]NI[PR][HN]S[HV]A[PS][NT]AF[AG]FQP[QR]A[LS]T58e⁃04020152W[EG]LG[NS]C[CP]WNNMPGVC81e⁃0402194LRR[RK][YC]LAEK14e⁃030
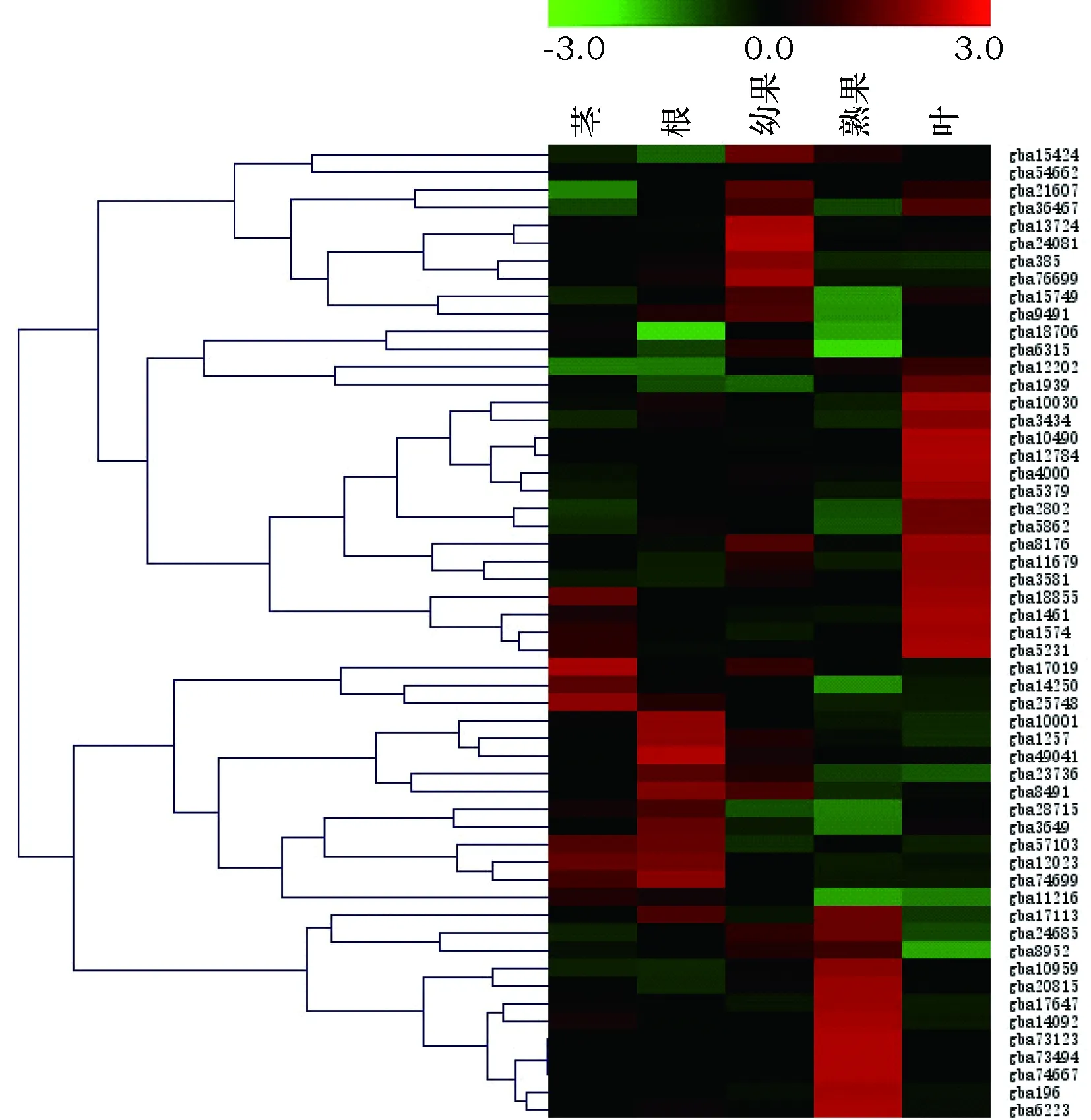
图5 不同组织中GbMYB基因的表达谱Fig.5 Expression profiles of GbMYB genes in Ginkgo biloba between different tissues 注:红色和绿色分别表示与对照相比表达上调和下调的GbMYB基因Note:Blocks with red and green indicate upregulated and downregulated transcript accumulation of GbMYB relative to the control, respectively.
拟南芥作为一种模式生物,关于其MYB转录因子的研究已经较为成熟。为进一步了解银杏MYB转录因子的功能我们用拟南芥的AtMYB蛋白和银杏GbMYB蛋白序列构建了系统进化树,发现GbMYB蛋白划分为16个亚家族。银杏中没有一个MYB蛋白序列与拟南芥的S12、S6、S19和S20亚家族聚在一起,可能是银杏在基因组进化过程中发生了基因的丢失现象。最新研究表明拟南芥S12亚家族来源于早期分化出的芸苔属植物基因复制事件[34]。在拟南芥中,这类家族调控芥子油苷的生物合成,而这类次级代谢产物主要分布在芸苔属植物中,所以在银杏中没有MYB转录因子跟其聚在一起,相似的研究结果在白梨[35]和大豆[31]中都有报道。另一方面,银杏的gba196,gba3518等序列没有与拟南芥的任何AtMYB亚家族聚合,而是单独聚在一起组成新的亚家族G16,这或许是该物种为适应环境而发生了基因组数量的增加,其机理还有待研究。我们对部分聚在一起的MYB转录因子进行了功能预测,为今后研
究银杏GbMYB 的功能奠定了基础。GbMYB蛋白保守基序的分析表明,银杏GbMYB的保守基序多达21个,各亚家族成员间的保守基序组成和分布都有所不同,表明GbMYB蛋白具有生物学上的多功能性和可塑性。进一步对银杏GbMYB的表达量进行聚类分析发现,不同基因在不同组织中的表达模式不同,表明银杏GbMYB参与调控各个组织的发育。感兴趣的是,G8亚族的成员可能参与类黄酮的合成,而且G8亚族的GbMYB基因在叶中有较高的表达量,推测可能与银杏叶中类黄酮含量较多相关[10]。
本研究为进一步全面解析银杏MYB转录因子的生物学功能提供了参考,尤其为银杏GbMYB对类黄酮物质合成的调控机理奠定了基础。银杏2.7亿年的生活史使其在生命进化过程中具有独特的历史地位,因此对其转录因子的研究也可为解析树木进化和生命进化的早期事件提供一定的理论依据。
4 结论
本研究为进一步全面解析银杏MYB转录因子的生物学功能提供了参考,尤其为银杏GbMYB对类黄酮物质合成的调控机理奠定了基础。银杏2.7亿年的生活史使其在生命进化过程中具有独特的历史地位,因此对其转录因子的研究也可为解析树木进化和生命进化的早期事件提供一定的理论依据。
[1]Rosinski J A, Atchley W R. Molecular evolution of the MYB family transcription factors:evidence for polyphyletic origin[J].Journal of Molecular Evolution, 1998, 46(1):74-83.
[2]Paz-Ares J, Ghosal D, Wlenand U, et al. The regulatory c1 locus of Zea mays encodes a protein with homology to myb proto-oncogene products and with structural similarities to transcriptional activators[J]. Embo Journal, 1987, 6(12):3553-3561.
[3]Perez-Rodriguez M, Jaffe F W, Butelli E, et al. Development of three different cell types is associated with the activity of a specific MYB transcription factor in the ventral petal of Antirrhinum majus flowers[J]. Development, 2005, 132(2):359-370.
[4]Higginson T, Li SF, Parish RW. At MYB103 regulates tapetum and trichome development in Arabidopsis thaliana[J]. Plant Journal, 2003, 35(2):177-192.
[5]Hartmann U, Sagasser M, Mehrtens F, et al. Differential combinatorial interactions of cis-acting elements recognized by R2R3-MYB, BZIP, and BHLH factors control light-responsive and tissue-specific activation of phenylpropanoid biosynthesis genes[J]. Plant Molecular Biology, 2005, 57(2):155-171.
[6]Jung C, Seo JS, Han SW, et al. Overexpression of At MYB44 enhances stomatal closure to confer abiotic stress tolerance in transgenic Arabidopsis[J]. Plant Physiology, 2008, 146(2):623-625.
[7]Goicoechea M, Lacombe E, Legay S, et al. EgMYB2, a new transcriptional activator from Eucalyptus xylem,regulates secondary cell wall formation and lignin biosynthesis[J].Plant Journal, 2005, 43(4):553-567.
[8]Qi L, Yang J, Yuan Y, et al. Overexpression of two R2R3-MYB genes from Scutellaria baicalensis induces phenylpro panoid accumulation and enhances oxidative stress resistance in transgenic tobacco[J]. Plant Physiology & Biochemistry, 2015, 94(4):235-243.
[9]Yuan Y, Qi L, Yang J, et al. A Scutellaria baicalensis, R2R3-MYB gene,SbMYB8, regulates flavonoid biosynthesis and improves drought stress tolerance in transgenic tobacco[J]. Plant Cell, Tissue and Organ Culture (PCTOC), 2015, 120(3):961-972.
[10]Feng Xu, Yingjing Ning, Weiwei Zhang, et al. An R2R3-MYB transcription factor as a negative regulator of the flavonoid biosynthesis pathway in Ginkgo biloba[J]. Functional & Integrative Genomics, 2014, 14(1):177.
[11]Yan J, Wang B, Zhong Y, et al. The soybean R2R3 MYB transcription factor GmMYB100 negatively regulates plant flavonoid biosynthesis[J]. Plant Molecular Biology, 2015, 89(1):35-48.
[12]Pérez-Rodríguez P, Riao-Pachón DM, Corrêa LG, et al. PlnTFDB: updated content and new features of the plant transcription factor database[J]. Nucleic Acids Research, 2009, 38:D822-829.
[13]Espley RV, Hellens RP, Putterill J, et al. Red colouration in apple fruit is due to the activity of the MYB transcription factor MdMYB10[J]. The Plant Journal,2007,49(3):414-427.
[14]van Beek T, Montoro P. Chemical analysis and quality control of Ginkgo biloba leaves, extracts, and phytopharmaceuticals[J]. Journal of Chromatography A, 2009, 1216(11):2002-2032.
[15]杨致荣,王兴春,薛金爱,等.药用植物长春花WRKY转录因子的鉴定及表达谱分析[J].生物工程学报,2013,29(6):785-802.
[16]Schultz J, Milpetz F, Bork P, et al. SMART, a simple modular architecture research tool: Identification of signaling domains[J].Proceedings of the National Academy of Sciences of the United States of America,1998,95(11): 5857-5864.
[17]Crooks G E, Hon G, Chandonia JM, et al. Web Logo: A sequence logo generator[J]. Genome Research, 2004, 14(6):1188-1190.
[18]Bailey TL, Johnson J, Grant CE,et al. The MEME suite[J].Nucleic Acids Res,2015,43(W1): W39-W49.
[19]成舒飞,端木慧子,陈超,等.大豆MYB转录因子的全基因组鉴定及生物信息学分析[J].大豆科学,2016,35(1):52-57.
[20]Dubos C, Stracke R, Grotewold E, et al. MYB transcription factors in Arabidopsis[J]. Trends in Plant Science, 2010, 15(10):573-81.
[21]Stracke R, Ishihara H, Huep G, et al. Differential regulation of closely related R2R3-MYB transcription factors controls flavonol accumulation in different parts of the Arabidopsis thaliana seedling[J]. Plant Journal,2007,50:660-677.
[22]Huang W, Khaldun A B, Chen J, et al. A R2R3-MYB Transcription Factor Regulates the Flavonol Biosynthetic Pathway in a Traditional Chinese Medicinal Plant, Epimedium sagittatum[J]. Frontiers in Plant Science, 2016, 7(e70778).
[23]Nakatsuka T, Saito M, Yamada E, et al. Isolation and characterization of GtMYBP3 and GtMYBP4, orthologues of R2R3-MYB transcription factors that regulate early flavonoid biosynthesis, in gentian flowers[J]. Journal of Experimental Botany, 2012, 63(18):6505-6517.
[24]Ryoung Shin, Adrien Y, Burch, et al. The Arabidopsis transcription factor MYB77 modulates auxin signal transduction[J]. Plant Cell, 2007, 19(8):2440-2053.
[25]Keller T, Abbott J, Moritz T, et al. Arabidopsis REGULATOR OF AXILLARY MERISTEMS1 controls a leaf axil stem cell niche and modulates vegetative development[J]. Plant Cell, 2006, 18(3):598-611.
[26]Müller D, Schmitz G, Theres K. Blind Homologous R2R3 Myb Genes Control the Pattern of Lateral Meristem Initiation in Arabidopsis[J]. Plant Cell, 2006, 18(3):586-597.
[27]Cominelli E, Galbiati M, Vavasseur A, et al. A guard-cell-specific MYB transcription factor regulates stomatal movements and plant drought tolerance[J].Current Biology, 2005, 15(13):1196-1200.
[28]Seo P J, Xiang F, Qiao M, et al. The MYB96 Transcription Factor Mediates Abscisic Acid Signaling during Drought Stress Response in Arabidopsis[J]. Plant Physiology, 2009, 151(1):275-289.
[29]Seo P J, Park C M. MYB96-mediated abscisic acid signals induce pathogen resistance response by promoting salicylic acid biosynthesis in Arabidopsis[J]. New Phytologist, 2010, 186(2):471-483.
[30]Reyes J L, Chua N H. ABA induction of miR159 controls transcript levels of two MYB factors during Arabidopsis seed germination[J]. Plant Journal for Cell & Molecular Biology, 2007, 49(4):592-606.
[31]Du H, Yang S S, Liang Z, et al. Genome-wide analysis of the MYB transcription factor superfamily in soybean[J]. BMC Plant Biology, 2012, 12(1):106.
[32]曹忠慧.苹果MYB转录因子家族的生物信息学分析及MdMYB121和MdSIMYB1在非生物胁迫响应中的作用[D].泰安:山东农业大学,2013.
[33]刘淑君.番茄MYB转录因子的鉴定及其表达分析[D].杭州:浙江师范大学,2012.
[34]Yanhui C,Xiaoyuan Y,Kun H,et al. The MYB transcription factor superfamily of Arabidopsis: expression analysis and phylogenetic comparison with the rice MYB family[J]. Plant Molecular Biology, 2006, 60(1):107-124.
[35]Xiaolong Li,Cheng Xue,Jiaming Li,et al.Genome-Wide Identification, Evolution and Functional Divergence of MYB Transcription Factors in Chinese White Pear (Pyrus bretschneideri). International Journal of Molecular Sciences, 2015, 16(12):28683-28704.
(编辑:武英耀)
Identification and characterization of MYB transcription factors inGinkgobiloba
Shi Xiaowen1, Sun Yan1, Ding Na1, Sun Huiying2, Wang Ting1, Jia Xiaoyun1*, Li Runzhi1*
(1.CollegeofAgriculture,ShanxiAgriculturalUniversity,Taigu030801,China; 2.ShanxiAcademyofAgriculturalSciences,Taigu030800,China)
[Objective] MYB is one of the largest families of transcription factors in plants and participates in kinds of biological processes, such as, plant development, cell morphogenesis and the synthesis of primary and secondary metabolites.Ginkgobilobais called “living fossil” in plant and contains many secondary metabolites,such as flavonoid. MYB transcription factors play an important role in flavonoid biosynthesis. In this study, MYB transcription factor ofginkgowere identified at the transcriptome level. The result will lay the foundation for functional study of GbMYB and identification of MYB from other species. [Methods] Firstly, MYB transcription factors inginkgowere screened and identified by BlASTP and PlantTFbcat. Sencondly, MEME and MEGA 7.0 software were used to analyze motifs and evolution ofginkgoMYB, respectively. Lastly, the expression patterns ofGbMYBin different tissues ofginkgowere analyzed by using MeV software. [Result] In our study, a total of 60 GbMYB were identified from 41151 proteins inginkgoand classified into three distinct groups (R1-MYB, R2R3-MYB and R1R2R3-MYB) according to the structure of MYB domain. Phylogenetic analysis showed these MYB genes were classified into 16 subgroups. Clearly, GbMYBs with the similar functions were gathered together. There were 21 different conserved motifs in GbMYB. Expression profiles analysis showed that the expression ofGbMYBhad tissue specificity.[Conclusion] Our study identified 60 GbMYB at the transcriptome level, which participate in the development of different tissues. The GbMYB was classed into 16 subfamilies by referring to AtMYB. The result of conserved motif analysis showed that GbMYB have diverse functions. Our research provided new message for comprehensively analyzing the structure and function of GbMYB and laid the foundation for the further study of GbMYB in the regulating of secondary metabolites.
MYB transcription factors,Ginkgobiloba, Flavonoid, Transcriptome
2016-12-15
2017-02-20
石晓雯 (1992-),女 (汉族),山西吕梁人,硕士研究生,研究方向:植物生物化学与分子生物学
*通信作者:贾小云,教授,博士生导师,Tel:13935439653;E-mail:gssjxy2001@hotmail.com;李润植,教授,博士生导师,Tel:13593105815:E-mail: rli2001@hotmail.com
国家自然科学基金(31101555);山西农业大学研究生创新项目(20143058);山西省高等学校优秀青年学术带头人项目(2012);山西人才引进与开发专项(614191)
S664.3;Q946.1
A
1671-8151(2017)05-0351-09
