T7噬菌体展示人肝癌cDNA文库筛选人CD8+T淋巴细胞特异结合分子
2017-06-09高凯丽李维娜薛晓畅张存郝强张伟张英起
高凯丽,李维娜,薛晓畅,张存,郝强,张伟,张英起
1.肿瘤生物学国家重点实验室;2.第四军医大学 药学系生物制药学教研室;陕西 西安 710032
T7噬菌体展示人肝癌cDNA文库筛选人CD8+T淋巴细胞特异结合分子
高凯丽1,2,李维娜1,2,薛晓畅1,2,张存1,2,郝强1,2,张伟1,2,张英起1,2
1.肿瘤生物学国家重点实验室;2.第四军医大学 药学系生物制药学教研室;陕西 西安 710032
目的:利用噬菌体表面展示技术从人肝癌T7 cDNA文库中筛选出能与人CD8+T淋巴细胞特异结合的蛋白分子。方法:以人CD8+T淋巴细胞为筛选靶标,对人肝癌T7 cDNA噬菌体肽库进行4轮“吸附-洗脱-扩增”生物淘选与富集,ELISA鉴定特异结合的噬菌体单克隆;对获得的阳性克隆裂解液进行PCR扩增及DNA测序,然后进行生物信息学分析,确定所编码的结合蛋白。结果:经过4轮筛选,结合人CD8+T淋巴细胞的噬菌体克隆得到富集,通过测序与同源性比对分析获得17个特异性结合的多肽序列;生物信息学分析发现阳性噬菌体克隆的同源蛋白中,可能与肝癌免疫逃逸相关的分子有Unc-51样自噬活化激酶1(ULK1)、一般受体磷酸肌醇相关支架蛋白1(GRASP)、中间α球蛋白抑制因子H1(ITIH1)、富含亮氨酸重复8家族成员D(LRRC8D)和CD164等。结论:从人肝癌T7 cDNA噬菌体肽库中筛选获得了能与人CD8+T淋巴细胞特异结合的分子,为阐明人肝癌发生发展过程中肿瘤免疫逃逸相关机制奠定了基础。
噬菌体展示技术;肝癌;CD8+T淋巴细胞;结合分子
恶性肿瘤严重危害着人类的健康和生命,已成为人类的重要死因。肿瘤免疫治疗是肿瘤治疗的一个有前景的新方向,其目的是激发或调动机体的免疫系统,增强肿瘤微环境的抗肿瘤免疫力,从而控制和杀伤肿瘤细胞。CD8+T淋巴细胞是机体免疫系统中的重要组成部分,也是主要的肿瘤浸润T淋巴细胞亚群[1],在机体抗肿瘤免疫应答中发挥了关键作用[2],可通过释放穿孔素和颗粒酶,直接杀伤肿瘤细胞。
近年来针对肿瘤免疫的机制研究发现,肿瘤患者体内存在免疫抑制机制[3],使肿瘤微环境中的免疫细胞的表型及功能发生改变,突出表现为T淋巴细胞处于抑制状态、免疫学活性降低、增殖能力减弱,导致机体免疫应答低下,从而妨碍机体对肿瘤的清除[4]。肿瘤微环境中CD8+T淋巴细胞功能受到抑制的现象已被广泛关注,但肿瘤究竟通过何种机制影响CD8+T淋巴细胞抗肿瘤免疫应答仍不清楚。
噬菌体表面展示技术常用于研究蛋白质之间的相互作用。T7噬菌体展示肽库的特点是通过把外源基因序列插入编码T7噬菌体包膜蛋白的基因,使外源基因编码的多肽与噬菌体包膜蛋白融合表达,并展示在噬菌体表面。通过亲和筛选,能够特异性地富集和筛选出含有目的多肽的噬菌体重组子,进一步获取其插入的编码核苷酸序列,从而从cDNA展示文库中分析鉴定出相互作用蛋白[5]。为了系统筛选肝癌细胞所表达的影响CD8+T淋巴细胞功能的肿瘤细胞相关分子,我们通过噬菌体表面展示这一高通量筛选技术,从人肝癌cDNA噬菌体展示肽库中筛选CD8+T淋巴细的相互作用分子,为进一步研究肿瘤细胞与CD8+T淋巴细胞相互作用从而引起肿瘤免疫微环境改变及免疫逃逸的新机制奠定基础。
1 材料与方法
1.1 材料
人肝癌T7 cDNA噬菌体展示文库、T7噬菌体宿主菌大肠杆菌BLT5403(Novagen公司);人淋巴细胞分离液(天津灏洋生物制品科技有限责任公司);MACS人CD8微珠(Mitenyi Biotec公司);FITC-抗人CD3单克隆抗体、APC-抗人CD8单克隆抗体(BD Pharmingen公司);RPMI1640培养基与小牛血清(Gibco公司);琼脂糖(博大科技公司);TaqDNA聚合酶(Promega公司);DNA提取试剂盒(TaKaRa公司);T7重组噬菌体中插入片段引物由南京金斯瑞生物科技有限公司合成,上游引物序列为5'-GGAGCTGTCGTATTCCAGTC-3',下游引物序列为5'-AACCCCTCAAGACCCGTT TA-3'。
1.2 人外周血CD8+T淋巴细胞的分选与体外培养
从第四军医大学西京医院血库领取健康志愿者外周血白膜,用RPMI1640培养基稀释后用淋巴细胞分离液进行密度梯度离心,吸取中间云雾状细胞层获得外周血单个核细胞。细胞计数后用美天旎公司磁珠分选试剂盒分离CD8+T淋巴细胞。
1.3 CD8+T淋巴细胞的表型鉴定
收集磁珠分选的CD8+T细胞,计数并调整细胞密度后加入离心管,每管1×106细胞。分别加入CD3、CD8单抗,4℃孵育30 min,流式细胞洗液洗涤固定后行流式细胞仪分析。
1.4 原始文库滴度测定
将大肠杆菌BLT5403接种于LB培养液,37℃、200 r/min培养至对数生长期;用气溶胶阻隔枪头取10μL噬菌体文库溶液,以LB培养液进行1/10梯度稀释,各梯度稀释产物各取100μL,加至含250μL BLT5403菌液的试管中,快速涡旋混匀,室温孵育5 min,加人3 mL 45℃顶层琼脂糖凝胶中混匀,倾至预热的LB培养板,37℃倒置培养;当噬菌斑长至1~2 mm时,计数平板上的噬菌斑生成数目,按下式计算噬菌体滴度:滴度=∑(各平板的噬菌斑个数×该平板的稀释率×10)/平板数。
1.5 人肝癌T7 cDNA噬菌体肽库亲和筛选
计数1×106个CD8+T淋巴细胞,加入含2% BSA的DPBS,室温条件下封闭2 h;TBS洗涤后加入10μL人肝癌T7 cDNA噬菌体肽库,孵育30 min;TBST洗涤去除非特异性结合的噬菌体,加入浓度为1%的SDS缓冲液200μL,振荡15 min进行特异性结合噬菌体洗脱,离心取50μL洗脱上清,加入BLT5403菌液中,37℃、200 r/min扩增至菌液澄清;分别对未扩增的洗脱液和扩增后噬菌体上清留样进行噬菌体滴度测定,进行下一轮筛选。共重复筛选4次,获得高度富集的噬菌体。
1.6 细胞ELISA初步鉴定特异性结合的噬菌体阳性克隆
新鲜分选的CD8+T淋巴细胞用3%脱脂奶粉于37℃封闭2 h,用封闭液稀释细胞并调整细胞密度,按1×106/孔接种于96孔酶联板,将噬菌体单克隆扩增上清分别加入各细胞孔及PBS孔(空白对照)中,37℃孵育1 h;TBST洗涤,1500 r/min离心15 min;加入T7 Fiber单克隆抗体,37℃孵育1 h,洗涤后加入HRP标记的羊抗小鼠二抗,37℃孵育0.5 h,洗涤后用邻苯二胺底物(OPD)显色,2 mol/L硫酸中止反应,酶标仪测定D490nm值。D490nm值高于阴性对照3倍以上为阳性克隆。
1.7 噬菌体单克隆的PCR扩增、测序
挑选40个经细胞ELISA鉴定能特异性结合CD8+T淋巴细胞的噬菌体单克隆,分别加入宿主菌中,37℃培养至彻底澄清,用10 mmol/L EDTA稀释后于65℃加热10 min进行裂解;以裂解液为模板,采用PCR对特异噬菌体的插入序列进行扩增(PCR条件:94℃预变性5 min;94℃变性1 min,50℃退火1 min,72℃延伸1 min,30个循环;72℃延伸10 min);通过1%琼脂糖凝胶电泳鉴定扩增结果,并送金斯瑞公司用 Applied Biosystems 3730-XL测序仪进行测序。
1.8 序列分析及蛋白功能预测
对筛选富集得到的噬菌体重组子PCR产物进行序列测定,通过BLASTn程序搜索NIH的GenBank数据库进行同源序列查找[6]。利用Inter Pro Scan(http://www.ebi.ac.uk/InterProScan/)及http: //www.expasy.org在线预测蛋白功能。
2 结果
2.1 CD8+T淋巴细胞的纯度检测
用免疫磁珠分选法从健康志愿者外周血白膜中获得人CD8+T淋巴细胞,采用流式细胞术对其进行纯度检测,结果如图1,所得CD8+T淋巴细胞纯度高于95%。
2.2 人肝癌cDNA文库的4轮生物淘选
为筛选出人肝癌中能与人CD8+T淋巴细胞特异结合的分子,以CD8+T淋巴细胞为筛选靶标,通过4轮“吸附-洗脱-扩增”生物淘选与富集,完成人肝癌T7噬菌体展示肽库的筛选。每轮的噬菌体投入量保持一致,第4轮与第1轮相比,富集了8.4倍(富集倍数=第4轮产出率/第1轮产出率,产出率=产出的噬菌体数量/投入的噬菌体数量),结果显示阳性噬菌体克隆有一定的有效富集(表1)。
2.3 细胞ELISA初步鉴定噬菌体阳性克隆
利用细胞ELISA对筛选后的噬菌体单克隆进行初步鉴定,检测其与CD8+T淋巴细胞的结合情况。随机挑选10个第3轮筛选后的单克隆,编号为1~10;随机挑选10个第4轮筛选后的单克隆,编号为11~20。结果如图2,除克隆3外,其余各克隆的CD8+T淋巴细胞实验组D值与PBS空白对照组D值之比大于3,提示它们能与CD8+T淋巴细胞较好地结合。第3轮筛选所得其余9个噬菌体克隆中有3个D值较高,第4轮筛选所得10个噬菌体克隆中有8个D值较高,说明筛选过程有良好的富集效果,4轮筛选后最终获得的是能与CD8+T淋巴细胞特异性结合的噬菌体阳性克隆。

图1 人CD8+T淋巴细胞纯度检测结果

表1 噬菌体富集结果
2.4 目的基因的PCR扩增
挑选40个经细胞ELISA鉴定能特异性结合CD8+T淋巴细胞的高亲和力噬菌体单克隆,转入宿主菌中扩增,以其裂解液为模板,用T7 Select引物进行PCR检测,结果显示大小不等的DNA条带。部分阳性克隆的电泳结果见图3。
2.5 DNA序列测定与同源性比对分析
对上一步得到的PCR产物进行序列测定,测序结果经BLASTn程序进行同源性搜索,排除重复序列,共筛选出17个与CD8+T淋巴细胞特异性结合的蛋白质,同源性为99%~100%。经文献检索及生物信息学分析,发现阳性噬菌体克隆的同源蛋白中,可能与肝癌免疫逃逸相关的分子有:Unc-51样自噬活化激酶1(ULK1)、一般受体磷酸肌醇相关支架蛋白1(GRASP)、中间α球蛋白抑制因子H1(ITIH1)、富含亮氨酸重复8家族成员D(LRRC8D)和CD164等。
3 讨论
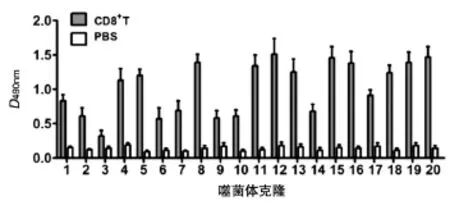
图2 ELISA鉴定噬菌体阳性克隆与CD8+T淋巴细胞的亲和力

图3 部分阳性噬斑PCR扩增产物
肿瘤组织局部免疫反应在抗肿瘤免疫过程中起重要作用,对肿瘤的发生、发展和转移具有重要影响。近年来,以免疫调节为基础的肿瘤免疫治疗因其良好的临床疗效,得到越来越多的重视和认可。然而研究发现,肿瘤细胞可以通过多种机制逃避免疫系统的监控、攻击,从而在体内生存,继而生长和转移,即肿瘤的免疫逃逸。
T细胞在免疫应答过程中具有关键作用,T细胞免疫在机体抗肿瘤过程中扮演重要角色。T淋巴细胞包括CD4+和CD8+T细胞。CD4+T细胞具有辅助细胞免疫和体液免疫应答的作用,CD8+T细胞则是重要的效应细胞,是抗肿瘤免疫反应过程中最主要的效应细胞,发挥着核心作用。CD8+T细胞具有细胞毒性作用,可释放穿孔素和颗粒酶,直接杀死靶细胞,导致靶细胞的死亡。
近年来在对肿瘤免疫逃避的机制研究中发现,肿瘤免疫微环境可以改变肿瘤的生物学特性、筛选适应微环境的肿瘤细胞存活,并通过分泌多种炎症因子、生长因子等构成免疫抑制网络,诱导免疫效应细胞的表型发生改变,负调控其免疫活性,使其无法有效清除体内肿瘤细胞;并且能促使具有抑制功能的免疫细胞在肿瘤内部不断蓄积,建立适宜的肿瘤微环境促进肿瘤进展。荷瘤机体中CD8+T细胞的免疫功能障碍是调控肿瘤逃逸免疫的重要机制,是限制肿瘤免疫治疗的关键因素。在肿瘤复杂的微环境中,肿瘤细胞表达、分泌大量分子,究竟是哪些表面分子或因子影响了CD8+T细胞的免疫功能,仍有待研究。筛选肿瘤细胞表达或分泌的能与人CD8+T淋巴细胞特异结合的分子,将有助于阐明人肝癌发生发展过程中诱导CD8+T淋巴细胞无能或功能障碍,从而促进肿瘤免疫逃逸的相关机制。
噬菌体展示技术是一种高效的分子相互作用筛选体系,具有库容大、高通量、快速、简便的特点,在生命科学理论和应用研究中都有广泛的应用[7-8]。Novagen公司的T7噬菌体载体能将外源基因序列插入编码T7噬菌体包膜蛋白的基因中,使得外源多肽或蛋白与噬菌体包膜蛋白融合表达并展示在噬菌体表面。通过3~5轮亲和筛选,能够从cDNA展示文库中特异性地富集和扩增能与靶标结合的噬菌体,分离和鉴定出阳性噬菌体重组子,获取插入序列。噬菌体展示肽库最常用的策略是以纯化的特定蛋白为靶标进行筛选,以整体细胞为靶标的筛选是一个新的方向,为相关研究提供了全新的思路[9]。
本课题的策略是以整个CD8+T细胞为靶标筛选人肝癌T7 cDNA噬菌体肽库,获得肿瘤细胞表达的能与CD8+T细胞特异结合的多肽。经过4轮筛选,保持一定的噬菌体投入量,回收率逐轮提高,表明阳性噬菌体克隆得到良好富集。利用细胞ELISA方法,对筛选后随机挑选的阳性克隆进行初步鉴定,获得了能与CD8+T细胞特异结合的高亲和力噬菌体阳性克隆;进而对高亲和力克隆进行DNA序列测定与同源性比对分析,排除重复序列,共筛选出17个与CD8+T淋巴细胞特异性结合的蛋白质,其中包括ULK1、GRASP、ITIH1、LRRC8D和CD164等有意义的分子。已知这些蛋白参与机体内多种生理、生化反应,推测可能与肝癌免疫逃逸相关。后续工作将以此为基础,进一步探讨这些分子对CD8+T细胞表型、分化发育、免疫功能、细胞毒活性等方面的影响,为阐明人肝癌发生发展过程中通过影响CD8+T淋巴细胞引起肿瘤免疫逃逸相关机制奠定基础,对提升肿瘤的免疫治疗效果具有潜在临床应用价值。
[1]Matkowski R,Gisterek I,Halon A.The prognostic role of tumor-infiltrating CD4 and CD8 T lymphocytes in breast cancer[J].Anticancer Res,2009,29(7): 2445-2451.
[2]Virgin H W,Wherry E J,Ahmed R.Redefining chronic viral infection[J].Cell,2009,138(1):30-50.
[3]Baitsch L,Fuertes-Marraco S A,Legat A,et al.The three main stumbling blocks for anticancer T cells [J].Trends Immunol,2012,33(7):364-372.
[4]Yi J S,Cox M A,Zajac A J.T-cell exhaustion:characteristics,causes and conversion[J].Immunology,2010, 129(4):474-481.
[5]Rosenberg A,Mierendorf R.T7 Select phage display system:a powerful new protein display system based on bacteriophage T7[J].Novation,1996,6:1-6.
[6]Altschul S F,Madden T L,Schaffer A A,et al. Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J].Nucleic Acids Res,1997,25(17):3389-3402.
[7]Samoylova T I,Morrison N E,Globa L P,et al.Peptide phage display:opportunities for development of personalized anticancer strategies[J].Anticancer Agents Med Chem,2006,6(1):9-17.
[8]Kehoe J W,Kay B K.Filamentous phage display in the new millennium[J].2005,105(11):4056-4072.
[9]Krag D N,Shukla G S,Shen G P,et al.Selection of tumor-binding ligands in cancer patients with phage display libraries[J]. Cancer Res, 2006,66(15):7724-7733.
Screening of Proteins Binding with Human CD8+T Lymphocytes with T7 Select Human Hepatoma cDNA Library
GAO Kai-Li1,2,LI Wei-Na1,2,XUE Xiao-Chang1,2, ZHANG Cun1,2,HAO Qiang1,2,ZHANG Wei1,2,ZHANG Ying-Qi1,2*
1.State Key Laboratory of Cancer Biology;2.Department of Biopharmaceutics,School of Pharmacy,Fourth Military Medical University;Xi'an 710032,China
*Corresponding author,E-mail:zhangyqh@fmmu.edu.cn
Objective:To screen human hepatoma T7 phage cDNA library for proteins binding with human CD8+T lymphocytes by using phage display technique.Methods:By using human CD8+T lymphocytes as the selective target,the T7 select human hepatoma cDNA library was biopanned for four times and positive phage clones were selected.The positive plaques were identified by ELISA and DNA sequencing.Homology analysis and bioinformatics analysis were used to investigate the function of the candidate molecules.Results:T7 phage cDNA inserts were acquired after 4 rounds of biopanning.We have obtained a series of tumor-associated molecules that can specifically bind human CD8+T lymphocytes,such as ULK1,GRASP,ITIH1,LRRC8D and CD164.Conclusion:We obtained some candidate molecules that may interact with human CD8+T lymphocytes through cDNA library phagedisplay method.The present study laid a foundation for further exploring the mechanism of the effect of cancer cells on CD8+T lymphocytes and targeted therapy for hepatoma.
phage display;hepatoma;CD8+T lymphocytes;binding molecules
R392.1;Q78
A
1009-0002(2017)03-0333-05
10.3969/j.issn.1009-0002.2017.03.018
2017-01-06
国家自然科学基金(81672800,81301785,81472649)
高凯丽(1992-),女,硕士研究生,(E-mail)1325202624@qq.com
张英起,(E-mail)zhangyqh@fmmu.edu.cn
