湖北省黄冈地区啮齿动物携带汉坦病毒M片段基因特征分析
2017-05-18刘冰玉倪向荣冉从盾陈禹铭刘东瀛杨占秋
刘冰玉,倪向荣,冉从盾,唐 辉,陈禹铭,刘东瀛,杨占秋
(武汉大学:1基础医学院医学病毒学研究所,病毒学国家重点实验室,2第一临床学院,3第二临床学院,湖北武汉430071)
·预防与转化医学·
湖北省黄冈地区啮齿动物携带汉坦病毒M片段基因特征分析
刘冰玉1,倪向荣2,冉从盾3,唐 辉2,陈禹铭2,刘东瀛1,杨占秋1
(武汉大学:1基础医学院医学病毒学研究所,病毒学国家重点实验室,2第一临床学院,3第二临床学院,湖北武汉430071)
目的:探讨湖北省黄冈地区野生啮齿动物自然感染汉坦病毒(hantavirus)情况及病毒基因分型特征.方法:2012~2013年秋冬季在湖北省黄冈地区的野外及居民区采用夹夜法捕鼠.对捕获的动物进行分类鉴定,取其肺脏提取RNA,合成Seoul病毒(SEOV)和汉滩病毒(HTNV)特异性引物,以RT⁃PCR方法扩增部分M片段核苷酸序列,对PCR产物进行测序,并进行系统发育和序列同源性分析.结果:共捕获啮齿类动物95只,其中褐家鼠75只(78.94%).6份(8.00%)褐家鼠标本SEOV阳性.系统发育分析关系显示当地SEOV属于A组,与Z37株同源性为95.80%~96.30%(核苷酸),97.90%~100%(氨基酸).黄胸鼠和黑线姬鼠SEOV和HTNV均为阴性.结论:湖北省黄冈地区的优势鼠种是褐家鼠,汉坦病毒型别主要是SEOV,其系统发育关系属于A组,且与疫苗株Z37同源性最高.该研究结果有助于为黄冈地区HFRS的预防提供理论依据.
汉坦病毒;啮齿动物;基因;系统发育分析
0 引言
汉坦病毒(Hantavirus,HV)属于布尼亚病毒科汉坦病毒属,为有包膜的负链RNA病毒,其基因组由大(L)、中(M)、小(S)三个片段组成,分别编码RNA聚合酶,包膜表面糖蛋白Gn和Gc,以及核衣壳蛋白.包膜表面糖蛋白Gn和Gc上有多个抗原表位,因此M片段有较大的进化压力[1],变异度较大.
HV感染人类后能引起肾综合征出血热(hemor⁃rhagic fever with renal syndrome,HFRS)和汉坦病毒肺综合征(hantavirus pulmonary syndrome,HPS).亚洲地区主要以HFRS为主[2].目前,在全世界范围内已发现HV有23个种和30个暂定的种[3].HV的自然宿主是啮齿动物,食虫目动物和蝙蝠[4-5].作为自然宿主,其被感染后无明显症状,病毒在体内繁殖,通过排泄物或气溶胶把病毒排出体外并被人类接受,导致感染[6].研究认为,自然宿主长期携带病毒而不患病,只不断向外排毒,是因为病毒长期在中间宿主上生活,为了适应宿主体内环境而相应改变了其遗传物质,完成病毒与自然宿主的共进化,最终导致了一种HV对应一种或亲缘相近的几种自然宿主.即在HV的进化中,共分化[7]和宿主转移[7]均起到重要作用[8].
在我国,HFRS主要由HV中的汉滩病毒(Han⁃taan virus,HTNV)和汉城病毒(Seoul virus,SEOV)两型病毒引起,这两型病毒的主要自然宿主分别是黑线姬鼠和褐家鼠.由于HV与宿主动物间的对应关系,宿主动物的类型及其感染情况决定了疫区流行病毒的型别和流行强度.因此,弄清当地流行的HV基因型别是采取有效防制策略的先决和必要条件.
近年来,每年全球约有30 000人感染HV而致病[9],中国是受HV危害最严重的国家[10],而湖北省又是HFRS流行区[11-13],黄冈地区则是其主要疫区之一,但是该疫区内流行的HV的基因型尚不明确.为了研究该地区宿主动物携带HV的阳性率和基因分型特点,本研究在2012~2013年捕获宿主动物,并对其携带的HV进行了M片段扩增、测序以及核酸和蛋白序列分析.
1 材料和方法
1.1 标本采集2012~2013年秋冬季在黄冈地区HFRS流行疫点周围的野外及居民区采用夹夜法,晚放晨取捕鼠.对捕获的鼠类经分类鉴定和登记后,无菌解剖采集鼠肺标本,放置于液氮中保存待检.
1.2 引物的设计及合成根据GenBank中HV基因序列,参考Liu等文献[13],针对HTNV、SEOV分别设计两对M片段特异性引物.SEOV引物(nt 53⁃490):SEO⁃M32F/SEO⁃M343R,SEO⁃M238F/M511R;HTNV(nt 61⁃496)引物:HTN⁃M40F/HTN⁃M282R和HTN⁃M255F/M517R(表1).

表1 汉坦病毒M片段特异性RT⁃PCR引物
1.3 RNA提取、反转录聚合酶链反应(RT⁃PCR)及测序用RNAprep Tissue Kit(Qiagen)从鼠肺中提取RNA.用随机引物和MMLV反转录酶(Promega)合成cDNA.用上述引物扩增部分M片段,并设置阳性对照(SEOV,HTNV病毒标准株),阴性对照(水).具体扩增条件为:94℃预变性5 min,94℃变性45 s,55℃退火45 s,72℃延伸45 s,共30个循环,最后于72℃延伸5 min.PCR产物用1%琼脂糖凝胶电泳后,用凝胶回收试剂盒(Qiagen)进行纯化.用ABI 3730自动测序仪测序.
1.4 系统发育分析用BioEdit软件进行基因序列比对及同源性分析,用MrBayes3.1软件进行种系发育分析,构建系统发育树.其他用于比较的HV序列来自于GenBank数据库.
2 结果
2.1 啮齿动物采集在黄冈地区居民区及野外共捕获啮齿类动物95只,其中褐家鼠75只,黄胸鼠14只,黑线姬鼠6只.褐家鼠占总数的78.94%,为当地的优势鼠种.
2.2 RT⁃PCR在75份褐家鼠中,有6份(8.00%)标本SEOV特异性RT⁃PCR阳性(图1),并进行PCR产物测序.全部褐家鼠标本HTNV特异性RT⁃PCR均为阴性.14份黄胸鼠和6份黑线姬鼠标本,SEOV和HTNV特异性RT⁃PCR均为阴性.
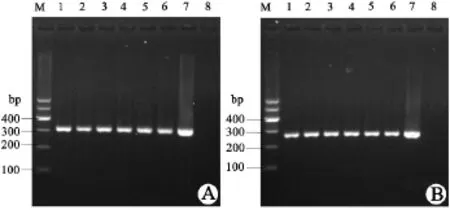
图1 SEOV阳性样本M片段PCR扩增结果
2.3 系统发育分析和同源性分析用SEOV部分M片段核苷酸序列构建的系统发育树见图2.黄冈地区6份SEOV阳性褐家鼠标本,编号分别为:MQ1⁃12,MQ1⁃14,MQ1⁃15,MQ2,MQ2⁃17,MQ2⁃20,在系统发育树中均属于SEOV的A组.其中4份标本(MQ1⁃12,MQ1⁃14,MQ1⁃15,MQ2⁃17)有较高的相似度(核苷酸序列相似度为98.80%~100%,氨基酸序列相似度为98.60%~100%)(表2).另2份标本(MQ2⁃2,MQ2⁃20)与这4份标本的相似度为核苷酸序列相似度为94.50%~95.60%,氨基酸序列相似度为97.20%~100%.黄冈地区SEOV与其他SEOV代表株(80⁃39,R22,Z37,L99)相比较,相似度为核苷酸序列94.50%~96.30%,氨基酸序列95.80%~100%,与Z37病毒同源性最高(核苷酸序列95.80%~96.30%,氨基酸序列97.90%~100%).
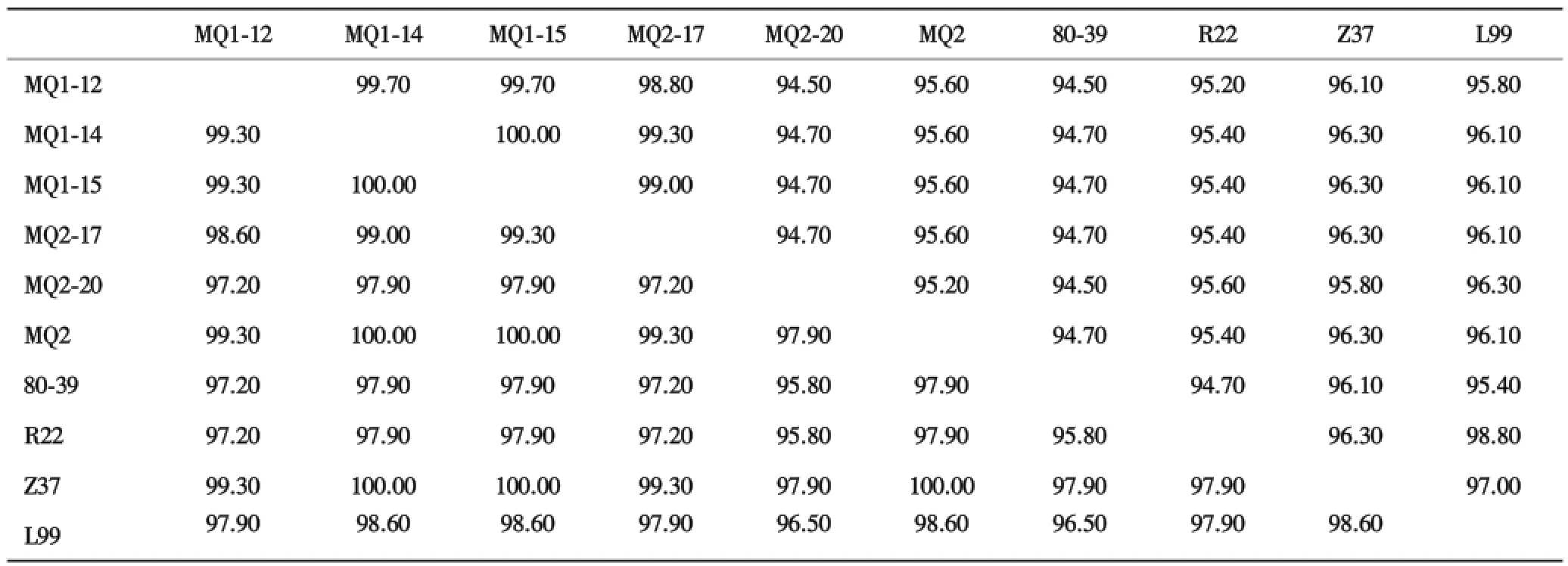
表2 黄冈地区啮齿动物携带SEOV与其他SEOV代表株的核苷酸、氨基酸序列相似度(%)
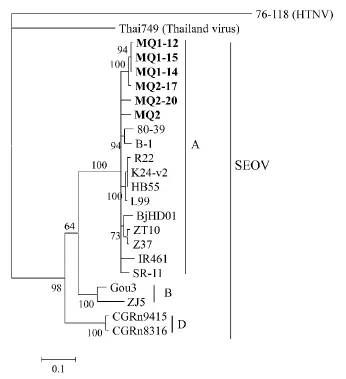
图2 SEOV部分M片段基因系统进化树
3 讨论
湖北省黄冈地区位于湖北省东部、大别山南麓、长江中游北岸.北接河南、东连安徽、南与鄂州、黄石、九江隔江相望.黄冈北部和东部为大别山低山丘陵,海拔多在500~800米;中部为海拔100~250米的丘陵岗地;南部为长江冲积平原,多湖泊.黄冈市自北向南逐渐倾斜、东北部与豫皖交界为大别山脉,主脊呈西北⁃东南走向.黄冈属亚热带大陆性季风气候,江淮小气候区,四季光热界线分明.其生态环境适于鼠类繁殖,是湖北省HFRS的主要疫区之一.
Zhang等[14]关于湖北省HFRS流行病学的研究表明,黄冈地区的HFRS年发病率,1980~1989年为(5~30)/10 000,1990~1999年和2000~2009年降低为(0~5)/10 000.湖北省HFRS空间⁃时间分布的分析表明,在2000~2003年黄冈最有可能为HFRS的聚集地.黄冈地区HFRS发病季节,1980~1989年为秋冬季>70%,1990~1999年和2000~2009年秋冬季发病占30%~70%.HTNV相关的HFRS发病高峰在秋冬季,而SEOV相关的HFRS发病高峰在春季.以上HFRS发病高峰时间的变化提示,在上世纪80年代,黄冈地区HFRS主要是由HTNV引起的,而从上世纪90年代到2009年,SEOV引起的HFRS比例升高.
本研究开展了黄冈地区啮齿动物携带HV的调查,发现当地优势鼠种为褐家鼠,其次还有黄胸鼠和黑线姬鼠.RT⁃PCR检测褐家鼠的HV携带率为8.00%,所携带病毒均为SEOV.本研究未检测到黄胸鼠和黑线姬鼠携带HV.提示黄冈地区HV的自然宿主以褐家鼠为主,病毒分型以SEOV为主.
Lin等[15]关于SEOV的分析表明,所有国外以及中国的大多数SEOV在系统发育树中属于A组,而中国山区的SEOV构成了B,C和D组.A组SEOV起源于中国,然后扩散到欧洲和世界范围.本研究对黄冈地区啮齿动物携带HV进行了部分M片段基因测序,系统发育分析.发现黄冈地区6只褐家鼠携带的SEOV均属于A组,而并未发现属于B、C和D组的SEOV.同源性分析提示黄冈地区SEOV与Z37病毒株同源性最高.
SEOV主要由褐家鼠携带,能引起轻型HFRS,导致人群隐性感染的几率高于HTNV.近几年HV流行病学研究表明我国某些其他地区也是以SEOV为主[16].这提示随着我国城镇化进程的加快,由褐家鼠携带的SEOV的流行有逐渐扩大的趋势.
本研究应用RT⁃PCR、基因测序和系统发育分析的方法,从分子生物学的角度证实了黄冈地区啮齿动物携带的HV以SEOV为主,从而为本地区HFRS的预防控制提供了科学依据.
[1]Sankar S,Upadhyay M,Ramamurthy M,et al.Novel insights on han⁃tavirus evolution:the dichotomy in evolutionary pressures acting on different hantavirus segments[J].PLoS One,2015,10(7):e0133407.
[2]Jiang H,Du H,Wang LM,et al.Corrigendum:hemorrhagic fever with renal syndrome:pathogenesis and clinical picture[J].Front Cell Infect Microbiol,2016,6:178.
[3]King AMQ,Lefkowitz EJ,Adams MJ,et al.Virus Taxonomy:classi⁃fication and nomenclature of viruses.ninth report of the international committee on taxonomy of viruses[M].San Diego:Elsevier,2012:725-741.
[4]Zhang YZ.Discovery of hantaviruses in bats and insectivores and the evolution of the genus Hantavirus[J].Virus Res,2014,187:15-21.
[5]Guo WP,Lin XD,Wang W,et al.Phylogeny and origins of hantavir⁃uses harbored by bats,insectivores,and rodents[J].PLoS Pathog,2013,9(2):e1003159.
[6]Kariwa H,Yoshimatsu K,Arikawa J.Hantavirus infection in East Asia[J].Comp Immunol Microbiol Infect Dis,2007,30(5-6):341-356.
[7]Plyusnin A,Sironen T.Evolution of hantaviruses:co⁃speciation with reservoir hosts for more than 100 MYR[J].Virus Res,2014,187:22-26.
[8]Ramsden C,Holmes EC,Charleston MA.Hantavirus evolution in relation to its rodent and insectivore hosts:no evidence for codiver⁃gence[J].Mol Biol Evol,2009,26(1):143-153.
[9]Watson DC,Sargianou M,Papa A,et al.Epidemiology of Hantavirus infections in humans:a comprehensive,global overview[J].Crit Rev Microbiol,2014,40(3):261-272.
[10]Zhang S,Wang S,Yin W,et al.Epidemic characteristics of hemor⁃rhagic fever with renal syndrome in China,2006-2012[J].BMC In⁃fect Dis,2014,14:384.
[11]Zhu N,Luo F,Chen Q,et al.Influence of HLA⁃DRB alleles on haemorrhagic fever with renal syndrome in a Chinese Han population in Hubei Province,China[J].Eur J Clin Microbiol Infect Dis,2015,34(1):187-195.
[12]Li JL,Ling JX,Liu DY,et al.Genetic characterization of a new subtype of Hantaan virus isolated from a hemorrhagic fever with renal syndrome(HFRS)epidemic area in Hubei Province,China[J].Arch Virol,2012,157(10):1981-1987.
[13]Liu J,Liu DY,Chen W,et al.Genetic analysis of hantaviruses and their rodent hosts in central⁃south China[J].Virus Res,2012,163(2):439-447.
[14]Zhang YH,Ge L,Liu L,et al.The epidemic characteristics and changing trend of hemorrhagic fever with renal syndrome in Hubei Province,China[J].PLoS One,2014,9(3):e92700.
[15]Lin XD,Guo WP,Wang W,et al.Migration of Norway rats resulted in the worldwide distribution of Seoul hantavirus today[J].J Virol,2012,86(2):972-981.
[16]Zhang YZ,Zou Y,Fu ZF,et al.Hantavirus infections in humans and animals,China[J].Emerg Infect Dis,2010,16(8):1195-1203.
Genetic analysis of M segment in hantavirus⁃es carried by wild rodents from the Huang⁃gang area,Hubei province
LIU Bing⁃Yu1,NI Xiang⁃Rong2,RAN Cong⁃Dun3,TANG Hui2,CHEN Yu⁃Ming2,LIU Dong⁃Ying1,YANG Zhan⁃Qiu11Institute of Medical Virology,State Key Laboratory of Virology,School of Basic Medical Sciences,2The First Clinical School,3The Second Clinical School,Wuhan University,Wuhan 430071,China
AIM:To study the infection and genotyping charac⁃teristics of hantavirus in wild rodents from the Huanggang area in Hubei province,China.METHODS:Rodents were captured in residential area and fields with night trapping method in Huang⁃gang,Hubei province during autumn and winter from 2012 to 2013.The species of rodents were identified.Then RNA was extracted from lung tissues.Partial M segments of Seoul virus(SEOV)and hantaan virus(HTNV)were detected by using specific RT⁃PCR.PCR products were sequenced.Phylogenetic relationship and sequence homology were analyzed by using Mr⁃Bayes 3.1 and BioEdit.RESULTS:A total of 95 rodents were captured,including 75(78.94%)Rattus norvegicus.Six(80%)Rattus norvegicus were identified to be SEOV positive.Phyloge⁃netic analysis revealed that the SEOV in Huanggang belongs to the phylogroup A.The nucleotide sequence identities between SEOV in Huanggang and stain Z37 were 95.80%to 96.30%,and amino acid sequence identities were 97.90%to 100%.Rattus flavipectus and Apodemus agrarius were both identified to be SEOV negative and HTNV negative.CONCLUSION:The dominant rodent spe⁃cies was Rattus norvegicus in Huanggang,Hubei province.The predominant hantaviruses carried by these rodents were SEOV,which belonged to phylogroup A,and were closely related with strain Z37.This research is helpful for the prevention of HFRS in Huanggang area,Hubei province,China.
hantavirus;rodent;gene;phylogenetic analysis
R373.9
A
2095⁃6894(2017)04⁃48⁃04
2017-01-12;接受日期:2017-01-28
国家自然科学基金资助项目(81371865,81402728)
刘冰玉.硕士.研究方向:汉坦病毒分子流行病学.
E⁃mail:563318929@qq.com
杨占秋.教授.研究方向:汉坦病毒进化与变异.
E⁃mail:zqyang@whu.edu.cn
