盐胁迫下盐穗木DNA聚合酶λ基因的克隆和表达分析
2017-04-13张冀杜驰张富春
张冀,杜驰,张富春
(新疆大学生命科学与技术学院/新疆生物资源基因工程重点实验室,乌鲁木齐 830046)
盐胁迫下盐穗木DNA聚合酶λ基因的克隆和表达分析
张冀,杜驰,张富春
(新疆大学生命科学与技术学院/新疆生物资源基因工程重点实验室,乌鲁木齐 830046)
【目的】开展盐胁迫下DNA损伤修复基因的表达研究,有助于揭示DNA损伤修复与盐生植物耐盐的相关性。【方法】根据盐胁迫下盐穗木转录组测序结果,利用RACE技术克隆获得了盐穗木DNA损伤修复基因HcDNA聚合酶λ(HcDNApolλ)基因,开放阅读框1 335 bp,编码444个氨基酸。【结果】保守结构域分析显示,HcDNApolλ基因属于DNA聚合酶X家族成员,系统进化分析显示HcDNApolλ为独立的分支,亚细胞定位于细胞核,无信号肽且不含跨膜区域的亲水蛋白。实时荧光定量PCR分析表明,随着NaCl胁迫浓度的增加,同化枝的HcDNApolλ基因的表达逐渐上调,在300 mmol/L NaCl处理 7和14 d时,HcDNApolλ的表达分别增加3和5倍。最后在700 mmol/L NaCl处理14 d时,HcDNApolλ的表达增加20倍,达到峰值。【结论】HcDNApolλ基因的表达受盐胁迫的诱导。
盐穗木;HcDNApolλ基因;盐胁迫;基因表达
0 引 言
【研究意义】植物扎根土壤由于不能移动以及对阳光的依赖性,使得其在维护基因组的完整性方面面临巨大的挑战,因为植物连续暴露在各种生物和非生物的胁迫之下[1],如细菌、病毒、紫外、电离辐射、高盐、化学诱变剂、自由基和体内的烷基化等作用, 各种诱导剂诱导的DNA损伤可能会导致DNA理化性质的改变,从而产生细胞毒害和遗传毒害作用[2]。因此植物只能依靠已经进化出高效广泛的DNA损伤检测和修复机制,来确保其基因组的稳定性进行物种延续。【前人研究进展】DNA双链断裂(DSBs)是DNA损伤中最有毒性的形式之一,由异常的复制和修复引起,因为双链的DNA都发生损伤,因此没有未损伤的DNA链作为复制修复的模板[3]。自发或者环境诱导的DNA双链断裂可能会导致细胞死亡、遗传不稳定和癌症。研究表明,DNA聚合酶X家族主要参与DNA的修复和重组[4],DNApol λ是DNA聚合酶X家族的成员,广泛存在于真核生物中,哺乳动物的DNApol λ是一个核酸外切酶缺陷的单一多肽的DNA聚合酶,与哺乳动物的DNA聚合酶β具有高度的序列同源性[5]。大量的研究表明,这两种聚合酶具有许多共同的生化活性,包括5’端的核糖核酸裂解酶的活性等,在DNA的碱基切除修复中起重要的作用。DNApol λ还有一个较强的作用在非同源的末端重组中连接双链断裂的DNA缺口。在动物细胞中的研究显示DNApol λ可以保护氧化的DNA损伤[6]。基因组广泛的序列分析显示:DNApol λ是植物中聚合酶家族的唯一成员,并且可能参与碱基切除修复,并且在DNA双链断裂的修复中起重要作用[7]。【本研究切入点】盐穗木(Halostachyscaspica)为多年生的藜科灌木,是典型的耐盐植物[8]。通过实验室对盐胁迫下盐穗木转录组测序分析[9],发现DNA损伤应激相关基因是一类上调表达的基因类群,因此选取DNA pol λ基因进行基因克隆和表达分析,期望阐明HcDNApolλ基因在DNA损伤修复中的功能及其与植物抗逆的相关性[10]。【拟解决的关键问题】克隆获得HcDNApolλ基因并进行生物信息学分析,探讨不同盐胁迫下HcDNApolλ基因在不同时间的表达模式,为明确HcDNApolλ基因在盐胁迫下的功能奠定基础。
1 材料与方法
1.1 材 料
于新疆五家渠103团野外盐碱地(E87.31°N44.29°)采集获得盐穗木种子,在实验室播种于花盆中,花土成分为:珍珠岩∶蛭石∶花土=1∶1∶3,并用双子叶植物营养液拌湿。温度控制在(24±2)℃,光照3 000 lx(350 μmol/(m2·s), 16 h/8 h:昼/夜),相对湿度为40%~60%条件下培养120 d左右,并淋浇以1/4 MS(Murashige and Skoog )培养液。
1.2 方 法
1.2.1 盐穗木的盐胁迫处理
选取0、100、300、500 和700 mmol/L 的NaCl胁迫处理,处理前48~72 h在托盘中加入1/4 MS培养液。待托盘中培养液吸收干净无多余水分,将胁迫所用不同浓度NaCl溶液分别淋浇在基质上,直至溶液充满基质溢出于托盘中。将花盆置于处理前的培养环境中,分别用含0、100、300、500 和700 mmol/L NaCl的1/4 MS的培养液培养,其中0 mmol/L NaCl处理作为对照,不同NaCl胁迫浓度处理下的盐穗木同化枝分别在0 h、24 h、72 h、7 d和14 d进行采集用于实时定量表达分析。
1.2.2 Total RNA提取和反转录
将不同浓度NaCl胁迫处理不同时间的盐穗木根和同化枝分别称量200 mg,用总RNA提取试剂盒(北京百泰克公司)提取总RNA,方法参照试剂盒说明书。用ND-2000和1.5%变性PAGE胶测定小RNA浓度和质量,每份组织3个重复。按照1 μg总RNA为模板进行逆转录(20 μL反应体系),以Takara Oligo(dT)作为引物用M-mLV反转录酶合成cDNA。程序:30℃ 10 min;42℃ 60 min;70℃ 15 min。以此cDNA 为模板进行PCR扩增。
1.2.3 盐穗木DNA聚合酶λ(HcDNApolλ)ORF的扩增
根据实验室前期对盐胁迫下盐穗木转录组的测序结果,克隆获得盐穗木DNA损伤修复基因HcDNApolλ的开放阅读框序列,设计上游引物5’GATTAGG AGCGAAGGTATCTGA 3’,下游引物5’ACGCACATCAAAGGTTTCTC3’,以cDNA为模板,进行HcDNApolλ开放阅读框的扩增。PCR反应扩增参数为94℃预变性5 min;94℃变性30 s,62℃退火40 s;72℃延伸90 s,35个循环;72℃延伸10 min,反应产物进行琼脂糖凝胶电泳检测。
按PCR产物纯化试剂盒(Omega) 说明书进行PCR产物的回收。回收的片段与pEASY-T5载体(北京全式金公司)连接,操作按试剂盒说明书进行。连接产物转化E.coliDH5α感受态细胞(北京全式金公司),菌液PCR鉴定出阳性克隆,送生工生物工程(上海)股份有限公司测序。
1.2.4 数据分析
利用DNASTAR 软件对已获得的HcDNApolλ的核酸序列进行分析并推导其氨基酸序列;利用ProtParam(http://web.expasy.org/protparam/)分析氨基酸序列的理化性;利用 TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)分析蛋白质的跨膜结构;利用Protscale(http:// web.expasy.org/protscale/)分析蛋白质亲疏水性;利用NCBI的保守结构域数据库CDD(http:// www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi?RID=1K7GNVX3015&mode=all)分析预测氨基酸序列的保守结构域;利用 PSORT II Prediction(http://psort.hgc.jp/form2.html)和WoLF PSORT(http://wolfpsort. org/)预测蛋白质的亚细胞定位;利用 TargetP(http://www. cbs.dtu. dk/services/TargetP/)分析蛋白潜在信号肽;利用NetPhos(http://www.cbs.dtu. dk/services/NetPhos/)分析蛋白质磷酸化位点;利用MEGA6.0 和DNAman 软件分析其同源性及构建系统发育树。
1.2.5 盐胁迫下HcDNApolλ基因表达检测
根据HcDNApolλ的cDNA序列设计qRT-PCR上游引物(5'-ACTCCTCGGCAATCTTCA TCAAA-3'),下游引物(5'-GAACGGTTTGGGATGGATGCT-3'),以Hcβ-Actin基因作为内参基因,设计上游引物(5'-CCAAAGGCCAACAGAGAGAAGA-3'),下游引物(5'- TGAGACACACC ATCACCAGA-3')。以0、100、300、500和700 mmol/L NaCl胁迫0 h、24 h、72 h、7 d和14 d的盐穗木的根和同化枝的cDNA为模板,取1.0 μL 的cDNA,参照QIAGEN试剂盒说明书。qRT-PCR反应参数为:94℃ 15 s;94℃ 30 s;58℃ 30s;72℃ 30s;40 cycles;72℃ 2 min。经ABI PRISM 7500实时定量PCR仪器检测,数据采用2-ΔΔCt法进行分析。
2 结果与分析
2.1HcDNApolλ基因的克隆及氨基酸同源性分析
利用RACE扩增获得HcDNApolλ基因的全长(图1),开放阅读框为1 335 bp,共编码444个氨基酸(图2),运用MEGA6.0软件对HcDNApolλ氨基酸序列构建系统进化树(图3),结果表明HcDNApolλ在进化上独树一支,与芝麻的亲缘关系最近。图1~3
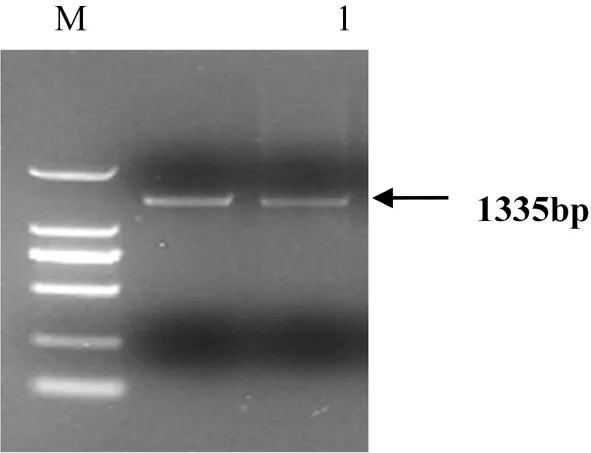
M:2000DNAmaker; 1-2 Amplification ofHcDNApolλORF
图1HcDNApolλ基因ORF扩增PCR产物
Fig.1 The produce of PCR amplification ofHcDNApolλgene

图2 DNA pol λ的核酸序列以及氨基酸序列
Fig.2 DNApolλ nucleotide acid sequence and amino acid sequence
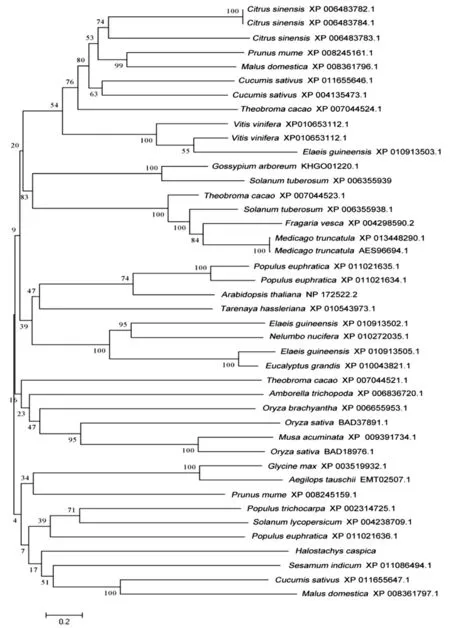
注:分支上的数字表示Bootstrap验证中基于1 000次重复该节点的可信度
Note: The number on the branches represent the reliability percent of bootstraps values based on 1,000 replications
图3 不同植物基于HcDNApolλ氨基酸序列的系统进化树
Fig.3 Phylogenetic tree of different plants based on amino acid ofHcDNApolλ
2.2HcDNApolλ的生物信息学
2.2.1HcDNApolλ氨基酸一级结构及理化性质预测分析
通过ExPASy ProtParam预测HcDNApolλ基因编码的氨基酸序列的组成和理化性质氨基酸残基数444个,正电荷氨基酸数59个,负电荷氨基酸数66个,分子量49 566.7 Da, 等电点pI=6.1, 半衰期h≥10,蛋白质不稳定指数为41.72。根据定义当不稳定系数分值小于40时,预测蛋白质比较稳定;当不稳定系数分值大于40时,则该蛋白质不稳定,因此HcDNApolλ为不稳定的蛋白。表1
2.2.2HcDNApolλ蛋白保守结构域预测
通过NCBI保守结构域数据库(Conserved Domain Database,CDD),分析HcDNApolλ的保守结构域,结果显示该蛋白属于DNA聚合酶X家族,有一个DNA聚合酶X超家族和一个DNA聚合酶β超家族,表明HcDNApolλ参与DNA损伤的修复,包括碱基切除修复、DNA双链断裂的修复和跨损伤的DNA合成等。
2.2.3HcDNApolλ亚细胞定位预测
应用Posort和WOLF POSORT两种工具预测了HcDNApolλ的亚细胞定位,预测结果分析显示,HcDNApolλ可能定位于细胞核、细胞质、线粒体、叶绿体和过氧化物酶体以及细胞骨架。表中显示的两种预测工具的结果一致,推测HcDNApolλ蛋白定位于细胞核的概率最高,同时细胞质和线粒体以及叶绿体中可能也有分布。表1
表1HcDNApolλ亚细胞定位预测
Table 1 Subcellular location of prochymosin ofHcDNApolλ

Posort亚细胞定位Posortsubcellularlocation概率Probability(%)WOLFPOSORT亚细胞定位WOLFPOSORTsubcellularlocation概率Probability(%)细胞核Nuclear43.5细胞核Nuclear5细胞质Cytoolasmic304细胞质Cytoolasmic3线粒体Mitochondria43线粒体Mitochondria2过氧化物酶体Microbody43叶绿体Chloroplast1高尔基体Golgi87过氧化物酶体Microbody1细胞骨架Cytoskeletal43
2.2.4HcDNApolλ信号肽的预测
通过SignalP4.1Server预测HcDNApolλ的信号肽,研究表明HcDNApolλ蛋白的N端1~70个氨基酸序列之间的信号肽分值D=0.100 注:C-score:原始剪切位点分值;S-score:信号肽分值;Y-score:综合剪切位点分值 图4HcDNApolλ信号肽预测 2.2.5HcDNApolλ跨膜结构域预测 跨膜结构域是膜内在蛋白与膜脂相结合的主要部位,它固着于细胞膜上起锚定作用,一般由20个左右的疏水氨基酸组成。通过Expasy软件中的TMHMM Server v.2.0工具预测HcDNApolλ编码氨基酸的跨膜螺旋区。预测结果显示,HcDNApolλ基因编码的氨基酸序列中不存在跨膜螺旋区,444个氨基酸残基全部在膜外,为非跨膜蛋白。图5 图5HcDNApolλ跨膜结构域预测 2.2.6HcDNApolλ的磷酸化位点和活性位点 磷酸化是生物体内一种普遍的调节方式,在细胞信号转导和蛋白调控过程中起着重要的作用。磷酸化位点分析表明,HcDNApolλ有33个丝氨酸、9个苏氨酸、2个酪氨酸磷酸化位点。利用NPS(Network Protein Sequence Analysis)的Prosite Scan 对HcDNApolλ进行活性位点分析。结果显示,HcDNApolλ共有9个活性位点分别是N-糖基化位点、cAMP和cGMP依赖的蛋白激酶磷酸化位点、蛋白激酶C磷酸化位点、酪蛋白激酶2磷酸化位点、酪氨酸蛋白激酶磷酸化位点、N-豆蔻酰化位点、ATP/GTP结合位点基序A、DNA聚合酶X信号,这些活性位点显示HcDNApolλ可能在多个通路过程中发挥作用。表2 表2HcDNApolλ在NPS中的活性位点预测 活性位点Activesite序列号Number基序类型Motiftype氨基酸序列Aminoacidsequence概率值ProbabilityN-糖基化位点N-glycosylationsitePS00001N-{P}-[ST]-{P}1114NVSR120-123NITE380-383NDTL5138e-03cAMP和cGMP依赖的蛋白激酶磷酸化位点PhosphorylationsitesofcAMPandcGMPdependentproteinkinasePS00004[RK](2)-x-[ST]107-110KRVS413-416KRGS1572e-03蛋白激酶C磷酸化位点PhosphorylationsitesofproteinkinaseCPS00005[ST]-x-[RK]25-27SEK51-53STR81-83TSK173-175SLR184-186TGK284-286SYR306-308SHK412-414SKR426-428TEK1423e-02酪蛋白激酶2磷酸化位点Phosphorylationsiteofcaseinkinase2PS00006[ST]-x(2)-[DE]16-19SSGE29-32SFSE62-65SSAE71-74TSDE84-87SFDE173-176SLRD188-191SKLE226-229TLED291-294SCGD300-303THPD334-337SEED340-343SGID378-381TGND426-429TEKE1482e-02酪氨酸蛋白激酶磷酸化位点Phosphorylationsiteoftyrosineproteinki⁃nasePS00007[RK]-x(2,3)-[DE]-x(2,3)-Y397-404RLDETGLYmin=4074e-04max=4083e-04N-豆蔻酰化位点N-myristoylationsitePS00008G-{EDRKHPFYW}-x(2)-[STAGCN]-{P}9-14GGNVSR68-73GTATSD169-174GIGKSL210-215GVGPAT402-407GLYLAT411-416GSKRGS415-420GSRGTA1397e-02ATP/GTP结合位点基序AMotifAofATP/GTPbindingsitePS00017[AG]-x(4)-G-K-[ST]283-290GSYR⁃RGKS7781e-05DNA聚合酶X信号XsignalofDNApolymerasePS00522G-[SG]-[LFY]-x-R-[GE]-x(3)-[SGCL]-x-D-[LIVM]-D-[LIVMFY](3)-x(2)-[SAP]283-302GSYR⁃RGKSSCGDMD⁃FVITHP1082e-11 2.3 qRT-PCR分析盐胁迫下HcDNApolλ基因在同化枝中的表达 采用实时荧光定量PCR(qRT-PCR)方法,以β-actin作为内参,分别检测不同盐浓度胁迫不同时间下盐穗木同化枝HcDNApolλmRNA转录水平的变化。结果表明,100 mmol/L NaCl胁迫处理后,HcDNApolλ在同化枝中的表达量和对照组相比没有明显的变化,而在300 mmol/L NaCl胁迫处理后,HcDNApolλ在同化枝中的表达明显上调,在处理7和14 d后表达量上调了约3和5倍。在300、500和700 mmol/L NaCl胁迫下,HcDNApolλ在同化枝中的表达量均在胁迫14 d 后达到最高,约是对照的5、15和20倍。图6 图6 盐穗木同化枝中HcDNApolλ响应盐胁迫的相对表达 DNA的复制、修复和重组对于活的有机体的遗传信息的复制、传递以及维护基因组的稳定性上起着十分重要的作用[11-12]。DNA依赖的DNA聚合酶在这些过程中起着至关重要的作用[13]。数据表明在人类的基因组中至少有13种类型的DNA聚合酶,根据其氨基酸序列的同源性,DNA聚合酶 可以被分为4个不同的家族(A、B、X和Y)[14]。大量的研究表明,DNA聚合酶X家族主要参与DNA的修复和重组途径[15-17],哺乳动物的DNA聚合酶X家族还包括其他的3个非复制型的聚合酶(聚合酶β,μ和TdT),聚合酶λ的结构主要包括3个功能域:a在N端的BRCT结构域,b在中心区域的DNA结合域,c在C端的DNA聚合酶域。生物化学的研究表明,人类的DNA聚合酶λ有3种酶的活性,DNA聚合酶活性、TdT酶活性以及5’脱氧核糖磷酸酶的活性[18]。尽管DNA聚合酶λ具有DNA聚合酶的活性,但是和复制型的聚合酶相比,它的持续合成的能力较低。这种酶的活性表明,它可能参与两种DNA损伤修复途径:碱基切除修复(BER)和非同源的末端连接途径(NHEJ)[19]。DNA聚合酶和dRT裂解酶的活性在短补丁的BER修复中合成小片段的DNA是必须的,而DNApol λ和XRCC4、lig4以及Ku70/Ku80复合体的相互作用也表明DNApol λ也参与NHEJ。DNApol λ 参与碱基切除修复(BER)通路,在DNA氧化损伤修复中具有重要作用[20]。同时在非同源末端链接(NHEJ)途径填补DNA末端的部分互补双链,起到修复DNA双链断裂中节点作用[21]。 植物基因组测序表明,DNA聚合酶λ是植物聚合酶X家族的唯一成员,可能参与BER途径和NHEJ途径[22]。拟南芥Atpolλ在DNA损伤修复中的功能已经有报道[23],紫外辐射可以诱导Atpolλ的表达,并且拟南芥DNApol λ的突变体表现出对紫外-B和MMC的敏感性,而且突变体还展现出对高盐和MMC处理的敏感性的增加。Atpolλ通过它的N端BRCT结构域可以和Atlig4以及AtXRCC4相互作用[24],表明植物的DNA pol λ在DNA损伤修复中起作用,并且可能通过NHEJ途径参与DSBs的修复。已有的研究表明高盐胁迫或者MMC处理可以诱导拟南芥的基因组的DNA 发生双链断裂,3种拟南芥polλ的突变体(atpol λ-1、atpol λ-2和atpol λ-3)表现出对高盐胁迫和MMC处理的高敏感性,而过表达Atpolλ则会增加突变体对于DNA双链断裂的修复效率[14]。 实验利用RACE技术从盐穗木中克隆获得HcDNApolλ基因,通过核苷酸序列比对显示该基因属于DNA聚合酶X家族的成员;氨基酸序列比对也表明该基因编码的蛋白质具有DAN聚合酶X家族的保守结构域,推测其可能在DNA损伤修复中起作用。HcDNApolλ的跨膜区分析,该蛋白不含跨膜区域且没有信号肽,属于胞内蛋白,亚细胞定位分析定位于细胞核。蛋白质的磷酸化修饰是生物体内重要的共价修饰方式之一,是真核生物细胞中一种普遍的调控手段。活性位点分析表明,HcDNApolλ编码的蛋白质具有多类磷酸化位点,这暗示着HcDNApolλ可能通过磷酸化和去磷酸化来实现活性的调节,实现对DNA双链断裂末端的识别以及缺口的填补。 研究发现在盐穗木中HcDNApolλ受到盐胁迫的诱导表达,特别是在700 mM NaCl 胁迫下,同化枝中的表达量与对照相比显著升高。表明盐穗木可能通过表达HcDNApolλ基因来识别并且修复基因组的DSB损伤,从而维护基因组的完整性来抵御外界的高盐碱以及干旱的胁迫,在基因组的水平上调节生长发育来提高对环境的适应性。 通过对盐穗木中获得的HcDNApolλ基因进行生物信息学分析,并探讨了盐胁迫下HcDNApolλ基因的表达规律,表明HcDNApolλ基因参与盐胁迫的应答。然而HcDNApolλ在盐穗木中盐胁迫下如何参与并修复DSB,这个过程是否与植物细胞周期检验点相关等一系列的问题还有待深入研究。盐穗木HcDNApolλ基因的克隆以及盐胁迫下的组织特异性表达分析,将为进一步研究盐穗木在盐胁迫下植物的抗逆生理机制与DNA损伤修复之间的关系奠定基础。 通过RACE技术克隆获得了盐穗木DNA损伤修复基因HcDNA聚合酶λ(HcDNApolλ)基因的开放阅读框为1 335 bp,编码444个氨基酸。保守结构域分析显示,HcDNApolλ基因属于DNA聚合酶X家族成员,亚细胞定位于细胞核,无信号肽且不含跨膜区域的亲水蛋白。实时荧光定量PCR分析表明,随着胁迫浓度的增加,HcDNApolλ的表达逐渐上调,在300 mmol/L NaCl胁迫处理7和14 d后表达量上调3和5倍,300、500和700 mmol/L NaCl胁迫下同化枝的表达量均在胁迫14 d 后达到最高,分别是对照的5、15和20倍均在14 d时并达到峰值。HcDNApolλ基因的表达受盐胁迫的诱导并影响DNA的损伤修复。 References) [1] Beard, W. A., & Wilson, S. H. (2006). Structure and mechanism of dna polymerase β.ChemInform, 408(21):91-107. [2] Brown, J. A., Pack, L. R., Sanman, L. E., & Suo, Z. (2011). Efficiency and fidelity of human dna polymerases λ and β during gap-filling dna synthesis.DNARepair, 10(1):24-33. [3]Taggart, D. J., Dayeh, D. M., Fredrickson, S. W., & Suo, Z. (2014). N-terminal domains of human dna polymerase lambda promote primer realignment during translesion dna synthesis.DnaRepair, (22):41-52. [4] Uchiyama, Y., Takeuchi, R., Kodera, H., & Sakaguchi, K. (2009). Distribution and roles of x-family dna polymerases in eukaryotes.Biochimie, 91(2):165-170. [5] 冉茂良, 高环, 尹杰, 等. 氧化应激与DNA损伤[J]. 动物营养学报, 2013, 25(10):2 238-2 245. RAN Mao-liang, GAO Huan, YIN Jie, et al. (2013). Oxidative Stress and DNA Injury[J].ChineseJournalofAnimalNutrition, 25(10):2,238-2,245.(In Chinese) [6] Resseguie, E. A., Staversky, R. J., Brookes, P. S., & O'Reilly, M. A. (2015). Hyperoxia activates atm independent from mitochondrial ros and dysfunction.RedoxBiology, (5):176-185. [7] Kondratova, A., Watanabe, T., Marotta, M., Cannon, M., Segall, A. M., & Serre, D., et al. (2015). Replication fork integrity and intra-sphase checkpoint suppress gene amplification.NucleicAcidsResearch, 43(5):2,678-2,690. [8]常 丹,张 霞,张富春.盐穗木HcPEAMT基因的克隆及表达分析[J].西北植物学报,2014,34(8):1 522-1 528. CHANG Dan, ZHANG Xia, ZHANG Fu-chun. (2014). Cloning and Expression Analysis of HcPEAMT Gene from Halostachys caspica [J].ActaBot.Boreal.-Occident.Sin, 34(8): 1,522-1,528. (In Chinese) [9]赵航,贾富强,张富春,等.盐胁迫下盐穗木差异表达基因的转录组信息分析[J]. 生物信息学,2014,12(2):90-98. ZHAO Hang, JIA Fu-qiang, ZHANG Fu-chun, et al. (2014). The transcriptome information analysis of differentially expressed genes of Halostachys caspica under salt stress [J].ChineseJournalofBioinformatics, 12(2): 90-98. (In Chinese) [10]杜驰. 盐胁迫下盐穗木DNA损伤应激通路中相关基因的表达与耐盐性分析[D]. 新疆大学硕士学位论文,2015. DU Chi. (2015).AnalysisofexpressionandsalttoleranceofDNAdamagestresspathway-relatedgenesintranscriptomeofHalostachyscaspicaundersaltstress[D]. Master Dissertation. Xinjiang University, Urumqi. (in Chinese) [11] Liu, W., Mazarei, M., Rudis, M. R., Fethe, M. H., Peng, Y., & Millwood, R. J., et al. (2013). Bacterial pathogen phytosensing in transgenic tobacco and arabidopsis plants.PlantBiotechnologyJournal, 11(1):43-52. [12] Loon, B. V., Markkanen, E., & Hübscher, U. (2010). Oxygen as a friend and enemy: how to combat the mutational potential of 8-oxo-guanine.DnaRepair, 9(6):604-616. [13]弓毅,杨劲.DNA聚合酶Y家族在跨损伤复制中的作用[J].生物医学工程学杂志,2013.30(1):213-216. GONG Yi, YANG Jin. (2013). The Biological Effect of Y-family DNA Polymerase on the Translesion Synthesis [J].JournalofBiomedicalEngineering, 30(1):213-216. (In Chinese) [14] Roy, S., Choudhury, S. R., Sengupta, D. N., & Kp., D. (2013). Involvement of atpol in the repair of high salt- and dna cross-linking agent-induced double strand breaks in arabidopsis.PlantPhysiology, 162(2):1,195-1,210. [15] Czarny, P., Pawlowska, E., Bialkowskawarzecha, J., Kai, K., & Blasiak, J. (2015). Autophagy in dna damage response.InternationalJournalofMolecularSciences, 16(2):2,641-2,662. [16] Ren Sun, S. E. L. W. (2012). Oxidative stress induced s-glutathionylation and proteolytic degradation of mitochondrial thymidine kinase 2.JournalofBiologicalChemistry, 287(287):24,304-24,312. [17]潘洪志, 李 蓉. DNA氧化损伤标志物8- 羟基脱氧鸟苷及其检测[J]. 中国卫生检验杂志, 2003, 13(4) : 404-405. PAN Hong-zhi, LI Rong.(2003). Detection of 8-hydroxy-2-deorygurnosine, a maker for oxidative DNA damage [J].ChinesejournalofHealthLaboratoryTechnology, 13(4): 404-405. (In Chinese) [18] Martin, M. J., Garcia-Ortiz, M. V., Gomez-Bedoya, A., Esteban, V., Guerra, S., & Blanco, L. (2013). A specific n-terminal extension of the 8 kda domain is required for dna end-bridging by human polμ and polλ.NucleicAcidsResearch, 41(19):9,105-9,116. [19] Yamtich, J., & Sweasy, J. B. (2010). Dna polymerase family x: function, structure, and cellular roles.BiochimicaEtBiophysicaActaProteins&Proteomics, 1,804(5):1,136-1,150. [20] Amoroso, A., Concia, L., Maggio, C., Raynaud, C., Bergounioux, C., & Crespan, E., et al. (2011). Oxidative dna damage bypass in arabidopsis thaliana requires dna polymerase λ and proliferating cell nuclear antigen 2.PlantCell, 23(2):806-822. [21] Martin, M. J., Garcia-Ortiz, M. V., Gomez-Bedoya, A., Esteban, V., Guerra, S., & Blanco, L. (2013). A specific n-terminal extension of the 8 kda domain is required for dna end-bridging by human polμ and polλ.NucleicAcidsResearch, 41(19):9,105-9,116. [22] Robertson, A. B., Klungland, A., Rognes, T., & Leiros, I. (2009). Dna repair in mammalian cells: base excision repair: the long and short of it.Cellular&MolecularLifeSciencesCmls, 66(6):981-993. [23] Gohlke, J., Scholz, C. J., Kneitz, S., Weber, D., Fuchs, J., & Hedrich, R., et al. (2013). Dna methylation mediated control of gene expression is critical for development of crown gall tumors.PlosGenetics, 9(2):113-127. Supported by:Special funds of Xinjiang Key Laboratory "The salt-stress responsive transcriptome of the halophyte Halostachys caspica and the functional identification of the major salt related genes"(2014KL001) and the National 973 Pre-Research Project "Physiological and molecular mechanisms of salt tolerance of desert halophytes in Xinjiang"(2012CB722204) Cloning and Expression Analysis ofHcDNApolλGene from Halostachys caspica under Salt Stress ZHANG Ji, DU Chi, ZHANG Fu-chun (XinjiangKeyLaboratoryofBiologicalResourcesandGeneticEngineering,CollegeofLifeScienceandTechnology,XinjiangUniversity,Urumqi830046,China) 【Objective】 The expression of DNA damage repair gene under salt stress is helpful to reveal the relationship between DNA damage repair and salt tolerance of halophyte.【Method】According to the transcriptome ofHalostachyscaspicaunder salt stress, a DNA damage repair gene DNA polymerase lambda (HcDNApolλ) was cloned fromHalostachyscaspicaby RACE. 【Result】Sequence analysis indicated thatHcDNApolλcontained an open reading frame of 1,335 bp, which encodes 444 amino acids. Conserved domain analysis showed thatHcDNApolλwas the number of DNA polymerase X family, and phylogenetic tree analysis indicated thatHcDNApolλwas an independent branch, which was an stable hydrophilic proteins sub-celled nuclear. Real time fluorescent quantitative PCR analysis showed that the expression ofHcDNApolλof assimilating branches gradually up-regulated with the increase of NaCl concentration. At 300 mmol/L NaCl treatments, the expression levels ofHcDNApolλwere increased 3 fold and 5 fold for 7 days and for 14 days, respectively. Finally, at 700 mmol/L NaCl treatment for 14 days, the expression levels ofHcDNApolλincreased 20 fold and reached the peak.【Conclusion】The expression ofHcDNApolλgene could be induced by salt stress. Halostachyscaspica;HcDNApolλgene; salt stress; expression analysis 10.6048/j.issn.1001-4330.2017.02.020 2016-11-18 新疆重点实验室专项资金“盐穗木盐胁迫响应的转录组研究及重要基因的功能鉴定”(2014KL001);国家“973”计划前期研究专项“新疆荒漠盐生植物耐盐的生理及分子机制”(2012CB722204) 张冀(1991-),男,硕士研究生,研究方向为植物分子生物学,(E-mail)153315267@qq.com 张富春(1962-),男,教授,博士,研究方向为分子生物学与基因工程,(E-mail)zfcxju@xju.edu.cn S188 A 1001-4330(2017)02-0361-10
Fig.4 Signal P prediction ofHcDNApolλ
Fig.5 Predicted transmembrane domain ofHcDNApolλ
Table 2 Scanning ofHcDNApolλfor site/signatures against proscan database in NPS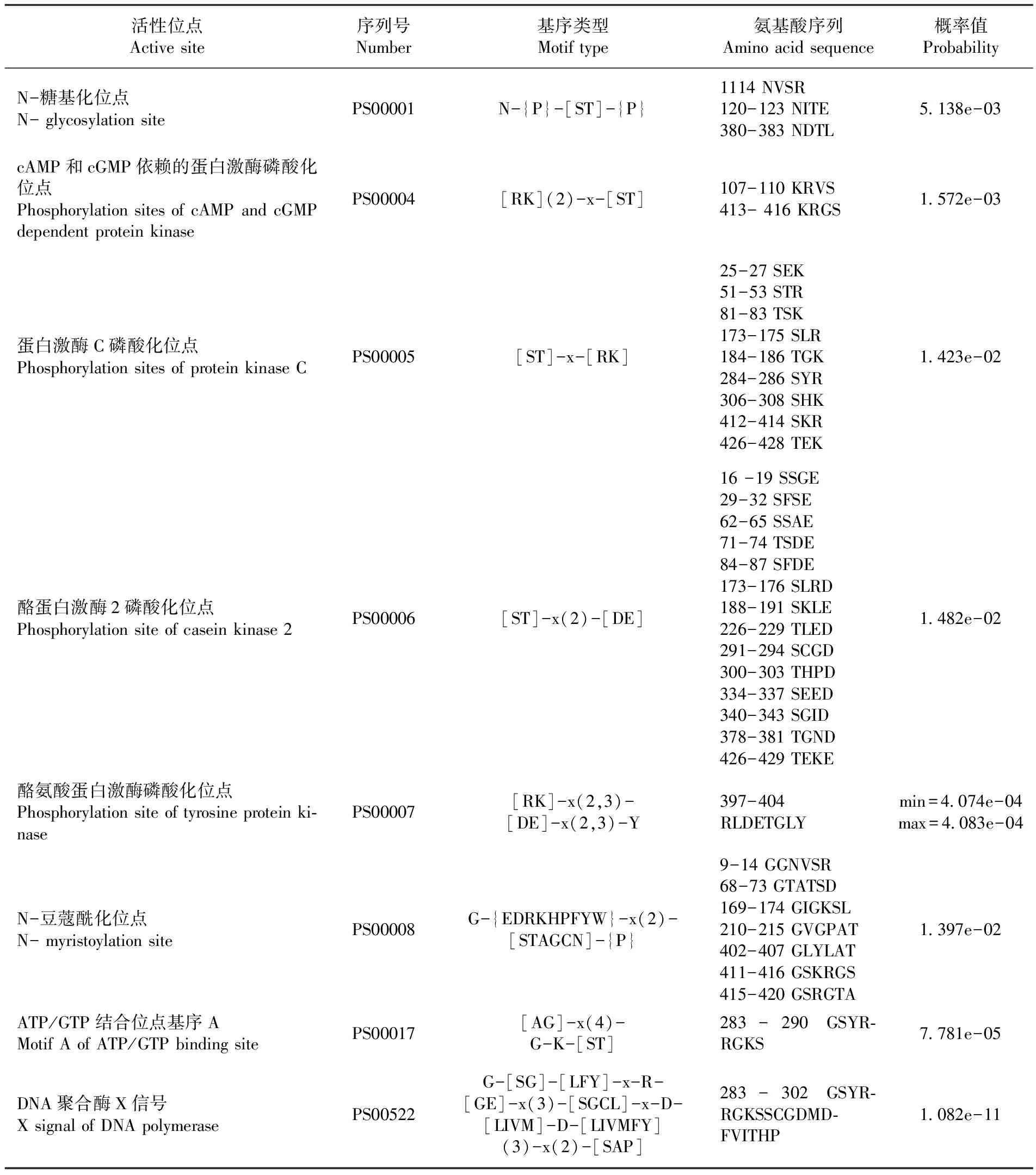

Fig.6 Relative expression ofHcDNApolλresponse to salt-stress3 讨 论
4 结 论
