光裸方格星虫野生与养殖群体线粒体控制区序列的遗传差异分析
2017-04-12周于娜彭银辉刘旭佳黄国强潘英蔡小辉
周于娜彭银辉刘旭佳黄国强潘 英蔡小辉,
(1. 广西大学动物科学技术学院, 南宁 530004; 2. 广西壮族自治区海洋研究所广西海洋生物技术重点实验室, 北海 536000; 3. 广东海洋大学广东省水产经济动物病原生物学及流行病学重点实验室, 湛江 524088)
光裸方格星虫野生与养殖群体线粒体控制区序列的遗传差异分析
周于娜1彭银辉2刘旭佳2黄国强2潘 英1蔡小辉2,3
(1. 广西大学动物科学技术学院, 南宁 530004; 2. 广西壮族自治区海洋研究所广西海洋生物技术重点实验室, 北海 536000; 3. 广东海洋大学广东省水产经济动物病原生物学及流行病学重点实验室, 湛江 524088)
基于线粒体控制区序列对光裸方格星虫(Sipunculus nudus Linnaeus, 1766)的2个养殖群体(营盘YP、竹林ZL)和4个野生群体(防城港FC、钦州QZ、大冠沙DG和越南海防YN)的91个个体进行遗传差异分析, 研究光裸方格星虫养殖和野生群体的遗传变异情况。结果显示: 获得的514 bp DNA序列中, 野生与养殖群体的多态性位点数分别为82和60, 均显示出对AT的偏倚性。共定义85个单倍型, 共享单倍型4个, 其中共享单倍型Hap5为原始单倍型, 营盘群体均为独享单倍型。各群体的单倍型多样性(Hd)相同, 野生群体的平均核苷酸多样性(Pi)(0.01531)略高于养殖群体(0.01514), 6个群体的遗传多样性水平依次为YN>YP>QZ>FC>ZL>DG。各群体间的遗传分化并不显著(P>0.05), 光裸方格星虫的遗传变异主要来自群体内个体间(99.08%), 同时未发现明显的地理谱系结构。研究表明, 光裸方格星虫野生群体的遗传多样性水平总体略高于养殖群体; 滩涂底播养殖方式较池塘养殖更利于维持光裸方格星虫遗传多样性; 各群体间不存在显著的遗传分化, 养殖群体正逐渐积累遗传变异, 但尚未足够以形成其独立的遗传结构。
光裸方格星虫; 线粒体控制区; 野生群体; 养殖群体; 遗传差异
光裸方格星虫(Sipunculus nudus Linnaeus)俗称“沙虫”, 隶属方格星虫目(Sipunculiformes)、方格星虫科(Sipunculidae)、方格星虫属(Sipunculus Linnaeus, 1766), 广泛分布于除北冰洋外其余三大洋沿岸[1]。光裸方格星虫的自然资源量在我国沿海自北向南呈递增的形式分布, 尤其盛产于广西海区[2]。光裸方格星虫因其味美、营养丰富且具有较高的药价性[3]而广受人们喜爱, 是北部湾重要经济类海产品之一。
20世纪80年代以来, 随着人们生活需求快速提高, 对光裸方格星虫自然资源的“挖掘”日益加重,种质资源逐渐出现衰退现象。为了保护和恢复自然资源, 自2004年起开启了人工繁育[4], 并在之后亲体培育[5]、苗种培育[6]等研究的基础上, 逐步完善了全人工养殖技术。光裸方格星虫人工养殖主要集中在广西沿海, 尤以北海海区为主[7], 其养殖模式主要分为滩涂养殖和池塘养殖两种[8]。随着光裸方格星虫人工养殖的迅速发展, 北海各海区人工养殖光裸方格星虫平均亩产量和公共滩涂人均采挖量均有显著增加[4]。
光裸方格星虫养殖业的快速发展取得了较为可观的生态和经济效益。然而, 光裸方格星虫养殖业快速发展趋生的自发引种、苗种异地交易等现象极易导致光裸方格星虫种质资源混杂, 遗传多样性下降, 严重威胁该产业的可持续发展。因此, 亟需开展相应的遗传多样性水平检测和种质资源评估的工作。目前, 已有许多关于光裸方格星虫的遗传学研究报道, 主要集中在运用RAPD、微卫星等分子标记对不同地理群体遗传多样性的探讨[9—13],但至今尚未有基于线粒体DNA控制区序列分析光裸方格星虫遗传背景的研究报道。本实验采用mtDNA控制区序列对光裸方格星虫不同野生地理群体及不同养殖模式下的养殖群体进行分子遗传学分析, 旨在揭示两者的遗传差异情况, 为评估种质资源和养殖业的可持续发展提供科学依据。
1 材料与方法
1.1 样品采集
本实验的4个野生群体分别采自中国防城港(FC)、钦州(QZ)、北海大冠沙(DG)和越南海防(YN), 2个养殖群体分别采自北海营盘(YP)和广西海洋研究所竹林海水增养殖试验基地(ZL)。所采集样本均为光裸方格星虫成体, 每个地理群体随机采集20—30尾。样本活体运回实验室, 取体壁肌肉于95%酒精中固定, -20℃保存备用, 每个地理群体随机抽取20个个体用于研究。
切取约30 mg组织, 利用TIANamp Marine A-nimals DNA Kit (北京天根)海洋动物组织基因组DNA试剂盒进行基因组DNA的提取, 经1.5%琼脂糖凝胶电泳检测所提DNA的完整性后, 4℃保存备用。
根据从GenBank下载光裸方格星虫线粒体基因组全长序列(Sipunculus nudus Linnaeus, FJ422961.1; KJ754934.1)设计控制区扩增简并引物: D-Loop 142上: 5′-CACGSCTCCCATTTTCYCTCCGATT-3′; D-Loop1180下: 5′-CCTGYKGTGCGKGTTAT TAARACAAG-3′, 并由南宁市天地杨生物科技有限公司合成。PCR反应体系为25 μL, 包含2.5 μL 10×PCR Buffer、2 μL MgCl2(25 mmol/L)、1 μL dNTPs (10 mmol/L)、0.2 μL Taq聚合酶(2.5 U/μL)、上下游引物(10 μmol/L)各1 μL、DNA模板1.5 μL,补充无菌水至25 μL。所用试剂均购自天根生化科技(北京)有限公司。PCR反应程序为: 94℃ 预变性5min, 然后94℃变性50s、55℃退火40s、72℃延伸60s, 共运行32个循环, 最后72℃下再延伸10min。所有PCR产物经1.5%琼脂糖凝胶电泳检测后, 送至上海立菲生物技术有限公司双向测序和拼接。
1.3 数据分析
所得拼接序列经MEGA6.0 (Molecular evolutionary genetics analysis)软件[14]进行比对, 并辅以人工校正, 确定序列比对长度; 再分析序列特征及计算各群体以及群体间的核苷酸组成; 基于Kimura双参数模型[15]计算遗传距离, 并构建系统聚类树; 基于同一模型, 以可口革囊星虫(Phascolosoma esculenta, GenBank: EF583817.1)为外类群构建单倍型邻接树(Neighbor-joining tree, NJ), 系统树经Bootstrap检验。利用DnaSP 5.0[16]确定单倍型和计算各个群体的遗传多样性水平。使用Arlequin 3.5[17]软件进行分子差异分析(AMOVA), 计算遗传分化系数Fst(F-statistics)并进行显著性统计。运用Network 4.6[18]软件构建单倍型的简约中介(Reduced-Median, MJ)网络图。
2 结果
2.1 序列特征
本研究所得光裸方格星虫线粒体DNA控制区序列共91条, 其中野生群体55条, 养殖群体36条。截取养殖和野生群体序列比对的同源片段, 约514 bp,用于比较分析。养殖群体序列中共检测出60个多态位点(Polymorphic site), 约占序列碱基总数的11.5%, 其中简约信息位点(Parsimony informative site) 31个, 单突变位点(Singleton variable site) 29个,平均转颠换比(Ts/Tv)为24.45。在野生群体中共检测到82个多态位点, 约占序列碱基总数的15.7%, 其中简约信息位点和单突变位点各占一半, Ts/Tv为9.30。对控制区序列的碱基组成分析如表 1所示,野生群体(A+T:67.8; G+C:32.2)的值与养殖群体(A+T:67.7; G+C:32.3)基本一致, 且各群体A+T含量均高于G+C。
2.2 群体遗传多样性
对不同发育阶段与叶功能性状的相关性分析显示(表2),样地1和样地2长柄双花木的发育状况一致,均与LT、LA呈显著正相关、与LWC、SLA、LPC呈显著负相关(P < 0.05);样地3和样地4的长柄双花木发育状况一致,均与LT、LA呈显著正相关、与LWC、SLA、LNC、LPC呈显著负相关(P < 0.01)。
91条序列共定义了85个单倍型, 其中共享单倍型只有4个(分别为Hap5、Hap32、Hap36、Hap46)。野生与养殖群体分别检测出了52和36个单倍型(表 2)。Hap5为4个野生群体的共享单倍型; Hap32和Hap36均为防城港与竹林群体的共享单倍型, Hap46为钦州与竹林群体的共享单倍型。营盘群体均为独享单倍型, 不存在与其他群体共享的单倍型。
统计各群体的遗传多样性参数如表 2所示, 各群体的单倍型多样性(Hd)均为1.000, 野生群体的平均单倍型多样性(0.996)略低于养殖群体(1.000); 野生群体的平均核苷酸多样性(Pi)和平均核苷酸差异数(K)(0.01531、7.77576)均略高于养殖群体(0.01514、7.69048); 综合核苷酸多样性(Pi)和平均核苷酸差异数(K), 各群体的遗传多样性大小为YN>YP>QZ>FC>ZL>DG。

表 1 光裸方格星虫控制区序列碱基组成Tab. 1 Nucleotide compositions of control region sequences in S. nudus (%)

表 2 光裸方格星虫群体的遗传多样性参数Tab. 2 Parameters of genetic diversity within S. nudus populations
2.3 遗传分化和聚类分析
利用MEGA6.0软件基于Kimura双参数模型计算群体间的遗传距离, 由表 3可知, 各群体间的遗传距离的值差异不大(0.0144—0.0160), 大冠沙(DG)与防城港(FC)群体的遗传距离最小(0.0144), 而营盘(YP)与防城港(FC)、钦州(QZ)和越南(YN)群体间的遗传距离最大, 均为0.0160。
两两群体间的分化指数Fst显示(表 3), 各群体间的遗传分化指数存在一定的差异, 但均未达到显著水平(P>0.05)。大冠沙(DG)群体与防城港(FC)群体间的遗传分化系数最低(-0.01314)而与竹林(ZL)群体的最高(0.03918)。运用Arlequin 3.5软件计算得出野生群体与养殖群体间的遗传分化指数为0.00623, 统计检验不显著(P>0.05)。
由分子差异分析(AMOVA)结果可知(表 4), 光裸方格星虫群体内个体间的遗传差异较大(99.08%),而野生和养殖群体间及各群体间的遗传差异均较小(0.45%; 0.47%)。基于群体间k2-p遗传距离的聚类分析显示(图 1), 6个光裸方格星虫群体共聚成两大支, 其中2个养殖群体(YP、ZL)独立聚成一大支;另一大支则由4个野生群体聚成, 亲缘关系较近的DG和FC、YN和QZ先各自聚成一小支, 再汇聚成一大支。
2.4 单倍型分析
光裸方格星虫群体的单倍型网络中介图显示(图 2), 各群体单倍型分散分布于各支, 无明显的与采样地点相对应的分支, 单倍型连接间的中间节点较多; 略呈星状分布趋势, Hap5位于中心位置, 且所占比重最大, 或可推测其为光裸方格星虫群体的原始单倍型。
基于光裸方格星虫群体单倍型构建的NJ系统树(图 3)表明, 野生和养殖群体的单倍型相互交错分布于各支, 没有形成明显的地理谱系结构。但有些养殖群体的独享单倍型也逐步形成了小支, 如Hap84和Hap69、Hap63和Hap73等。
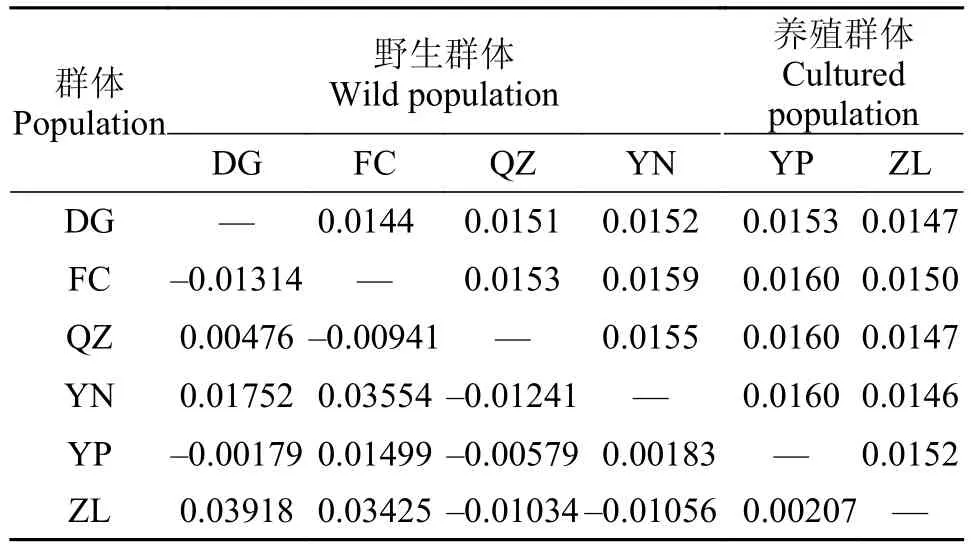
表 3 光裸方格星虫两两群体间的分化指数Fst(下三角)和遗传距离(上三角)Tab. 3 Pairwise Fst(below diagonal) and genetic distance (above diagonal) among S. nudus populations
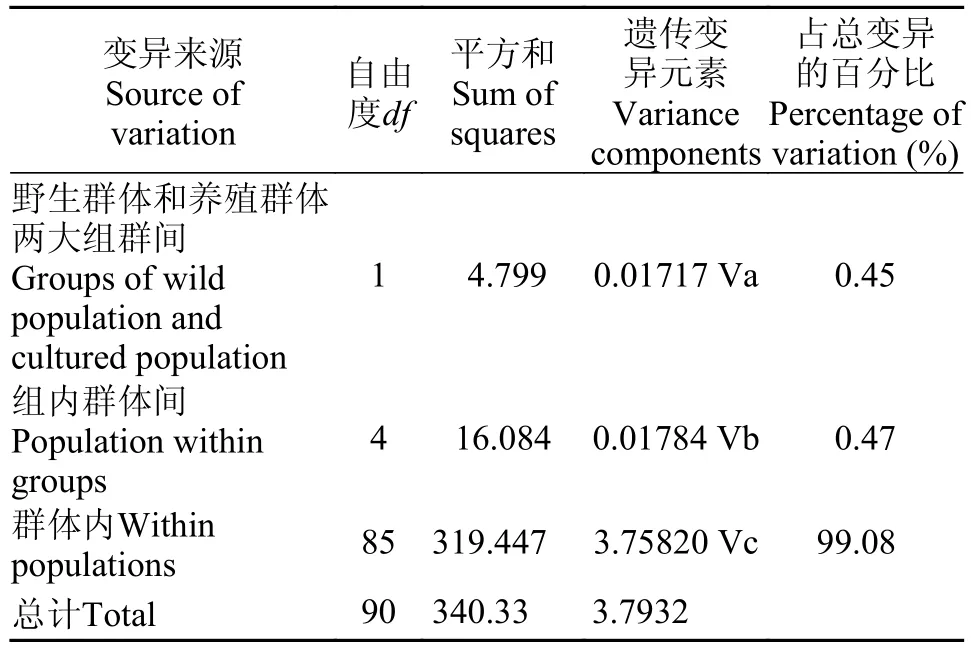
表 4 光裸方格星虫群体的分子方差分析Tab. 4 Analysis of molecular variance (AMOVA) for the S. nudus populations
3 讨论
3.1 光裸方格星虫野生与养殖群体的遗传多样性水平分析
在本研究中, 野生群体和养殖群体碱基组成的A+T含量均高于G+C, 这与许多海洋生物如河鲈(Perca fluviatilis)[19]、中国对虾(Fenneropenaeus chinensis)[20]等相似。此外, 光裸方格星虫如其他环节动物和昆虫等一样有着消极的GC偏倚率[21]。
遗传多样性水平是评价物种资源的重要指标,水平的高低与物种适应性、生存能力、进化潜能等成正比[22]。在群体遗传学研究中, 通常以单倍型多样性(Hd)和核苷酸多样性(Pi)来衡量种群的遗传多样性水平的高低[23]。本研究结果显示, 各群体具有相同的单倍型多样性(Hd), 而野生群体的平均核苷酸多样性(Pi)和平均核苷酸差异数(K)(0.01531; 7.77576)均略高于养殖群体(0.01514; 7.69048), 表明了光裸方格星虫野生群体的遗传多样性水平总体上略高于养殖群体。在水生生物研究中也有类似的报道, 如中华绒螯蟹(Eriocheir sinensis)[24]和中国对虾[20]。养殖群体亲本基数足够大、养殖与野生群体之间存在着基因交流等原因, 而使得养殖群体的遗传多样性水平尚未出现显著降低。

图 1 基于群体间遗传距离构建的NJ树Fig. 1 NJ tree based on genetic distance between populations
光裸方格星虫群体的遗传多样性水平依次为YN>YP>QZ>FC>ZL>DG, 其中营盘养殖群体具有较高的遗传多样性水平。究其原因, 一是营盘群体放养苗种来源广, 同时光裸方格星虫浮游幼体的迁徙特性促进了基因交流, 导致该群体遗传多样性较高; 二是其养殖方式的差异影响了遗传多样性的水平。营盘群体采自滩涂放养, 而竹林群体则是池塘养殖。前者养殖环境的开放性较强, 开阔的人工滩涂养殖较池塘封闭养殖更有利于资源的恢复。滩涂底播养殖在采挖时, 捕大留小, 即保证了一定数量的亲本, 使其可在滩涂上自行繁育, 对资源有保护和恢复作用。国内采用底播增殖模式以恢复或增加生态资源的报道已有不少, 如刺参(Apostichopus japonicas)[25]的底播增殖有助于恢复或增加刺参资源。此外, 皱纹盘鲍(Haliotis discus hannai)[26]、魁蚶(Scapharca broughtonii)[27]、鳞砗磲(Tridacna squamosa)[28]等也有类似的报道。

图 2 光裸方格星虫群体控制区序列单倍型中介网络图Fig. 2 Median-joining network of S. nudus populations based on control region sequences
竹林养殖群体因累代人工繁育活动且缺乏与外界的基因交流而表现出了较低的遗传多样性水平, 但仍略高于大冠沙野生群体, 这是由于竹林群体历代选择繁育的亲本基数大, 选择压力高, 使其人工繁育后代可保持较高的遗传多样性水平。但竹林养殖群体的人工后代种质状况仍有待继续观察和研究。

图 3 基于光裸方格星虫群体控制区序列构建的NJ系统树Fig. 3 NJ tree of S. nudus populations based on control region sequences
大冠沙野生群体的遗传多样性最低。该海区尚未进行人工放苗养殖活动, 且长期过度采挖、非法高压水枪的使用等可能使该区域的方格星虫种质资源出现一定程度上的衰退。
3.2 光裸方格星虫野生群体与养殖群体的遗传结构分析
NJ树显示, 光裸方格星虫的养殖和野生群体分别聚类为两大支。郭昱嵩等[10]在对北部湾光裸方格星虫地理群体研究中发现聚在一起的群体其地理位置相邻, 而本研究中并无明显类似现象。钦州与越南群体聚类为一支, 这极大可能是由于钦州曾从越南引种而引起[12]。
遗传距离研究结果显示, 大冠沙与防城港群体亲缘关系最近, 而营盘养殖群体与防城港、越南、钦州野生群体的同源性最低。遗传分化指数结果显示, 各群体间存在着一定的分化, 但均不显著(P>0.05), 且养殖群体与野生群体间尚未存在显著分化(0.00623; P>0.05); 其中大冠沙群体与防城港群体遗传分化最低, 与竹林养殖群体的遗传分化较高。肖明松等[29]发现长吻(Leiocassis longirostris)野生群体与养殖群体间有着较小遗传变异, 这与本研究结果相似。光裸方格星虫的遗传变异主要来自群体内个体间(99.08%), 群体间的遗传差异较低。由上述可知, 光裸方格星虫各群体间已经存在着不显著的分化, 主要是养殖群体与野生群体间的遗传分化, 但分化的强度不大。
光裸方格星虫的单倍型中介网络图和系统树均未检测到明显的地理谱系结构。类似的结果在条石鲷(Oplegnathus fasciatus)[23]和中国近海褐牙鲆(Paralichthys olivaceus)[30]中也有报道。在本研究中, 网络图的单倍型连接间的中间节点较多, 暗示尚有许多隐藏单倍型未被检测出来; 单倍型略呈星状分布趋势, 表明光裸方格星虫历史上出现过种群扩张事件[18]。Hap5为4个野生群体的共享单倍型,中介网络图也显示其位于中心位置, 频率高, 并且与其连接突变的单倍型最多, 据此可推测其为光裸方格星虫的原始单倍型。值得注意的是, 营盘群体的单倍型均为独享单倍型, 且Hap84、Hap69等养殖群体的独享单倍型也逐步形成了独立小支, 这些现象提示了养殖群体正在积累遗传变异, 但尚未足够以形成其独立的遗传结构。
基于本研究结果, 我们认为今后应继续坚持较高选择强度的人工选繁亲本策略, 尽可能保证一定的亲本数量和质量, 并开展人工良种选育工作。对于公共海区的自然群体资源, 采取更为明确、科学、合理的资源保护及恢复措施, 从而保证光裸方格星虫的野生资源遗传多样性处于一个良好的水平。
[1]Li F L, Zhou H, Wang W. A checklist of Sipuncula from the China coasts [J]. Journal of Ocean University of Qingdao, 1992, 22(2): 72—88 [李凤鲁, 周红, 王玮. 中国沿海星虫动物门名录. 青岛海洋大学学报, 1992, 22(2): 72—88]
[2]Lan G B, Liao S M, Yan B. Effect of water temperature on larval development and metamorphosis of Sipunculus nudus [J]. Journal of Fisheries of China, 2007, 31(5): 633—638 [兰国宝, 廖思明, 阎冰. 水温对方格星虫幼体发育及变态的影响. 水产学报, 2007, 31(5): 633—638]
[3]Chen X X, Lin X Y, Lu C Y, et al. Advances on research of Sipunculus [J]. Marine Science, 2008, 32(6): 66—70 [陈细香, 林秀雁, 卢昌义, 等. 方格星虫属动物的研究进展. 海洋科学, 2008, 32(6): 66—70]
[4]Yang J L, Zou J, Jiang Y, et al. Demonstration and extension of artificial culture technology of Sipunculus nudus [J]. Journal of Aquaculture, 2013, 34(12): 19—21 [杨家林, 邹杰, 蒋艳, 等. 方格星虫全人工养殖技术示范推广.水产养殖, 2013, 34(12): 19—21]
[5]Zou J, Peng H J, Jiang Y, et al. An experiment on probody cultivation of Sipunculus nudus [J]. Fishery Modernization, 2010, 37(3): 30—33 [邹杰, 彭慧婧, 蒋艳,等. 方格星虫亲体培育试验. 渔业现代化, 2010, 37(3): 30—33]
[6]Liu T M, Feng Q Y, Yang M Q, et al. Artificial propagation and seed breeding technology of Sipunculus nudus [J]. Journal of Tropical Biology, 2013, 4(1): 88—93 [刘天密, 冯全英, 杨明秋, 等. 光裸方格星虫人工繁殖及苗种培育技术. 热带生物学报, 2013, 4(1): 88—93]
[7]Peng Y H, Huang G Q, Liu X J, et al. Advances in germplasm research and artificial culture of Sipunculus nudus [J]. Journal of Guangxi Academy of Sciences, 2015, 31(1): 9—15 [彭银辉, 黄国强, 刘旭佳, 等. 方格星虫种质资源及人工增养殖研究进展. 广西科学院学报, 2015, 31(1): 9—15]
[8]Li J W, Zhu C B, Xie X Y, et al. Research progress on breeding, aquaculture and development of Sipunculus nudus [J]. South China Fisheries Science, 2014, 10(5): 94—98 [李俊伟, 朱长波, 颉晓勇, 等. 方格星虫的繁育、养殖及研究开发进展. 南方水产科学, 2014, 10(5): 94—98]
[9]Du X, Chen Z, Deng Y, et al. Comparative analysis of genetic diversity and population structure of Sipunculus nudus as revealed by mitochondrial COI sequences [J]. Biochemical Genetic, 2009, 47(11—12): 884—891
[10]Guo Y S, Wang Q H, Li X L, et al. Genetic diversity in three wild stock of Sipunculus nudus Linnaeus, 1766 in Beibu Gulf [J]. Journal of Fishery Sciences of China, 2012, 19(1): 62—69 [郭昱嵩, 王庆恒, 黎幸连, 等. 北部湾光裸星虫3个地理群体遗传多样性. 中国水产科学, 2012, 19(1): 62—69]
[11]Ning Y, Wu Q S, Xu D H, et al. Genetic structure and population differentiation of Sipunculus nudus in China based on sequence analyses of mitochondrial COI gene [J]. Journal of Fujian Fisheries, 2012, 34(2): 91—98 [宁岳, 巫旗生, 徐德华, 等. 不同地理群体裸体方格星虫遗传结构及种群分化研究. 福建水产, 2012, 34(2): 91—98]
[12]Song Z K, Liu T, Yang J L, et al. Genetic diversity and genetic structure of Sipunculus nudus in coastal Guangxi area [J]. Fisheries Science, 2011, 30(12): 749—753 [宋忠魁, 刘婷, 杨家林, 等. 广西沿海裸体方格星虫群体遗传多样性及遗传分化. 水产科学, 2011, 30(12): 749—753]
[13]Wang Q H, Du X D, L K. Genetic diversity of Sipunculus nudus as revealed by RAPD [J]. Marine Fisheries Research, 2006, 27(3): 57—61 [王庆恒, 杜晓东, 李康. 光裸星虫遗传多样性的RAPD分析. 海洋水产研究, 2006, 27(3): 57—61]
[14]Tamura K, Stecher G, Peterson D, et al. MEGA6: molecular evolutionary genetics analysis version 6.0 [J]. Molecular Biology and Evolution, 2013, 30(12): 2725—2729
[15]Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences [J]. Journal of Molecular Evolution, 1980, 16(2): 111—120
[16]Rozas J, Sanchez-DelBarrio J C, Messeguer X, et al. DnaSP, DNA polymorphism analyses by the coalescent and other methods [J]. Bioinformatics, 2003, 19(18): 2496—2497
[17]Excoffier L, Laval G, Schneider S. Arlequin (version3.0): an integrated software package for population genetics data analysis [J]. Evolutionary Bioinformatics Online, 2005, 1: 47—50
[18]Bandelt H J, Forster P, Sykes B C, et al. Mitochondrial portraits of human populations using median networks [J]. Genetics, 1995, 141(2): 743—753
[19]Wu Fe, Hu W G, Wang C H, et al. Genetic diversity of the cultivated and natural Perca fluviatilis [J]. Acta Hydrobiologica Sinica, 2016, 40(1): 181—188 [武菲, 胡文革, 王翠华, 等. 河鲈养殖与野生群体遗传多样性比较分析. 水生生物学报, 2016, 40(1): 181—188]
[20]Zhang H, Gao T X, Zhuang Z M, et al. Comparative analysis of the mitochondrial DNA control region between the cultured and wild population of Chinese shrimp (Fenneropenaeus chinensis) [J]. Journal of Fisheries of China, 2010, 34(8): 1149—1155 [张辉, 高天翔, 庄志猛, 等. 中国对虾养殖群体与野生群体线粒体控制区序列的比较.水产学报, 2010, 34(8): 1149—1155]
[21]Mwinyi A, Meyer A, Bleidorn C, et al. Mitochondrialgenome sequence and gene order of Sipunculus nudus, give additional support for an inclusion of Sipuncula into Annelida [J]. BMC Genomics, 2009, 10(1): 1—16
[22]Liu P. Application of DNA-marking on exploitation and conservation of germplasm resource of marine life [J]. Journal of Fishery Sciences of China, 2000, 7(2): 86—89 [刘萍. DNA标记技术在海洋生物种质资源开发和保护中的应用. 中国水产科学, 2000, 7(2): 86—89]
[23]Xiao Z Z, Xiao Y S, Ren G J, et al. Comparative analysis of genetic variation of cultured and wild rock bream Oplegnathus fasciatus population based on mtDNA control region [J]. Oceanologia et Limnologia Sinica, 2013, 44(1): 249—254 [肖志忠, 肖永双, 任桂静, 等. 条石鲷(Oplegnathus fasciatus)养殖群体与野生群体线粒体控制区序列遗传变异研究. 海洋与湖沼, 2013, 44(1): 249—254]
[24]Liu Q, Liu H, Wu X G, et al. Genetic variation of wild and cultured populations of Chinese mitten crab from the Yangtze, Huanghe, and Liaohe river basins using microsatellite marker [J]. Oceanologia et Limnologia Sinica, 2015, 46(4): 958—968 [刘青, 刘皓, 吴旭干, 等. 长江、黄河和辽河水系中华绒螯蟹野生和养殖群体遗传变异的微卫星分析. 海洋与湖沼, 2015, 46(4): 958—968]
[25]Wang W M, Ji Y E, Liu H L, et al. Research on the bottom sowing propagation technology of Apostichopus japonicas in shallow sea [J]. Journal of Aquaculture, 2012, 33(3): 16—17 [王卫民, 纪玉娥, 刘红蕾, 等. 刺参浅海底播增殖技术研究. 水产养殖, 2012, 33(3): 16—17]
[26]Liu Z C, Wang Z Q. The introduction of artificial bottom sowing propagation technology of Haliotis discus hannai [J]. China Fisheries, 1992, (8): 35 [柳忠传, 王尊清. 皱纹盘鲍人工底播增殖技术简介. 中国水产, 1992, (8): 35]
[27]Zhang Q X. Artificial bottom sowing propagation technology of Scapharca broughtonii [J]. Marine Science, 1991, 15(6): 3—4 [张起信. 魁蚶的人工底播增殖. 海洋科学, 1991, 15(6): 3—4]
[28]Li Y P, Li G Y, Li J J. An bottom sowing propagation experiment of Tridacna squamosa in Paracel Islands [J]. Scientific Fish Farming, 2015, (9): 44—45 [李育培, 李广毅, 李家积. 西沙群岛海域鳞砗磲底播养殖试验. 科学养鱼, 2015, (9): 44—45]
[29]Xiao M S, Cui F, Kang J, et al. Analysis on sequence polymorphism of the mitochondrial DNA control region and population genetic diversity of the cultivated and natural Chinese longsnout catfish (Leiocassis longirostris) [J]. Acta Hydrobiologica Sinica, 2013, 37(1): 90—99 [肖明松, 崔峰, 康健, 等. 长吻养殖群体与野生群体遗传多样性分析. 水生生物学报, 2013, 37(1): 90—99]
[30]Song N, Zhang X, Gao T. Genetic variability in eight cultured and two wild populations of Japanese flounder, Paralichthys olivaceus, based on the mitochondrial DNA control region [J]. Journal of the World Aquaculture Society, 2011, 42(4): 512—521
GENETIC VARIATION ANALYSIS ON WILD AND CULTURED POPULATIONS OF SIPUNCULUS NUDUS INFERRED FROM MTDNA CONTROL REGION SEQUENCES
ZHOU Yu-Na1, PENG Yin-Hui2, LIU Xu-Jia2, HUANG Guo-Qiang2, PAN Ying1and CAI Xiao-Hui2,3
(1. College of Animal Science and Technology of Guangxi University, Nanning 530004, China; 2. Guangxi Key Laboratory of Marine Biotechnology, Guangxi Institute of Oceanology, Beihai 536000, China; 3. Guangdong Provincial Key Laboratory of Pathogenic Biology and Epidemiology for Aquatic Economic Animals, Zhanjiang 524088, China)
Sipunculus nudus is one of the commercially important inshore demersal species. Understanding the population genetic of this species is a critical component of conservation management and artificial reproduction. However, few studies were reported about the genetic variation between wild and cultured population. The current study employed ninety-one mitochondrial DNA control region sequences to analyze the genetic variation of two cultured (Yingpan and Zhulin in Beihai) and four wild (Daguansha, Qinzhou, Fangchenggang in China and Haiphong in Vietnam) populations of S. nudus. Results showed that sixty and eighty-two polymorphic sites were detected in cultured and wild populations based on the 514 bp sequence, respectively. A negative GC-skew was found in mtDNA control region sequences of S. nudus. Eighty-five haplotypes were identified from control region sequences, among which 4 haplotypes were shared by all populations except Yingpan. According to the Median-joining network, the Hap5 was the chief ancestral haplotype of S. nudus. The average nucleotide diversity (Pi) (0.01514) of cultured populations was slightly lower than the wild populations (0.01531), and all populations had the same haplotype diversity (Hd). The order of genetic diversity for six populations was: YN>YP>QZ>FC>ZL>DG. No significant (P>0.05) genetic differentiations were detected among populations in terms of the genetic distance and fixation index (Fst). The AMOVA analysis indicated that the genetic variance mainly appeared within populations (99.08%). Non-significant genealogical structures were found from NJ and Median-joining network analyses among populations. The genetic diversity of wild population was slightly higher than the cultured populations. The bottom sowing culture was more beneficial to maintain high level genetic diversity of S. nudus. Meanwhile, the genetic differentiations existed among populations were not significant, and the cultured population accumulated genetic variations try to form its own genetic structure.
Sipunculus nudus; mtDNA control region; Wild population; Cultured population; Genetic variation
Q75; S917.4
A
1000-3207(2017)02-0384-07
10.7541/2017.47
2016-05-14;
2016-07-11
国家自然科学基金(31160532); 广西科学研究与技术开发计划项目(桂科攻14121006-2-1); 广西自然科学基金(2015GXNSFBA 139081); 广西科学院基本科研业务费(15YJ22HYS13)资助 [Supported by the National Natural Science of China Foundation (31160532); Research Program of Science Technology Department of Guangxi Province (Gui S&T Tackle 14121006-2-1); Natural Science Foundation of Guangxi Province of China (2015GXNSFBA139081); Basic Scientific Research Service Fee of Guangxi Academy of Sciences (15YJ22HYS13)]
周于娜(1992—), 女, 广西南宁人; 在读硕士研究生; 主要从事海洋经济动物遗传与育种研究。E-mail: nnzhouyuna@163.com
彭银辉(1981—), E-mail: pyinhui@163.com; 潘英(1968—), E-mail: yingpan@gxu.edu.cn
