卵巢成熟期和退化期星斑川鲽脑垂体和下丘脑组织的转录组比较分析
2017-04-12江王丽娟王波郑凤荣王长海
周 江王丽娟王 波郑凤荣王长海
(1. 南京农业大学资源与环境学院, 江苏省海洋生物学重点实验室, 南京 210095; 2. 中华人民共和国盐城出入境检验检疫局,盐城 224002; 3. 国家海洋局第一海洋研究所, 青岛 260061)
卵巢成熟期和退化期星斑川鲽脑垂体和下丘脑组织的转录组比较分析
周 江1王丽娟2王 波3郑凤荣3王长海1
(1. 南京农业大学资源与环境学院, 江苏省海洋生物学重点实验室, 南京 210095; 2. 中华人民共和国盐城出入境检验检疫局,盐城 224002; 3. 国家海洋局第一海洋研究所, 青岛 260061)
为了解星斑川鲽(Starry Flounder, Platichthys stellatus)脑组织基因表达与卵巢发育调控的关系, 发掘相关功能基因, 研究提取卵巢成熟期和退化期星斑川鲽雌鱼脑垂体和下丘脑组织总RNA, 运用HiSeq 2000高通量测序技术进行转录组测序分析。测序结果经拼接组装后共获得30640条Unigenes, Blast同源性比对显示, 其中24128条Unigenes获得注释; 经eggNog功能注释后29137条Unigenes序列分为26类, 分别涉及信号转导、翻译机制等生理生化过程。KEGG pathway数据比对显示, 卵巢成熟期涉及98种代谢途径, 135条序列表达上调; 卵巢退化期涉及192种代谢途径, 648条序列表达上调。Unigenes表达量及表达差异分析表明在卵巢成熟期334条序列表达上调, 卵巢退化期987条序列表达上调。获得功能注释的Unigene中, 408条涉及生殖调控和内分泌调控, 参与生殖调控的信号分子有1508条。试验采用实时定量 PCR研究了涉及生殖调控和内分泌调控基因促性腺激素释放激素(GnRH)、神经激肽B(NKB)、促性腺激素(GtH)、促滤泡激素(FSH)、肿瘤转移抑制因子(Kiss)以及催乳素释放肽受体(PrRPR)在卵巢两个不同发育时期脑垂体和下丘脑的表达情况, 结果表明除FSH外, 其余均在卵巢退化期时期脑组织中表达升高, 与转录组测序结果趋势一致。
星斑川鲽; 脑组织; 转录组测序; 实时定量PCR
星斑川鲽(Starry Flounder, Platichthys stellatus)又称星突江鲽, 隶属鲽形目鲽科川鲽属, 为广盐、冷水性种类, 在中国、朝鲜、韩国、日本、俄罗斯及美国太平洋沿岸海域均有分布, 具有广温广盐、耐受性强、易于驯化和营养丰富等特点, 是理想的集约化养殖对象[1]。近年来, 虽然星斑川鲽人工繁育技术有所突破[2], 但对于其卵巢发育的调控机制却了解不多。目前, 国内外有关星斑川鲽的研究报道多集中在其生长[3], 生物学地位[4,5]、生态分布以及营养价值[1]等方面, 少数学者的研究涉及了星斑川鲽的种群遗传结构和生理、生化等方面[6—8], 我国于2004年开始对星斑川鲽亲鱼培育技术进行研究[9]之后相继对人工育苗和工厂化养殖技术[10,11]、胚胎发育[12]、幼鱼形态发育与生长[13]进行了研究,对于星斑川鲽繁殖相关分子机制研究还未见报道。由于鱼类配子发生和性腺成熟是通过神经内分泌系统经由脑-垂体-性腺轴调控得以实现, 其结果表现为性腺发育周期性变化, 下丘脑分泌促性腺激素释放激素, 垂体随后分泌促性腺激素, 性腺接受调控分泌相应的性激素和孕激素, 随后性腺发育,成熟后进入生殖阶段, 完成繁殖后即退化。繁殖和退化过程的完成经过了许多结构和功能的转变, 了解调控这些转变的因子是研究星斑川鲽人工繁殖的关键。
对鱼类卵巢发育的调控及影响的早期研究多从内分泌角度出发, 但转录组学分析技术的应用为研究硬骨鱼类卵巢发育及排卵的分子调控机制提供了新的工具[14,15]。迄今为止, 有关卵巢发育转录表达的研究工作较多地集中在模式鱼类, 如斑马鱼(Danio rerio)、鲦(Pimephales promelas)[16,17], 尽管近期在养殖鱼类, 如花条鲈(Morone saxatilis)、橄榄比目鱼(Paralichthys olivaceus)等也有关于卵巢转录组研究报道, 但这些研究或者采用的定量效率较弱:如基因芯片技术、抑或是各期卵巢的混合样品, 其关注的依然是卵巢器官特异基因表达的特性分析, 而非对不同成熟期的卵巢转录特征进行分析[18, 19]。
转录组测序技术可以获得特定细胞在某一状态下所能转录出来的所有RNA, 包括非编码RNA和信使RNA (mRNA), 可高通量地获得基因表达的RNA水平有关信息, 通过获得研究对象基因组转录区域的信息, 鉴定转录发生位点, 可变剪切等, 可对基因进行精确的定量分析[20—22]。目前, 转录组测序技术已逐步取代基因芯片技术, 成为从全基因组水平研究基因表达的主流方法。为了详细了解星斑川鲽的繁殖机理和遗传特点, 本研究借助高通量测序技术对卵巢成熟期和退化期星斑川鲽脑垂体和下丘脑组织进行了分析, 为今后我国星斑川鲽人工繁殖技术水平提高以及杂交育种和品种改良研究提供技术支持。
1 材料与方法
1.1 实验材料及总RNA提取
试验用星斑川鲽取自日照海洋水产资源增殖站, 随机取样卵巢成熟期和退化期健康星斑川鲽3龄雌性亲鱼各10尾(1053.6±42.5) g, 分别取脑垂体和下丘脑组织后迅速投入液氮转入-80℃保存待用。总RNA提取方法参照Trizol Reagent (Invitrogen)说明书进行, 以1.0%琼脂糖凝胶电泳和紫外分光光度计检验总RNA质量。
1.2 样品前处理及测序
将卵巢成熟期和退化期亲鱼脑垂体和下丘脑组织RNA检测浓度和纯度后, 各选取5条质量最佳的亲鱼RNA作为混合测序样品。根据测得的RNA浓度, 将符合要求的RNA样品等量混合至新离心管中, 再次检测混合后RNA样品的浓度和纯度。混合后RNA样品置于1.5 mL离心管中送上海派森诺公司测序。
将提取的卵巢成熟期和退化期的星斑川鲽脑垂体和下丘脑总RNA用带有Oligo (dt)的磁珠富集mRNA, 由反转录酶和随机引物将片段化的mRNA合成cDNA第一链, 第二链合成使用缓冲液、dNTPs、RNase H和DNA polymerase Ⅰ, 经核酸外切酶和聚合酶进行末端修复, 连接接头, 分离纯化, 扩增DNA文库。采用Picogreen和荧光分光光度计定量文库, Agilent 2100控制PCR富集片段质量, 验证DNA文库质量, 样品稀释至4—5 pmol/L后上机测序。
利用HiSeq 2000平台获得星斑川鲽卵巢成熟期和退化期脑垂体和下丘脑组织图像序列信息, 然后转化为相应核苷酸序列, 进行可信度分析及质量评估。去除低质量序列, 运用Velvet和Oases软件进行组装拼接, blast聚类得到Unigene(http://www.ebi.ac. uk/~zerbino/velvet; http://www.ebi.ac.uk/~zerbino/ oases)[23,24]。
1.3 序列比对、eggNOG功能注释、KEGG代谢通路分析及表达差异分析
将所得序列与SwissProt、nr、Non Redudant数据库进行序列比对, 统计与数据库中已收录基因具有高度相似性的序列, 获取最佳注释(E-value<10-5); 将所有序列在eggNOG数据库中进行比对(E-value<10-5), 获得表达序列的NOG功能注释及分类,收集建立基因同源组群, 包括COG和KOG数据(http://www.ncbi.nlm.nih.gov/COG; http://eggnog. embl.de/)[25]。
使用RPKM值(Reads per kb per million reads),计算基因表达量, 2-fold法统计成熟期和退化期序列RPKM值差异(定义: 一条序列的RPKM值在成熟期大于或等于退化期的2倍, 则为成熟期表达上调,反之则为退化期表达上调。若两时期RPKM值差异不超过2倍, 则为表达无差异) 。全部序列在KEGG pathway数据库中进行比对, 获得相关序列代谢通路注释及在代谢通路中的具体位置信息(http://www. genome.jp/kegg/)[26,27]。
1.4 GnRH、NKB、GtH、Kiss、FSH及PrRPR在不同卵巢发育时期实时定量PCR分析
为验证RNA测序结果, 将进行转录组测序时提取的卵巢成熟期和退化期星斑川鲽脑垂体和下丘脑组织总RNA各取3个样本, 使用Prime ScriptTMII 1st strand cDNA synthesis Kit试剂盒(TaKaRa)反转录得到cDNA。按照Real-time PCR说明书FastStart Universal SYBR Green Master (ROX)进行Real-time PCR反应, 所用引物如表 1, 目的基因Ct值取3个平行均值, 管家基因β-actin为内参, 目的基因Ct值与内参基因的Ct值之间差异称为ΔΔCt, 2-ΔΔCt值即为样品的该基因的相对表达水平[28], 设成熟期为对照,即成熟期样品的值均设为1倍, PCR反应条件为: 95℃, 10min; 95℃, 15s; 60℃, 60s; 72℃, 1min, 40个循环。
2 结果
2.1 序列注释及分析
拼接组装得到的30640条Unigenes经Blast同源比对NCBI数据库, 其中24128条获得同源序列注释,占总数的78.75% (E-value<10-5)。将全部Unigenes进行eggNOG注释后, 归入适当的eggNOG簇。在功能已知的29137条Unigenes中, 按功能不同将其分为26大类, 其中涉及信号转导机制的Unigenes所占比例最大, 为21.20%, 其次是涉及基本功能和转录机制的Unigenes各占9.29%和8.84% (图 1)。利用RPKA值差异显著性(P<0.05)计算两个卵巢发育时期基因表达量, 并用2-fold法进行数据统计分析差异表达基因分析(图 2)。在获得的转录组序列数据中, 成熟期表达上调的序列有334条, 退化期表达上调的序列有987条。

表 1 实时定量PCR引物序列Tab. 1 Sequences of Real-time PCR primers
2.2 差异表达unigene的GO富集分析
在两期表达上调的1321条Unigenes与GO数据库进行比对的结果显示如图 3所示, 三大功能分类分别包含了6、9和37个功能分类。在细胞组分分组中, 两时期脑组织差异表达基因在细胞、细胞内和细胞组分类型比例最高, 分别为22.60%、19.21%、19.21%和21.69%、18.81%和17.08%。在分子功能分组中, 两时期脑组织差异表达基因在分子功能、离子结合所占比例较高, 为50.43%、27.35%和44.95%和29.62%。在生物学过程分组中, 两时期脑组织差异表达基因在生物学过程、生物合成过程和细胞氮代谢过程所占比例较高, 分别为16.13%、8.06%、7.66%和17.69%、7.85%和6.62%。差异表达基因的GO功能富集分析表明, 在星斑川鲽卵巢发育过程中, 蛋白质等营养物质的积累, 包括钙磷等微量元素在体内的变化以及因此导致的胞内胞外一系列信号应答机制和相关激素的合成、释放、调控生殖发育作用为此期间主要的神经内分泌行为。
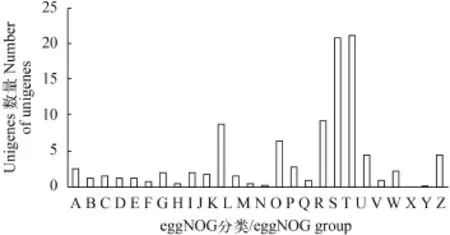
图 1 Unigenes eggNOG功能分类Fig. 1 eggNOG function classification of unigenesA. RNA加工与修饰RNA processing and modification; B. 染色质结构与变化Chromatin structure and dynamics; C. 能量产生与转化Energy production and conversion; D. 细胞周期调控与分裂,染色体重排Cell cycle control, cell division, chromosome partitioning; E. 氨基酸运输与代谢Amino acid transport andmetabolism; F. 核苷酸运输与代谢Nucleotide transport and metabolism; G. 碳水化合物运输与代谢Carbohydrate transport and metabolism; H. 辅酶运输与代谢Coenzyme transport and metabolism; I. 脂类运输与代谢Lipid transport and metabolism; J.翻译, 核糖体结构与生物合成Translation, ribosomal structure and biogenesis; K. 转录Transcription; L. 复制、重组与修复Replication, recombination and repair; M. 胞壁/膜生物发生Cell wall/memberance/envelope biogenesis; N. 细胞运动Cell motility; O. 蛋白质翻译后修饰与转运, 分子伴侣Posttranslational modification, protein turnover, chaperones; P. 无机离子运输与代谢Inorganic ion transport and metabolism; Q. 次生产物合成, 运输及代谢Secondary metabolites biosynthesis, transport andcatabolism; R. 一般功能基因General function prediction only; S. 功能未知Function unknown; T. 信号传导机制Signal transduction mechanisms; U. 胞内分泌与膜泡运输Intracellular trafficking, secretion, and vesicular transport; V. 防御机制Defense mechanisms; W. 细胞外结构Extracellular; X. 未确定Undetermined; Y.核酸结构Nuclear structure; Z. 细胞骨架Cytoskeleton
2.3 KEGG pathway注释及富集分析
KEGG (Kyoto Encyclopedia of Genes and Genomes)数据库记录细胞中基因产物的功能以及基因产物的相互作用网络, 基于KEGG通路的分析有助于我们进一步了解基因的生物学功能以及所有可能的代谢途径, 而且可以对催化各步反应的酶进行全面的注解。对全部序列进行KEGG代谢通路数据库比对和注释, 结果表明成熟期涉及98种代谢途径, 表达上调的序列有135条; 退化期涉及192种代谢途径, 表达上调的序列有648条(图 4)。结合差异表达基因的KEGG功能注释结果, 进行Pathway显著性富集分析。共得到显著富集的Pathway包括免疫系统、神经系统、信号分子相互作用、信号转导、内分泌系统、细胞通讯等代谢通路。此外,一些参与生殖、生长和发育的功能基因分入卵母细胞减数分裂(Oocyte meiosis)和促性腺激素释放激素信号通路(GnRH signaling pathway)(表 2)。通过对注释结果进一步统计分析, 查找到与生殖发育调控以及内分泌相关的基因有408条, 不同分类的生殖发育调控以及内分泌相关的基因及数目如表3所示。参与生殖调控与内分泌相关的信号分子及基因数目主要有钙调蛋白98条、磷酸酯酶534条、蛋白激酶798条及环腺苷酸 87条, 共计1508条。

图 2 卵巢发育不同时期序列表达差异Fig. 2 Unigenes different expression in different ovary deve-lopment stages
2.4 GnRH、NKB、GtH、Kiss、FSH及Pr-RPR在不同卵巢发育时期的脑组织中的表达验证
图 5所示, 生长激素释放激素GnRH、促性腺激素GtH、促滤泡激素FSH、PrRPR、神经激肽BneurokininB及Kisspeptin基因在卵巢不同发育时期的表达不同, GnRH、NKB、GtH、Kis和PrRPR在卵巢退化期的表达水平高, PrRPR的表达水平在退化期超过了成熟期的5倍, 而FSH则在卵巢成熟期的表达水平较高, 与转录组测序结果趋势一致, 尽管表达量存在一定差异。
3 讨论
目前, RNA测序技术在鱼类的研究中已经发挥了重要作用, 郭威[29]利用该技术进行了黄鳝脑及性腺转录组分析及差异基因筛选, 发现在3种不同性腺发育时期黄鳝间, 差异表达基因多集中在性腺中;程云英[30]对6月龄奥里亚罗非鱼雌雄性腺经行了转录组测序, 发现二者中大部分基因表达模式相一致; Zucchi等[31]使用微阵列杂交的方法研究了雌性斑马鱼孕酮诱导后脑和卵巢中基因变化情况。星斑川鲽为我国新兴的经济海水鱼类, 具有重要的经济价值, 但该鱼分子遗传基础研究较为薄弱, 基因组序列信息缺乏, 本研究采用HiSeq 2000高通量测序技术对卵巢发育不同时期的星斑川鲽的脑组织进行测序分析, 有望充实星斑川鲽的公共EST数据资源, 挖掘更多具有潜在价值的基因并更好地理解星斑川鲽生殖和发育的分子生物学机制。
3.1 卵巢差异表达基因的分析
本次测序共获得30640条Unigenes, 24128Uni-genes能获得生物信息学注释。其中408条与生殖调控相关, 而参与生殖调控的信号分子则有1508条。Unigenes的KEGG pathway数据比对分析,卵巢成熟期涉及98种代谢途径, 表达上调的序列有135条; 卵巢退化期涉及192种代谢途径, 表达上调的序列有648条。Unigenes表达量及表达差异分析表明在卵巢成熟期基因表达上调的334条Unigenes,在卵巢退化期表达上调987条Unigenes。

图 3 Unigenes的GO分类Fig. 3 GO classification of unigenes

图 4 KEGG信号通路中的unigenes数量分布Fig. 4 The distribution of unigenes in KEGG pathway

表 2 星斑川鲽卵巢发育相关基因Tab. 2 The Genes involved in ovary development of Platichthys stellatus

表 3 生殖调控相关的基因Tab. 3 The genes involved in procreation regulation

图 5 GnRH、FSH、NKB、GtH、Kis、PrRPR在卵巢成熟期和退化期脑组织中的表达Fig. 5 The expression of GnRH, NKB, FSH, GtH, Kiss and PrRPR during the stage of ovary maturation and stage of ovary degeneration
星斑川鲽的卵巢发育是属于非同步型, 繁殖期分批成熟多次产卵, 即卵巢内含有至少两种处于不同发育阶段的卵母细胞群, 在第一次排卵结束后卵巢中还存留相当部分的待成熟卵母细胞。据本研究结果显示, 在第一次排卵前, 差异表达基因中包括促进葡萄糖转运蛋白、蛋白磷酸酶、微管蛋白、过氧化物酶、果糖-二磷酸醛缩酶、谷氨酰转肽酶、铁传递蛋白、二氢蝶啶还原酶等基因, 涉及的信号通路包括蛋白质、脂肪代谢、钙磷代谢, 而有关促性腺激素释放激素、促性腺激素的调控基因等只是常态表达, 这说明了在此期间, 卵巢发育需要大量营养物质及微量元素的支持而相关的性激素只是缓慢平稳的上升; 排卵行为产生是以促性腺激素分泌到峰值为先导, 与此相对应, 在本研究中,星斑川鲽雌鱼排卵过程中差异表达基因包括促性腺激素释放激素受体、神经肽Y、胰岛素样生长因子、电压门控钙离子通道、孕激素受体等一批配合排卵过程调控的基因表达都变为上调状态, 据对金鱼、鲤等的研究表明, 促性腺激素释放激素刺激促性腺激素释放依赖于细胞外钙离子的有效性, 白细胞介素等免疫因子参与排卵过程的调控[32—35]。
采用实时定量PCR的方法对与生殖调控相关的基因进一步的研究发现, FSH基因在星斑川鲽性腺成熟期时的脑组织中表达较高, 而GtH、PrRPR、NKB、GnRH以及Kiss则在退化期的表达水平较高。Levavi等[36]在鲑科鱼中的研究表明, FSH主要在性腺发育早期起调控作用, 并不参与排卵的调控,而LH主要参与配子成熟和排出, Rocha等[37]在舌齿鲈中的研究发现, FSHR强烈表达于卵黄生成期和早期卵黄生成阶段, 但并不表达于成熟卵细胞, 真鲷(Pagrus major) LHβ转录本在两性配子发生到排出始终保持较高水平[38]。这些结果表明GtHs在硬骨鱼生殖调控中的功能存在一定的种属特异性。
3.2 与卵巢发育相关的信号通路
从本次转录组测序中, 胰岛素样生长因子(IGF1)、表皮生长因子受体(EGFR)、细胞周期蛋白依赖性激酶(Cyclin Dependent Kinase, CDK)、细胞分裂周期因子(cell division cycle, cdc)、p38 丝裂原活化蛋白激酶(p38 mitogen-activated protein kinase)、Jun氨基末端激酶(Jun N-terminal kinase)、细胞外信号调节激酶1/2 (ERK1/2)等一系列关键酶和受体调节因子蛋白激酶A (PKA)都被发现。其中细胞外信号调节激酶(Extra-cellular signal-regulated kinase, ERK)信号通路、p38 丝裂原活化蛋白激酶(p38 mitogen-activated protein kinase, p38)信号通路、Jun氨基末端激酶(Jun N-terminal kinase, JNK)信号通路和细胞外信号调节激酶5 (extracellular-regulated kinase 5, ERK5)信号通路[39]是丝裂原活化蛋白激酶(MAPK)信号转导途径的子通路, 各子途径相互交错联系并作用, 形成一个巨大的信息传递网。据研究, MAPK信号通路在海星(Asterina miniata)卵母细胞减数分裂过程发挥重要作用, 即MAPK途径维持卵母细胞减数分裂MI期的稳定, 但也人有认为MI期停滞与MAPK途径无关, 但MAPK会影响排卵后的MI期的长短[40]。Roux等[41]发现MAPK通路会影响紫海胆(Strongylocentrotus purpuratus)精子和卵子的结合, 由此可见, MAPK信号通路对卵子发生、精子形成和性腺发育都有着显著的功能。
[1]Liu S L, Wang B, Liu Z H, et al. Analysis and evaluation of the nutrition of platichthys stellatus [J]. Progress in Fishery Sciences, 2009, 30(6): 18—24 [刘世禄, 王波, 刘振华, 等. 星斑川鲽的营养分析与评价. 渔业科学进展, 2009, 30(6): 18—24]
[2]Wang B, Wang Z L, Sun P X, et al. The development prospect of the breeding conditions of Platichthys stellatus [J]. Fishery Modernization, 2006, 5: 16—18 [王波, 王宗灵, 孙丕喜, 等. 星斑川鲽的养殖条件及发展前景. 渔业现代化, 2006, 5: 16—18]
[3]Campana S E, Neilson J D. Daily growth increments in otoliths of starry flounder (Platichthys stellatus) and the influence of some environmental variables in their production [J]. Canadian Journal of Fisheries and Aquatic Sciences, 2011, 39(7): 937—942
[4]Ma A J, Zhuang Z M, Li C, et al. The biologic characteristics of starry flounder (Platichthy stelltus Pallas) [J]. Marine Fisheries Rresearch, 2006, 27(5): 91—95 [马爱军, 庄志猛, 李晨, 等. 星突江鲽生物学特性及养殖前景.海洋水产研究, 2006, 27(5): 91—95]
[5]Qi G S, Li D, Chen S Q, et al. Morphological characteristics and internal structure of Platichthys stellatus Pallas [J]. Journal of Fishery Sciences of China, 2008, 15(1): 1—11 [齐国山, 李迪, 陈四清, 等. 星突江鲽的形态特征及内部结构研究. 中国水产科学, 2008, 15(1): 1—11]
[6]You F, Wu Z H, Li J, et al. RAPD analysis of genetic diversity in cultured stock of Platichthys stellatus [J]. Advances in Marine Science, 2007, 25(1): 73—78 [尤锋, 吴志昊, 李军, 等. 星斑川鲽(Platichthys stellatus)养殖群体的RAPD分析. 海洋科学进展, 2007, 25(1): 73—78]
[7]Park J Y, Kijima A. Genetic variability and differentiation within and between the stone flounder (Kareius bicoloratus) and the starry flounder (Platichthys stellatus) [J]. Tohoku Journal of Agricultural Research, 1991, 41(3—4): 69—82
[8]Borsa P, Blanquer A, Berrebi P. Genetic structure of the flounders Platichthys flesus and P. stellatus at different geographic scales [J]. Marine Biology, 1997, 129(2): 233—246
[9]Liu Z H, Wang S X, Wang B, et al. Study on the propagation controlling techniques of Platichthys stellatus [J]. Shandong Fisheries, 2007, 24(12): 1—2 [刘振华, 王森勋, 王波, 等. 星斑川鲽亲鱼生殖调控技术研究. 齐鲁渔业, 2007, 24(12): 1—2]
[10]Zheng C B, Jiang Q P. Essential of artificial of platichthys stellatus (Pallas) [J]. Moedern Fisheries Information, 2006, 21(12): 31—33 [郑春波, 姜启平. 星斑川鲽Platichthys stellatus (Pallas)人工育苗技术要点. 现代渔业信息, 2006, 21(12): 31—33]
[11]Zheng C B, Jiang Q P. Industrial aquaculture technology of Starry Flounder Platichthys stellatus [J]. Fisheries Science and Technology Information, 2007, 34(3): 34—35 [郑春波, 姜启平. 星斑川鲽工厂化养殖技术. 水产科技情报, 2007, 34(3): 34—35]
[12]Wang B, Liu Z H, Sun P X, et al. The morphological observation on the embryonic development of Starry Flounder, Platichthys stellatus [J]. Haiyang Xuebao, 2008, 30(2): 130—136 [王波, 刘振华, 孙丕喜, 等. 星斑川鲽胚胎发育的形态观察. 海洋学报, 2008, 30(2): 130—136]
[13]Liu Z H, Wang B, Yao Z G, et al. Morphological development and growth of larval and juvenile fish of Starry Flounder, Platichthys stellatus [J]. Advances in Marine Science, 2008, 26(1): 90—97 [刘振华, 王波, 姚振刚, 等.星斑川鲽仔、稚、幼鱼的形态发育与生长. 海洋科学进展, 2008, 26(1): 90—97]
[14]Clelland E, Peng C. Endocrine/paracrine control of zebrafish ovary development [J]. Molecular and Cellular Endocrinology, 2009, 312(1-2): 42—52
[15]Lubzens E, Young G, Bobe J, et al. Oogenesis in teleosts: how fish eggs are formed [J]. General and Comparative Endocrinology, 2010, 165(3): 367—389
[16]Li Y, Chia J M, Bartfai R, et al. Comparative analysis of the testis and ovary transcriptomes in zebrafish by combining experimental and computational tools [J]. Com Parative and Fuctional Genomics, 2004, 5(5): 403—418
[17]Wang R L, Bencic D C, Garcia-Reyero N, et al. Natural variation in fish transcriptomes: comparative analysis of the fathead minnow (Pimephales promelas) and zebrafish (Danio rerio) [J]. Plos One, 2014, 9(9): e114178
[18]Chapman R W, Reading B J, Sullivan C V. Ovary transcriptome profiling via artificial intelligence reveals a transcriptomic fingerprint predicting egg quality in striped bass, Morone saxatilis [J]. Plos One, 2014, 9(5): e96818
[19]Fan Z, You F, Wang L, et al. Gonadal transcriptome analysis of male and female olive flounder (Paralichthys oli-vaceus) [J]. Biomed Research International, 2014, 2014(10): 1153—1166
[20]Hua W P, Zhang Y, Song J, et al. Denovo transcriptome sequencing in Salvia miltiorrhiza to identify genes involved in the biosynthesis of active ingredients [J]. Genomics, 2011, 98(4): 272—279
[21]Zheng M G, Tian J H, Yang G P, et al. Transcriptome sequencing, annotation and expression analysis of Nannochloropsis sp. at different growth phases [J]. Gene, 2013, 523(2): 117—121
[22]Zhang Z, Li B, Wang Y G, et al. The microflora structure in digestive tract of half-smooth tongue sole (Cynoglossus semilaevis Gunther) cultured in outdoor pond basing on high-through sequencing technique [J]. Acta Hydrobiologica Sinica, 2015, 39(1): 38—45 [张正, 李彬, 王印庚,等. 基于高通量测序的池塘养殖半滑舌鳎消化道菌群的结构特征分析. 水生生物学报, 2015, 39(1): 38—45]
[23]Grabherr M G, Haas B J, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome [J]. Nature Biotechnology, 2011, 29(7): 644—652
[24]Radakovits R, Jinkerson R E, Fuerstenberg S I, et al. Draft genome sequence and genetic transformation of the oleaginous alga: Nannochloropsis gaditana [J]. Nature Communications, 2012, 3(2): 686
[25]Tatusov R L, Fedorova N D, Jackson J D, et al. The COG database: an updated version includes eukaryotes [J]. BMC Bioinformatics, 2003, 4(1): 41
[26]Mortazavi A, Williams B A, Mccue K, et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq [J]. Nature Methods, 2008, 5(7): 621—628
[27]Kanehisa M, Araki M, Goto S, et al. KEGG for linking genomes to life and the environment [J]. Nucleic Acids Research, 2008, 36: D480—D484
[28]Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCtmethod [J]. Methods, 2001, 25(4): 402—408.
[29]Guo W. Screening of Different Express Sex-related Genes and Changes of Serum Hormones Level in Monopterus albus [D]. Thesis for Master of Science. Huazhong Agricultural University, Wuhan. 2014 [郭威. 黄鳝性别差异表达基因的筛选及血清激素水平的变化. 硕士学位论文, 华中农业大学, 武汉. 2014]
[30]Cheng Y Y. Comparative analyses of gonadal transcriptomes from two closely related tilapiines at 180 dah and bioinfonnatic analyses of NR super family [D]. Thesis for Master of Science. Southwest University, Chongqing. 2015 [程云英. 两种罗非鱼6月龄性腺转录组比较及NR超家族生物信息学分析. 硕士学位论文, 西南大学,重庆. 2015]
[31]Zucchi S, Castiglioni S, Fent K. Progesterone alters global transcription profiles at environmental concentrations in brain and ovary of female zebrafish (Danio rerio) [J]. Environmental Science & Technology, 2013, 47(21): 12548—12556
[32]Richards J S, Liu Z L, Shimada M. Immune-like mechanisms in ovulation [J]. Trends in Endocrinology & Metabolism, 2008, 19(6): 191—196
[33]Liu Z L, Shimada M, Richards J S. The involvement of the toll-like receptor family in ovulation [J]. Journal of Assisted Reproduction Genetics, 2008, 25(6): 223—228
[34]Bobe J, Goetz F W. A tumor necrosis factor decoy receptor homologue is up-regulated in the brook trout (Salvelinus fontinalis) ovary at the completion of ovulation [J]. Biology of Reproduction, 2000, 62(2): 420—426
[35]Bobe J, Goetz F W. Molecular cloning and expression of a TNF receptor and two TNF ligands in the fish ovary [J]. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2001, 129(2—3): 475—481
[36]Levavi S B, Bogerd J, Mananos E L, et al. Perspectives on fish gonadotropins and their receptors [J]. General and Comparative Endocrinology, 2010, 165(3): 412—437
[37]Rocha A, Gomez A, Zanuy S, et al. Molecular characterization of two sea bass gonadotropin receptors: cDNA cloning, expression analysis, and functional activity[J]. Molecular and Cellular Endocrinology, 2007, 272(1—2): 63—76
[38]Gen K, Yamaguchi S, Okuzawa K, et al. Physiological roles of FSH and LH in red seabream, Pagrus major [J]. Fish Physiology and Biochemistry, 2003, 28(1): 77—80
[39]Junttila M R, Li S P, Westermarck J. Phosphatase-mediated crosstalk between MAPK signaling pathways in the regulation of cell survival [J]. Faseb Journal, 2008, 22(4): 954—965
[40]Usui K, Hirohashi N, Chiba K. Involvement of mitogenactivating protein kinase and intracellular pH in the duration of the metaphase I (MI) pause of starfish oocytes after spawning [J]. Development Growth & Regeneration, 2008, 50(5): 357—364
[41]Roux M M, Townley I K, Raisch M, et al. A functional genomic and proteomic perspective of sea urchin calcium signaling and egg activation [J]. Developmental Biology, 2007, 300(1): 416—433
COMPARISON ANALYSIS ON TRANSCRIPTOME IN THE PITUITARY GLAND AND HYPOTHALAMUS OF STARRY FLOUNDER (PLATICHTHYS STELLATUS) AT THE OVARY MATURATION AND DEGRADATION STAGES
ZHOU Jiang1, WANG Li-Juan2, WANG Bo3, ZHENG Feng-Rong3and WANG Chang-Hai1
(1. Jiangsu Provincial Key Laboratory of Marine Biology, College of Resources and Environmental Science, Nanjing Agricultural University, Nanjing 210095, China; 2. Yancheng Entry-exit Inspection and Quarantine Bureau of the People’s Republic of China, Yancheng 224002, China; 3. The First Institute of Oceanography, State Oceanic Administration, Qingdao 260061, China)
To investigate the relationship of genes expression between brain tissue and ovary development, and explore more functional genes of Platichthys stellatus, total RNA from the mixed sample of pituitary gland and hypothalamus at the ovary maturation and degradation stages were isolated and sequenced by HiSeq 2000 high throughput sequencing technology. After assembly and splicing, 24128 unigenes from a total of 30640 unigenes were annotated based on the blast homology alignment. All 29137 unigenes with eggNog annotations were divided into 26 categories that were involved in physiological processes, such as signaling transduction, translation mechanism, etc. The results from KEGG pathway analysis showed that all unigenes at the ovary maturation stage were involved in metabolic pathways with 135 up-regulated sequences, while the unigenes at the ovary degradation stage were associated with 192 metabolic pathways with 648 up-regulated sequences. Moreover, 334 differentially expressed genes (DEGs) were up-regulated at the ovary maturation stage, while 987 up-regulated DEGs were observed at the ovary degradation stage. Total 408 unigenes were involved in reproductive and endocrine regulation, and 1508 unigenes were involved in signaling molecules. The expression of some reproduction-associated DEGs including growth hormone releasing hormone (GnRH), neurokinin B (NKB), gonadotropin (GtH), follicle stimulating hormone (FSH), kisspeptin (Kiss), and prolactin-releasing peptide receptor (PrRPR) were verified by qRT-PCR. All genes except FSH were consistent with the results of the transcriptome sequencing.
Platichthys stellatus; Brain tissue; Transcriptome sequencing; qRT-PCR
S961.6
A
1000-3207(2017)02-0346-10
10.7541/2017.42
2016-05-26;
2016-07-11
国家高技术发展计划“863项目”(2012AA10A413、2012AA10A408); 国家海洋公益性行业科研专项(201305005)资助[Supported by the National High-tech R&D Program “863 Program” (2012AA10A413, 2012AA10A408); Public Science and Technology Research Funds Projects of Ocean (201305005)]
周江(1979—), 男, 湖北荆州人; 博士研究生; 主要从事海洋生物学研究。E-mail: zhoujiang768@126.com
王长海(1962—), 男, 山东烟台人; 教授、博士生导师; 主要从事海洋生化工程研究。E-mail: chwang@njau.edu.cn
