中华鳖vasa基因克隆及在卵母细胞中的表达分析
2017-04-12张飘逸李伟朱新平洪孝友陈昆慈徐红艳
张飘逸李 伟朱新平洪孝友陈昆慈徐红艳
(1. 中国水产科学研究院珠江水产研究所, 农业部热带亚热带水产资源利用与养殖重点实验室, 广州 510380; 2. 上海海洋大学水产与生命学院, 上海 201306)
中华鳖vasa基因克隆及在卵母细胞中的表达分析
张飘逸1,2李 伟1朱新平1,2洪孝友1,2陈昆慈1,2徐红艳1,2
(1. 中国水产科学研究院珠江水产研究所, 农业部热带亚热带水产资源利用与养殖重点实验室, 广州 510380; 2. 上海海洋大学水产与生命学院, 上海 201306)
研究利用中华鳖为研究模型进行爬行类生殖细胞发育分化成熟等生物学研究, 克隆了中华鳖vasa基因的cDNA序列, 全长3865 bp, 包括5′端非编码区90 bp, 3′端非编码区1699 bp, 开放阅读框长2076 bp, 共编码691个氨基酸。中华鳖Vasa氨基酸序列包含DEAD-box家族蛋白8个保守保守功能域, 在N末端有4个RGG重复序列和2个GG富集区, 与小鼠Vasa蛋白的同源性较高(72%)。荧光定量PCR的结果表明, 中华鳖vasa mRNA主要精巢和卵巢中表达, 其他体组织中均难检测到表达。卵巢冰冻切片原位杂交结果显示: 中华鳖vasa mRNA在生殖细胞中特异表达; 在卵子发生过程中的不同发育期卵母细胞中呈现动态的变化。即vasa mRNA在初级卵母细胞及生长期卵母细胞中表达最强, 且均匀分布在细胞质中, 随着卵母细胞的逐渐增大, 信号逐渐减弱, 直至在成熟的卵母细胞中几乎检测不到表达信号, 说明vasa可能在中华鳖早期卵母细胞发育中起重要作用。同时, vasa基因可作为中华鳖生殖细胞分子标记物, 根据其mRNA的表达水平来鉴别不同发育时期的卵母细胞。研究结果为进一步开展中华鳖胚胎生殖细胞发育及配子生成, 特别是研究中华鳖, 乃至爬行类原始生殖细胞(Primordial Germ Cells, PGCs)的起源、迁移、分化等研究奠定了基础。
vasa; 中华鳖; 生殖细胞; 卵子发生
Vasa蛋白是一种ATP依赖的RNA解旋酶, 属于DEAD-box (Asp-Glu-Ala-Asp)家族。vasa基因最早发现于果蝇(Drosophila melanogaster)中, 是一个母源遗传基因, 在生殖细胞分化中起重要作用[1]。vasa基因在动物中高度保守, 其同源基因在无脊椎动物和脊椎动物中得以广泛研究, 包括果蝇(Drosophila melanogaster)[2]、家蚕(Bombyx mori)[3]、小鼠(Mus musculus)[4]、人(Homo sapiens)[5]、鸡(Gallus gallus)[6]、斑马鱼(Danio rerio)[7]、青鳉(Oryzias latipes)[8]、异育银鲫(Carassius auratus gibelio)[9]、金目鲈(Lates calcarifer)[10]、牙鲆(Paralichthys olivaceus)[11]、红耳龟(Trachemys scripta)[12]、蜥蜴(Podarcis sicula)[13]等。除线虫等一些特例物种外, vasa基因仅限在生殖细胞中特异性表达, 因此被作为研究生殖细胞的分子标记物, 广泛应用于生物的配子发生和原生殖细胞(Primordial Germ Cells, PGCs)起源、迁移、分化等方面的研究[14]。Vasa蛋白参与生殖系mRNA的翻译调控[15], 是生殖细胞分化所必需的。在果蝇中, vasa基因突变造成雌性果蝇不育, 卵子发生过程中存在严重缺陷[16]。在老鼠中, vasa基因功能丧失会使雄性生殖细胞的增殖和分化受阻, 导致雄性老鼠不育, 而雌性仍然是可育的[17]。更重要的是, 研究表明vasa基因产物在动物卵母细胞中的表达及分布模式与动物胚胎PGCs的起源方式密切相关[18]。例如, 在一些模式生物中,包括果蝇、斑马鱼、爪蟾, vasa基因产物在卵子发生过程中的各期卵母细胞中均有表达, 并随着卵母细胞的发育成熟, vasa基因产物逐渐集中分布到卵母细胞的特定区域-生殖质区, 与其他生殖质因子相互作用形成生殖质颗粒, 随后在胚胎发育过程中,生殖质颗粒决定PGCs的形成[15,16,19]。而在哺乳动物中, vasa基因产物只在早期卵母细胞中有表达,在晚期卵母细胞中检测不到而且也没有发现生殖质颗粒的存在[20], 因此哺乳动物的PGCs起源方式完全不同于鱼类的, 其PGCs是由胚外外胚层细胞分泌的信号分子诱导部分多能外胚层细胞发育形成的, 仅依靠细胞间的交互作用, 即细胞非自主的方式, 不受任何母源因子的调控[21]。总之, 通过vasa等生殖细胞标记基因, 各种低等无脊椎动物及哺乳类的生殖细胞的发育及分化都得以广泛深入的研究。而爬行类, 特别是龟鳖类的生殖细胞发育模式还鲜有报道。爬行类是一支从古两栖类分化出来的产羊膜卵的类群, 与鸟类哺乳类共称为羊膜动物。而龟鳖类是现存于地球上的爬行动物中最古老的一类, 是产无羊膜卵动物演变成产羊膜卵动物实现进化飞跃的联结点[22]。对其生殖细胞发育的研究, 不论在动物生殖生物学还是生殖进化领域都有很深远的意义。
中华鳖(Pelodiscus sinensis), 隶属于爬行纲(Repitlia)、龟鳖目(Testudinata)、鳖科(Tironychidae)、鳖属(Peodiscus)。中华鳖具有很高的药用、营养及观赏价值, 因此受到广大养殖户和消费者的青睐, 是淡水养殖的重要组成部分。但是近几十年来, 受环境破坏、滥捕等影响中华鳖野生资源相当匮乏, 加上养殖场育种不规范, 养殖品种的种质也日趋退化[23,24]。然而, 仅仅通过传统选育技术进行种质资源保护及改良已无法跟上日渐加剧的种质退化速度, 因此结合传统选育和现代细胞基因工程等育种新技术, 进行龟鳖类种质资源的保护与良种培育显得十分迫切。迄今为止, 研究者们利用组织学、细胞学等方法对龟鳖类生殖细胞的发育和分化做了大量的结构学及形态学研究[25—27], 而对其生殖细胞发育分化的分子机制尚未有清楚的了解, 导致龟鳖类动物生殖细胞工程育种、保种等的相关研究在国内外的开展都极其有限。
本研究主要进行了中华鳖vasa基因cDNA全序列的克隆分析, 并初步分析了vasa转录本在中华鳖卵子发生过程中的表达及分布模式, 研究结果为将来进一步探讨中华鳖, 乃至爬行类生殖细胞的起源及迁移以及理清动物生殖细胞发育方式在物种进化过程中的演变等奠定了基础。
1 材料与方法
1.1 实验材料
实验用的中华鳖均采自广州省惠州市财兴实业有限公司中华鳖养殖场, 取1、2、3冬龄生长良好的中华鳖雌雄各3只。将实验用中华鳖颈动脉放血, 掀开腹甲, 采集卵巢、精巢、心脏、肝脏、脾、肾、脑、血液, 用1xPBS (DEPC水配)稍稍清洗, 快速投入液氮中冷冻, 并放于-80℃冰箱中保存,用于总RNA提取。另取一部分精巢和卵巢组织, 用4%多聚甲醛(PFA)固定及甲醇脱水保存备用。
本实验所用的PCR引物由上海生工生物技术有限公司合成。RNA保存及提取试剂TriPure isolation Reagent、合成探针及杂交所用的digoxigenin (DIG) RNA Labeling Kit、Blocking Reagent、Anti-Digoxigenin-AP、Anti-Digoxigenin-POD、NBT/ BCIP Stock均购自罗氏(Roche)公司, RNase-free DNase Ⅰ、pGEM-T Easy载体购自Promega公司, M-MLV Frist Strand cDNA Synthesis Kit, Gel Extraction Kit为OMEGA公司产品, TransStart Tip Green qPCR SuperMix购自全式金公司, Taq酶、限制性内切酶、pMD19-T载体购自TaKaRa公司产品,荧光染料及抗淬灭封片剂来自Life公司。其余试剂均为国产分析纯。
1.2 实验方法
总RNA提取和单链cDNA合成采集的体组织和性腺组织样品用HiPure Total RNA Kit (Magen)试剂盒提取总RNA, 使用Nano Q分光光度计检测总RNA的浓度和纯度, 并用1%琼脂糖凝胶电泳检测RNA的完整性, -80℃保存。取1 μg总RNA用M-MLV Frist Strand cDNA Synthesis Kit (OMEGA)合成第一条链cDNA, 操作按照试剂盒说明书进行。
中华鳖vasa基因全长cDNA克隆根据NCBI (XM_006139555.1)上预测的中华鳖vasa基因序列,设计特异性引物Ps vasaF和Ps vasaR (表 1)进行PCR扩增反应, 克隆获得中华鳖vasa cDNA序列。PCR反应条件为: 94℃ 3min; 98℃ 10s, 66℃ 40s, 72℃4min共38个循环, 72℃ 5min。PCR产物用1%琼脂糖凝胶电泳, 确定目的片段后用Gel Extraction Kit (OMEGA)胶回收, 克隆到pMD19-T (TaKaRa)载体中, 重组质粒转化至DH5α感受态细胞中, 经蓝白筛选挑取阳性克隆测序。根据获得的Ps vasa cDNA序列, 使用Vector NTI Advance 11软件预测氨基酸序列。使用NCBI数据库上的BLASTN进行核苷酸序列比对, BLASTP进行氨基酸序列比对并同多种脊椎动物进行序列对比分析。采用MEGA 6软件,按邻近法(Neighbour-Joining, NJ)构建系统进化树,用自展法(Bootstrap)进行1000次检验。
荧光定量PCR根据测序所得的中华鳖vasa cDNA序列, 设计一对特异的定量引物PSvas1PS-vas2 (表 1), 以中华鳖ef1α为内参基因, 设计引物ef1αF和ef1αR (表 1)。取1冬龄个体的精巢、卵巢、心、肝脏、脾、肾、脑、血液的总RNA, 用反转录试剂盒合成cDNA, 用该cDNA作为模板, 进行PCR扩增, 每组设3个重复。扩增反应在Applied Biosystems StepOnePlus Real-Time PCR Systems仪器上进行, 采用2-ΔΔCt法来计算相对表达量, 反应程序: 50℃2min, 95℃预变性2min; 95℃ 15s, 60℃ 30s, 72℃30s, 40个循环。溶解曲线程序: 95℃ 15s, 60℃ 30s, 95℃ 15s。

表 1 中华鳖vasa基因克隆和mRNA表达引物Tab. 1 Primers used for vasa gene cloning and expression analysis of Chinese Soft-shell Turtle
原位杂交RNA探针制备选取中华鳖vasa cDNA序列3′端非编码区(3′UTR)中的一段约1.3 kb的序列, 设计引物3′vasaF和3′vasaR (表 1)。将这段序列连接到pGEM-T载体中, 筛选阳性克隆提取质粒。使用限制性内切酶将载体质粒线性化, 加入乙醇和醋酸钠沉淀过夜。沉淀经75%乙醇洗涤干燥后溶于DEPC水中。采用digoxigenin (DIG) RNA Labeling Kit, Roche体外转录正反义探针, 操作步骤按照说明书进行。
原位杂交操作剪刀剪取小块的性腺组织,放入4%PFA中4℃固定过夜。用DEPC水配置的甲醇溶液梯度脱水(25%、50%、75%和100%), 保存在100%甲醇中-20℃保存。取一小块组织用DEPC水配置的甲醇溶液梯度复水(75%、50%、25%), 然后用1×PBS洗涤2次, 最后放入30%蔗糖溶液中4℃过夜。用OCT包埋组织块, 冷冻切片机切成6—8 μm厚的切片贴于防脱载玻片上。杂交具体过程依照已有报道进行[9]。化学显色采用BCIP/NBT显色液进行显色, 荧光显色则用TSATMPlus Fluorescence Systems (NEN Life Science)显色, 碘化丙啶(PI)染核, 抗淬灭封固剂(Gold Antifade reagent, Invitrogen)封片, 显微观察拍照。
2 结果
2.1 中华鳖vasa cDNA克隆及序列分析
实验克隆得到中华鳖vasa cDNA全序列与NCBI GenBank预测的DDX4序列的一致性达~99% (XM_006139555.1), cDNA全长3865 bp, 5′端非编码区(5′UTR)90 bp, 3′端非编码区(3′UTR)1699 bp, 开放阅读框(Open reading frame, ORF)长2076 bp, 编码691个氨基酸。氨基酸序列包含DEAD-box家族蛋白8个保守功能域, 分别是AQTGSGKT (motifⅠ)、PTRELI (motifⅠa)、TPGR (motifⅠb)、DEAD (motifⅡ)、SAT (motif Ⅲ)、RGLD (motif Ⅴ)、HRIGRTG (motif Ⅵ)、GYSKLTPVQ (motif Q); 在N末端有4个RGG重复序列和2个GG(甘氨酸)富集区。因此将克隆得到的中华鳖cDNA序列命名为Psvasa , 并提交NCBI GenBank (登录号: KT934805)。将推导出的中华鳖Vasa氨基酸序列与几种代表性脊椎动物相应的序列进行同源性比对, 结果显示中华鳖Vasa氨基酸序列与小鼠(Mus musculus)同源性较高为72%, 与鸭嘴兽(Ornithorhynchus anatinus)、安乐蜥(Anolis carolinensis)的同源性均为69%, 与鸡(Gallus gallus)、热带爪蟾(Xenopus tropicalis)、金目鲈(Lates calcarifer)、斑马鱼(Danio rerio)、果蝇(Drosophila melanogaster)的同源性分别在59%至42%之间(未发表数据)。运用MEGA 6软件构建NJ系统进化树(图 1), 结果表明中华鳖PsVasa序列属于Vasa家族, 处于爬行类与哺乳类之间, 并与哺乳类Vasa同源性更高。
2.2 中华鳖vasa mRNA组织表达特征
以中华鳖ef1α基因作为内参基因, 以Psvasa基因特异性引物作为扩增引物, 利用qRT-PCR检测Psvasa转录本在不同组织中的表达水平。结果显示: Psvasa mRNA在精巢和卵巢中均有较高水平的表达, 但在心、肝脏、脾、肾、脑、血液中几乎检测不到表达信号(图 2)。这说明中华鳖vasa基因和其他很多动物一样, 仅在性腺中表达。
2.3 中华鳖vasa mRNA在卵子发生过程中的表达分布
通过对中华鳖不同发育期卵巢的冰冻切片进行化学原位杂交分析, 发现Psvasa mRNA仅在生殖细胞的细胞质中有表达, 在体细胞中未检测到表达信号。而且在卵子发生过程中的早期卵母细胞中可检测到很强的Psvasa mRNA信号: 在卵原细胞中具有较强的表达, 在初级卵母细胞期表达最强, 且信号在整个细胞质中均匀分布, 生长期卵母细胞信号主要集中在核周区域且随着卵母细胞逐渐发育,卵母细胞核周区域信号也逐渐减弱(图 3A、B), 在成熟期卵母细胞中几乎检测不到信号(图 3C)。
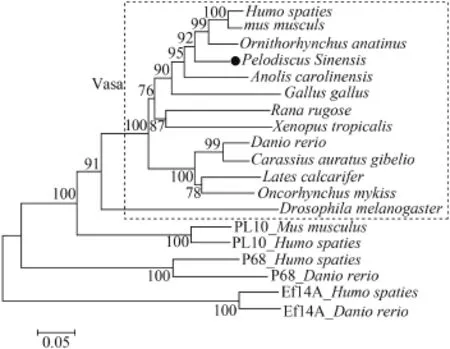
图 1 NJ法构建系统进化树Fig. 1 Phylogenetic tree generated with the neighbor-joining method所用氨基酸序列包括All amino acid sequences including: 中华鳖(Pelodiscus Sinensis) Vasa, ALK02502; 人(Humo spaties) Vasa, AAF86585; 小鼠(Mus musculus) Vasa, AAI37602.1; 鸭嘴兽(Ornithorhynchus anatinus) Vasa, NP_001233782; 蜥蜴(Anolis carolinensis) Vasa, ENSACAT00000015036; 鸡(Gallus gallus) Vasa, NP_990039.2; 蛙(Rana rugosa) Vasa, AB372211; 爪蟾(Xenopus tropicalis) Vasa, NP_001016823; 斑马鱼(Danio rerio) Vasa, AAI29276.1; 银鲫(Carassius auratus gibelio) Vasa, AAV70960; 金目鲈(Lates calcarifer) Vasa, AHG95980.1; 虹鳟(Oncorhynchus mykiss) Vasa, BAA88059; 果蝇(Drosophila melanogaster) Vasa, CAA31405.1; 鼠(Mus musculus) PL10, NP_149068; 斑马鱼(Danio rerio) PL10(NP571016); 人(Humo spaties) P68, NP_004387; 斑马鱼(Danio rerio) P68(AAI59199);人(Humo spaties) Efl4A(NP_001407); 斑马鱼(Danio rerio) Efl4A (AAI53508)虚线框内为Vasa蛋白家族

图 2 中华鳖vasa基因转录本的组织特异性表达分析Fig. 2 The mRNA level of vasa in multiple tissuesHeart: 心脏, Liver: 肝脏, Spleen: 脾, Kidney: 肾, Brain: 脑, Blood: 血液, Testis: 精巢, Ovary: 卵巢; ** P<0.01
通过高灵敏度荧光原位杂交进行进一步验证,结果显示: Psvasa mRNA的荧光杂交信号与化学杂交法的基本一致。在卵原细胞、初级卵母细胞、生长期卵母细胞中均有表达, 卵原细胞中信号较强,在初级卵母细胞及生长期卵母细胞最开始阶段信号最强, 且均匀分布在细胞质中, 之后主要集中分布在生长期卵母细胞的核周区域(图 4)。
3 讨论
3.1 中华鳖vasa cDNA的鉴定
本研究在中华鳖中扩增得到了一个编码DEAD-box家族蛋白的cDNA序列, 比对结果表明, 扩增获得的cDNA序列比GenBank (XM_006139555.1)中序列极为一致, 达99%相似性。然而, 本研究扩增获得的cDNA序列编码的蛋白序列与DEAD-box家族蛋白具有更高的同源性(未发表数据), 其编码的蛋白与很多已知的其他物种的Vasa蛋白一样包含DEAD-box家族特有的8个功能域[28,29], 而且其N末端具有多个RGG重复序列和GG (甘氨酸)富集区,推测与RNA的结合有关[30], 并与DEAD-box家族中的其他蛋白有相互作用[31,32]。因此本研究获得的cDNA序列, 编码的蛋白命名为PsVasa。同时, 通过DEAD家族基因蛋白同源性比对及系统进化树分析, 结果表明PsVasa归属于Vasa蛋白一支(图 1), 而非DEAD-box家族的其他蛋白如PL10、PL68等。在系统进化树上, PsVasa居于蜥蜴与哺乳类之间,与哺乳动物(小鼠、鸭嘴兽)的同源性更高, 预示着中华鳖PsVasa在生殖细胞中的调控作用更接近哺乳类, 其生殖细胞的发育方式也可能更类似于哺乳类的。
3.2 中华鳖vasa 基因转录本在卵母细胞中的表达定位分析
迄今为止, 关于vasa基因的组织特异性表达及功能在研究界仍有许多争议。在已报道的绝大多数物种中, vasa基因主要作用于生殖细胞的发育,在性腺组织中特异表达[9,33]。与此相似, 在本研究的中华鳖中, 荧光定量RT-PCR结果显示, Psvasa mRNA在性腺(卵巢和精巢)中高表达, 在体组织中表达很低甚至难以检测到。而在某些物种中的研究显示, vasa基因在性腺外的体组织中也有少量表达, 如非洲爪蟾[34]和线虫[35]的体细胞, 虹鳟的脑和心脏[11], 黑斑蛙的脑、肌肉、肾脏和心脏[36]均检测到了vasa基因的表达。据分析vasa可能不仅调控生殖细胞的发育, 还通过调节有关mRNAs的转录表达, 来维持细胞的全能性[34,35]。总之, vasa基因的功能, 特别是随着脊椎动物的进化而发生的演变, 还需要大量的研究工作加以验证及阐明。

图 3 化学原位杂交法分析卵巢中vasa转录本的表达Fig. 3 The vasa mRNA level in adult ovary by chemical in situ hybridization (CISH)卵巢切片组织RNA探针杂交, 包括反义探针(A-C)和正义探针(D)。阴性对照没有信号。在卵原细胞、初级卵母细胞、生长期卵母细胞中均有表达, 卵原细胞中信号较强, 在初级卵母细胞及生长期卵母细胞最开始阶段中信号最强, 且均匀分布在细胞质中, 之后在生长期卵母细胞的核周区域表达较强, 而卵周区域的信号较弱, 且随着卵母细胞逐渐发育, 卵母核周区域信号也逐渐减弱, 在成熟期卵母细胞中几乎检测不到信号。Ⅲ-Ⅴ为初级卵母细胞期; Ⅵ-Ⅸ为生长期卵母细胞; Ⅹ为成熟期卵母细胞Adult ovary cryosections were hybridized withantisense (A-C) and sense (D) RNA probes. The signals were developed by alkaline phosphatase (AP) staining (purple). No signal is detected by sense probe. The Psvasa mRNA signals strongly appeared in oogonia and increased to peak from primary oocyte to early growing oocyte stage and displayed a uniform distribution. As oocyte grown into larger size, the signal in the perinuclear cytoplasm became much stronger, however, the signal in oocyte became weak when vitellogenesis further develop. Primary oocytes are stageⅢ-Ⅴ; Growing oocytes are stageⅥ-Ⅸ; Mature oocytes are stage Ⅹ
脊椎动物的配子发生是一个复杂的过程, 包括生殖干细胞的自我更新和分化, 减数分裂以及减数分裂后形态上的变化。RT-PCR结果已表明vasa基因在中华鳖性腺中特异表达, 且卵巢组织冰冻切片原位杂交分析结果显示vasa mRNA只在生殖细胞中表达, 因此vasa基因也可以作为生殖细胞特异性标记分子来研究中华鳖卵子发生过程及卵母细胞分化成熟机制。中华鳖的卵子发生主要分为4个时期: 卵原细胞期、初级卵泡期、生长卵泡期、成熟卵泡期, 其中生长卵泡期又分为小生长期和大生长期, 卵黄积累开始于大生长期[25]; 也有研究人员将其划分为卵黄发生前期和卵黄发生期, 又根据透明带、滤泡细胞等结构特点将整个过程细分为10个期[37]。本实验参照这后一种方法对卵子进行分期, Psvasa在卵子发生中的生殖细胞中呈现动态的变化: 在卵原细胞中起始表达, 在初级卵母细胞及生长期卵母细胞最早期中表达量升高且在整个细胞质内均分分布, 之后Psvasa在生长期卵母细胞的核周区域信号聚集, 随着卵母细胞逐渐增长, 核周区域的信号也慢慢减弱, 直至在接近成熟的卵母细胞中几乎检测不到Psvasa表达信号(图 3)。
Psvasa在初级卵母细胞及生长期卵母细胞最早期中表达量最高可能与这时期卵母细胞中RNA,蛋白质等物质大量积累有关。而在生长期卵母细胞中Psvasa表达逐渐减弱可能和该时期卵母细胞体积不断变大, Psvasa mRNA相对密度减少或者是Psvasa mRNA表达水平下降等因素有关。Psvasa mRNA在雌性生殖细胞中的表达谱与巴西红耳龟中的表达较为相似。巴西红耳龟vasa mRNA在初级卵母细胞中表达最强且信号均匀分布在细胞质中, 卵黄生成进行中的卵母细胞里, 其信号也有体现出核周的信号要稍强于胞质的其他区域的现象,而且随着生长期卵母细胞的增大, 细胞质中的信号也逐渐减弱[12]。但与鱼类vasa基因表达分布模式存在很大的不同, 例如在银鲫中vasa mRNA在整个卵子发生过程中均有表达, 表达水平呈现先升高后降低的模式[9], 银鲫vasa mRNA表达最强是在卵黄生成前和卵黄生成早期卵母细胞中并且同样也是均匀分布在胞质中, 而中华鳖中表达水平最高是在初级卵母细胞以及生长期卵母细胞的最早期。中华鳖从生长期开始信号就主要集中在核周围区域,但银鲫中信号集中在核周围是在卵黄生成中期的卵母细胞中。随着卵黄发生的继续进行, 两者在胞质中的信号都慢慢减弱, 在成熟的银鲫卵母细胞中, vasa信号定位于卵母细胞特定区域, 特别是在金目鲈生长期卵母细胞中发现vasa基因主要集中分布于巴氏小体(Balbiani body, BB)[10], 而中华鳖卵母细胞中没有发现BB结构, 而且其成熟期卵母细胞中甚至没有检测到Psvasa信号。Psvasa mRNA在卵母细胞中的表达模式与哺乳类卵母细胞vasa基因产物的表达方式接近, 小鼠vasa 基因产物在卵母细胞中均匀分布且无生殖质结构的存在[20], 从而决定了小鼠PGCs的起源方式与鱼类的不同[18]: 小鼠PGCs不受任何母源因子的调控, 是依靠细胞间的交互作用, 即细胞非自主的方式形成的, 属后成论模式。而鱼类PGCs是有vasa等生殖质因子调控形成的, 属先成论模式。本研究发现中华鳖vasa转录本在其卵母细胞中的表达模式不同于鱼类, 而类似于哺乳类的, 暗示中华鳖PGCs的形成不是由生殖质调控的先成模式, 至于是否行驶哺乳类的后成模式, 还需要进一步研究证明。

图 4 荧光原位杂交法分析卵巢中vasa转录本的表达分布Fig. 4 The expression of vasa mRNA level in adult ovary by fluorescence in situ hybridization (FISH)卵巢切片组织RNA探针杂交。Psvasa mRNA仅在生殖细胞中表达, 在体细胞中未见表达。而且在卵子发生过程中的早期卵母细胞中可检测到很强Psvasa mRNA信号, 在卵原细胞中具有较强的表达, 在初级卵母细胞期表达最强, 且信号在整个细胞质中均匀分布, 生长期卵母细胞中信号主要集中在核周区域。Ⅲ-Ⅴ为初级卵母细胞期; Ⅵ-Ⅸ为生长卵母细胞期; Ⅹ为成熟期卵母细胞。vasa信号(绿色); PI染核(红色); 比例尺, 100 μmAdult ovary cryosections were hybridized with RNA probes. The vasa signals were developed by TSA kit (Green) and the nuclear was stained with PI (Red). The Psvasa mRNA signals were strong in oogonia and reached to peak level from primary oocyte to early growing oocyte stage and displayed a uniform distribution. As growing oocytes grown into larger sizes, the signals in the perinuclear cytoplasm became much stronger, while the signals in oocytes became weak with the vitellogenesis fruther develop. Primary oocytes are stage Ⅲ-Ⅴ; Growing oocytes are stage Ⅵ-Ⅸ; Mature oocytes are stage Ⅹ; Scale bar, 100 μm
Psvasa mRNA特异于生殖细胞的表达模式及在卵子发生过程中呈现的时间和空间上的动态变化, 预示Psvasa对中华鳖的卵子发生过程起重要的调控作用, 同时可作为可靠的分子标记物来区分不同发育时期的卵母细胞。更为重要的是本研究结果为将来通过Psvasa标记分子进行中华鳖PGCs的起源、迁移、分化等方面的研究奠定了基础。
[1]Mehrnoush Dehghani, Paul Lasko. C-terminal residues specific to Vasa among DEAD-box helicases are required for its functions in piRNA biogenesis and embryonic patterning [J]. Development Genes and Evolution, 2016, 226: 401—412
[2]Mehrnoush Dehghani, Paul Lasko. In vivo mapping of the functional regions of the DEAD-box helicase Vasa [J]. Biology Open, (2015) 4, 450—46
[3]Cao G, Zhang Y, Xue R, et al. Alternative splicing, expression patterns and promoter characters of vasa-like gene from the silkworm, Bombyx mori [J]. Molecular Biology Reports, 2012, 39(5): 5967—5976
[4]Reunov A A, Reunova Y A. In mouse oocytes the mitochondrion-originated germinal body-like structures accumulate mouse Vasa homologue (MVH) protein [J]. Zygote, 2015, 23(4): 1—6
[5]Castrillon D H, Quade B J, Wang T Y, et al. The humanVASA gene is specifically expressed in the germ cell lineage [J]. Proceedings of the National Academy of Sciences of the United States of America, 2000, 17(97): 9585—9590
[6]Tsunekawa N, Naito M, Sakai Y, et al. Isolation of chicken vasa homolog gene and tracing the origin of primordial germ cells [J]. Development, 2000, 127(12): 2741—2750
[7]Hartung O, Forbes M M, Marlow F L. Zebrafish vasa is required for germ-cell differentiation and maintenance [J]. Molecular Reproduction and Development, 2014, 81(10): 946—961
[8]Yuan Y, Li M, Hong Y. Light and electron microscopic analyses of Vasa expression in adult germ cells of the fish medaka [J]. Gene, 2014, 545(1): 15—22
[9]Xu H, Gui J, Hong Y. Differential expression of vasa RNA and protein during spermatogenesis and oogenesis in the gibel carp (Carassius auratus gibelio), a bisexually and gynogenetically reproducing vertebrate [J]. Developmental dynamics: an official publication of the American Association of Anatomists, 2005, 233(3): 872—882
[10]Xu H, Lim M, Dwarakanath M, et al. Vasa identifies germ cells and critical stages of oogenesis in the Asian seabass [J]. International Journal of Biological Sciences, 2014, 10(2): 225—235
[11]Wu X, Wang Z, Jiang J, et al. Cloning, expression promoter analysis of vasa gene in Japanese flounder (Paralichthys olivaceus) [J]. Comparative Biochemistry and Physiology Part B Biochemistry and Molecular Biology, 2014, 167(1): 41—50
[12]Bachvarova R F, Crother B I, Manova K, et al. Expression of Dazl and Vasa in turtle embryos and ovaries: evidence for inductive specification of germ cells [J]. Evolution and Development, 2009, 11(5): 525—534
[13]Maurizii M G, Cavaliere V, Gamberi C, et al. Vasa protein is localized in the germ cells and in the oocyte-associated pyriform follicle cells during early oogenesis in the lizard Podarcis sicula [J]. Development Genes and Evolution, 2009, 219(7): 361—367
[14]Chen Y D, Zou Z H, Wang Y L, et al. Progress in studies of vasa gene [J]. Chinese Journal of Zoology, 2010, 45(4): 173—180 [陈玉冬, 邹志华, 王艺磊, 等. vasa基因研究进展. 动物学杂志, 2010, 45(4): 173—180]
[15]Lasko P. The DEAD-box helicase Vasa: evidence for a multiplicity of functions in RNA processes and developmental biology [J]. Biochimica et Biophysica Acta, 2013, 1829(8): 810—816
[16]Hay B, Jan L Y, Jan Y N. Localization of vasa, a component of Drosophila polar granules, in maternal-effect mutants that alter embryonic anteroposterior polarity [J]. Development, 1990, 109(2): 425—433
[17]Tanaka S S, Toyooka Y, Akasu R K, et al. The mouse homolog of Drosophila Vasa is required for the development of male germ cells [J]. Genes and Development, 2000, (14): 841—853
[18]Xu H Y, Li M Y, Gui J F, et al. Fish germ cells [J]. Science China: Life Sciences, 2010, 53(4): 435—446
[19]Hay B, Jan L Y, Jan Y N. A protein component of Drosophila polar granules is encoded by vasa and has extensive sequence similarity to ATP-dependent helicases [J]. Cell, 1988, 55(4): 577—587
[20]Toyooka Y, Tsunekawa N, Takahashi Y, et al. Expression and intracellular localization of mouse VASA-homologue protein during germ cell development [J]. Mechanisms of Development, 2000, 93(1—2): 139—149
[21]Extavour C G, Akam M. Mechanisms of germ cell specification across the metazoans: epigenesis and performation [J]. Development, 2003, 130(24): 5869—5884
[22]Shine R. Reptiles [J]. Current Biology, 2013, 23(6): 227—231
[23]Huang L Y, He Z Y, Ding S H, et al. Current status and countermeasures for protecting and utilizing on softshelled turtle (Pelodiscus sinensis) germplasm resources in China [J]. Journal of Ningbo University, 2005, 18(2): 183—186 [黄丽英, 何中央, 丁诗华, 等. 中华鳖种质资源的研究现状及保护、利用对策. 宁波大学学报: 理工版, 2005, 18(2): 183—186]
[24]Zhou T, Li P P. Chelonian species diversity and current status in China [J]. Sichuan Journal of Zoology, 2007, 26(2): 463—467 [周婷, 李丕鹏. 中国龟鳖物种多样性及濒危现状. 四川动物, 2007, 26(2): 463—467]
[25]Hei N, Yang P, Yang Y, et al. Fine structural observation on the oogenesis and vitellogenesis of the Chinese softshelled turtle (Pelodiseus sinensis) [J]. Zygote, 2009 (18): 109—120
[26]Zhu D Y. Morphological and histological obervation of gonadal development in soft-shelled turtle, Trionyx sinensis [J]. Acta Hydrobiologica Sinica, 2009, 33(5): 924—930 [朱道玉. 中华鳖性腺的发生与发育研究. 水生生物学报, 2009, 33(5): 924—930]
[27]Zhang L, Yang P, Bian X, et al. Modification of sperm morphology during long-term sperm storage in the reproductive tract of the Chinese soft-shelled turtle, Pelodiscus sinensis [J]. Scientific Reports, 2015, (5): 16096
[28]Rocak S, Linder P. DEAD-box proteins: the driving forces behind RNA metabolism [J]. Molecular Cell Biology, 2004, 5(3): 232—241
[29]Tanner N K. The newly identified Q motif of DEAD box helicases is involved in adenine recognition [J]. Cell Cycle, 2003, 2(1): 18—19
[30]Bartfai R, Orban L. The vasa locus in zebrafish: multiple RGG boxes from duplications [J]. DNA Cell Biology, 2003, (22): 47—54
[31]Benz J, Trachsel H, Baumann U. Crystal structure of the ATPase domain of translation initiation factor 4A from Saccharomyces cerevisiae-the prototype of the DEADbox protein family [J]. Structure, 1999, 7(6): 671—679
[32]Gibson T J, Thompson J D. Detection of dsRNA-binding domains in RNA helicase A and Drosophila maleless: implications for monomeric RNA helicases [J]. Nucleic Acids Research, 1994, 22(13): 2552—2556
[33]Castrillon D H, Quade B J, Wang T Y, et al. The human VASA gene is specifically expressed in the germ cell lineage [J]. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(17): 9585—9590
[34]Ikenishi K, Tanaka T S. Spatio-temporal expression of Xenopus vasa homolog, XVLG1, in oocytes and embryos: The presence of XVLG1 RNA in somatic cells as well as germline cells [J]. Development Growth and Differentiation, 2000, 42(2): 95—103
[35]Seydoux G, Strome S. Launching the germline in Caenorhabditis elegans: regulation of gene expression in early germ cells [J]. Development, 1999, 126(15): 3275—3283
[36]Jia R, Nie L W, Wang N, et al. Molecular cloning and expression patterns of the Vasa gene from Rana nigromaculata (Amphibia: Anura) [J]. Zoologia, 2009, 26(26): 316—322
[37]Hei N, Yang P, Yang Y, et al. Fine structural observation on the oogenesis and vitellogenesis of the Chinese softshelled turtle (Pelodiseus sinensis) [J]. Zygote, 2010, 18(2): 109—120
CLONING AND EXPRESSION ANALYSIS OF VASA IN CHINESE SOFT-SHELL TURTLE OOCYTES
ZHANG Piao-Yi1,2, LI Wei1, ZHU Xin-Ping1,2, HONG Xiao-You1,2, CHEN Kun-Ci1,2and XU Hong-Yan1,2
(1. Key Laboratory of Tropical & Subtropical Fishery Resource Application & Cultivation of Ministry of Agriculture, Pearl River Fisheries Research Institute, Chinese Academy of Fishery Sciences, Guangzhou 510380, China; 2. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306, China)
vasa encodes a DEAD-box RNA helicase and is a highly conserved germ cell marker across animal phyla. vasa is necessary for germ cell development during embryogenesis and gametogenesis. The Chinese soft-shell turtle (Pelidiscus sinensis) was used as a model to study germ cell development and differentiation in reptiles. Here a full vasa cDNA was cloned, 3865 bp in total, containing a 5′-UTR of 90 bp, a 3′-UTR of 1699 bp and an open reading frame (ORF) of 2076 bp, encoding a protein of 691 aa. The deduced amino acid sequence contains 8 conserved motifs of DEAD-box family protein, and possesses 4 RGG repeats and 2 GG repeats. The predicted protein is 72% identical in sequence to its homologs from mouse. The vasa mRNA was detected exclusively in the gonads of both sexes in softshell turtle. Chromogenic and fluorescent RNA in situ hybridization revealed that the vasa mRNA was restricted to germ cells: It was highly expressed from primary oocyte to early growing oocyte and uniformly distributed in oocyte cytoplasm. In oocytes at the late stage, the vasa mRNA was perinuclear distributed and decreased in oocytes with the vitellogenesis proceeding. The vasa mRNA signal was undetectable in the mature oocytes. The findings indicated that vasa gene also played an important role in oogenesis in the Chinese soft-shell turtle. Hence turtle vasa is a reliable germ cell marker to identify female germ cells at different stages during oogenesis. These results provide a theoretical basis to study the germ cell development and differentiation during embryogenesis and /or gametogenesis, especially to investigate the initiation and migration of primordial germ cells (PGCs) in Chinese soft-shell turtle and reptiles.
vasa gene; Chinese soft-shell turtle; Germ cell; Oogenesis
Q786
A
1000-3207(2017)02-0306-08
10.7541/2017.37
2016-04-18;
2016-07-18
广东省科技计划项目-国际科技合作领域(2016A050502029); 广东省科技计划项目(2014A020208037); 引进国际先进农业科学技术‘948’项目(2015-Z019)资助 [Supported by Science and Technology Projects of Guangdong Province (2016A050502029); Science and Technology Projects of Guangdong Province (2014A020208037); 948 Foundation of Ministry of Agriculture (2015-Z019)]
张飘逸(1991—), 女, 浙江东阳人; 硕士研究生; 主要研究方向为水产种质资源与遗传育种。E-mail: zhangpiaoyi00000 @126.com
朱新平, 博士, 研究员; E-mail: zhuxinping_1964@163.com; 徐红艳, 博士, 研究员; E-mail: xuhyzqh@163.com
