丙型肝炎病毒蛋白对肝细胞RASSF2 mRNA表达的影响及其机制
2017-03-31陈炜冯德云李波王颖
陈炜,冯德云,李波,王颖
(中南大学湘雅医院 病理科,湖南 长沙 410008)
丙型肝炎病毒(hepatitis C virus,HCV)感染是肝细胞癌发生的重要诱因之一,据统计,全世界有2亿多人感染了HCV病毒,约占了世界人口的3.3%。HCV蛋白受宿主和病毒蛋白酶的作用被切割形成至少10种蛋白,包括4种结构蛋白(核心蛋白Core、包膜蛋白E1和E2,还有p7离子通道)和6种非结构蛋白(NS2、NS3、NS4A、NS4B、NS5A、NS5B)。随着对肿瘤发病机制认识的深入,表观遗传学改变在肿瘤发生中的作用已日渐受到重视。DNA甲基化是表观遗传学的重要表现形式,是调节基因组功能的重要手段,RASSF2通过启动子甲基化在多种肿瘤中失活[1-7]。它作为一种潜在的抑癌基因,具有促进凋亡和细胞周期停滞及抑制细胞生长的作用[8-9]。RASSF2有3个转录本,分别为RASSF2A、RASSF2B和RASSF2C,仅RASSF2A转录本的启动子区有CpG岛。研究表明,RASSF2A以GTP依赖的方式与K-RAS直接结合,促进细胞凋亡、细胞周期停滞和抑制细胞生长[8],在肺癌、胃癌、乳腺癌及胰腺癌中存在RASSF2A启动子甲基化[1,10-11]。5-杂氮-2'-脱氧胞苷(5-aza-dC)是一种核苷酸类似物,可抑制甲基化转移酶的活性,使DNA去甲基化,体外实验表明,5-aza-dC对胰腺癌、鼻咽癌、胃癌及结肠癌等多种细胞有显著生长抑制作用[12-15],推测HCV蛋白可能是通过诱导RASSF2A基因启动子甲基化,抑制其转录,从而异常激活RAS信号通路,参与肝细胞恶性转化。本研究观察了分别表达NS3、Core、NS5A的QSG7701肝细胞株(NS3/QSG7701、Core/QSG7701、NS5A/QSG7701)中RASSF2 mRNA表达水平的改变,并探讨RASSF2A启动子甲基化在HCV蛋白所致的肝细胞癌中的作用。
1 材料与方法
1.1 主要材料与试剂
稳定表达Core、NS3的人源肝细胞系Core/QSG7701和NS3/QSG7701由本实验室构建及保存;稳定表达NS5A的人源肝细胞系NS5A/QSG7701由中南大学湘雅二医院传染科龚国忠教授惠赠;其中L02细胞株购自中南大学细胞生物学研究室;RT-PCR试剂盒、DNA提取试剂盒、逆转录试剂盒均购自TaKaRa公司;EZDNA甲基化修饰试剂盒购自天漠科技有限公司;5-aza-dC购自Sigma公司;NS3、NS5A、β-Actin单克隆抗体以及Core多克隆抗体均购自Abcam公司;辣根过氧化物酶标记山羊抗兔和羊抗鼠均购自中杉金桥;AnnexinV/PI双染试剂盒购自联科生物;细胞增殖与毒性检测试剂盒购自七海复泰。RASSF2基因的PCR引物由上海生物工程有限公司合成(正义:CTA TGG CTC TGT CAC CAA CG,反义:GCT TCT GTT TCT CAC CAC TCG,长度128 bp),GAPDH引物(正义:GTG AAC CAT GAG AAG TAT GAC AAC,反义:CAT GAG TCC TTC CAC GA TAC C,长度123 bp);RASSF2A启动子甲基化引物(正义:GTT CGT CGT CGT TTT TTA GGC G,反义:AAA AAC CAA CGA CCC CCG CG,长度108 bp),RASSF2A启动子未甲基化引物(正义:AGT TTG TTG TTG TTT TTT AGG TGG,反义:AAA AAA CCA ACA ACC CCC ACA,长度108 bp)。
1.2 主要方法
1.2.1 RT-PCR 按照细胞中提取RNA的步骤所示取出生长良好的细胞,用PBS清洗3次,1 mL的TRIzol试剂吹打,200 μL的三氯甲烷孵育5 min,500 μL的异丙醇孵育10 min,75%的乙醇吹打混匀,离心后静置干燥5 min。加入10 μL的DEPC水,吹打混匀,取2 μL RNA样本,紫外分光光度计检测RNA样本的浓度和纯度,再根据TAKARA公司的逆转录试剂盒所示配制基因组DNA去除的反应液,共 10 μL,反应条件为 42 ℃ 2 min,4 ℃存放。配制20 μL的逆转录反应液,反应条件为37 ℃15 min,85 ℃ 5 s,4 ℃存放,根据康维世纪PCR试剂盒进行扩增,扩增条件为94 ℃预变性2 min,94 ℃变性 30 s,61 ℃退火 30 s,72 ℃延伸 30 s,72 ℃终延伸2 min,共35个循环,扩增结束后取出样本,配制好2%的琼脂糖凝胶,直接进行电泳。
1.2.2 甲基化特异性PCR(MSP) 细胞培养好后取出,胰酶消化后,用1 mL的PBS吹打,离心后加入200 μL的PBS重悬细胞,根据TAKARA公司的全基因组DNA提取试剂盒所示,最终洗脱DNA,再取20 μL DNA按照天漠公司的甲基化修饰试剂盒所示加入CT Conversion Reagent,放到PCR仪上,设置程序为98 ℃ 10min,64 ℃ 2.5 h,接着继续按试剂盒所示最终洗脱DNA,按照康维世纪盒所示配制50 μL PCR扩增反应液,设置反应条件为94 ℃预变性2 min,94 ℃变性30 s,61 ℃退火30 s,72 ℃延伸30 s,72 ℃终延伸2 min,共35个循环,扩增后取反应产物,配制好2%的琼脂糖凝胶后,进行电泳。
1.2.3 Western blot 首先按步骤提取细胞中的总蛋白,准备4 ℃离心机,用冰的PBS洗涤3次,每瓶细胞中加入100 μL的细胞裂解混合物,冰上裂解30 min,离心后得到蛋白,用BCA法测定样本OD值,每管加入25 μL的标准品和200 μL已配制好的工作液,37 ℃孵育30 min,568 nm处测定OD值。然后每管加入22.5 μL的双蒸水、2.5 μL的待测蛋白及200 μL的分析液,37 ℃孵育30 min,测OD值计算蛋白浓度,再按比例将蛋白与上样缓冲液混合,沸水5 min,按步骤配好分离胶和积层胶后,将Maker、混合物和上样缓冲液加入到凝胶孔中,连接电泳装置,65 V电压50 min,110 V电压60 min,完毕后取出凝胶,配制好转膜缓冲液,并加入甲醇,将凝胶和滤纸直接放入到转膜缓冲液中,PDVF膜甲醇浸泡15 s,双蒸水平衡5 min,放到转膜缓冲液里。安装三明治结构,从下到上依次为滤纸→PVDF膜→凝胶→滤纸。15 V恒压45 min。配制好封闭液,PVDF膜放到封闭液中,摇晃4 h。接着一抗孵育(摇床2 h,4 ℃冰箱过夜),TBST洗涤3次,每次10 min,二抗孵育4 h,TBST洗涤3次,每次10 min。打开显像仪,设置曝光程序为每5s曝光1次,共曝100张。
1.2.4 MTT 待细胞生长良好后,PBS洗3次,胰酶消化吹打细胞并计数。每孔加入2 500个细胞,设置5个复孔,且每株细胞分别为对照组和实验组,培养箱中培养24、48、72 h后,避光加入20 μL(5 mg/mL)的MTT试剂,继续培养4 h。加入150 μL的DMSO,摇晃20 min后,酶标仪490 nm处测其OD值。
1.2.5 流式细胞术 取生长良好的细胞,胰酶消化后分组,再加入不同浓度药物,贴壁生长后用预冷PBS洗涤3次,并重悬。再按照试剂盒所示加入相应试剂,上机前加入10 μL的PI,避光孵育10 min。加入400 μL的1×结合缓冲液,上机并记录结果。
1.3 统计学处理
采用PASW Statistics 18软件进行统计分析,实验数据用均数±标准差(±s)表示,计量资料组间比较采用独立样本t检验,P<0.05认为差异具有统计学意义。
2 结 果
2.1 NS3/QSG7701、Core/QSG7701、NS5A/QSG7701细胞中相应蛋白表达
提取分别转染空载体pRcCMV、NS3、NS5A和Core表达质粒的QSG7701细胞的总蛋白,检测NS3、Core、NS5A蛋白的表达,结果显示转染了NS3、NS5A和Core表达质粒的细胞均有相应蛋白的表达(图1)。
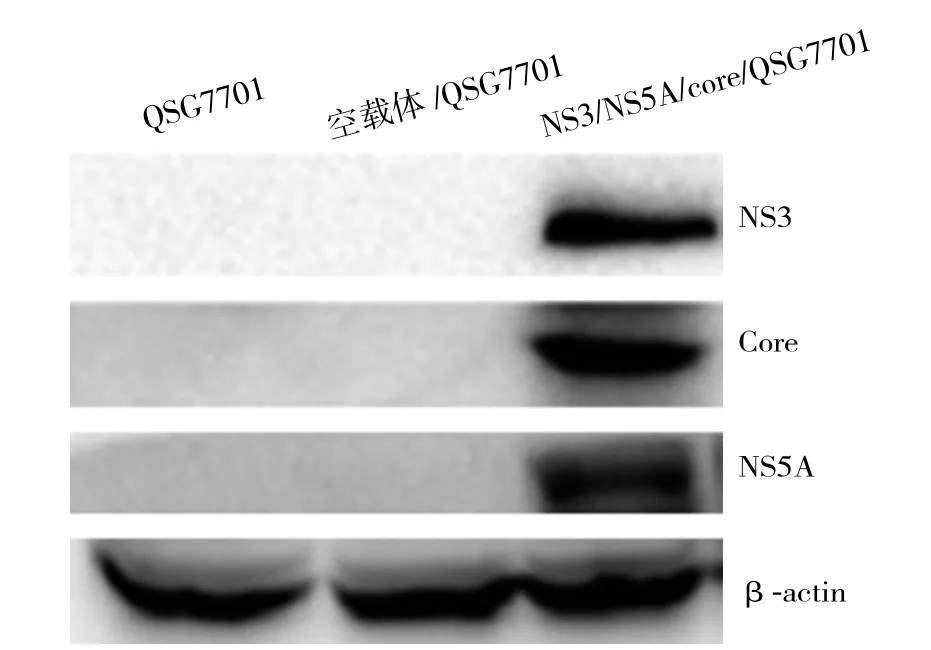
图1 NS3、Core、NS5A蛋白检测Figure1 Detection of expressions of NS3, Core and NS5A proteins
2.2 L02、Core/QSG7701、NS3/QSG7701及 NS5A/QSG7701细胞中RASSF2 mRNA表达
提取L02、Core/QSG7701、NS3/QSG7701及NS5A/QSG7701细胞的总RNA,检测每株细胞RASSF2 mRNA的相对表达量,与L02细胞(RASSF2 mRNA表达量:0.582±0.066)比较,Core/QSG7701(RASSF2 mRNA表达量:0.027±0.004)、NS3/QSG7701(RASSF2 mRNA表达量:0.146±0.074)及NS5A/QSG7701(表达水平0.094±0.014)3株细胞的RASSF2 mRNA的表达均明显降低(P=0.000、0.002、0.000)(图2)。

图2 RASSF2 mRNA表达检测Figure 2 Detection of RASSF2 mRNA expressions
2.3 RASSF2A启动子甲基化检测
提取NS3/QSG7701、Core/QSG7701及NS5A/QSG7701细胞的DNA,进行甲基化的修饰,检测该3株细胞系的RASSF2A启动子的甲基化状态,显示3株细胞RASSF2A启动子均发生了完全甲基化(图3)。

图3 MSP结果 M:甲基化特异性扩增;U:非甲基化特异性扩增Figure 3 Results of MSP M: Methylated-specific methylation;B: Unmethylated-specific methylation
2.4 5-aza-dC处理对各细胞RASSF2 mRNA表达的影响
每株细胞设置3组,分别为未加药物的对照组、5 μmol/L 5-aza-dC实验组和10 μmol/L 5-azadC实验组,培养4 d,检测各组细胞RASSF2 mRNA的表达。结果示:NS3/QSG7701细胞株中,与对照组比较,5 μmol/L 5-aza-dC实验组和10 μmol/L 5-aza-dC实验组RASSF2 mRNA的表达明显增多(P=0.000、0.000);Core/QSG7701细胞株中,与对照组比较,5 μmol/L 5-aza-dC实验组和10 μmol/L 5-aza-dC实验组RASSF2 mRNA的表达明显增多(P=0.023、0.000);NS5A/QSG7701细胞中,与对照组比较,5 μmol/L 5-aza-dC实验组和10 μmol/L 5-aza-dC实验组RASSF2 mRNA的表达无明显变化(P=0.170、0.270)(图4)。
2.5 5-aza-dC处理对NS3/QSG7701和Core/QSG7701细胞生物学行为的影响
将NS3/QSG7701和Core/QSG7701细胞分为未加药物的对照组和10 μmol/L 5-aza-dC处理的实验组,NS3/QSG7701细胞株对照组与实验组凋亡率分别为(11.68±1.05)%、(27.60±1.57)%,差异有统计学意义(P=0.000);Core/QSG7701细胞株对照组与实验组凋亡率分别为(20.38±1.62)%、(56.79±1.92)%,差异有统计学意义(P=0.000)(图5)。同时,于10 μmol/L 5-aza-dC处理0、24、48、72 h后检测细胞的增殖,与对照组比较,NS3/QSG7701细胞72 h后,细胞的增殖明显降低,差异有统计学意义(P<0.05)(表1);Core/QSG7701细胞24、48、72 h时间点,细胞的增殖均明显受到抑制,差异有统计学意义(均P<0.05)(表2)。

图4 5-aza-dC处理后各细胞RASSF2 mRNA的表达 A:NS3/QSG7701细胞;B:Core/QSG7701细胞;C:NS5A/QSG7701细胞Figure 4 RASSF2 mRNA expressions in each type of cells after 5-aza-dC treatment A: NS3/QSG7701 cells; B: Core/QSG7701 cells;C: NS5A/QSG7701 cells

图5 流式细胞术检测细胞凋亡Figure 5 Cell apoptosis detected by flow cytometry
表1 5-aza-dC处理对NS3/QSG7701细胞增殖的影响(OD,±s)Table 1 Effect of 5-aza-dC treatment on proliferation in NS3/QSG7701 cells (OD,±s)

表1 5-aza-dC处理对NS3/QSG7701细胞增殖的影响(OD,±s)Table 1 Effect of 5-aza-dC treatment on proliferation in NS3/QSG7701 cells (OD,±s)
注:1)与0 h比较Note: 1) Comparison with 0-h value
时间 对照组 实验组 P1)0 h 0.09±0.008 0.09±0.008 —24 h 0.453±0.029 0.375±0.045 0.065 48 h 1.260±0.108 1.116±0.003 0.082 72 h 1.458±0.028 1.273±0.017 0.010
3 讨 论
HCV基因组大约有9.6 kb,编码约含3 000个氨基酸的多聚蛋白,能被病毒或宿主细胞的酶剪切成至少10种结构蛋白和非结构蛋白,这些蛋白在病毒的复制和致病过程中起着重要作用[16]。研究[17]表明,表达HCV相关蛋白的质粒转染至正常肝细胞后,能促进肝细胞无限增殖,并导致其恶性转化。DNA甲基化是表观遗传学中最常见的现象,约60%的人类基因启动子区含有CpG岛。CpG岛甲基化使染色体结构发生变化,高度螺旋化,凝缩成团,失去转录活性,从而导致转录抑制。目前发现启动子高甲基化的基因有Rb、P16INK4a、VHL、hMLH,这些启动子基因的高甲基化与细胞周期、凋亡、DNA的修复以及血管生成有关[18-21]。有研究[22-24]显示抑癌基因RASSF2A基因失活最重要的机制是由于其启动子发生了甲基化,在结肠癌的细胞系、结肠腺瘤以及结肠癌中常见RASSF2A启动子甲基化,而正常的结肠黏膜则否。
表2 5-aza-dC处理对Core/QSG7701细胞增殖的影响(OD,±s)Table 2 Effect of 5-aza-dC treatment on proliferation in Core/QSG7701 cells (OD,±s)

表2 5-aza-dC处理对Core/QSG7701细胞增殖的影响(OD,±s)Table 2 Effect of 5-aza-dC treatment on proliferation in Core/QSG7701 cells (OD,±s)
注:1)与0 h比较Note: 1) Comparison with 0-h value
时间 对照组 实验组 P1)0 h 0.09±0.008 0.09±0.008 —24 h 0.394±0.005 0.354±0.012 0.006 48 h 1.150±0.017 0.999±0.028 0.001 72 h 1.417±0.058 1.136±0.029 0.002
既往研究[25]表明表观遗传学的变化是可逆的,DNA甲基转移酶的抑制剂可以改变DNA启动子甲基化的状态,使其发生去甲基化,5-aza-dC是一种DNA甲基转移酶抑制剂,当DNA复制时,它可结合到5-甲基胞嘧啶的位置,使DNA甲基转移酶失活,达到去甲基化的作用,使甲基化的抑癌基因重新活化,抑制细胞生长或促进细胞凋亡。5-aza-dC除了能够诱导DNA去甲基化,还可以使组蛋白去甲基化,它还可还原甲基化或乙酰化所诱导的染色质重塑[26]。在基因转录沉默过程中,组蛋白乙酰化和DNA甲基化之间相互作用,启动子CpG岛的甲基化使局部的组蛋白去乙酰化,组蛋白乙酰化保护DNA不发生甲基化[27]。
本研究检测了正常肝细胞L02和NS3/QSG7701、Core/QSG7701及NS5A/QSG7701细胞系中RASSF2 mRNA的表达,结果显示,与正常的肝细胞L02相比,NS3/QSG7701、Core/QSG7701及NS5A/QSG7701细胞的RASSF2 mRNA的表达均下降,表明NS3、Core和NS5A蛋白能抑制RASSF2 mRNA的表达;MSP检测该3株细胞系中RASSF2A基因启动子甲基化的状态,发现RASSF2A基因启动子均发生了完全甲基化。为了进一步分析RASSF2 mRNA的表达的下降是否与RASSF2A启动子的高甲基化有关,用不同浓度的去甲基化药物5-aza-dC处理了NS3/QSG7701、Core/QSG7701、NS5A/QSG7701细胞,结果发现5-aza-dC处理后,NS3/QSG7701和Core/QSG7701细胞RASSF2 mRNA的表达明显升高,而NS5A/QSG7701细胞RASSF2 mRNA的表达没有明显改变。这说明HCVNS3和Core可能通过RASSF2A启动子甲基化,发挥其转录抑制作用,而NS5A/QSG7701细胞RASSF2 mRNA表达降低,可能除了启动子甲基化外,还有其他表观遗传学修饰参与了转录沉默。
细胞增殖和细胞凋亡是反映细胞生物学行为的主要指标之一,有研究[28-31]证实同为RASSF家族的RASSF5作为RAS效应因子,连接RAS与HIPPO信号通路,在调控蛋白的稳定性和促进衰老的过程中发挥着重要作用,抑癌基因RASSF2A很可能是通过与K-Ras相互作用发挥其促进细胞增殖和抑制细胞凋亡的功能[32],本研究结果显示,10 μmol/L 5-aza-dC处理NS3/QSG7701和Core/QSG7701细胞后,能抑制细胞增殖和促进细胞凋亡,表明HCVNS3和Core可能是通过促进RASSF2A启动子甲基化导致其表达下调,从而在HCV致癌过程中通过影响肝细胞的生物学行为参与肝细胞癌的发生。
[1] Cooper WN, Dickinson RE, Dallol A, et al. Epigenetic regulation of the ras effector/tumour suppressor RASSF2 in breast and lung cancer[J]. Oncogene, 2008, 27(12):1805–1811.
[2] Donninger H, Hesson L, Vos M, et al. The Ras effector RASSF2 controls the PAR-4 tumor suppressor[J]. Mol Cell Biol, 2010,30(11):2608–2620. doi: 10.1128/MCB.00208–09.
[3] Schagdarsurengin U, Richter AM, Hornung J, et al. Frequent epigenetic inactivation of RASSF2 in thyroid cancer and functional consequences[J]. Mol Cancer, 2010, 9:264. doi: 10.1186/1476–4598–9–264.
[4] Guo W, Dong Z, Cui J, et al. Aberrant hypermethylation of RASSF2 in tumors and peripheral blood DNA as a biomarker for malignant progression and poor prognosis of esophageal squamous cell carcinoma[J]. Clin Exp Metastasis, 2016, 33(1):73–85.
[5] Perez-Janices N, Blanco-Luquin I, Torrea N, et al. Differential involvement of RASSF2 hypermethylation in breast cancer subtypes and their prognosis[J]. Oncotarget, 2015, 6(27):23944–23958.
[6] Gharanei S, Brini AT, Vaiyapuri S, et al. Rassf2 methylation is a strong prognostic marker in younger age patients with ewing sarcoma[J]. Epigenetics, 2013, 8(9):893–898. doi: 10.4161/epi.25617.
[7] Guerrero-Setas D, Pérez-Janices N, Blanco-Fernandez L, et al.RASSF2 hypermethylation is present and related to shorter survival in squamous cervical cancer[J]. Mod Pathol, 2013, 26(8):1111–1122. doi: 10.1038/modpathol.2013.32.
[8] Vos MD, Ellis CA, Elam C, et a1. RASSF2 is a novel K-Ras-specific effector and potential tumor suppressor[J]. J Biol Chcm, 2003,278(30):28045–28051.
[9] Zhang Z, Sun D, Van do N, et a1. Inactivation of RASSF2A by promoter methylation correlates with lymph node metastasis in nasopharyngeal carcinoma[J]. Int J Cancer, 2007, 120(1):32–38.
[10] Maruyama R, Akino K, Toyota M, et al. Cytoplasmic RASSF2A is a proapoptotic mediator whose expression is epigenetically silenced in gastric cancer[J]. Carcinogenesis, 2008, 29(7):1312–1318. doi:10.1093/carcin/bgn060.
[11] Zhao L, Cui Q, Lu Z, et a1. Aberrant methylation of RASSF2A in human pancreatic ductal adenocarcinoma and its relation to clinicopathologic features[J]. Pancreas, 2012, 41(2):206–211. doi:10.1097/MPA.0b013e318223d1a5.
[12] Malumbres M, Barbacid M. RAS oncogenes:the first 30 years[J].Nat Rev Cancer, 2003, 3(6):459–465. doi: 10.1038/nrc1097.
[13] Murata H, Tsuji S, Tsujii M, et al. Promoter hypermethylation silences cyclooxygenase-2 (Cox-2) and regulates growth of human hepatocellular carcinoma cells[J]. Lab Invest, 2004, 84(8):1050–1059.
[14] Schagdarsurengin U, Wilkens L, Steinemann D, et al. Frequent epigenetic inactivation of the RASSF1A gene in hepatocellular carcinoma[J]. Oncogene, 2003, 22(12):1866–1871.
[15] Jaenisch R, Bird A. Epigenetic regulation of gene expression: how the genome integrates intuinsic and environmental signals[J]. Nat Genet, 2003, 33(Suppl):245–254.
[16] 董金玲, 高萍, 刘顺爱, 等. HCV core/NS3/NS5A对内源性IFN-β表达影响及其调控机制初步研究[J]. 中国肝脏病杂志:电子版,2013, 5(1):1–5. doi:10.3969/j.issn.1674–7380.2013.01.001.Dong JL, Gao P, Liu SA, et al. Effects and regulation mechanism of HCV core/NS3/NS5A on expression of IFN-β in HepG2 cells[J].Chinese Journal of Liver Diseases:Electronic Version, 2013, 5(1):1–5. doi:10.3969/j.issn.1674–7380.2013.01.001.
[17] 何琼琼, 肖旭贤, 冯德云, 等.丙 型肝炎病毒NS3基因对人肝细胞生物学特性的影响[J]. 生物技术通报, 2007, (4):135–140.doi:10.3969/j.issn.1002–5464.2007.04.033.He QQ, Xiao XX, Feng DY, et al. Effect of Hepatitis c Virus NS3 Gene on Biological Charictarization of Human Hepatocyte[J].Biotechnology Bulletin, 2007, (4):135–140. doi:10.3969/j.issn.1002–5464.2007.04.033.
[18] Herman JG, Baylin SB. Gene silencing in canoer in association with promoter hypermcthylation[J]. N Engl J Med, 2003, 349(21):2042–2054.
[19] Sakai T, Toguchida J, Ohtani N, et a1. Allele-specific hypermethylation of the retinoblastoma tumor-suppressor gene[J].Am J Hum Genet, 1991, 48(5):880–888.
[20] Herman JG, Latif F, Weng Y, et a1. Silencing of the VHL tumor suppressor gene by DNA methylation in renal carcinoma[J]. Proe Natl Acad Sci U S A, 1994, 91(21):9700–9704.
[21] Herman JG, Merlo A, Mao L, et a1. Inactivation of the CDKN2/p16/MTS1 gene is frequently associated with aberrant DNA methylation in all common human cancers[J]. Cancer Res, 1995,55(20):4525–4530.
[22] Akino Km, Toyota M, Suzuki H, et a1. The Ras effector RASSF2 is a novel tumor-suppressor gene in human colorectal cancer[J].Gastroenterology, 2005, 129(1):156–169.
[23] Hesson LB, Wilson R, Morton D, et a1. CpG island promoter hypermethylation of a novel Ras-effector gene RASSF2A is an early event in colon carcinogenesis and correlates inversely with K-ras mutations[J]. Oncogene, 2005, 24(24):3987–3994.
[24] Park HW, Kang HC, Kim IJ, et a1. Correlation between hypermethylation of the RASSF2A promoter and K-ras/BRAF mutations in microsatellite-stable colorectal cancers[J]. Int J Cancer,2007, 120(1):7–12.
[25] Momparler RL. Epigenetic therapy of cancer with 5-aza-2'-deoxycytidine (decitabine)[J]. Semin Oncol, 2005, 32(5):443–451.
[26] Nguyen CT, Weisenberger DJ, Velicescu M. Histone H3-lysine 9 methylation is associated with aberrant gene silencing in cancer cells and is rapidly reversed by 5-aza-2'-deoxycytidine[J]. Cancer Res, 2002, 62(22):6456–6461.
[27] Vaissière T, Sawan C, Herceg Z. Epigenetic interplay between histone modifications and DNA methylation in gene silencing[J]. Mutat Res, 2008, 659(1/2):40–48. doi: 10.1016/j.mrrev.2008.02.004.
[28] Khokhlatchev A, Rabizadeh S, Xavier R, et a1. Identification of a novel Ras-regulated proapoptotic pathway[J]. Curr Biol, 2002,12(4):253–265.
[29] Barnoud T, Donninger H, Clark GJ. Ras Regulates Rb via NORE1A[J]. J Biol Chem, 2016, 291(6):3114–3123. doi: 10.1074/jbc.M115.697557.
[30] Donninger H, Calvisi DF, Barnoud T, et a1. NORE1A is a Ras senescence effector that controls the apoptotic/senescent balance of p53 via HIPK2[J]. J Cell Biol, 2015, 208(6):777–789. doi: 10.1083/jcb.201408087.
[31] Schmidt ML, Donninger H, Clark GJ. Ras regulates SCF(β-TrCP)protein activity and specificity via its effector protein NORE1A[J].J Biol Chem, 2014, 289(45):31102–31110. doi: 10.1074/jbc.M114.594283.
[32] Clark J, FreemanJ, Donninger H. Loss of RASSF2 Enhances Tumorigencity of Lung Cancer Cells and Confers Resistance to Chemotherapy [J]. Mol Biol Int, 2012, 2012:705948. doi:10.1155/2012/705948.
