基于RNA-seq数据大规模开发中华蜜蜂幼虫的SSR分子标记
2017-03-16熊翠玲付中民王鸿权侯志贤童新宇李汶东郑燕珍陈大福
熊翠玲,张 璐,付中民,王鸿权,侯志贤,童新宇,李汶东,郑燕珍,陈大福,郭 睿
(福建农林大学蜂学学院,福州350002)
基于RNA-seq数据大规模开发中华蜜蜂幼虫的SSR分子标记
熊翠玲*,张 璐*,付中民,王鸿权,侯志贤,童新宇,李汶东,郑燕珍,陈大福**,郭 睿**
(福建农林大学蜂学学院,福州350002)
中华蜜蜂是东方蜜蜂的一个亚种,也是我国的特有蜂种。本研究基于已获得的中华蜜蜂幼虫肠道转录组数据预测SSR分子标记,并进行SSR位点的信息分析和SSR引物的发掘。利用MISA软件对幼虫肠道转录组中数据组装得到的43557条 unigenes进行搜索,共预测出13448个SSR位点,它们分布于7763条 unigene中,其中最主要的重复类型为二核苷酸重复(58.03%),其次为三核苷酸重复(28.23%)和四核苷酸重复(9.72%)。二核苷酸重复中的基序主要是 AT/AT(占总量的30.4%)。对于所有的SSR位点,利用Primer Premier 5软件成功设计出21627 对引物,随机选取 48 对引物对5个不同来源的中华蜜蜂幼虫肠道样品进行SSR位点扩增,共有15对成功扩增出符合预期的目的片段。研究结果表明利用转录组数据大规模开发中华蜜蜂幼虫的SSR引物是可行的,本研究开发出的SSR引物为研究中华蜜蜂分子遗传学奠定了基础。
RNA-seq;中华蜜蜂;幼虫肠道;SSR分子标记
简单重复序列 (simple sequence repeat, SSR)也称微卫星 (microsatellite),由1-6 个核苷酸碱基为重复单元 (motif)组成。SSR分子标记具有多态性高、重复性好、实验操作容易及呈共显性遗传等特点 (Jarneetal., 1996),因此广泛应用于动植物及微生物等学科领域。然而,SSR分子标记的特异性在一定程度上限制了其引物的通用性 (Glennetal., 2005)。对于基因序列未知的物种,SSR标记的开发较为困难。SSR分子标记的传统开发方法以基因文库构建法 (包括SSR富集文库)为主 (Hamiltonetal., 1999; Zaneetal., 2002),但费时费力且效率低下。利用公共数据库 (如NCBI、EMBL和DDBJ)中的共享基因序列挖掘SSR分子标记也是一种较好的方法,但对于非模式生物或新物种而言,有限的基因序列资源严重阻碍SSR分子标记的开发。第二代高通量测序技术的迅速发展和应用为规模化遗传变异检测 (O’Neilletal., 2013)和分子标记位点开发 (Daveyetal., 2011; Yuetal., 2011; Pandeyetal., 2013)带来了宝贵机遇。然而,基于高通量测序数据挖掘SSR分子标记仍面临着大数据对生物信息学方法和技术的挑战。东方蜜蜂Apiscerana(简称东蜂)分布广泛,但各地不同的蜜源和气候条件使各地的东蜂在形态和种性上存在诸多差异。中华蜜蜂Apisceranacerana(简称中蜂)起源于古喜马拉雅山系的热带、亚热带雨林,已适应我国各种生态地理条件,广泛分布在长江流域的亚热带常绿阔叶林区和稻茶区 (段成鼎, 2003)。目前,中蜂己经分化为不同的地理系,并在长期进化中形成其独特的适应性 (杨冠煌, 1983)。较之西方蜜蜂Apismellifera(简称西蜂),中蜂具有抗螨害、耐寒、善利用零星蜜粉源等优点。
西蜂基因组早在2006年就已完成测序并公布 (Honeybee Genome Sequencing Consortium, 2006),为蜜蜂行为、遗传进化和基因功能研究提供了重要的参考信息,也有力推动了其SSR分子标记的开发和应用。2015年,韩国的研究人员公布了东方蜜蜂的基因组(Park D, 2015),但当时并没有公布基因位置及功能注释信息。目前,中蜂可利用的SSR分子标记极少,有限的基因序列资源极大地限制了中蜂微卫星标记的开发,中蜂种群遗传学研究受到严重制约。此前,梁勤等(2009)利用6对微卫星DNA标记对福建省4个中蜂群体进行遗传多样性分析,评估群体内的遗传变异和群体间的遗传分化。徐新建(2013)等应用10个微卫星DNA标记对海南岛11个地点和大陆2个地点中蜂分析表明,海南中蜂多样性丰富,岛屿和邻近大陆种群发生了明显的遗传分化。本课题组利用RNA-seq技术对中蜂幼虫肠道进行转录组测序,成功组装了中蜂幼虫肠道的参考转录组并对其进行了功能及代谢通路注释 (未发表数据),可为中蜂的微卫星标记开发提供重要的参考信息。
本研究基于已获得的中蜂幼虫肠道RNA-seq数据,利用MISA软件搜索所有unigenes中的SSR位点信息,并通过Primer Premier 5软件设计特异性引物,进而对5个不同来源的中蜂幼虫肠道样品的SSR位点进行PCR扩增,研究结果将为中蜂分子遗传学的相关研究奠定基础,也为其它非模式生物或新物种的SSR分子标记挖掘提供方法借鉴。
1 材料与方法
1.1 转录组数据
中蜂幼虫肠道的转录组测序委托广州基迪奥生物科技有限公司进行,测序平台为Illumina Hiseq 2500,采用双端测序 (PE125),RNA-seq共获得43577 条 unigenes。本研究测得的转录组数据已上传美国国家生物技术信息中心 (NCBI)SRA数据库,SRA号:SRA456721。
1.2 供试蜜蜂幼虫
本研究用于验证SSR引物的中蜂幼虫取自四川、辽宁、福建、江西和广东。每个来源的中蜂幼虫分别剖取肠道,液氮速冻后转移至-80℃保存备用。使用组织DNA提取试剂盒 (TIANamp Genomic,中国),提取各肠道样品的总DNA,-20℃保存备用。
1.3 SSR 鉴定
利用软件MISA (http://pgrc.ipk-gatersleben. de/misa/)搜索unigenes的SSR分子标记,标准如下:二核苷酸重复≥6次,三核苷酸重复≥5次,四核苷酸重复≥5次,五核苷酸重复≥5次和六核苷酸重复≥5次。
1.4 SSR 引物设计与验证
根据MISA的输出结果,利用Primer Premier 5(PREMIER Biosofe Int., Palo Alto, CA)对每一个含有16 bp碱基重复的SSR设计特异性引物。设置目的片段必须包含SSR起始-3 bp,终止+6 bp,扩增片段大小为100-300 bp。引物的长度设置为18-25 bp,最适长度为 22 bp,引物最大允许有一个不能识别的碱基。设置引物的退火温度 (Tm)为55℃-65℃,最适 Tm为58℃,上下游引物间的Tm差异最大允许3℃,引物末端稳定性最大为250。引物由上海生工生物工程有限公司进行合成。为验证设计引物的稳定扩增,随机挑选 48对引物进行验证。PCR 反应体系为20 μL:其中中蜂幼虫肠道总DNA模板 1 μL,Mixture 10 μL,上下游引物(10 μmol/L)各 1 μL,无菌水补至20 μL。反应程序如下:94℃预变性5 min;94℃变性50 s,55℃退火30 s,72℃延伸30 s,共33个循环;72℃延伸10 min。PCR 产物经 4% 琼脂糖凝胶电泳检测。
2 结果与分析
2.1 中华蜜蜂幼虫肠道转录组中 SSR 位点的分布特点
利用MISA软件对中蜂幼虫肠道转录组中43577 条 unigenes 进行搜索,共预测出13448个SSR位点,它们分布于7763 条 unigenes 中,出现频率 (含有SSR的 unigene 数量与总 unigene 数量之比)为17.82%。这些SSR基序包含1-5 bp 的串联重复序列。在中华蜜蜂幼虫肠道转录组数据中,SSR基序的重复类型中总数最多的是二核苷酸重复,达SSR总数的58.03%;其次是三核苷酸重复和四核苷酸重复,分别占SSR 总数的28.23%和9.72%;五核苷酸和六核苷酸重复的数量很少 (2.53%和 1.49%)(表 1)。中蜂幼虫肠道转录组 SSR 重复单元的重复次数分布在4-24次。二核苷酸重复次数分布在6-24次,三核苷酸重复次数分布在5-24次,四核苷酸重复次数分布在4-24次,五核苷酸重复次数分布在4-12次,六核苷酸重复次数分布在4-10次,其中,重复次数最多的为6次,占24.22%。对SSR 基元出现频率进行统计,结果显示二核苷酸重复中主要是 AT/AT 基元,占总量的30.04%,AG/CT 基元其次,占 22%,其它基元百分比超过2% 的只有AC/GT、AAC/GTT、AAG/CTT、AAT/ATT、AGC/CTG、AGG/CCT和AAAG/CTTT,其比例分别为4.7%、2.9%、5.2%、7.2%、2.4%、2.9%和3% (图 1)。三核苷酸中ACT/AGT基序最少,占SSR总数的1.3% (图1)。
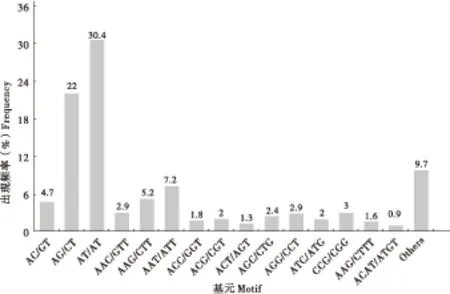
图1 基于基序类型中 SSRs 在中华蜜蜂幼虫肠道转录组中的出现频率Fig.1 Frequency distribution of SSRs based on motif types in Apis cerana cerana larval gut transcriptome

重复类型Repeattype重复数Numberofrepeats4567891011121314>=15合计Total百分比(%)Percentage二核苷酸Dinucleotide--232016091203874595335122149863478045803三核苷酸Triucleotide-15537975791681762602249443811137972823四核苷酸Tetranucleotide6673259412046111697327430972五核苷酸Pentanucleotide2165344382541--326253六核苷酸Hexaucleotide1334723625-1--2201149总计Total1016197832572314143110658813701806113875713448-百分比(%)Pertancage7561471242217211064792655275134045103562--
2.2 中华蜜蜂幼虫肠道 SSRs引物设计
利用Primer Premier 5软件进行SSR引物的批量设计,针对7209个 unigenes共设计出21627对SSR引物。随机挑选48对引物对国内5个不同来源的中蜂幼虫样品进行扩增,PCR产物的琼脂糖凝胶电泳结果显示,共有15对引物扩增成功 (表2),并且扩增片段与预期片段大小基本一致 (图2),有效扩增的比例达31.25%。上述结果表明基于RNA-seq数据开发SSR引物的方法可行,这些SSR位点多态性的高低有待于进一步的试验评估。

图2 不同来源中华蜜蜂幼虫肠道样品 15 个SSR位点的PCR扩增产物电泳Fig.2 Agarose gel electrophoresis for PCR amplification of 15 SSR loci in Apis cerana cerana larval gut samples from different regions in China注:A-E,不同SSR引物在各中蜂幼虫肠道样品中SSR位点扩增产物电泳;S,四川来源的中蜂幼虫肠道样品; L,辽宁来源的中蜂幼虫肠道样品; F,福建来源的中蜂幼虫肠道样品; J,江西来源的中蜂幼虫肠道样品; G,广东来源的中蜂幼虫肠道样品; M, DNA marker;SSR1-15分别代表15对 SSR 引物,引物序列见表2。Note:A-E, Agose gel electrophoresis for PCR amplification of SSR loci in each A. c. cerana larval gut sample using different SSR primers;S, A. c. cerana larval gut sample from Sichuan province; L, A. c. cerana larval gut sample from Liaoning province; F, A. c. cerana larval gut sample from Fujian province; J, A. c. cerana larval gut sample from Jiangxi province; G, A. c. cerana larval gut sample from Guangdong Province; M, DNA marker; SSR1-15 represent 15 pairs of SSR primer and the sequences are shown in Table 2.
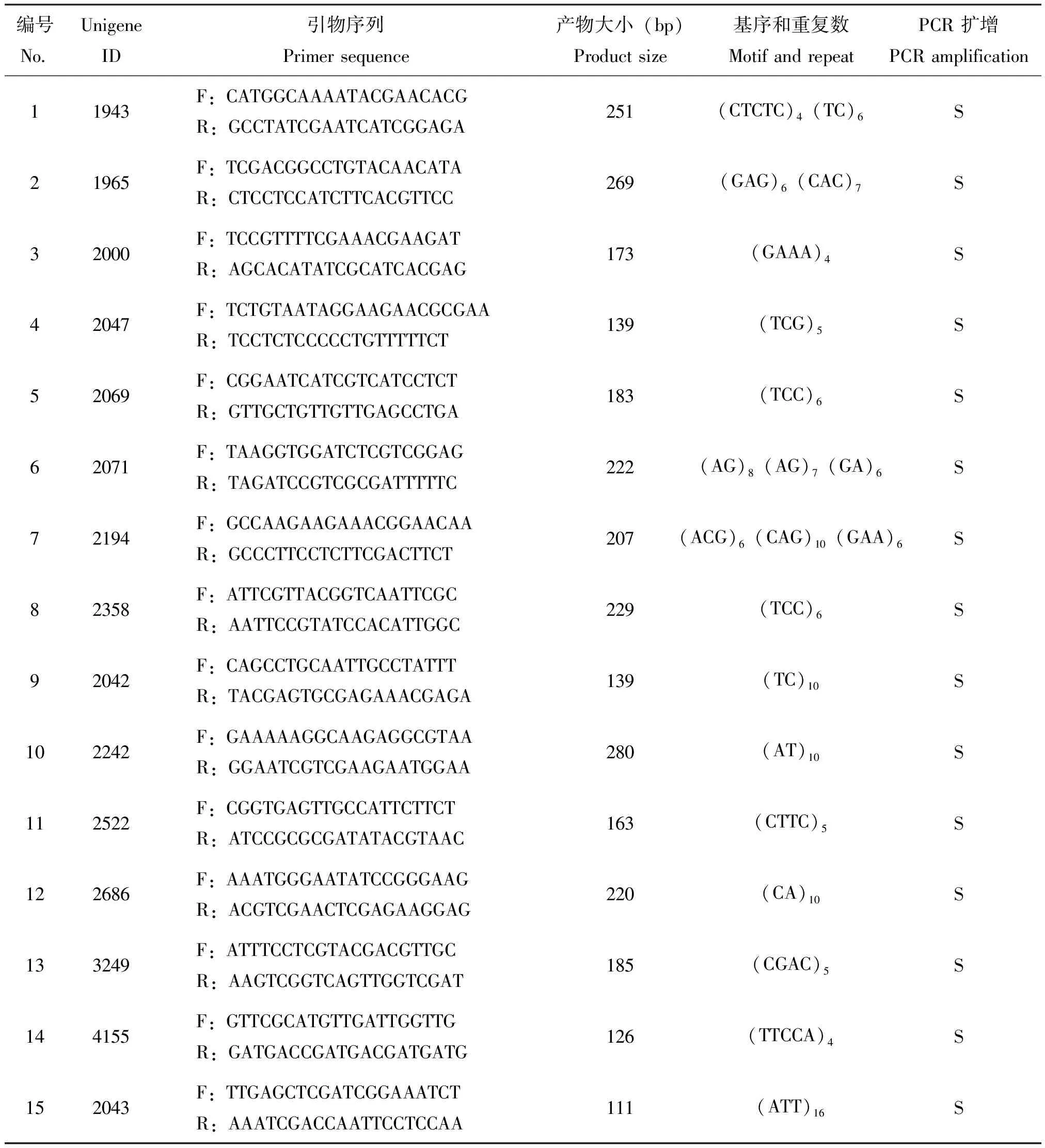
编号NoUnigeneID引物序列Primersequence产物大小(bp)Productsize基序和重复数MotifandrepeatPCR扩增PCRamplification11943F:CATGGCAAAATACGAACACGR:GCCTATCGAATCATCGGAGA251(CTCTC)4(TC)6S21965F:TCGACGGCCTGTACAACATAR:CTCCTCCATCTTCACGTTCC269(GAG)6(CAC)7S32000F:TCCGTTTTCGAAACGAAGATR:AGCACATATCGCATCACGAG173(GAAA)4S42047F:TCTGTAATAGGAAGAACGCGAAR:TCCTCTCCCCCTGTTTTTCT139(TCG)5S52069F:CGGAATCATCGTCATCCTCTR:GTTGCTGTTGTTGAGCCTGA183(TCC)6S62071F:TAAGGTGGATCTCGTCGGAGR:TAGATCCGTCGCGATTTTTC222(AG)8(AG)7(GA)6S72194F:GCCAAGAAGAAACGGAACAAR:GCCCTTCCTCTTCGACTTCT207(ACG)6(CAG)10(GAA)6S82358F:ATTCGTTACGGTCAATTCGCR:AATTCCGTATCCACATTGGC229(TCC)6S92042F:CAGCCTGCAATTGCCTATTTR:TACGAGTGCGAGAAACGAGA139(TC)10S102242F:GAAAAAGGCAAGAGGCGTAAR:GGAATCGTCGAAGAATGGAA280(AT)10S112522F:CGGTGAGTTGCCATTCTTCTR:ATCCGCGCGATATACGTAAC163(CTTC)5S122686F:AAATGGGAATATCCGGGAAGR:ACGTCGAACTCGAGAAGGAG220(CA)10S133249F:ATTTCCTCGTACGACGTTGCR:AAGTCGGTCAGTTGGTCGAT185(CGAC)5S144155F:GTTCGCATGTTGATTGGTTGR:GATGACCGATGACGATGATG126(TTCCA)4S152043F:TTGAGCTCGATCGGAAATCTR:AAATCGACCAATTCCTCCAA111(ATT)16S
3 结论与讨论
此前,本课题组利用RNA-seq技术对中蜂幼虫肠道进行深度测序,成功组装并注释其参考转录组。本研究利用生物信息学方法从中蜂幼虫肠道的转录组数据中预测出多达13448个SSR位点,这些SSR位点种类丰富,以二核苷酸重复类型为主,其次为三核苷酸重复。其它昆虫如黄粉虫Tenebriomolitor(朱家颖, 2013)、扶桑绵粉蚧Phenacoccussolenopsis(罗梅, 2014)和沙葱萤叶甲Galerucadaurica(张鹏飞, 2016)等的相关研究结果显示单核苷酸重复是最主要的重复类型,其次为三核苷酸重复;沟眶象Eucryptorrhynchuschinensis(武政梅等, 2016)、二点委夜蛾Athetislepigone(Lietal., 2013)及云南切梢小蠹Tomicusyunnanensis(袁远等, 2014)等昆虫的SSR以三碱基重复为主。这与本研究的结果略有不同,以二核苷酸重复类型为主可能是中蜂SSR分子标记的特征。在表达序列标签(ESTs)中,多数情况下单核苷酸重复最多,其次为三核苷酸重复。三核苷酸重复是昆虫EST-SSR 中的核心重复类型,因为三核苷酸核心基元极少产生编码框滑动突变现象,其在编码区较其它重复基元类型更加稳定 (Wangetal., 2012)。本研究中,中蜂幼虫的SSR出现频率为17.82%,高于沟眶象(武政梅等, 2016)、黑翅土白蚁Odontotermesformosaanus(9.98%)(Huangetal., 2012)及烟粉虱Bemisiatabaci(5.07%)(Xieetal., 2012)等昆虫中SSR的出现频率。推测较高的SSR出现频率是中蜂幼虫的特征之一。在动植物的转录组或基因组中,GC/CG是二碱基重复的SSR中的稀有重复基元。本研究发现GC/CG核心基元数量仅为1个,与前人研究结果类似 (张琳琳, 2008;张鹏飞, 2016)。
Tkahashi等(2009)曾通过传统方法开发出12个东蜂微卫星标记,其中8个具有高多态性。郭慧萍等(2016)利用8个微卫星标记对秦巴山区17个样点共979个蜂群的中蜂遗传多态性进行评估,并以长白山中蜂和阿坝中蜂作为外群进行种群遗传分化分析。总体而言,目前已有的中蜂SSR分子标记数量十分有限,严重制约中蜂遗传进化与种群遗传研究。本研究中,设计合成的特异性SSR引物多达21627对,为了验证上述SSR引物,随机挑选其中的48对对国内5个不同来源的中蜂幼虫样品的SSR位点进行PCR扩增,结果显示有15对引物可有效扩增出SSR位点,有效扩增率达31.25%,说明基于RNA-seq数据预测出的SSR位点只有部分真实存在,然而,通过此法开发出的可用于多态性分析的SSR位点数目仍非常可观。本研究中,SSR位点扩增产物的电泳采用的是琼脂糖凝胶法,此法较聚丙烯酰胺凝胶电泳 (PAGE)法分辨率低,上述SSR位点多态性的确定有待于进一步研究。
综而述之,利用以RNA-seq为代表的二代测序技术挖掘SSR分子标记具有通量高、速度快和效率高的优点,解决了传统开发方法的瓶颈问题,使微卫星标记的大规模开发成为现实。中蜂是非模式生物,其基因组信息刚刚公布 (Park, 2015),本研究说明基于高通量测序数据开发非模式生物的SSR分子标记是一种快速高效的途径。目前,以单分子测序技术为代表的三代测序技术逐渐兴起,SSR分子标记开发及功能基因定位将更为方便快捷。
References)
Fries I, Wei H, Shi W,etal. Grooming behavior and damaged mites(Varroajacobsoni)inApisceranaceranaandApismelliferaligustica.[J].Apidologie, 1996, 27 (1): 3-11.
Gong YF,Zhang QK . Classification and Evolution ofApis[M].Fuzhou: Fujian Science and Technology Press, 2000, 21-26. [龚一飞, 张其康. 蜜蜂分类与进化 [M]. 福州:福建科学技术出版社, 2000, 21-26]
Guo HP, Zhou SJ, Zhu XJ,etal. Population genetic analysis ofApisceranaceranafrom the Qing-Daba mountain areas base on microsatellite DNA[J].ActaEntomologicaSinica, 2016, 59 (3): 337-345. [郭慧萍, 周姝婧, 朱翔杰, 等. 秦巴山区中华蜜蜂种群微卫星DNA遗传分析 [J]. 昆虫学报, 2016, 59 (3): 337-345]
Guo R, Wang S, Xue R,etal. The gene expression profile of resistant and susceptibleBombyxMoristrains revealsCypovirus-associated variations in host gene transcript levels [J].Appl.MicrobiolBiotechnol., 2015, 99 (12): 5175-87.
Honeybee Genome Sequencing Consortium. Insights into social insects from the genome of the honeybeeApismellifera[J].Nature, 2006, 443: 931-949.
Huang Q, Sun P, Zhou X,etal. Characterization of head transcriptome and analysis of gene expression involved in caste differentiation and aggression inOdontotermesformosanus( Shiraki)[J].PLoSONE, 2012, 7 (11): e50383.
Lin Z, Page P, Li L,etal. Go east for better honey bee health:Apisceranais faster at hygienic behavior thanA.mellifera[J].PLoSONE, 2016, 11 (9): e0162647.
Li HL. Construction of cDNA Libraries from Head of Bees, Cloning, Expression and Subcellular Localization of the Antenna Specific Protein Gene in the Chinese Honeybee,Apisceranacerana[D]. Zhejiang University, 2007. [李红亮. 中华蜜蜂头部ests文库构建和主要触角特异蛋白基因克隆、定位及其表达鉴定 [D]. 2007, 浙江大学]
Liang Q, Zhang LQ. Analysis of genetic diversity of fourApisceranaceranapopulations in Fujian province with microsatellite markers [J].JournalofFujianAgricultureandForestryUniversity(Natural Science Edition), 2009, 38 (4): 388-392. [梁勤, 张立卿. 利用微卫星标记分析福建4个中华蜜蜂群体的遗传多样性 [J]. 福建农林大学学报(自然科学版),2009, 38 (4):388-392]
Luo M, Zhang H, Bin SY,etal. High-throughput discovery of SSR genetic markers in the mealybug,Phenacoccussolenopsis(Hemiptera: Pseudococcidae), from its transcriptome database [J].ActaEntomologicaSinica, 2014, 57 (4): 395-400. [罗梅, 张鹤, 宾淑英, 等.基于转录组数据高通量发掘扶桑绵粉蚧微卫星引物 [J]. 昆虫学报, 2014, 57(4):395-400]
Novaes E, Drost DR, Farmerie WG,etal. High-throughput gene and SNP discovery inEucalyptusgrandis, an uncharacterized genome [J].BMCGenomics, 2008, 9 (1): 312.
Park D, Jung JW, Choi BS,etal. Uncovering the novel characteristics of Asian honey bee,Apiscerana, by whole genome sequencing [J].BMCGenomics, 2015, 16 (1): 1-16.
Peng YS. Geographical races ofApisceranaFabricius in China and their distribution. Review of Recent Chinese Publications and a Preliminary Statistical Analysis [J].Apidologie, 1989, 20 (1): 9-20.
Peng YS, Fang YZ, Xu SY,etal. The resistance mechanism of the Asian honey bee,ApisceranaFabr, to an ectoparasitic mite,VarroajacobsoniOudemans[J].ScienceOpenResearch, 1987, 49 (1).
Takahashi JI, Shimizu S, Koyama S,etal. Variable microsatellite loci isolated from the Asian honeybee,Apiscerana(Hymenotera: Apidae)[J].MolecularEcologyResources, 2009, 9 (3): 819.
Wang Q, Sun LX, Xiao PX,etal. Study on technology for indoor artificial feeding ofApisceranaceranalarvae [J].ShangdongAgricultureScience, 2009 (11): 113-116. [王倩, 孙亮先, 肖培新, 等. 室内人工培育中华蜜蜂幼虫技术研究 [J]. 山东农业科学, 2009 (11): 113-116]
Werner R. Co-adaptation ofApisceranaFabr. andVarroajacobsoniOud [J].Apidologie, 1999. 30 (2-3): 97-110.
Wei DD, Shi JX, Zhang XX,etal. Analysis of microsatellite loci fromBactroceradorsalisbased on transcriptome dataset. [J].ChineseJournalofAppliedEcology, 2014, 25 (6): 1799-1805. [魏丹丹, 石俊霞, 张夏瑄, 等. 基于转录组数据的桔小实蝇微卫星位点信息分析 [J]. 应用生态学报, 2014, 25 (6): 1799-1805]
Wu ZM, Gao P, Wen JB. Characteristic analysis of microsatellite inEucryptorrhynchuschinensistranscriptome [J].JournalofEnvironmentalEntomology, 2016, 38 (5): 979-983. [武政梅, 高朋, 温俊宝. 沟眶象转录组微卫星特征分析 [J]. 环境昆虫学报, 2013, 56 (5): 554-560]
Xie W, Meng QS, Wu QJ,etal. Pyrosequencing theBemisiatabacitranscriptome reveals a highly diverse bacterial community and a robust system for insecticide resistance[J].PLoSONE, 2012, 7 (4): e35181.
Xu XJ, Zhou SJ, Zhu XJ,etal. Microsatellite DNA analysis of genetic diversity ofApisceranaceranain Hainan island, southern China [J].ActaEntomologicaSinica, 2013, 56 (5): 554-560. [徐新建, 周姝婧, 朱翔杰, 等. 海南岛中华蜜蜂遗传多样性的微卫星DNA分析 [J]. 昆虫学报, 2013, 56 (5): 554-560]
Yuan Y, Zhang LF, Wu GX,etal. High-throughput discovery microsatellites inTomicusyunnanenss(Coleoptera: Scolytonae)[J].JournalofEnvironmentalEntomology, 2014, 36 (2): 166-170. [袁远, 张丽芳, 吴国星, 等. 云南切梢小蠹微卫星的高通量发掘[J]. 环境昆虫学报, 2014, 36 (2): 166-170]
Yu YL, Zhou SJ, Xu XJ,etal. Analysis on genetic diversity ofApisceranaceranain Changbai mountains [J].JournalofFujianAgricultureandForestryUniversity(Natural Science Edition), 2013, 42 (6): 643-647. [于瀛龙, 周姝婧, 徐新建, 等. 长白山中华蜜蜂(Apisceranacerana)遗传多样性分析 [J]. 福建农林大学学报(自然科学版), 2013, 42 (6): 643-647]
Zhou BF, Zhu XJ. The protection ofApisceranacerana. In:China Apicultural Conference on Sustainable Development & Apicultural Technology and Ecology [C]. 2008. [周冰峰, 朱翔杰. 论中华蜜蜂种质资源的保护.见:中国蜂业科技可持续发展学术研讨会暨蜂业科技与生态研讨会[C]. 2008]
Zhu JY, Wu GX, Yang B. High-throughput discovery of SSR genetic markers in the yellow mealworm beetle,Tenebriomolitor(Coleoptera: Tenebrionidae), from its transcriptome database [J].ActaEntomologicaSinica, 2013, 56 (7): 724-728. [朱家颖, 吴国星, 杨斌. 基于转录组数据高通量发掘黄粉甲微卫星引物 [J]. 昆虫学报, 2013, 56 (7): 724-728]
Zhang LL, Wei ZM, Lian ZM,etal. Abundance of microsatellites in the entire genome and EST ofTriboliumcastaneum[J].ChineseBulletinofEntomology, 2008, 45 (1): 38-42. [张琳琳, 魏朝明, 廉振民, 等. 赤拟谷盗全基因组和EST 中微卫星的丰度 [J]. 昆虫知识, 2008, 45 (1): 38-42]
Zhang PF,Zhou XR,Pang BP,etal.High-throughput discovery of microsatellite markers inGalerucadaurica(Coleoptera: Chrysomelidae)from a transcriptome database [J].ChineseJournalofAppliedEntomology,2016, 53 (5):1058-1064. [张鹏飞, 周晓榕, 庞保平, 等. 基于转录组数据高通量发掘沙葱萤叶甲微卫星引物 [J]. 应用昆虫学报, 2016, 53 (5): 10581064]
Large-scale development of SSR primers forApisceranaceranalarvae based on its RNA-seq datasets
XIONG Cui-Ling*,ZHANG Lu*,FU Zhong-Min,WANG Hong-Quan,HOU Zhi-Xian,TONG Xin-Yu,LI Wen-Dong,ZHENG Yan-Zhen,CHEN Da-Fu**,GUO Rui**
(College of Bee Science, Fujian Agriculture and Forestry University, Fuzhou 350002, China)
Apisceranaceranais a subspecie ofApiscerana, and it’s also an unique honeybee specie in China. In the present study, SSR molecular markers forA.c.ceranawere predicted based on the transcriptome datasets ofA.c.ceranalarval gut previously obtained. Further, we performed infromation analysis of SSR loci and development of SSR primers. First, MISA software was used to search 43557 unigenes assembled from RNA-seq data of eastern honeybee larval gut, and a total of 13448 SSR loci were found, which distributed in 7763 unigenes. Among these SSR loci, the main repeat types were di-nucleotide repeats (58.03%), followed by tri-nucleotide repeats (28.23%)and finally tetra-nucleotide repeats (9.72%). The AT/AT motif was the most abundant (30.4%)in di-nucleotide repeats. In total, 21627 pairs of primers were designed from all SSRs. 48 pairs of SSR primers were randomly selected and used to amplify the SSR loci inA.c.ceranalarva gut samples from five different regions in China, and 15 SSR primer pairs could amplify the target fragments with expected size. Our findings suggested that large-scale development ofA.c.ceranalarvae’s SSR primers on basis of transcriptome data is feasible, additionally, the 15 SSRs primers developed here can lay a foundation for study on molecular genetics ofA.c.cerana.
RNA-seq;Apisceranacerana; larval gut; SSR
现代农业产业技术体系建设专项资金(CARS-45-KXJ7);福建农林大学科技发展资金(KF2015123);国家自然科学基金(30800806)
Received:2016-12-08;接收日期Accepted:2017-01-16
Q963;S891
A
1674-0858(2017)01-068-07
熊翠玲,张璐,付中民,等.基于RNA-seq数据大规模开发中华蜜蜂幼虫的SSR分子标记[J].环境昆虫学报,2017,39(1):68-74.
*共同第一作者简介:熊翠玲,女,1977年生,福建永定人,实验师,研究方向为蜜蜂保护学,E-mail: pandacl@sina.com;张璐,女,1996年生,河南濮阳人,本科生,研究方向为蜂学,E-mail: m17805949180@163.com
**通讯作者Authors for correspondence, E-mail: fafu_ruiguo@126.com; dfchen826@163.com
