小麦TILLING突变体检测中基因组DNA提取方法的优化
2017-03-01徐继法赵吉强FUZeyuJAMESONPaula宋建成
徐 艳,徐继法,陈 磊,赵吉强,FU Zeyu,JAMESON Paula E ,宋建成
(1.烟台大学生命科学学院,山东烟台 264005; 2.School of Biological Sciences,University of Canterbury,Christchurch,New Zealand)
小麦TILLING突变体检测中基因组DNA提取方法的优化
徐 艳1,徐继法1,陈 磊1,赵吉强1,FU Zeyu2,JAMESON Paula E2,宋建成1
(1.烟台大学生命科学学院,山东烟台 264005; 2.School of Biological Sciences,University of Canterbury,Christchurch,New Zealand)
为了优化小麦高通量TILLING突变体扫描所需基因组DNA提取的方法,对利用SDS法、改良SDS法、CTAB法、改良CTAB法、AxyPrep DNA提取试剂盒、Qiagen DNeasy Plant Mini Kit试剂盒以及再生Qiagen硅胶柱方法提取的小麦基因组DNA进行了系统比较。结果表明,尽管7种方法所提取DNA的纯度及琼脂糖凝胶电泳检测结果均良好,但再生Qiagen硅胶柱方法、CTAB法和改良SDS法的DNA浓度显著高于其他方法。根据小麦细胞分裂素氧化酶基因1( TaCKX1)的DNA序列设计引物,以新鲜DNA样品用于PCR扩增大于1 000 bp的片段时,后4种方法扩增效果优于前3种方法;DNA在-20 ℃下保存100 d后重新进行PCR扩增时,则只有Qiagen试剂盒和再生Qiagen硅胶柱2种方法提取的DNA能扩增出大于1 000 bp的片段。采用本实验室与新西兰坎特伯雷大学合作建立的再生硅胶柱与自配提取液提取小麦基因组DNA,既保证了DNA的高质量,又降低了使用试剂盒的成本,为小麦TILLING库的构建及其他相关试验提供了一种经济有效的DNA提取方法。
小麦;TILLING;基因组DNA;提取方法
小麦是世界各地广泛种植的主要粮食作物之一。其全基因组大小高达17 000 Mb,约是人类基因组的5倍多,水稻基因组的36倍,而且80%的基因组序列为冗余重复序列[1]。同时,其异源六倍体的特性决定了性状遗传的复杂性,每个基因常含有三个拷贝。因此,利用转基因技术进行小麦基因功能研究和农艺性状的遗传改良具有较大难度[2]。定向诱导基因组局部突变 (targeting induced local lesions IN genomes,TILLING)技术是20世纪90年代末期由美国Fred Hutchinson癌症研究中心Steven Henikoff团队和华盛顿大学Luca Comai团队发展起来的一种反向遗传学研究方法[3-7]。该技术结合了化学诱变、PCR技术和高通量定向筛选的方法,已被广泛应用于遗传学研究,是功能基因组研究中用于发现遗传多样性和大规模筛选基因突变的有效手段[4-5]。目前,该技术已成功地应用于小麦的基因组研究和育种实践[8-11]。提取数千个突变体植株的高质量基因组DNA是TILLING技术中的基础环节,由于突变群体DNA库建成后需要长期保存,以便随时满足不同研究人员及不同研究项目对目的基因突变进行扫描检测的需要。因此,所提取DNA的质量将直接影响后续PCR等步骤的结果乃至TILLING群体突变体筛选鉴定成败[12-14]。
目前,常用的植物基因组DNA提取方法,如CTAB(十六烷基三甲基溴化铵)法、SDS(十二烷基磺酸钠)法、PVP(聚乙烯吡咯烷酮)法等,不仅提取效率和DNA的质量存在较大差异[15-21],而且我们在进行小麦TIILING群体突变体筛选过程中发现,这些方法所提取的许多DNA样品经一段时间保存后,PCR扩增效果发生明显衰减,甚至经常导致扩增完全失败,严重影响了TILLING突变体的扫描效果和效率。而采用国内外公认效果较好的Qiagen等试剂盒,尽管保证了所提取DNA的高质量,但往往因DNA得率较低和提取成本太高等缺陷,难以满足TIILING突变体筛选对大量DNA样品的需要。本研究对采用CTAB法及其改良法、SDS法及其改良法、两种DNA提取试剂盒以及新建立的提取方法所提取的小麦叶片基因组DNA进行系统比较,旨在获得快速、简便、低成本提取小麦基因组DNA的方法,为TILLING技术应用过程中大量DNA样品的提取提供可靠保障。
1 材料与方法
1.1 植物材料
DNA提取材料为实验室水培至展开叶长10 cm左右的野生型济麦22幼苗,选取新鲜叶片剪成1 cm后混匀。
1.2 试验试剂
Qiagen DNA提取试剂盒购自德国Qiagen公司。AxyPrep基因组DNA小量试剂盒购自康宁生命科学有限公司。其他试验用试剂均为分析纯。1×TE buffer、Binding buffer、Washing buffer Ⅰ、Washing buffer Ⅱ和Elution buffer的配制参照Fu等[22]的方法。
1.3 基因组DNA的提取方法
本研究共采用了7种基因组DNA提取方法,每种方法重复3次。
1.3.1 SDS法
取新鲜小麦叶片,在液氮中研磨成粉末,称取0.05 g于1.5 mL离心管中;加入500 μL SDS提取液,混匀,室温静置10 min;65 ℃水浴30 min,间或混匀;冰浴5 min,加入20 μL 2% β-巯基乙醇,混匀后静置5 min,室温12 000 r·min-1下离心5 min;取450 μL上清于新的1.5 mL离心管中,加入500 μL氯仿-异戊醇(24∶1),混匀后静置5 min,室温12 000 r·min-1下离心5 min;取400 μL上清于新的1.5 mL离心管(如果杂质含量仍然很高,可以重复该步1~2次),加入等体积-20 ℃预冷的异丙醇,混匀,于-20 ℃冰箱,静置30 min;4 ℃ 12 000 r·min-1下离心10 min,弃上清,用70%乙醇洗沉淀2次,凉风吹干或37 ℃烘干;加入50 μL的1×TE buffer溶解沉淀。
1.3.2 改良SDS法
对上述SDS法进行两方面的改进:(1)提取缓冲液中加入2% PVP和20 μL 2% β-巯基乙醇。(2)水浴后加入500 μL氯仿-异戊醇(24∶1),混匀静置,离心;取450 μL上清于新的1.5 mL离心管中,加入等体积-20 ℃预冷的异丙醇,混匀。后面步骤同1.3.1。
1.3.3 CTAB法
取新鲜小麦叶片,在液氮中研磨成粉末,称取0.05 g于1.5 mL离心管中;加入500 μL 60 ℃预热的CTAB缓冲液和10 μL β-巯基乙醇,混匀;于60 ℃水浴30 min,间或混匀;冰浴15 min,加入500 μL氯仿-异戊醇(24∶1),混匀后冰浴15 min,间或混匀,室温10 000 r·min-1下离心5 min;取450 μL上清于另一1.5 mL离心管,加入等体积-20 ℃预冷的异丙醇,混匀;-20 ℃静置30 min,4 ℃ 10 000 r·min-1下离心10 min;弃上清,用70%乙醇洗沉淀2次,凉风吹干或37 ℃烘干;用50 μL的1×TE buffer溶解沉淀。
1.3.4 改良CTAB法
取新鲜小麦叶片,在液氮中研磨成粉末,称取0.05 g于1.5 mL离心管中;加入500 μL 65 ℃预热的CTAB缓冲液和10 μL β-巯基乙醇,混匀;于65 ℃水浴1 h,间或混匀;加入500 μL氯仿-异戊醇(24∶1),不停混匀10 min,室温10 000 r·min-1下离心5 min;取450 μL上清于另一2.0 mL离心管,加入1 500 μL(至少是上清液2倍)的-20 ℃预冷的异丙醇,混匀;于-20 ℃静置1 h,4 ℃ 10 000 r·min-1下离心5 min;弃上清,加入500 μL的1×TE buffer于室温溶解30 min;至白色沉淀完全溶解后,加入500 μL氯仿-异戊醇(24∶1),轻轻混匀10~20 min,室温10 000 r·min-1下离心5 min;取450 μL上清于新的2.0 mL离心管,加入1.5 mL -20 ℃预冷无水乙醇和60 μL醋酸钠,混匀,于-20 ℃过夜(至少2 h);4 ℃ 12 000 r·min-1下离心5 min,弃上清,用70%乙醇洗沉淀2次,凉风吹干或37 ℃烘干;加入50 μL的1×TE buffer溶解沉淀。
1.3.5 AxyPrep DNA提取试剂盒法
DNA的提取严格按照试剂盒的操作说明进行。
1.3.6 Qiagen DNeasy Plant Mini Kit提取试剂盒法
DNA的提取严格按照试剂盒的操作说明进行。
1.3.7 再生Qiagen硅胶柱提取法
取新鲜小麦叶片,在液氮中研磨成粉末,称取0.05 g于1.5 mL离心管中;加入500 μL 85 ℃预热的CTAB缓冲液,混匀后于85 ℃水浴10 min,间或混匀;加入500 μL氯仿-异戊醇(24∶1),混匀,室温12 000 r·min-1下离心5 min;取450 μL上清于新的1.5 mL离心管,加入750 μL Binding buffer,混匀;每次移取700 μL混合液于经1~10次再生的Qiagen硅胶柱(再生方法:将已使用过的硅胶柱于1 mol·L-1HCl浸泡12 h后,转移至0.1 mol·L-1HCl浸泡2~3 d,最后用无菌水离心过滤清洗3遍),12 000 r·min-1下离心1 min,弃滤液;加入700 μL Washing buffer Ⅰ,12 000 r·min-1下离心1 min,重复一次;加入800 μL Washing buffer Ⅱ,12 000 r·min-1下离心1 min,弃滤液,12 000 r·min-1离心2 min;将硅胶柱置于新的1.5 mL离心管,加入50 μL 65 ℃预热的Elution buffer,静置1 min,12 000 r·min-1离心1 min,可重复一次。
1.4 DNA的质量检测
1.4.1 微量分光光度计检测
采用微量分光光度计(NanoVue)测定以上所提取DNA的A260/A280、A260/A230和浓度值。
1.4.2 琼脂糖凝胶电泳检测
取DNA溶液1 μL,用1.0%琼脂糖凝胶在130 V的电压下电泳20 min,由溴化乙锭染色,用Gel Doc XR+全自动凝胶成像系统(Bio-RAD公司)拍照检测DNA质量。
1.4.3 PCR扩增检测
细胞分裂素氧化酶对小麦的生长发育和产量有重要影响,本实验室目前正以细胞分裂素氧化酶基因1 ( TaCKX1)为目的基因对TILLING群体进行突变体筛选,在筛选过程中发现常规方法提取的DNA用于扩增大于1 000 bp的片段时常无法扩增出目的条带。因此,本试验以扩增 TaCKX1基因来检测DNA的质量。根据 TaCKX1基因的DNA序列,采用primer 5.0设计引物(表1)。PCR反应体系为10 μL,含10×PCR buffer 1 μL、25 mmol·L-1MgCl20.7 μL、100 ng·μL-1的DNA模板0.4 μL、10 μmol·L-1的上下游引物各0.4 μL、10 mmol·L-1dNTPs 0.1 μL、TaqDNA聚合酶0.2 μL、ddH2O 6.8 μL。PCR扩增程序:94 ℃预变性5 min;94 ℃变性30 s,57 ℃退火30 s,72 ℃延伸70 s,30个循环;72 ℃延伸10 min。扩增完成后,取等量的PCR产物,用1.0%琼脂糖凝胶在130 V电压下电泳15~20 min,根据条带大小和清晰度比较PCR产物的质量。
2 结果与分析
2.1 不同方法所提取DNA的微量分光光度计检测结果
表2为7种方法提取的DNA的纯度和浓度。由表2可见,采用不同方法提取小麦基因组DNA,A260/A280比值都在1.8~2.0之间,A260/A230比值都接近2.0,说明所提取的DNA纯度都很好;而再生Qiagen硅胶柱法、CTAB法和改良SDS法的DNA浓度都显著高于其他方法。其中再生Qiagen硅胶柱法提取的浓度最高,比浓度位居第二的CTAB法提高50.2%,比Qiagen DNeasy Plant Mini Kit提取试剂盒法提高234.4%。
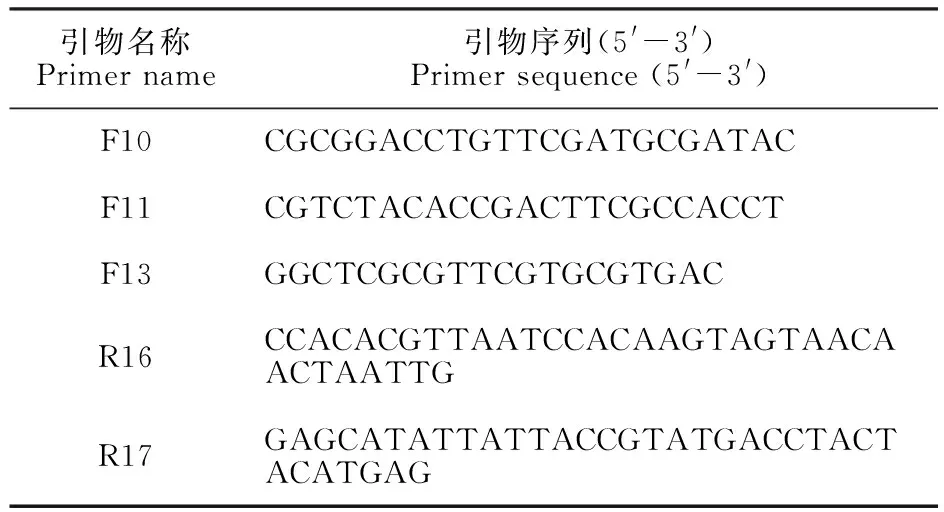
表1 用于扩增 TaCKX1基因的引物Table 1 Primers of TaCKX1 gene

表2 不同方法所提取DNA的纯度和浓度Table 2 Purity and concentration of DNA extracted using different methods
用SPSS 17对各浓度进行差异显著性分析,各浓度后不同小写字母表示差异在0.01水平上显著。
Significance analysis of concentrations was conducted using SPSS 17; The lowercases following the concentrations indicated the significant differences at 0.01 levels.
2.2 不同方法所提取DNA的琼脂糖凝胶电泳检测结果
对DNA进行琼脂糖凝胶电泳检测,结果如图1,且多次电泳均得到相似结果。由图1可知,7种方法所提取的DNA完整性均较好,无RNA和其他杂质污染。在上样量都为1 μL的情况下,SDS法和Qiagen硅胶柱法所提取DNA条带亮度较其他方法的低,说明DNA浓度较低;改良SDS法、CTAB法和AxyPrep试剂盒法所提取的DNA主带下方有拖带现象,说明DNA有部分降解;改良CTAB法、Qiagen试剂盒法和再生Qiagen硅胶柱法提取的DNA条明亮,无拖带现象,DNA质量较好。
2.3 新鲜DNA的PCR扩增检测结果
将三个上游引物和两个下游引物进行两两组合,可扩增出6种大小不同的PCR产物,F10/R16为1 078 bp,F10/R17为1 150 bp,F11/R16为970 bp,F11/R17为1 042 bp,F13/R16为659 bp,F13/R17为731 bp。以7种方法提取的DNA为模板进行PCR扩增,进行3次重复试验,PCR产物经琼脂糖凝胶电泳检测,结果均如图2所示。其中SDS法、改良SDS法和CTAB法只能扩增出较小的片段,对于大于1 000 bp的片段扩增困难;改良CTAB法、AxyPrep试剂盒法、Qiagen试剂盒法和再生Qiagen硅胶柱法能够扩增出较大片段。
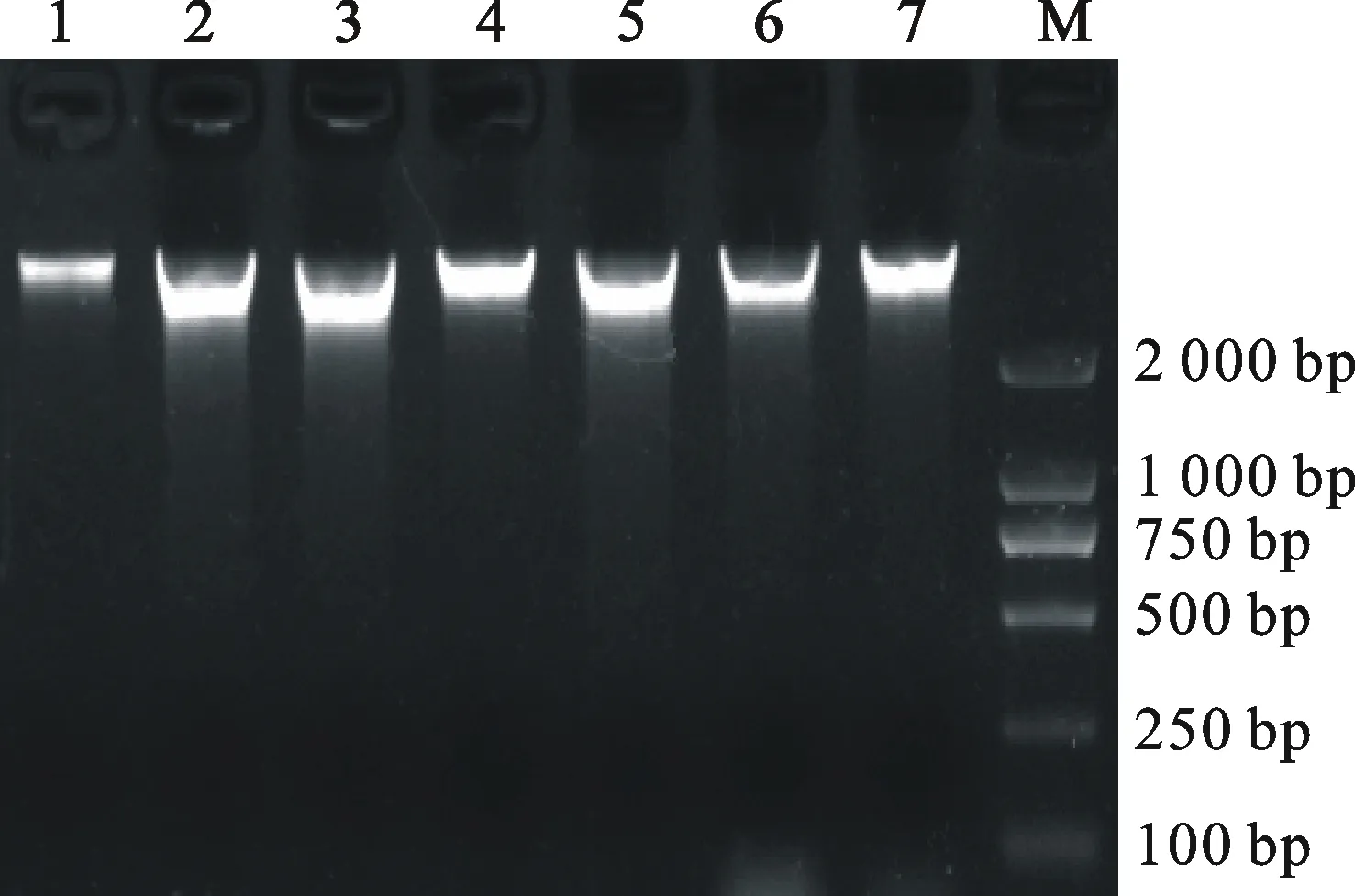
M:DL2000;1:SDS法;2:改良SDS法;3:CTAB法;4:改良CTAB法;5:AxyPrep DNA试剂盒法;6:Qiagen DNA试剂盒法;7:再生Qiagen硅胶柱法。图2和图3中同。
M: DL2000; 1: SDS method; 2: Modified SDS method; 3: CTAB method; 4: Modified CTAB method; 5:AxyPrep DNA column method; 6:Qiagen DNeasy Plant Mini Kit method; 7: Regenerated Qiagen column method.The same in Fig.2 and Fig.3.
图1 7种方法所提取DNA的琼脂糖凝胶电泳结果
Fig.1 Agarose gel electrophoresis result of DNA extracted by seven methods
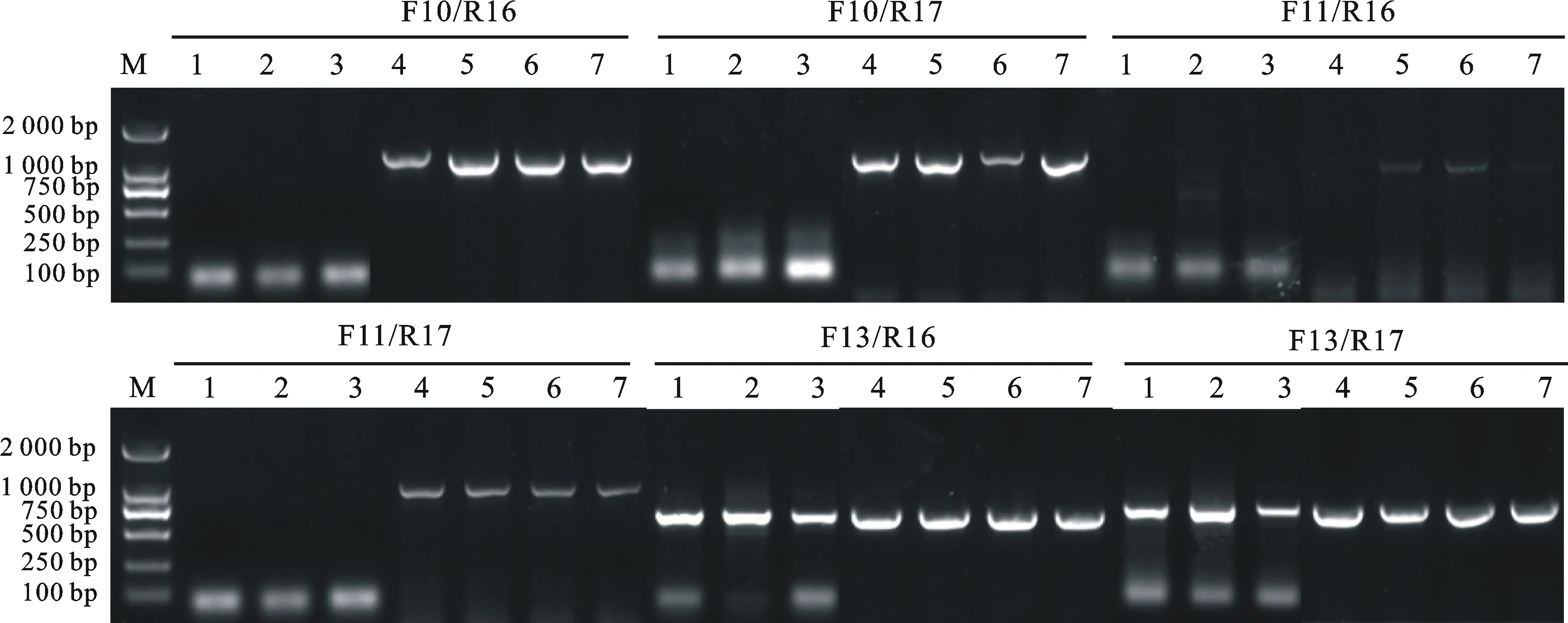
图2 以不同方法所提取DNA为模板的PCR扩增产物琼脂糖凝胶电泳结果
2.4 于-20 ℃存放100 d后DNA的PCR扩增检测结果
TILLING技术中所提取的DNA需要长期存放,以便随时对目的基因进行扫描筛选,但长期存放会影响DNA的质量,进而影响后续的PCR结果和突变体鉴定的效率。图3为DNA于-20 ℃存放100 d后再进行检测的结果,多次电泳得到相似结果。由图可见,DNA存放一段时间后都有轻微降解,其中改良CTAB法降解最严重,其他方法提取的DNA主带仍清晰明亮。为进一步鉴定本研究所提取DNA的质量,将能够较好扩增出大片段的4种DNA于-20 ℃存放100 d后再进行PCR扩增,产物经琼脂糖凝胶电泳检测,结果如图4。由图4可见,存放100 d后,对于两对扩增小片段引物,4种方法所提取的DNA都有较好的扩增效果;而对于四对扩增大片段引物,则只有Qiagen法和再生Qiagen硅胶柱法能扩增出清晰明亮的条带,改良CTAB法和AxyPrep法扩增结果不佳。
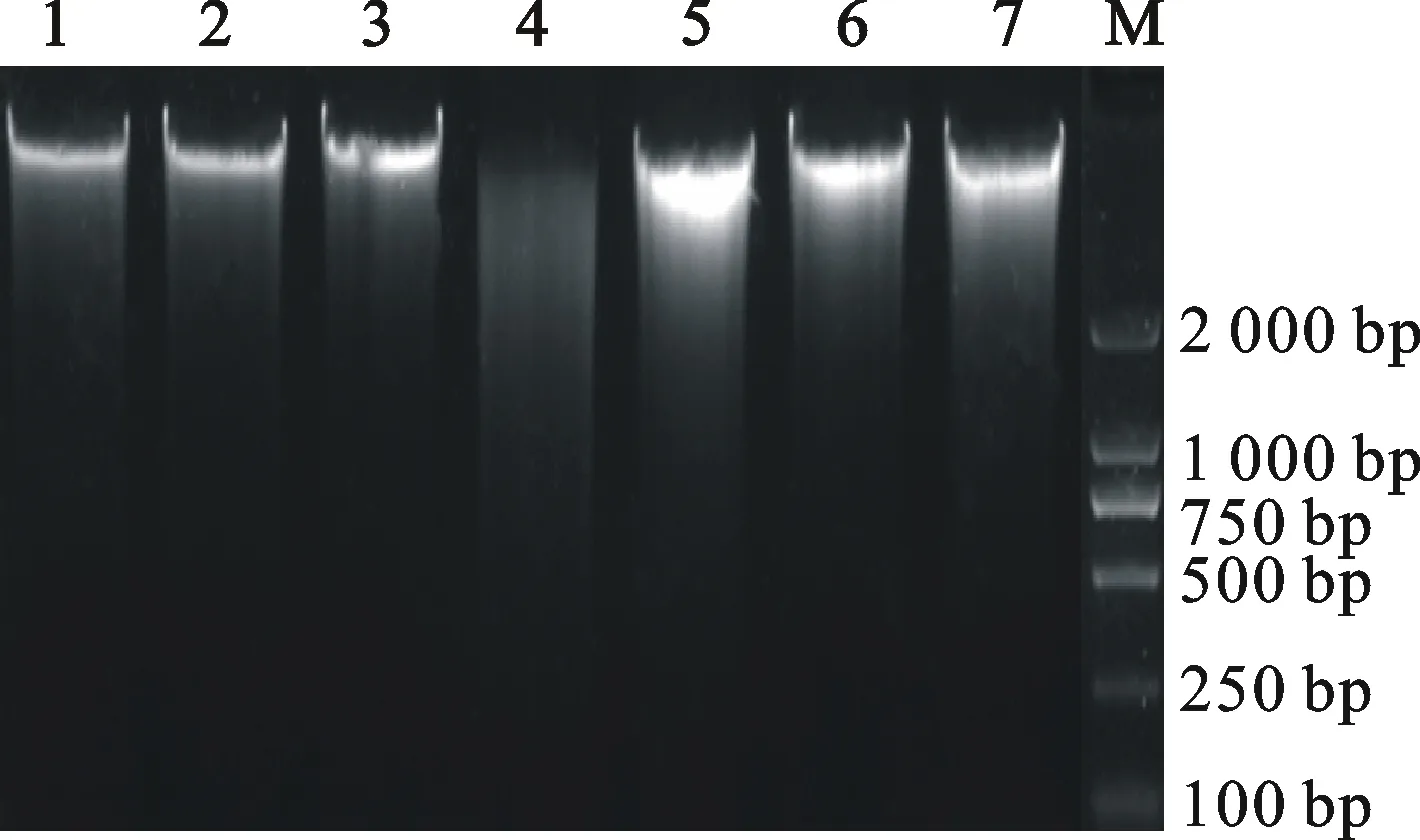
图3 7种方法所提取DNA经-20 ℃存放100 d后的琼脂糖凝胶电泳结果

M:DL2000;1:改良CTAB法;2:AxyPrep试剂盒法;3:Qiagen试剂盒法;4:再生Qiagen硅胶柱法。
M: DL2000; 1: Modified CTAB method; 2:AxyPrep DNA column method; 3:Qiagen DNeasy Plant Mini Kit method; 4: Regenerated Qiagen column method.
图4 -20 ℃存放100 d后的DNA经PCR扩增后所得产物的琼脂糖凝胶电泳结果
Fig.4 Agarose gel electrophoresis result of PCR products with DNA template stored in -20 ℃ for 100 days
3 讨 论
高质量的DNA是TILLING技术成功应用的基础。由于小麦基因组庞大,遗传基础复杂,且植株中含有较多的多酚、蛋白质、脂类等干扰物质,常规方法提取出的DNA往往不能满足PCR的要求。尤其是由于用于TILLING突变体鉴定的DNA往往需要进行长达数年甚至更长时间的保存,对DNA的质量要求非常高。尽管国内外研究者对DNA提取中的杂质去除进行了很多研究[23-26],但在本研究条件下效果均不够理想。本文比较了7种不同的DNA提取方法,其中SDS法、改良SDS法和CTAB法简单快速,但所得DNA常无法扩增出大于1 000 bp的产物,此结果与本实验室利用TILLING群体对多个目标基因突变体筛选试验过程中所遇到的情况是一致的。改良CTAB法和AxyPrep试剂盒法所得DNA能够较好的扩增出产物,但长期储存后扩增效果差,并且改良CTAB法耗时过长,所用试剂多,不适合TILLING技术进行大规模基因组DNA样品的提取。Qiagen试剂盒法和再生Qiagen硅胶柱法所提取的DNA质量高,储存一段时间后能扩增出清晰明亮的条带,可满足TILLING技术对DNA质量的要求。然而,Qiagen试剂盒的费用较高,不适合科研经费不足的研究者或需要大批量提取DNA样品的TILLING技术。而采用本实验室与新西兰坎特伯雷大学Jameson教授实验室合作建立的再生硅胶柱法,通过HCl浸泡及无菌水洗涤等简单程序对使用过的硅胶柱进行回收再生,可使Qiagen试剂盒硅胶柱重复利用10次以上[22],可以大幅度降低成本。而且所提取的小麦基因组DNA具有得率高、质量好、PCR扩增成功率高等优点,能很好地满足TILLING技术中大量提取DNA样品的需求。
[1]BRENCHLEY R,SPANNAGL M,PFEIFER M,etal.Analysis of the bread wheat genome using whole-genome shotgun sequencing [J].Nature,2012,491:705.
[2] 叶兴国,陈 明,杜丽璞,等.小麦转基因方法及其评述 [J].遗传,2011,33(5):422.
YE X G,CHEN M,DU L P,etal.Description and evaluation of transformation approaches used in wheat [J].Hereditas,2011,33(5):422.
[3]COLBERT T,TILL B J,TOMPA R,etal.High-throughput screening for induced point mutations [J].PlantPhysiology,2001,126(2):480.
[4]HENIKOFF S,TILL B J,COMAI L.TILLING.Traditional mutagenesis meets functional genomics [J].PlantPhysiology,2004,135(2):630.
[5]TILL B J,REYNOLDS S H,GREENE E A,etal.Large-scale discovery of induced point mutations with high-throughput TILLING [J].GenomeResearch,2003,13(3):524.
[6]GREENE E A,CODOMO C A,TAYLOR N E,etal.Spectrum of chemically induced mutations from a large-scale reverse-genetic screen inArabidopsis[J].Genetics,2003,164(2):731.
[7]MCCALLUM C M,COMAI L,GREENE E A,etal.Targeting induced local lesions IN genomes(TILLING) for plant functional genomics [J].PlantPhysiology,2000,123(2):439.
[8]UAUY C,PARAISO F,COLASUONNO P,etal.A modified TILLING approach to detect induced mutations in tetraploid and hexaploid wheat [J].BMCPlantBiology,2009,9:114.
[9]SLADE A J,FUERSTENBERG S I,LOEFFLER D,etal.A reverse genetic,nontransgenic approach to wheat crop improvement by TILLING [J].NatureBiotechnology,2005,23(1):75.
[10]KOORNNEEF M,DELLAERT L W,VAN DER VEEN J H.EMS- and radiation-induced mutation frequencies at individual loci inArabidopsisthaliana(L.)Heynh. [J].MutationResearch,1982,93(1):109.
[11]SIMMONDS J,SCOTT P,BRINTON J,etal.A splice acceptor site mutation in TaGW2-A1,increases thousand grain weight in tetraploid and hexaploid wheat through wider and longer grains [J].Theoretical&AppliedGenetics,2016,129:1.
[12] 董建力,王敬东,惠红霞,等.小麦幼叶DNA微量快速提取方法的改进 [J].麦类作物学报,2007,27(3):475.
DONG J L,WANG J D,HUI H X,etal.Improvement of the method for rapid DNA extraction from wheat leaflet [J].JournalofTriticeaeCrops,2007,27(3):475.
[13] 田再民,龚学臣,张利增.小麦DNA提取方法的比较 [J].湖北农业科学,2009,25(4):22.
TIAN Z M,GONG X C,ZHANG L Z.Comparison of several kinds of wheat DNA extraction methods [J].HubeiAgriculturalSciences,2009,25(4):22.
[14]LIN H Y,CHIANG J W,SHIH Y C.Detection of genetically modified soybeans by PCR method and immunoassay kits [J].JournalofFood&DrugAnalysis,2001,9(3):160.
[15] 刘 萍,代红军,张立杰,等.DNA提取方法与植物种类的效应比较 [J].宁夏农林科技,1998(1):15.
LIU P,DAI H J,ZHANG L J,etal.Mutual effects between various extraction methods and plant species [J].NingxiaJournalofAgricultureandForestryScienceandTechnology,1998(1):15.
[16] 陈 璋,朱秀英,卢 勤.三种快速提取植物DNA改良方法的比较 [J].遗传,1990(2):10.
CHEN Z,ZHU X Y,LU Q.Comparison of three improved rapid extraction methods for plant DNA [J].Hereditas,1990(2):10.
[17] 李永明.实用分子生物学方法手册[M].北京:科学出版社,1998:176.
LI Y M.Practical Molecular Biomethods Handbook [M].Beijing:Science Press,1998:176.
[18] 顾红雅.植物基因与分子操作[M].北京:北京大学出版社,1995:19.
GU H Y.Plant Genetic and Molecular Operation[M].Beijing:Peking University Press,1995:19.
[19] 王小利.小麦基因组DNA提取方法比较研究 [J].实验技术与管理,2012,29(10):39.
WANG X L.Study on comparison of methods of extracting genome DNA from wheat [J].ExperimentalTechnologyandManagement,2012,29(10):39.
[20] 王 勇,袁 华,刘国磊,等.改进的碱裂解法大规模提取质粒DNA [J].数理医药学杂志,2010,23(5):519.
WANG Y,YUAN H,LIU G L,etal.Improved alkaline lysis method for large-scale extraction of plasmid DNA [J].JournalofMathematicalMedicine,2010,23(5):519.
[21] 戴 剑,洪德林,张大栋,等.一种快速高效的DNA提取方法研究 [J].麦类作物学报,2011,31(3):437.
DAI J,HONG D L,ZHANG D D,etal.Study on a rapid,high efficient DNA extraction method [J].JournalofTriticeaeCrops,2011,31(3):437.
[22]FU Z Y,SONG J C,JAMESON P E.A rapid and cost effective protocal for plant genomic DNA isolation using regenerated silica columns in combination with CTAB method [J].JournalofIntegrativeAgriculture,2016,doi:10.1016/S2095-3119(16)61534-4.
[23] 陈林杨,宋敏舒,查红光,等.一种改良的植物基因组DNA通用提取方法 [J].植物分类与资源学报,2014,36(3):375.
CHEN L Y,SONG M S,CHA H G,etal.A modified protocol for plant genome DNA extraction [J].PlantDiversityandResources,2014,36(3):375.
[24] 李金璐,王 硕,于 婧,等.一种改良的植物DNA提取方法 [J].植物学报,2013,48(1):72.
LI J L,WANG S,YU J,etal.A modified plant DNA extraction method [J].ChineseBulletinofBotany,2013,48(1):72.
[25] 刘 欣,祝长青,王毅谦,等.大豆转基因检测中DNA提取方法的比较研究 [J].食品科学,2013,34(4):199.
LIU X,ZHU C Q,WANG Y Q,etal.Methodology comparison of DNA extraction from soybean for detection of genetically modified organism [J].FoodScience,2013,34(4):199.
[26] 吴艳艳,代德艳,蔡春梅.一种改良的大豆DNA提取方法 [J].大豆科学,2015,34(1):112.
WU Y Y,DAI D Y,CAI C M.A modified UREA protocol for soybean DNA extraction [J].SoybeanScience,2015,34(1):112.
Optimization of Genomic DNA Extraction Methods for Mutant Screen in a Wheat TILLING Population
U Yan1,XU Jifa1,CHEN Lei1,ZHAO Jiqiang1,FU Zeyu2,JAMESON Paula E2,SONG Jiancheng1
(1.School of Life Sciences,Yantai University,Yantai,Shandong 264005,China; 2.School of Biological Sciences,University of Canterbury,Christchurch,New Zealand)
In order to optimize the genomic DNA extraction methods for efficient mutant screening in wheat TILLING population,seven different DNA extraction methods,such as SDS method,modified SDS method,CTAB method,modified CTAB method,AxyPrep DNA column method,Qiagen DNeasy Plant Mini Kit method and regenerated Qiagen column method,were compared for their effectiveness on genomic DNA extraction.The results showed that the DNA concentration varied among different extraction methods,and the DNA concentration of regenerated Qiagen column method,CTAB method,and modified SDS method were higher than that of the other method.The quality of DNA extracted by all the seven methods in terms of purity and integrity were satisfied when detected with agarose gel electrophoresis.However,when these DNA samples were used as PCR template to amplify products more than 1 000 bp in length,the amplification efficiency of the modified CTAB method,AxyPrep DNA column method,Qiagen DNeasy Plant Mini Kit method and regenerated Qiagen column method was much better than that of the other three methods.After storing the DNA samples in -20 ℃ for 100 days,the target gene sequences longer than 1 000 bp could only be amplified from the DNA samples extracted using Qiagen DNeasy Plant Mini Kit method and regenerated Qiagen column method.The newly developed method combining the regenerated Qiagen column with home-made solutions allowed us to extract high quality genomic DNA from a wheat TILLING population,with much higher DNA concentration and lower cost than the Qiagen DNeasy Plant Mini Kit.This will provide an efficient and economical genomic DNA extraction method for TILLING procedure in wheat.
Wheat; TILLING; Genome DNA; Extraction methods
时间:2017-01-16
2016-10-05
2016-12-24
国家自然科学基金项目(31371616)
E-mail:xuyan540769161@163.com
宋建成(E-mail:Jcsong88@yahoo.com)
S512.1;S330
A
1009-1041(2017)02-0185-07
网络出版地址:http://www.cnki.net/kcms/detail/61.1359.S.20170116.1833.010.html
