外引大麦SSR标记遗传多样性及其与农艺性状的关联分析
2017-03-01贾建磊王晋民吕仲昱朱惠琴杨莉娜李宗仁
赖 勇,贾建磊,王晋民,任 龙,吕仲昱,朱惠琴,马 辉,杨莉娜,李宗仁
(1.青海大学农牧学院,青海西宁 810016; 2.三江源生态与高原农牧业国家重点实验室,青海西宁 810016;3.青海大学现代教育技术中心,青海西宁 810016; 4.甘肃隆源农业科学研究所,甘肃兰州 730070)
外引大麦SSR标记遗传多样性及其与农艺性状的关联分析
赖 勇1,2,贾建磊1,2,王晋民1,任 龙3,吕仲昱4,朱惠琴1,马 辉1,杨莉娜1,李宗仁1
(1.青海大学农牧学院,青海西宁 810016; 2.三江源生态与高原农牧业国家重点实验室,青海西宁 810016;3.青海大学现代教育技术中心,青海西宁 810016; 4.甘肃隆源农业科学研究所,甘肃兰州 730070)
为了掌握引进种质资源遗传状况,以55份国外大麦品种为材料,利用SSR分子标记分析了其遗传多样性及性状与标记的关联关系。结果表明,54对SSR标记从55份材料中共检测出154个等位基因、167种基因型。基因多样性变化范围为0.103~0.708,平均值为0.448。PIC变化范围为0.098~0.651,平均值为0.382。不同材料间的遗传相似系数(GS)变化范围为0.533~0.940,平均值为0.722。在GS值约为0.682处可将55份材料分为3类,分别包含49、2、4份材料。群体遗传结构分析将供试材料分成3大亚群,各包含9、24、22份材料。基于GLM模型的关联分析,在P<0.01水平下共检测到16对与株高、穗长、穗下茎长、千粒重和全生育期等5个农艺性状相关联的标记,对性状的解释率的变化范围为6.3%~33.1%。其中,5对标记同时与多个农艺性状相关联。
大麦;SSR标记;遗传多样性;关联分析
大麦是具有食用、饲用及酿造等多种用途的重要谷类作物,在我国主要种植于青藏高原地区。随着人工选择的加剧,亲本遗传基础狭窄成为制约育种进程的主要问题之一[1]。通过对引进种质资源的遗传多样性分析,可以有效地指导亲本组合配制,提高大麦育种效率。在遗传多样性分析的基础上,发掘其中的有利基因,对于后期的分子标记辅助选择具有重要意义。基于连锁不平衡的关联分析目前已成为基因发掘研究的主要方法之一,具有不需构建分离群体、能够有效分析等位基因对性状的贡献率等优点[2-4]。分子标记技术在种质资源的遗传多样性研究及有利基因发掘中已被广泛应用。其中,SSR因其多态性高、数量丰富、重复性好、呈共显性、操作简单等优点[5-7],被广泛应用于各类作物的遗传多样性和有利基因发掘研究中[8-11]。国内外关于大麦遗传多样性的报道较多[12-14]。同时在基因发掘研究方面,Kraakman等[15]利用AFLP和SSR标记进行关联分析,找到部分与抽穗期、株高、叶锈抗性等性状相关联的主效QTL。Sun等[16]以42对SSR进行标记与性状的关联分析,发掘出与株高、穗长、千粒重等重要农艺性状相关联的标记位点。Ivandic等[17]通过SSR标记与性状的关联分析,发掘出与大麦抗白粉病相关联的位点。这些研究表明,关联分析是农艺性状、抗病、抗胁迫等相关基因发掘的有效手段。本研究以引进的55份国外大麦品种为材料,以SSR标记技术分析其遗传多样性以及性状与标记的关联关系,为后期大麦新品种选育过程中的亲本组合配制和分子标记辅助选择提供一定的理论依据。
1 材料与方法
1.1 供试材料及其农艺性状的测定
供试材料为55份国外引进大麦品种,包括19份丹麦品种、12份匈牙利品种、12份德国品种以及少量其他国家栽培品种(表1)。所有材料于2014年3-7月和2015年3-7月连续两个生长季种植于青海大学农牧学院实验地,每份材料种2行,行长1 m,行间距20 cm,管理同一般大田。于分蘖期采幼嫩叶片,-80 ℃保存备用。于成熟期,每个品种随机抽取10个单株,测定株高、穗长、穗下茎长、千粒重和全生育期等农艺性状,然后取平均值。
1.2 SSR标记分析
总基因组DNA的提取采用CTAB法[18]。选用54对均匀分布于大麦1H-7H染色体上的SSR标记(表2)进行多态性扫描。PCR扩增体系:2×MasterMix( BIOTEKE,PR1701) 5 μL,上下游引物( 10 μmol·L-1) 各1 μL,模板DNA(60 ng·μL-1) 1 μL,加ddH2O至10 μL。PCR扩增程序:94 ℃预变性5 min;94 ℃变性50 s,64~55 ℃(每个循环降低1 ℃)退火50 s,72 ℃延伸50 s,10个循环;94 ℃变性50 s,55 ℃退火50 s,72 ℃延伸50 s,30个循环;72 ℃延伸5 min。PCR产物用8.0%非变性聚丙烯酰胺凝胶电泳分离,银染显色。
1.3 数据统计分析
以二进制和基因型记录SSR分析结果:同一位点上具有相同迁移率的条带记为1,无带记为0,构建二元矩阵;以字母A、B、C等代表不同的等位基因,记录基因型。用NTSYS-pc软件计算遗传相似系数(genetic similarity coefficient,GS),并按非加权配对法(UPGMA)和SHAN程序聚类分析。用Powermarker 3.25软件分析等位基因数、基因型数、基因多样性以及多态性信息量(polymorphism information content,PIC)。用Structure 2.3.1软件分析群体遗传结构,估计最佳群体组群数,参数设置参考赖 勇等[19]。当K值持续增大时,通过计算ΔK的方法确定K值[20]。采用Tassel 2.1软件中一般线性模型(general linear model,GLM)进行关联分析,GLM分析中以Q值作为协变量进行回归分析。
2 结果与分析
2.1 供试材料的农艺性状
由表1可知,55份材料在株高、穗长、穗下茎长、千粒重和全生育期等性状上均有一定的差异。株高变化范围为62.5~124.0 cm,变异系数为0.177,以德国品种NFC Tripple的株高最低,丹麦品种H.dist. V. trif的株高最高;穗长变化范围为5.8~10.7 cm,变异系数为0.120,以丹麦品种Svakof. Mari的穗长最长,日本品种Kantougohere的穗长最短;穗下茎长的变化范围为15.3~44.5 cm,变异系数为0.278,以丹麦品种H.dist. V. trif的穗下茎最长,匈牙利品种M-01/40的穗下茎最短;千粒重变化范围为31.7~60.3 g,变异系数为0.098,以阿富汗品种Loeal的千粒重最大;全生育期变化范围为90~109 d,变异系数为0.048,以波兰品种Pocnodzenie Dania、阿富汗品种Loeal和叙利亚品种Briggs-Research Ⅰ的生育期最短,德国品种PASADENA的生育期最长。
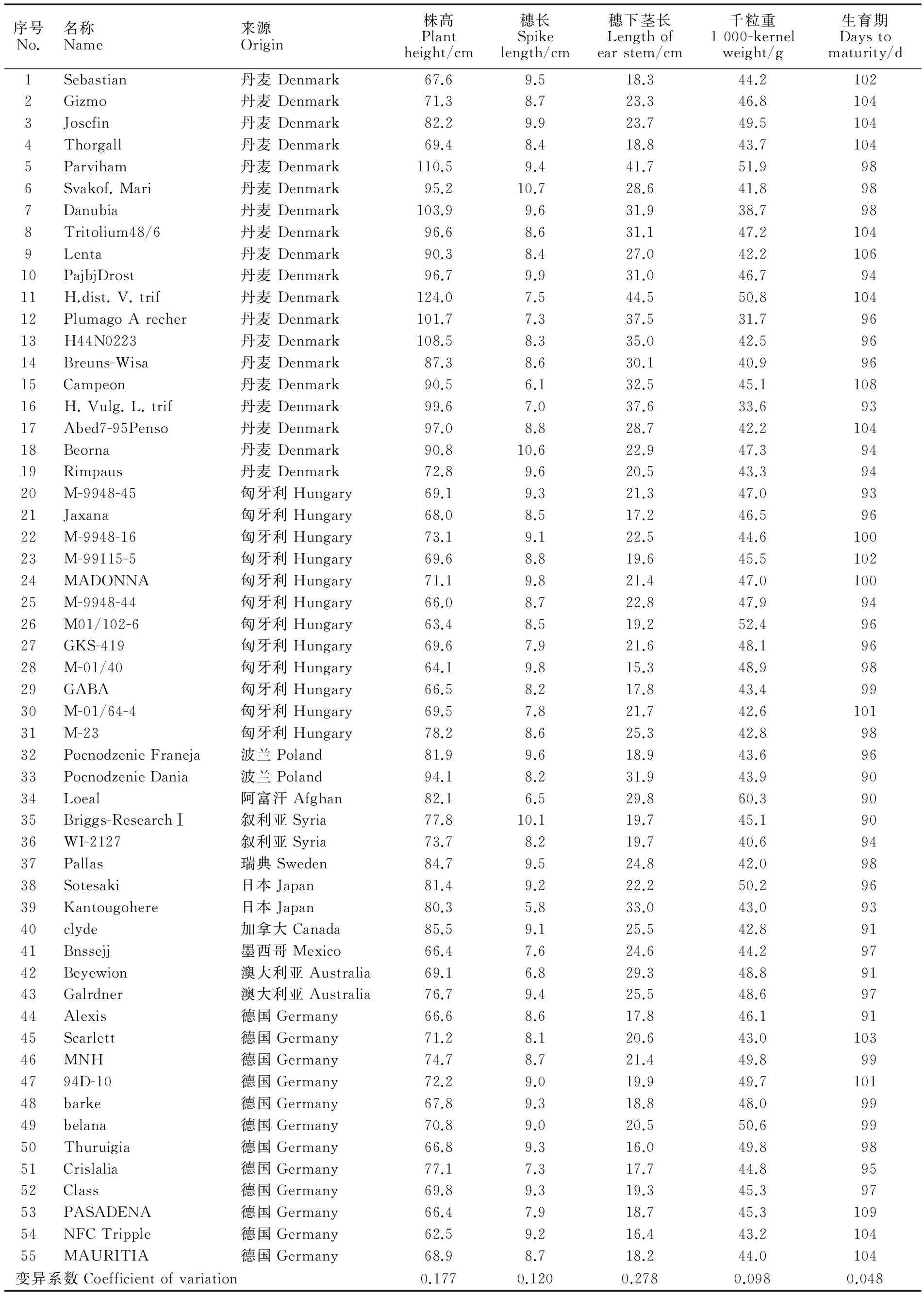
表1 国外引进大麦品种名称、来源及农艺性状调查结果Table 1 Name,origin and agronomic traits of barley accessions from abroad
2.2 SSR标记分析
通过多态性扫描分析,54对SSR标记从55份引进材料中共检测出等位基因154个、基因型167种,平均每对标记可检测到2.9个等位基因、3.1种基因型,所有标记均表现出一定的多态性(表2)。等位基因变化范围为2~6,基因型变化范围为2~7,其中标记Bmag223检测到的等位变异最多,为6个;标记MGB402检测到基因型最多,有7种。基因多样性指数变化范围为0.103~0.708,平均值为0.448。PIC值变化范围为0.098~0.651,平均值为0.382。基因多样性指数与PIC值是遗传多样性的重要指标,二者结果存在一致性。
2.3 遗传相似性系数及聚类分析
55份引进大麦材料间的GS值变化范围为0.533~0.940,平均值为0.722。丹麦品种嘉士伯3号与嘉士伯4号间的GS值最大,为0.940,说明它们之间的亲缘关系较近,不适合作为杂交组合。丹麦品种Parviham与阿富汗品种Loeal间的GS值最小,为0.533,说明它们之间的亲缘关系较远,可以考虑作为杂交组合进行配置。总的来看,来源地相同的材料间GS值较大,来源地不同的材料间GS值较小,遗传差异分析结果与来源地差异基本相符合。
按UPGMA法进行聚类分析(图 1)表明,55份大麦材料在GS值为0.682水平被聚成3类。第一大类群来源最丰富,包括大部分丹麦品种、匈牙利品种和德国品种,同时还包括少量其他国家品种;波兰品种Pocnodzenie Dania与加拿大品种加72大麦被聚到第二类;Parviham等4个丹麦品种被聚为第三类。
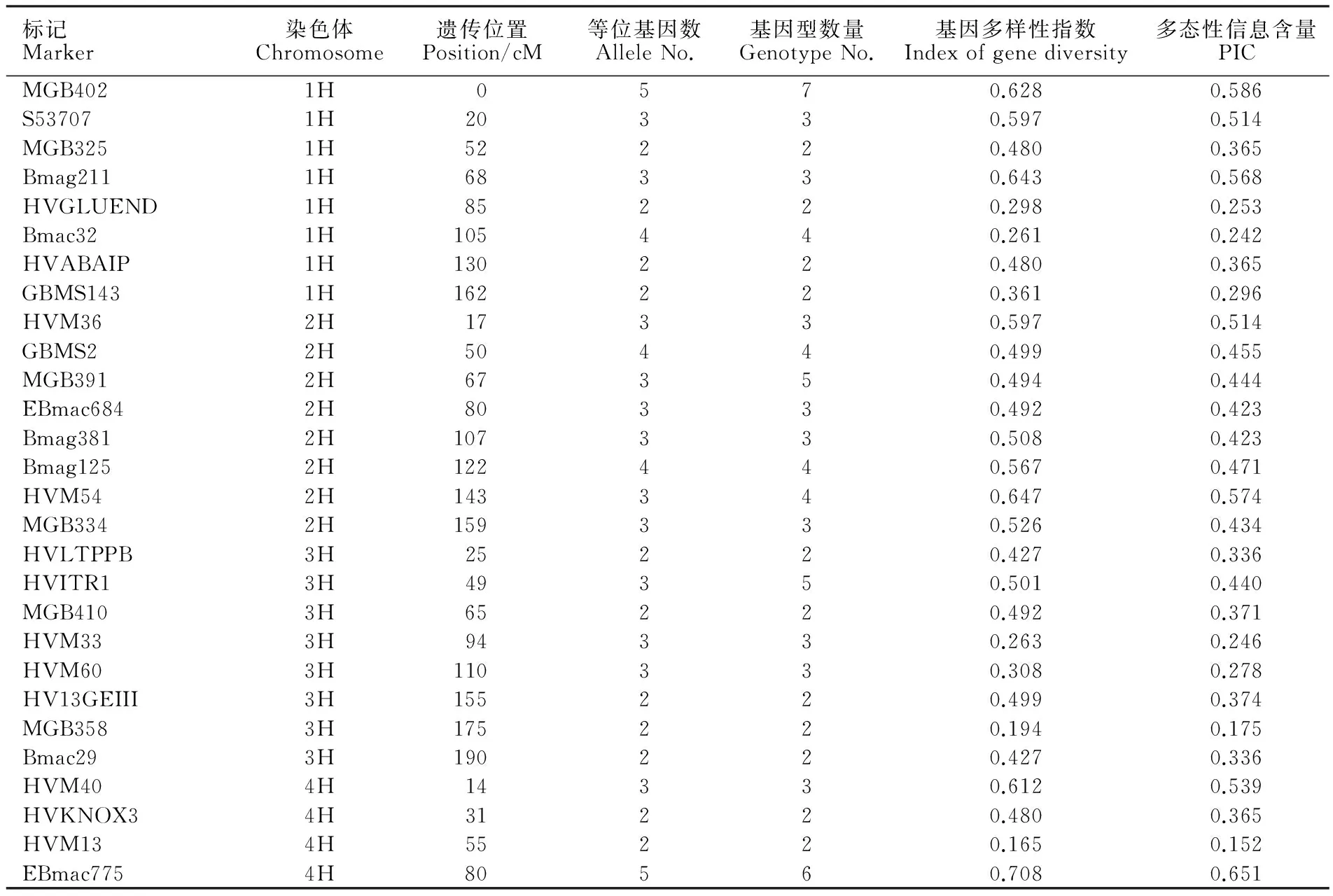
表2 54对SSR标记的遗传多样性分析Table 2 Genetic diversity analysis of 54 SSR markers
(续表2 Continued table 2)
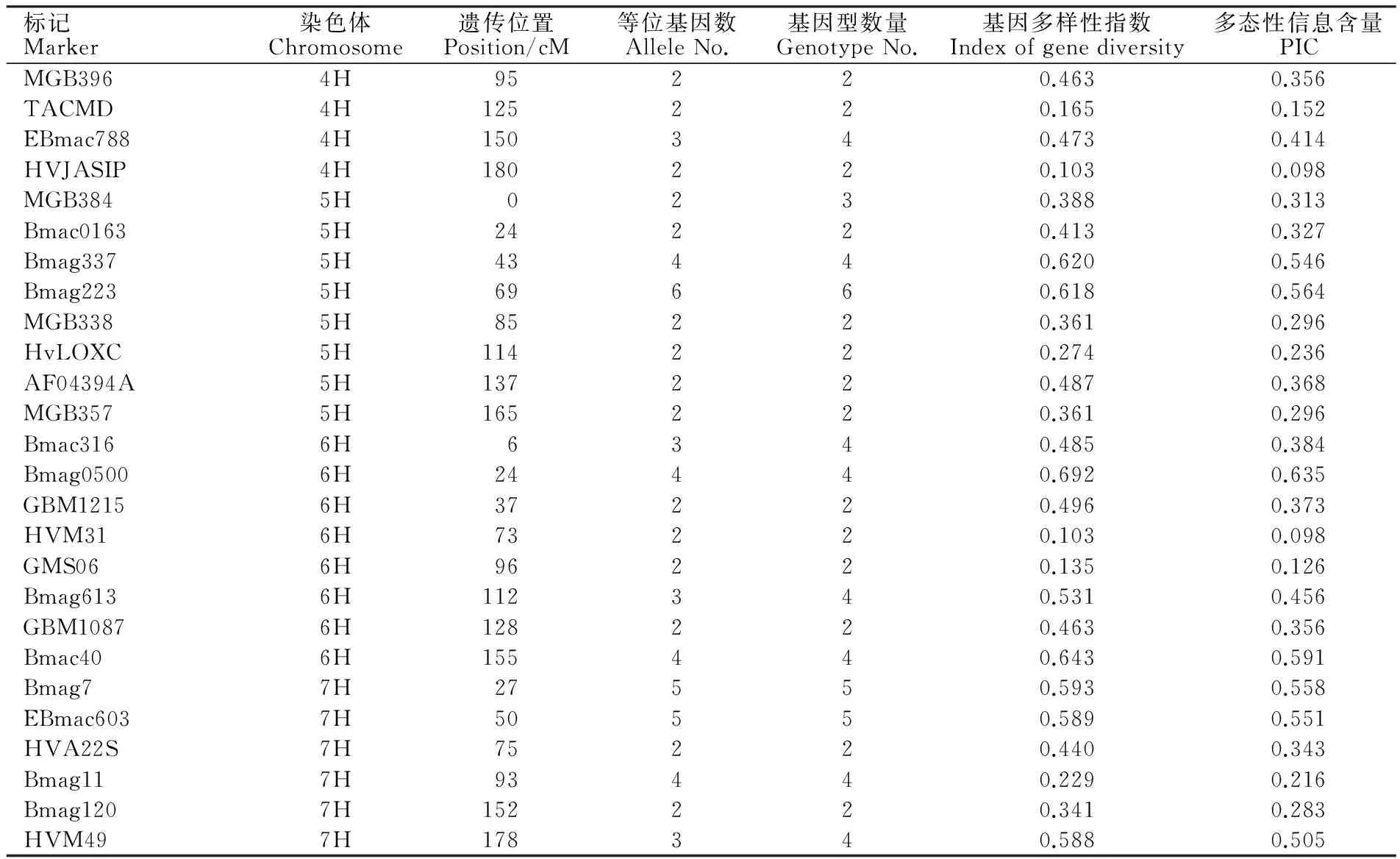
标记Marker染色体Chromosome遗传位置Position/cM等位基因数AlleleNo.基因型数量GenotypeNo.基因多样性指数Indexofgenediversity多态性信息含量PICMGB3964H95220.4630.356TACMD4H125220.1650.152EBmac7884H150340.4730.414HVJASIP4H180220.1030.098MGB3845H0230.3880.313Bmac01635H24220.4130.327Bmag3375H43440.6200.546Bmag2235H69660.6180.564MGB3385H85220.3610.296HvLOXC5H114220.2740.236AF04394A5H137220.4870.368MGB3575H165220.3610.296Bmac3166H6340.4850.384Bmag05006H24440.6920.635GBM12156H37220.4960.373HVM316H73220.1030.098GMS066H96220.1350.126Bmag6136H112340.5310.456GBM10876H128220.4630.356Bmac406H155440.6430.591Bmag77H27550.5930.558EBmac6037H50550.5890.551HVA22S7H75220.4400.343Bmag117H93440.2290.216Bmag1207H152220.3410.283HVM497H178340.5880.505
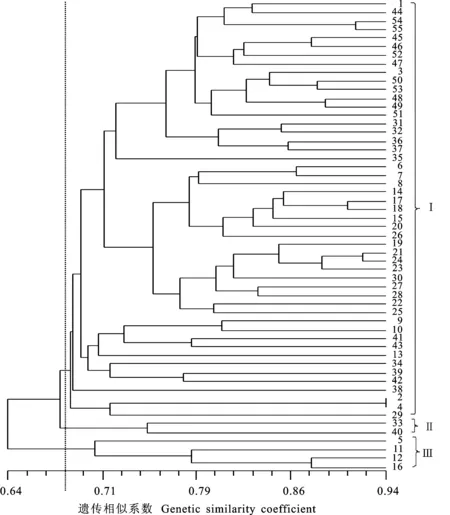
图1 基于54对SSR标记数据的55份大麦材料聚类图
2.4 群体遗传结构分析
通过54对标记获得的基因型数据,对55份材料进行群体遗传结构分析。由图2可知,当K=3时,ΔK值最大,因此将55份材料分成3个亚群。第一亚群(P1)材料数最少,包括Parviham在内的9份丹麦品种;第二亚群(P2)含有24份材料,包括部分丹麦品种、匈牙利品种和所有德国品种;第三亚群(P3)含有22份材料,也包括了部分丹麦品种、匈牙利品种以及少量其他国家的品种。
2.5 关联分析
从表3可知,在P<0.01水平下,有16对标记与各性状相关联,对表型变异的解释率范围为6.3%~33.1%,其中,与株高关联的标记有4对,与穗长相关联的标记有6对,与穗下茎长相关联的标记有4对,与千粒重相关联的标记有6对,与全生育期相关联的标记有2对,即有5对标记同时与多个性状相关联;而在P<0.001水平下,仅有其中6对标记与各性状相关联。
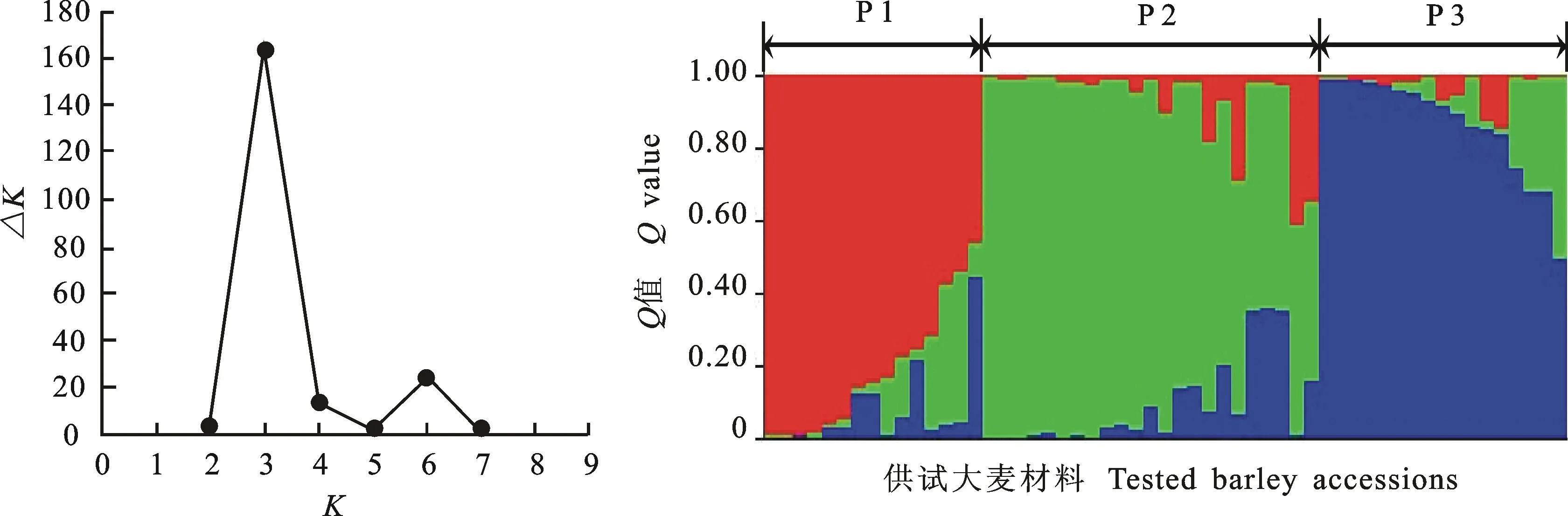
图2 55份外引大麦材料的群体遗传结构

标记Marker染色体Chromosome图距Position/cM表型解释率Explainedphenotypicvariation/%株高Plantheight穗长Spikelength穗下茎长Lengthofearstem千粒重1000-kernelweight全生育期DaystomaturityHVGLUEND1HL8514.56.3GBMS22HS5017.619.225.2EBmac6842HL8014.115.0Bmag1252HL12222.3HVM333HL9416.228.6HVM603HL11033.1HV13GEIII3HL15521.3Bmac293HL1904.8EBmac7754HL8019.222.4EBmac7884HL1509.3Bmac3166HS629.1Bmag05006HS2421.5GBM12156HS3714.2GMS066HL9612.3Bmac406HL15520.4Bmag77HS2710.9
下划线标注的数字表示在P<0.001水平下关联标记对表型的解释率。
The underlined number indicates the explained phenotypic variation of SSR markers related to agronomic traits at 0.001 level.
3 讨 论
张赤红等[21-22]研究表明,我国大麦存在遗传基础狭窄的问题。引进种质资源是丰富遗传基础的有效手段。本研究引进大麦品种均来源于其他国家,与国内尤其是青海省的大麦品种存在较大遗传差异,从一定程度上丰富了育种基础。但是由于引进大麦均为栽培品种,经过大量人工选择而来,这些品种之间的遗传差异较小。通过54对SSR标记的分析,55份引进大麦品种间的GS值变化范围为0.533~0.940,平均值为0.722,整体仍然较高,亲缘关系不够远。引进的55大麦品种中,二者间GS值较大的,例如嘉士伯3号与嘉士伯4号(GS=0.940),不作为杂交组合考虑,主要与青海本地材料进行杂交。结合表型数据,德国品种NFC Tripple株高仅有62.5 cm,可以作物降低大麦株高、增强抗倒伏能力的亲本材料;同时丹麦品种H.dist. V. trif的株高为124.0 cm,比NFC Tripple高61.5 cm,因此二者可以配置组合构建分离群体进行株高遗传分析。千粒重是重要的产量相关性状,阿富汗品种Loeal的千粒重达到60.3 g,可以作为后期改良大麦千粒重的亲本材料。
遗传多样性分析是后期进行关联分析发掘种质有利基因的基础[23-25]。本研究以54对SSR标记对引进的55份大麦材料进行遗传多样性分析,确定各材料在不同位点上所含有的等位变异及基因型,比较了它们的遗传背景差异,为后期进行关联分析发掘引进种质当中的有利等位基因奠定了基础。基于哈迪-温伯格平衡数学模型的遗传结构分析,将55份大麦材料分成了3个亚群,不存在人为因素。因此,在后期关联分析中,群体遗传结构分析的结果可以避免人为划分亚群的干扰,提高关联的准确性[11,26-27],计算得到的Q值作为协变量用于关联分析。通过GLM模型关联分析,在P<0.01水平下,共有16对与株高、穗长、穗下茎长、千粒重和全生育期相关联的标记。关于大麦穗长QTL定位的报道较少,主要位于2H、3H、4H、5H和7H这5条染色体上[28-31]。本研究中检测到的穗长相关联标记Bmac40位于6H染色体的长臂上,可能是新的穗长QTL位点,但需要进一步验证其真实性。穗下茎长的相关QTL还未见报道,本研究共检测到4对相关联标记,对于这些结果的准确性,仍需要后期进一步试验的验证。株高是育种过程中的重要农艺性状,与作物的抗倒伏能力显著相关[32-33]。大麦株高的QTL大量被定位到1H-7H染色体长、短臂上[34-36],说明大麦株高的相关QTL没有集中于某一条染色体或某条染色体的某一端。本研究同时通过关联分析,检测出HVGLUEND、GBMS2、EBmac684和EBmac775等4对标记与株高相关联,分别位于大麦1H、2H和4H染色体上,并且HVGLUEND和GBMS2在P<0.001水平下与株高相关联。由于这4对标记未见与株高相关联的报道,需要进一步验证才能作为后期辅助选择的候选标记。Lacaze等[37]将大麦千粒重QTL定位于大麦1H-7H染色体,其中在2H染色体的52~62.3 cM和6H染色体的102~125.4 cM区间内均有一个显著QTL,本研究中的千粒重相关联标记GBMS2和GMS06分别位于这两个区间附近,说明这一结果较可靠,GBMS2和GMS06可以作为千粒重分子标记辅助选择的候选标记。关于生育期的QTL被定位于1H、3H、4H、5H和6H[38-39]。本研究检测到的生育期相关联标记HV13GEIII和GBM1215分别位于3H和6H染色体,但是与Korff等[38]、Tinker等[39]的结果不在同一区间,是否为新的QTL位点有待进一步研究验证。
[1] TANKSLEY S D,MCCOUCH S R.Seed banks and molecular maps:unlocking genetic potential from the wild [J].Science,1997,277(5329):1063.
[2] FLINT-GARCIA S A,THOMSBERRY J M,BUCKLER I V.Structure of linkage disequilibrium in plants [J].AnnualReviewofPlantBiology,2003,54(4):357.
[3] ZONDERVAN K T,CARDON L R.The complex interplay among factors that influence allelic association [J].NatureReviewsGenetics,2004,5(2):89.
[4] GUPTA P K,RUSTGI S,KULWAL P L.Linkage disequilibrium and association studies in higher plants:present status and future prospects [J].PlantMolecularBiology,2005,57(4):461.
[5] SAGHAI-MAROOF M A,BIYASHEV R M,YANG G P,etal.Extraordinarily polymorphic microsatellite DNA in barley:species diversity,chromosomal locations,and population dynamics [J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica,1994,91(12):5466.
[6] WEBER J L.Informativeness of human(dC-dA)n.(dG-dT)npolymorphism [J].Genomics,1990,7(4):524.
[7] WANG Z,WEBER J L,ZHONG G,etal.Survey of plant short tandem DNA repeats [J].TheoreticalandAppliedGenetics,1994,88(1):1.
[8] 段艳凤,刘 杰,卞春松,等.中国88个马铃薯审定品种SSR指纹图谱构建与遗传多样性分析[J].作物学报,2009,35(8):1451.
DUAN Y F,LIU J,BIAN C S,etal.Construction of fingerprinting and analysis of genetic diversity with SSR markers for eighty-eight approved potato cultivars(SolanumtuberosumL.) in China [J].ActaAgronomicaSinica,2009,35(8):1451.
[9] 匡 猛,杨伟华,许红霞,等.中国棉花主栽品种DNA指纹图谱构建及SSR标记遗传多样性分析[J].中国农业科学,2011,44(1):20.
KUANG M,YANG W H,XU H X,etal.Construction of DNA fingerprinting and analysis of genetic diversity with SSR markers for cotton major cultivars in China [J].ScientiaAgriculturaSinica,2011,44(1):20.
[10] MACCAFERRI M,SANGUINETI M C,ENRICO N,etal.Population structure and long-range linkage disequilibrium in a durum wheat elite collection[J].MolecularBreeding,2005,15(3):271.
[11] FLINT-GARCIA S A,THUILLET A C,YU J M,etal.Maize association population:a high-resolution platform for quantitative trait locus dissection [J].PlantJournal,2005,44(6):1054.
[12] HAMZA S,HAMIDA W B,REBAI A,etal.SSR-based genetic diversity assessment among Tunisian winter barley and relationship with morphological traits [J].Euphytica,2004,135(1):107.
[13] WANG A,YU Z,DING Y.Genetic diversity analysis of wild close relatives of barley from Tibet and the middle east by ISSR and SSR markers [J].ComptesRendusBiologies,2009,332(4):393.
[14] MALYSHEVA-OTTO L,GANAL M W,LAW J R,etal.Temporal trends of genetic diversity in European barley cultivars(HordeumvulgareL.) [J].MolecularBreeding,2007,20(4):309.
[15] KRAAKMAN A T W,MARTNEZ F,MUSSIRALIEV B,etal.Linkage disequilibrium mapping of morphological,resistance,and other agronomically relevant traits in modern spring barley cultivars [J].MolecularBreeding,2006,17(17):41.
[16] SUN D F,REN W B,SUN G L,etal.Molecular diversity and association mapping of quantitative traits in tibetan wild and worldwide originated barley(HordeumvulgareL.) germplasm [J].Euphytica,2011,178(1):31.
[17] IVANDIC V,HACKETT C A,NEVO E,etal.Analysis of simple sequence repeats(SSRs) in wild barley from the fertile crescent:associations with ecology,geography and fowering time [J].PlantMolecularBiology,2002,48(5-6):511.
[18] PATERSONA H,BRUBAKER C L,WENDEL J F.A rapid method for extraction of cotton(Gossypiumspp.)genomic DNA suitable for RFLP or PCR analysis [J].PlantMolecularBiologyReporter,1993,11(2):122.
[19] 赖 勇,孟亚雄,王 晋,等.大麦遗传多样性及连锁不平衡分析[J].作物学报,2013,39(12):2154.
LAI Y,MENG Y X,WANG J,etal.Genetic diversity and linkage disequilibrium analysis in barley [J].ActaAgronomicaSinica,2013,39(12):2154.
[20] EVANNO G,REGNAUT S,GOUDET J.Detecting the number of clusters of individuals using the software STRUCTURE:a simulation study[J].MolecularEcology,2005,14(8):2611.
[21] 张赤红,张 京.大麦品种资源遗传多样性的SSR标记评价[J].麦类作物学报,2008,28(2):214.
ZHANG C H,ZHANG J.Genetic diversity assessment of barley germplasm resources using SSR markers [J].JournalofTriticeaeCrops,2008,28(2):214.
[22] 刘志敏,金 能,吕 超,等.大麦种质资源的SSR遗传多样性分析[J].麦类作物学报,2011,31(5):839.
LIU Z M,JIN N,LÜ C,etal.Genetic diversity analysis of barley varieties by SSR[J].JournalofTriticeaeCrops,2011,31(5):839.
[23] HARRIS B P,STOKESBRY K D E.The spatial structure of local surficial sediment characteristics on Georges Bank,USA[J].ContinentalShelfResearch,2010,30(17):1840.
[24] WANG M L,ZHU C S,BARKLEY N A,etal.Genetic diversity and population structure analysis of accessions in the US historic sweet sorghum collection[J].TheoreticalandAppliedGenetics,2009,120(1):13.
[25] KLINE J B,MOORE D J,CLEVENGER C V.Activation and association of the Tec tyrosine kinase with the human prolactin receptor:Mapping of a Tec/Vav-receptor binding site [J].MolecularEndocrinology,2001,15(5):832.
[26] ZHANG Z W,ERSOZ E,LAI C Q,etal.Mixed linear model approach adapted for genome-wide association studies[J].NatureGenetics,2010,42(4):355.
[27] PRICEA L,ZAITLEN N A,REICH D,etal.New approaches to population stratification in genome-wide association studies [J].NatureReviewsGenetics,2010,11(7):459.
[28] BAGHIZADEH A,TALEEI A R,NAGHAVI M R.QTL analysis for some agronomic traits in barley(HordumvulgareL.) [J].InternationalJournalofAgricultrueandBiology,2007,9(2):372.
[29] HORI K,KOBAYASHI T,SHIMIZU A,etal.Effcient construction of a high-density linkage map and its application to QTL analysis in barley [J].TheoreticalandAppliedGenetics,2003,107(107):806.
[30] SAMERI M,TAKEDA K,KOMATSUDA T.Quantitative trait loci controlling agronomic traits in recombinant inbred lines from a cross between oriental- and occidental-type barley cultivars [J].BreedingScience,2006,56(3):243.
[31] WANG J,YANG J,MCNEIL D L,etal.Identifcation and molecular mapping of a dwarfing gene in barley(HordeumvulgareL.) [J].Euphytica,2010,175(3):331.
[32] 章忠贵,刘斌美,许 学,等.水稻株高突变系的农艺性状与抗倒伏研究[J].核农学报,2010,24(3):430.
ZHANG Z G,LIU B M,XU X,etal.Agronomic characters and lodging resistance of plant height mutants of rice [J].ActaAgriculturaeSinica,2010,24(3):430.
[33] 肖应辉,罗丽华,闫晓燕,等.水稻品种倒伏指数QTL分析[J].作物学报,2005,31(3):348.
XIAO Y H,LUO L H,YAN X Y,etal.Quantitative trait locus analysis of lodging index in rice(OryzasativaL.) [J].ActaAgronomicaSinica,2005,31(3):348.
[34] MARQUEZ-CEDILLO L A,HAYES P M,KLEINHOFS A,etal.QTL analysis of agronomic traits in barley based on the doubled haploid progeny of two elite North American varieties representing different germplasm groups [J].TheoreticalandAppliedGenetics,2001,103(4):625.
[35] TEULAT B,BORRIES C,THIS D.New QTLs identified for plant water status,water-soluble carbohydrate and osmotic adjustment in a barley population grown in a growth-chamber under two water regimes [J].TheoreticalandAppliedGenetics,2001,103(1):161.
[36] KORFF M,WANG H,LEON J,etal.AB-QTL analysis in spring barley:II.Detection of favourable exotic alleles for agronomic traits introgressed from wild barley(H.vulgaressp.spontaneum) [J].TheoreticalandAppliedGenetics,2006,112(7):1221.
[37] LACAZE X,HAYES P M,KOROL A.Genetics of phenotypic plasticity:QTL analysis in barley,Hordeumvulgare[J].Heredity,2009,102(2):168.
[38] KORFF M,GRANDO S,DEL G A,etal.Quantitative trait loci associated with adaptation to mediterranean dryland conditions in barley[J].TheoreticalandAppliedGenetics,2008,117(5):653.
[39] TINKER N A,MATHER D E,ROSSNAGEL B G,etal.Regions of the genome that affect agronomic performance in two-row barley [J].CropScience,1996,36(4):1053.
Analysis of Genetic Diversity and Association with Agronomic Traits in Barley (HordeumvulgareL.)Introduced from Abroad Using SSR Markers
LAI Yong1,JIA Jianlei1,WANG Jinming1,REN Long2,LÜ Zhongyu4,ZHU Huiqin1,MA Hui1,YANG Lina1,LI Zongren1
(1.Agriculture and Animal Husbandry Colleague of Qinghai University,Xining,Qinghai 810016,China; 2.State Key Laboratory of Plateau Ecology and Agriculture,College of Agriculture and Animal Husbandry,Qinghai University,Xining,Qinghai 810016,China; 3.Modern Education Technology Centre of Qinghai University,Xining,Qinghai 810016,China; 4.Gansu Longyuan Research Institute of Agricultural Science,Lanzhou,Gansu 730070,China)
In order to understand the genetic information of introduced germplasm resources,55 barley accessions from abroad were introduced and genetic diversity and association with agronomic trait was analyzed by SSR markers. A total of 154 alleles were detected from these accessions. There were 164 genotypes among these accessions based on 54 SSR markers. The gene diversity ranged from 0.103 to 0.708,and the PIC ranged from 0.098 to 0.651.The genetic similarity(GS) among these accessions was from 0.533 to 0.940,with the average of 0.722. The accessions were divided into three subgroups at the GS level of 0.682,in which there were 49,2 and 4 materials,respectively. Based on the analysis of population structure,55 accessions were composed of three subpopulations,in which there were 9,24 and 22 materials,respectively. There were 16 markers associated with plant height,spike length,length of ear stem,thousand kernel weight,and days to maturity,based on the general line model(GLM). The rate of explanation on the phenotype with marker ranged from 0.063 to 0.331. And five markers were associated with more than one trait.
Barley; SSR markers; Genetic diversity; Association analysis
时间:2017-01-16
2016-06-23
2016-01-05
国家自然科学基金地区基金项目(31660429)
E-mail:xlaiyong@163.com
李宗仁(E-mail:lizongrenqd@126.com)
S512.3;S330
A
1009-1041(2017)02-0197-08
网络出版地址:http://www.cnki.net/kcms/detail/61.1359.S.20170116.1833.014.html
