小麦LEAsm基因及其启动子的克隆与功能研究
2017-03-01李孟军李亚青张士昌史占良
李孟军,李亚青,张士昌,张 楠,史占良
(石家庄市农林科学研究院,河北石家庄 050041)
小麦LEAsm基因及其启动子的克隆与功能研究
李孟军,李亚青,张士昌,张 楠,史占良
(石家庄市农林科学研究院,河北石家庄 050041)
胚胎发育晚期丰富蛋白(LEA)是一个小的高亲水性的蛋白家族,该蛋白家族在逆境胁迫下大量积累,保护植物免受逆境胁迫。LEA蛋白可分为7组,其中重复的11-氨基酸基序是第3组 LEA蛋白的特征。为深入分析第3组LEA蛋白在小麦响应逆境胁迫中的作用机制,利用芯片技术从小麦表达谱中筛选出一个渗透胁迫诱导表达的第3组LEA蛋白基因TaLEAsm,然后根据该基因序列设计引物筛选石麦15的BAC文库,获得1个含有该基因的BAC单克隆,以该BAC单克隆质粒为模板,通过BAC延伸测序克隆了TaLEAsm基因及其启动子序列,并对TaLEAsm序列特征、表达模式和启动子功能进行了初步分析。结果表明,TaLEAsm基因序列仅含有1个105 bp的内含子,其开放读码框长675 bp,编码224个氨基酸。TaLEAsm含有10个11-氨基酸重复序列,属于第3组 LEA蛋白。低温、高盐和渗透胁迫均诱导TaLEAsm基因上调表达,但在根和叶中表达模式不同。在TaLEAsm基因起始密码子上游1 500 bp序列中,预测含有14个逆境响应顺式元件。在拟南芥中,TaLEAsm基因启动子能够启动GUS基因表达,渗透胁迫诱导GUS基因明显上调表达。以上结果表明,TaLEAsm为小麦脱水响应基因,其启动子为渗透胁迫诱导启动子。
小麦;TaLEAsm;启动子;克隆;功能研究
胚胎发育晚期丰富蛋白(LEA)是生物体内与渗透调节有关的蛋白家族,在植物胚胎发育晚期大量积累,响应多种非生物逆境胁迫,如干旱、盐害和低温等[1]。Dure等[2]首次在棉花子叶中发现LEA蛋白。根据氨基酸序列保守结构域和亲水性,植物LEA蛋白可分为7组,除第5组LEA蛋白具有疏水特征外,其余6组均为亲水蛋白[3]。第3组LEA蛋白通常含有多拷贝的由11个氨基酸组成的基序 (TAQAAKEKAGE),其分子量差异通常是由于11个氨基酸基序的拷贝数造成的[4]。研究表明,主要农作物第3组LEA蛋白的11-氨基酸基序以多拷贝串联形式存在,其长度占蛋白序列长度的40%~60%,带电荷/极性氨基酸和疏水氨基酸数目分别占重复序列氨基酸总数的71%和29%[5]。
植物第3组LEA蛋白的积累与多种非生物逆境之间(低温、干旱、盐害等)存在很高的相关性。小麦第3组LEA-L2蛋白的组成型表达能够显著提高拟南芥的抗冻害能力[6]。转大麦 HVA1的桑树(Morusindica)和水稻对干旱和高盐的耐受性明显提高[7-8]。干旱和高盐诱导水稻幼苗中 OsLEA19a上调表达,其编码蛋白可能参与水稻对干旱和高盐的抗性反应[9]。干旱、低温胁迫、盐胁迫和外源ABA处理能够提高小麦 TaLEAL3的表达量,表明其可能参与了小麦逆境胁迫响应[10]。外源ABA、干旱、低温、高盐和高温等均诱导腊梅 CpLEA的上调表达[11]。在烟草中过量表达 ZmLEA3能够提高烟草的抗渗透胁迫和氧化胁迫的能力[12]。在玉米茎中 ZmLEA3的表达受到低温、渗透胁迫、外源ABA和H2O2诱导后显著提高[13]。
逆境胁迫诱导表达启动子使抗逆基因在特定时间和空间表达,提高植物抗逆性的同时,可减少抗逆基因组成型表达给植物带来的负面效应[14]。与CaMV35S启动子相比较,诱导性启动子RD29A启动 TaEXPB23在烟草中表达,转基因烟草对水分胁迫耐受性提高,同时降低了 TaEXPB23对烟草的负面影响[14]。拟南芥 CBF4上游序列CBF4P在干旱胁迫条件下转录活性高于CaMV35S启动子[15]。水杨酸和低温处理可诱导葡萄(Vitispseudoreticulata)VpSTS启动子驱动GUS基因表达,起始密码子上游162 bp对于该启动子的组成型表达和诱导表达模式必不可少[16]。干旱胁迫诱导水稻 Oshox24的启动子强烈上调表达,同时该启动子还受到脱落酸的诱导[17]。 OsDhn1启动子受到干旱诱导,但不受伤害、冷害、盐害和外源ABA处理等诱导[18]。高盐胁迫使拟南芥 AtPUB18启动子驱动的GUS基因表达量显著升高[19]。逆境胁迫诱导基因 ZmRXO1上游1 576 bp启动子序列可驱动GUS基因在烟草中高水平表达,其中582 bp(-582 to -1)的序列能够在激素(MeJA、GA、ABA)、干旱、低温诱导下驱动GUS基因表达[20]。
LEA 蛋白基因在非生物逆境中的作用已在多种作物中得到证实[6-13],小麦第3组LEA蛋白基因,特别是其启动子的克隆及其功能分析,不仅有利于对其作用机制的深入探讨,而且也为利用第3组LEA 蛋白基因提高作物抗逆性奠定基础。本研究利用Affymetrix小麦表达谱芯片分析石麦15渗透胁迫的诱导表达谱,发现1个第3组LEA蛋白基因TaLEAsm受渗透胁迫诱导高丰度表达。通过PCR筛选石麦15基因组BAC文库,结合BAC克隆测序了TaLEAsm基因及其启动子,并对TaLEAsm序列特征、表达模式和启动子功能等进行了初步分析,以期为深入研究和利用TaLEAsm进行小麦抗逆遗传改良奠定基础。
1 材料与方法
1.1 材 料
供试材料为冬小麦品种石麦15,由石家庄市农林科学研究院小麦研究所提供。挑选大小基本一致的饱满种子在光照培养箱中用自来水培养至二叶一心期,改用1/2 Hoagland培养液继续培养7 d,培养温度为22±1 ℃,每天光照14 h。选取生长发育一致的幼苗20~25株置培养皿中,分别进行16% PEG6000(1/2 Hoagland配制)、200 mmol·L-1NaCl(1/2 Hoagland配制)和4 ℃(春化箱)处理,并于各处理的0、1、3、6、9、12、24和48 h取根和叶片,取样期间保持全天光照,所取样品液氮速冻后于-70 ℃冰箱保存。试验设置3个重复。
石麦15基因组BAC文库由1 000 000个以上克隆组成,保存在1 020个混合池中,克隆插入片段平均长度为85 kb,空载率为2.5%,保存于石家庄市农林科学研究院生物技术实验室。
1.2 石麦15的BAC文库筛选
根据小麦TaLEAsmcDNA序列(GenBank:AY148490)设计TaLEAsmF1/TaLEAsmR1(TaLEAsmF1:5′-GGTCGTGTTCCAAGAAA CC-3′;TaLEAsmR1:5′-AGCAAAGTAAATTA AGCGCC-3′)和TaLEAsmF2/TaLEAsmR2(TaLEAsmF2:5′-GTTCCAAGAAACCAAAA TGG-3′;TaLEAsmR2:5′-ACATGCGTCTAGT GATTCCTG-3′)两对嵌套引物对石麦15 BAC文库进行筛选。 PCR 扩增程序:94 ℃ 3 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,30个循环。TaqDNA聚合酶为2×TaqPCR StarMix(GenStar)。
1.3TaLEAsm基因gDNA、cDNA及启动子序列的获得
以含有TaLEAsmBAC单克隆质粒为模板,根据BAC单克隆质粒上已知TaLEAsm序列设计测序引物,用BigDye○RTerminator v3.1 Cycle Sequencing Kit(ABI)进行测序PCR反应,用ABI 3730 XL DNA Analyzer进行测序。采用primer walking方法进行步移,直至获得基因完整序列及其启动子序列。用RNAiso Plus(TaKaRa) 提取根和叶片总RNA。用polyATract○RmRNA isolation system III (Promega) 分离mRNA。用PrimerScriptTM1st Strand cDNA Synthesis Kit (TaKaRa)合成第一链cDNA。PCR扩增引物为TaLEAsmF1/TaLEAsmR1。PCR产物克隆使用DNA A-Tailing Kit和pMD18-T vector (TaKaRa)。
1.4TaLEAsm基因的表达模式分析
实时定量PCR反应采用SYBR○RPremix ExTaqTMII(TaKaRa)。上游引物:5′-CACCAG GAATCACTAGACGCAC-3′;下游引物:5′-CAAATGAGCTGAAATCACGAAAG-3′。PCR扩增程序:95 ℃ 2 min;95 ℃ 30 s,59 ℃ 30 s,72 ℃ 30 s,40个循环。以小麦β-actin基因为内参,反应在ABI Prism7500荧光定量PCR仪上运行,3次重复。采用2-ΔΔCt法进行定量分析。
1.5TaLEAsm基因启动子表达载体的构建与拟南芥的转化
根据序列比对结果确定TaLEAsm基因启动子序列,在此基础上设计引物TaLEAsm-proF/TaLEAsm-proR(TaLEAsm-proF:5′-CGGG ATCCGTGCGTGTCGGAAGAACTC-3′,下划线部分为BamHI酶切位点;TaLEAsm-proR:5′-CCAAGCTTTGGAACACGACCTGGAATC-3′,下划线部分为HindIII酶切位点),扩增区域为起始密码子ATG上游从11 bp到1 647 bp。TaLEAsm基因启动子表达载体构建使用pCAMBIA1831Z载体。20 μL PCR反应体系包含2×Pfu PCR MasterMix 10 μL(TIANGEN)、上下游引物(10 μmol·L-1)各1 μL、模板质粒DNA (20 ng·L-1)2 μL及ddH2O 6 μL。PCR 扩增程序:94 ℃预变性5 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸4 min,30个循环;72 ℃延伸8 min。酶切片段连接使用T4 DNA ligase(NEB)。转化菌株分别使用大肠杆菌DH5α和农杆菌GV3101。通过BamHⅠ和HindⅢ酶切和测序对构建的启动子表达载体进行验证。以拟南芥生态型Columbia-0为受体,采用花序浸染法进行转化[21]。转基因拟南芥鉴定采用PCR和GUS染色的方法,经过连续培养,启动子功能验证采用T3代纯合体进行。
1.6 GUS组织化学染色
转基因拟南芥苗(T3代)在抗性平板上生长10 d后,选取生长一致的幼苗,分别于1/2MS培养基、16% PEG8000(1/2MS培养基配制)、400 mmol·L-1Mannitol(1/2MS培养基配制)在培养室中处理12 h(22 ℃,16 h光照;16 ℃,8 h黑暗)。用不含X-Gluc的GUS染色液洗涤转基因拟南芥植株后,转移到Eppendorf管中,加入适量GUS染色液浸没全部植株,37 ℃保温8 h,然后去除染色液,用95%乙醇37 ℃脱色2~3 h,组织染色完毕后保存于75%乙醇中,4 ℃保存。使用变倍体视显微镜(Nikon SMZ1000)观察,并用数码相机(Nikon DXM1200F)拍照记录。
2 结果与分析
2.1TaLEAsm引物特异性分析
Affymetrix小麦表达谱芯片分析表明,小麦cDNA序列(GenBank No. AY148490)渗透胁迫后上调表达,其中叶片表达量在渗透胁迫后3 h、9 h和27 h分别为对照的138.3倍、71.3倍和45.6倍,根系分别为对照的42.4倍、28.5倍和45.8倍。NCBI Blastx比对显示,该基因为第3组LEA蛋白编码基因,命名为TaLEAsm。根据TaLEAsmcDNA序列设计嵌套引物,分别以石麦15基因组DNA和cDNA为模板,使用高保真酶进行PCR和RT-PCR扩增,分别随机挑取6个克隆测序。测序结果表明,PCR产物仅含有TaLEAsm,嵌套引物均为TaLEAsm特异性引物(图1)。
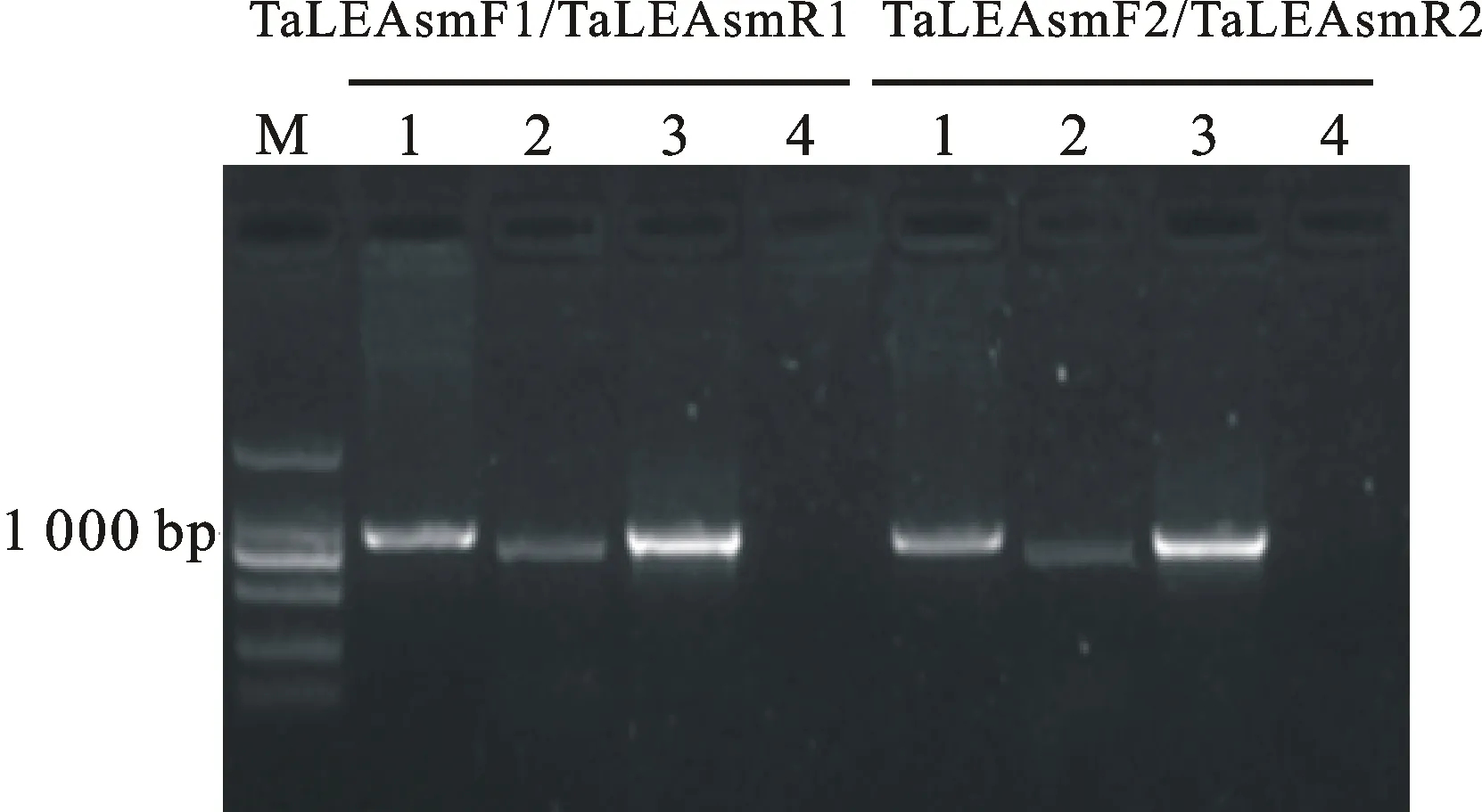
M:DL2000;1:石麦15的DNA;2:石麦15的cDNA;3:BAC质粒;4:ddH2O。
M:DL2000; 1:DNA from Shimai 15; 2:cDNA from Shimai 15; 3:BAC plasmid; 4:ddH2O.
图1TaLEAsm基因PCR引物特异性检测
Fig.1 Specific detection of PCR primers forTaLEAsmgene
2.2TaLEAsm基因gDNA和cDNA序列扩增结果
以石麦15基因组BAC文库混合池质粒为模板,采用引物TaLEAsmF1/TaLEAsmR1,利用PCR技术对文库进行筛选,获得阳性混合池4个(pool 269、pool 479、pool 588和pool 721)。PCR产物测序表明,4个混合池中TaLEAsm基因序列一致。通过对混合池pool 269进一步筛选,分离出含有TaLEAsm的BAC单克隆。脉冲场凝胶电泳显示,该克隆插入片段75 kb左右。
根据TaLEAsmcDNA序列设计测序引物,以含有该序列的BAC单克隆质粒为模板,通过5次测序反应,经序列组装,获得1条长为3 846 bp 的DNA序列。NCBI blastx分析表明,该序列中含有完整的TaLEAsm。TaLEAsm基因cDNA序列和gDNA序列分别长为843 bp和948 bp,该基因仅含有1个105 bp的内含子(图2)。TaLEAsm读码框长为675 bp,编码一个含有224个氨基酸的蛋白,分子量为23.2 kD。TaLEAsm具有10个重复的11-氨基酸基序(TAQAAKEKAGE),这些重复的基序以串联重复形式分3组分布在TaLEAsm序列中,长度占蛋白质全序列的54%[4](图3)。序列比对表明,单子叶植物第3组LEA基因在11-氨基酸基序重复区域保守性低,11-氨基酸基序数目的不同导致该区域序列长度的变化(图4)。

图2 TaLEAsm基因结构
2.3TaLEAsm基因的表达模式分析
分析TaLEAsm基因在低温、高盐、PEG胁迫处理下的小麦幼苗根和叶中的表达,发现其受这些胁迫诱导表达,但表达模式有所不同(图5)。在高盐条件下,根中TaLEAsm基因表达量明显高于叶中,二者相差3倍以上;而在PEG渗透胁迫条件下,叶中TaLEAsm基因表达量高于根中,二者相差也在3倍以上。低温、高盐、PEG胁迫处理下,叶中TaLEAsm基因在24 h时表达量达到峰值;而根中TaLEAsm基因在12 h时表达量达到峰值。说明TaLEAsm基因为低温、高盐和PEG诱导表达基因;在高盐和PEG胁迫下,TaLEAsm基因在根和叶中可能存在不同的调控机制。
2.4TaLEAsm基因启动子的克隆与序列分析
TaLEAsm基因转录起始点为A,位于起始密码子上游23 bp处,符合转录起始点的一般特点。通过延伸测序获得TaLEAsm基因起始密码子ATG上游5′侧翼序列2 290 bp。采用PlantCARE在线预测TaLEAsm基因ATG上游1 500 bp序列的顺式作用元件,该启动子序列中含有14个与逆境响应相关的顺式作用元件,包括ABRE(5)、CE3(2个)、CGTCA-motif(2个)、HSE(2个)、TGACG-motif(2个)和motif IIb(1个)(表1),说明TaLEAsm基因可能与小麦逆境胁迫反应有关。
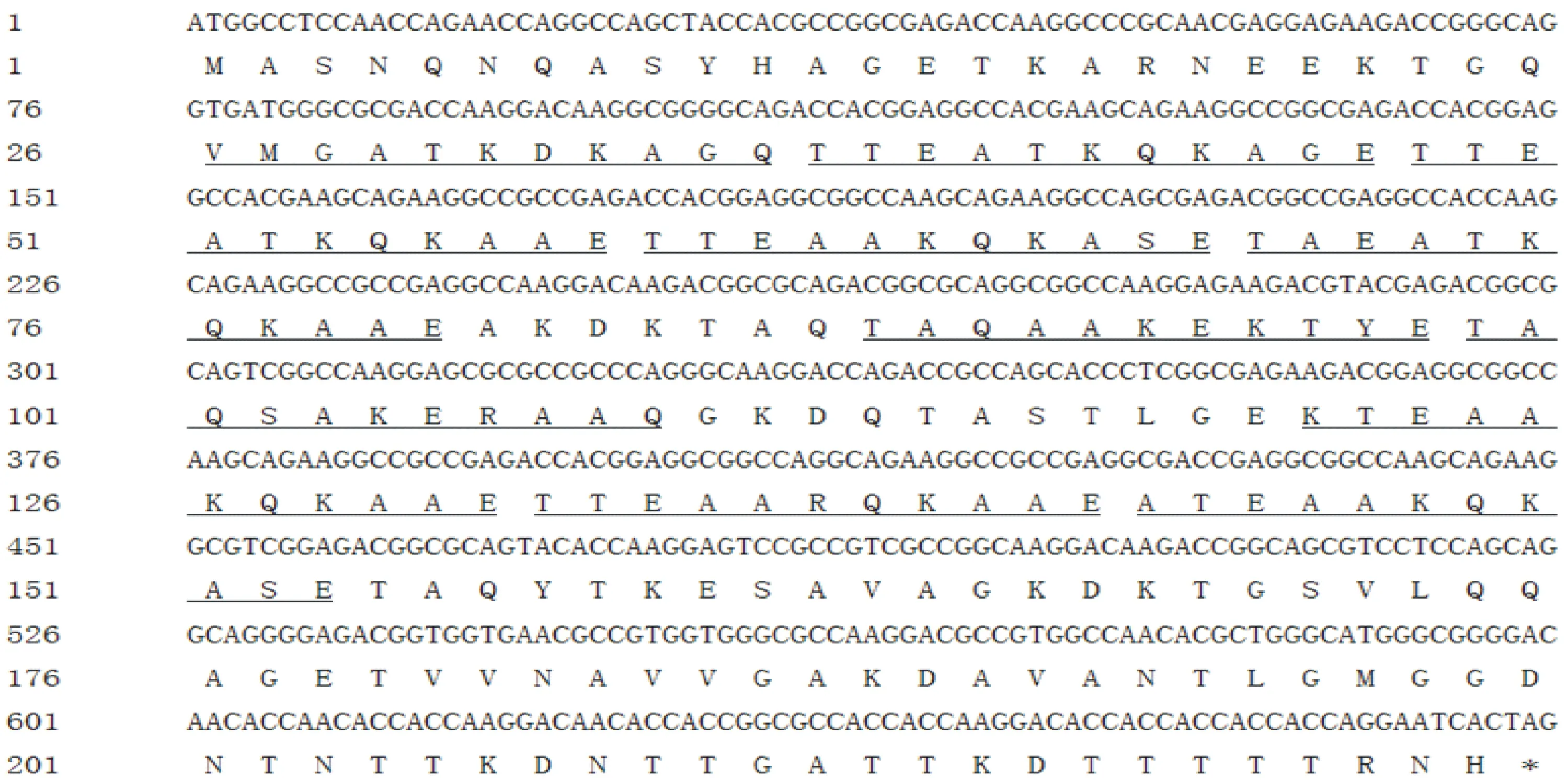
11-氨基酸基序用细线标出。 The motif of eleven amino acids is marked by thin line.

图4 单子叶植物第3组LEA蛋白的序列比对
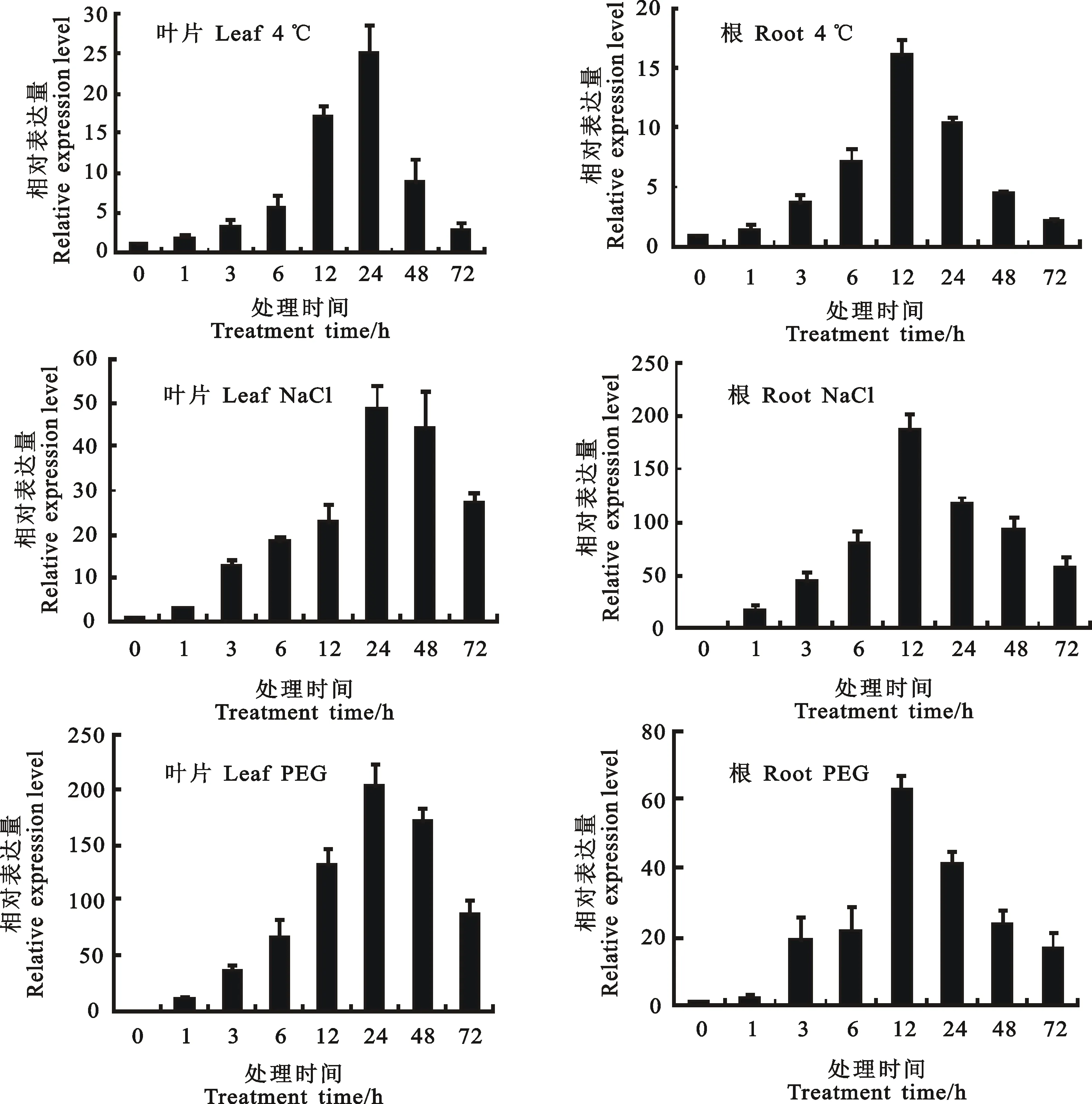
图5 TaLEAsm基因在不同胁迫处理下的表达模式
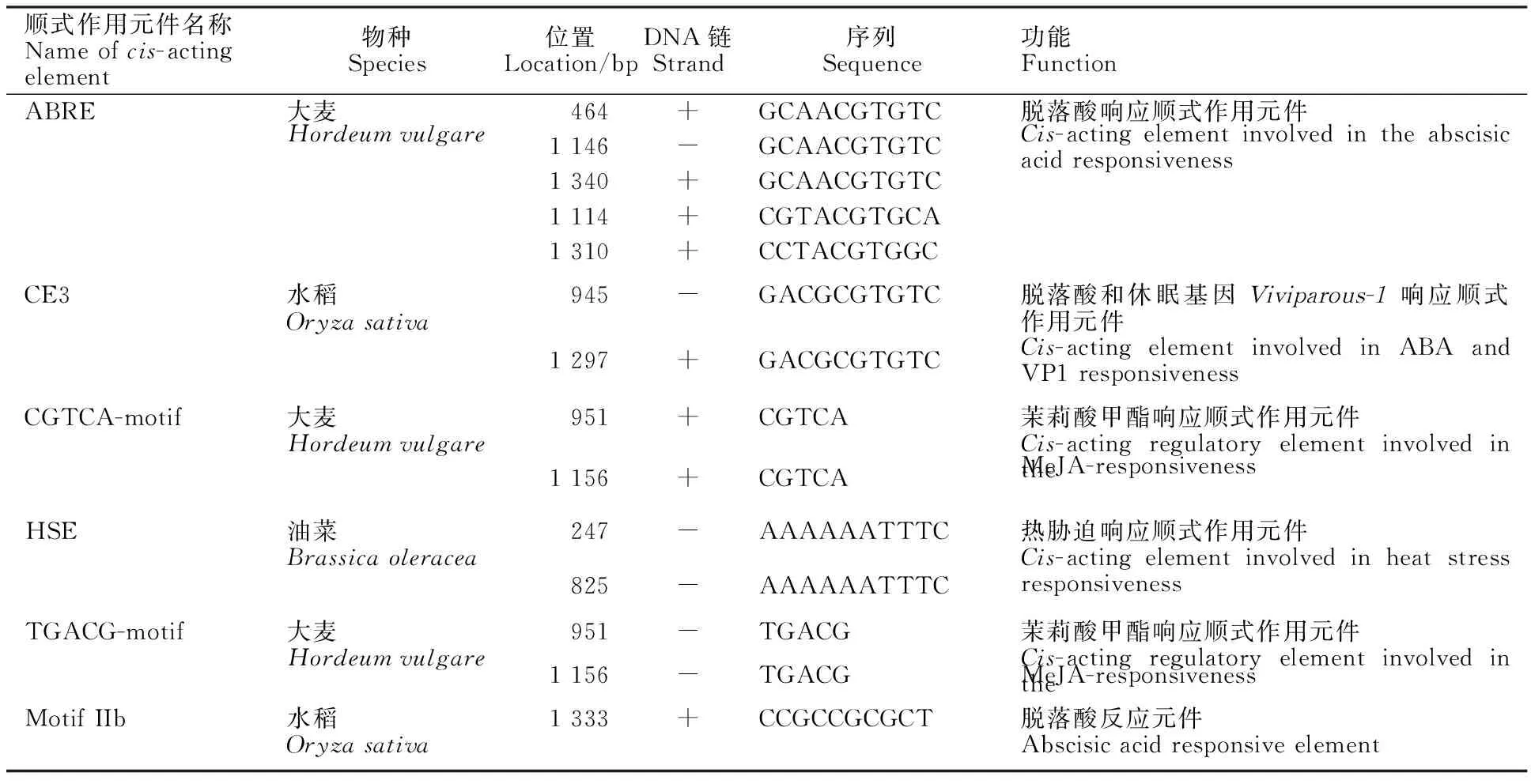
顺式作用元件名称Nameofcis-actingelement物种Species位置Location/bpDNA链Strand序列Sequence功能FunctionABRE大麦464+GCAACGTGTC脱落酸响应顺式作用元件Hordeumvulgare1146-GCAACGTGTCCis-actingelementinvolvedintheabscisicacidresponsiveness1340+GCAACGTGTC1114+CGTACGTGCA1310+CCTACGTGGCCE3水稻Oryzasativa945-GACGCGTGTC脱落酸和休眠基因Viviparous-1响应顺式作用元件1297+GACGCGTGTCCis-actingelementinvolvedinABAandVP1responsivenessCGTCA-motif大麦Hordeumvulgare951+CGTCA茉莉酸甲酯响应顺式作用元件Cis-actingregulatoryelementinvolvedinthe1156+CGTCAMeJA-responsivenessHSE油菜Brassicaoleracea247-AAAAAATTTC热胁迫响应顺式作用元件Cis-actingelementinvolvedinheatstressresponsiveness825-AAAAAATTTCTGACG-motif大麦Hordeumvulgare951-TGACG茉莉酸甲酯响应顺式作用元件Cis-actingregulatoryelementinvolvedinthe1156-TGACGMeJA-responsivenessMotifIIb水稻Oryzasativa1333+CCGCCGCGCT脱落酸反应元件Abscisicacidresponsiveelement
2.5TaLEAsm基因启动子的功能分析
以含有TaLEAsm基因的BAC质粒为模板,经PCR扩增获得了TaLEAsm起始密码子ATG上游5′侧翼序列1 637 bp,构建了启动子表达载体(TaLEAsmPro::GUS)。采用花序转化法转化拟南芥,选取T3代纯合体进行GUS表达分析。1/2MS培养基、16% PEG6000和400 mmol·L-1Mannitol处理的拟南芥幼苗在子叶中均能观察到显示GUS活性的蓝色。与在1/2MS培养基生长的拟南芥幼苗相比,16% PEG6000和400 mmol·L-1Mannitol处理均导致拟南芥幼苗子叶的蓝色明显加深(图6)。GUS染色结果表明,TaLEAsm基因启动子存在组成型表达,渗透胁迫处理能够明显提高其表达水平,因此,TaLAEsm启动子为渗透胁迫诱导型启动子。

图6 TaLEAsmPro::GUS转基因拟南芥GUS染色
3 讨 论
植物在生长发育过程中,面临着多种非生物逆境胁迫,在长期进化过程中,植物形成了一系列的抵御机制,从而减轻或消除各种逆境胁迫对其生长发育的影响。已有研究表明,逆境胁迫诱导植物第3组LEA蛋白基因上调表达,转第3组LEA蛋白基因能够提高植物的抗逆性[6-13,22]。低温、高盐和干旱等非生物逆境胁迫均诱导小麦第3组LEA蛋白基因 TaLEAL3、 TaDRLea3-2和 TaLEAsm的上调表达,但其诱导表达谱呈现不同特征[10,23],这些结果表明,第3组LEA蛋白基因可能通过不同机制参与小麦逆境胁迫响应。
第3组LEA蛋白的11-氨基酸基序可形成兼性α-螺旋,其亲水表面与过量的离子“螯合”,从而减轻胞质中高浓度离子对细胞的毒害。其疏水面可结合膜结构或蛋白,从而维持膜结构稳定和阻止蛋白聚集变性[4]。作物第3组LEA蛋白的11-氨基酸基序的拷贝数相差较大,少则5个(棉花D-7),多则27个(胡萝卜DC8)[5]。小麦TaLEAsm属于典型的第3组LEA蛋白基因,编码蛋白含有10个11-氨基酸基序,这些基序以串联重复形式分3组分布在TaLEAsm序列中,占蛋白长度的一半以上。第3组LEA蛋白11-氨基酸基序与植物抗逆性有关,但其拷贝数与植物抗逆性强弱之间是否相关缺乏试验证据,尚待深入研究。
TaLEAsm和 TaLEAL3启动子含有的逆境响应相关的顺式作用元件不同,前者为ABRE、CE3等,而后者为ABRE、DRE、DRE/CRT 和DPBF 等,这表明TaLEAsm和 TaLEAL3可能通过依赖ABA的信号转导途径参与小麦逆境胁迫响应,同时也为二者对不同逆境的响应差异提供了佐证。
第3组LEA蛋白参与植物多种非生物逆境胁迫响应,随着对其研究的不断深入,第3组LEA蛋白有望在作物抗逆分子育种中发挥更为重要的作用。TaLEAsm及其启动子的克隆为小麦抗逆转基因育种提供了新的选择[15-20,24]。TaLEAsm对低温、盐害和干旱胁迫响应强烈,其启动子为干旱强诱导型启动子,暗示该基因能够提高小麦的非生物逆境抗性,但其逆境响应机制尚需深入探讨。
[1] 杨 杞,尹佳佳,王 颖,等.柠条锦鸡儿 CkLEA1基因克隆及表达分析[J].中国生物工程杂志,2013,33(5):93.
YANG Q,YIN J J,WANG Y,etal.Cloning and expression analysis of CkLEA1 gene inCaraganakorshinskiiKom [J].ChinaBiotechnology,2013,33(5):93.
[2]DURE L,CHLAN C.Developmental biochemistry of cottonseed embryogenesis and germination:XII.Purification and properties of principal storage proteins [J].PlantPhysiology,1981,68(1):180.
[3]XIONG L,ZHU J K.Molecular and genetic aspects of plant responses to osmotic stress [J].Plant,Cell&Environment,2002,25(2):131.
[4]DURE L.A repeating 11-mer amino acid motif and plant desiccation [J].PlantJournal,1993,3:363.
[5] 孙海丹,兰 英,刘 昀,等.LEA蛋白质11-氨基酸基序与植物抗旱性[J] .东北师范大学学报(自然科学版),2004,36(3):85.
SUN H D,LAN Y,LIU Y,etal.11-amino acid motif in late embryogenesis abundant protein (LEA) and plant desiccation tolerance [J].JournalofNortheastNormalUniversity(NaturalScienceEdition),2004,36(3):85.
[6]NDONG C,DANYLUK J,WILSON K E,etal.Cold-regulated cereal chloroplast late embryogenesis abundant-like proteins:molecular characterization and functional analyses [J].PlantPhysiology,2002,129:1368.
[7]LAL S,GULYANI V,KHURANA P.Overexpression of HVA1 gene from barley generates tolerance to salinity and water stress in transgenic mulberry (Morusindica) [J].TransgenicResearch,2008,17:651.
[8]XU D,DUAN X,WANG B,etal.Expression of a late embryogenesis abundant protein gene, HVA1,from barley confers tolerance to water deficit and salt stress in transgenic rice [J].Plantphysiology,1996,110:249.
[9] 胡廷章,吴应梅,陈再刚,等.水稻 OsLEA19a基因的克隆、表达及生物信息学分析[J].华北农学报,2011,26(6):73.
HU T Z,WU Y M,CHEN Z G,etal.Molecular cloning,expression character and bioinformatics analysis of OsLEA19a from rice [J].ActaAgriculturaeBoreali-Sinica,2011,26(6):73.
[10] 闵东红,赵 月,陈 阳,等.小麦胁迫相关基因 TaLEAL3的克隆及分子特性分析[J].作物学报,2012,38(10):1847.
MIN D H,ZHAO Y,CHEN Y,etal.Isolation and molecular characterization of stress-related TaLEAL3 gene in wheat [J].ActaAgronomicaSinica,2012,38(10):1847.
[11] 马 婧,孙文婷,王 晶,等.蜡梅胚胎晚期丰富蛋白基因CpLEA的克隆及表达分析[J].园艺学报,2014,41(8):1663.
MA J,SUN W T,WANG J,etal.Cloning and expression analysis of a late embryogenesis abundant protein geneCpLEAfromChimonanthuspraecox[J].ActaHorticulturaeSinica,2014,41(8):1663.
[12]LIU Y,WANG L,XING X,etal. ZmLEA3,a multifunctional group 3 LEA protein from maize (ZeamaysL.),is involved in biotic and abiotic stresses [J].PlantandCellPhysiology,2013,54:944.
[13]LIU Y,LIANG J,SUN L,etal.Group 3 LEA protein, ZmLEA3,is involved in protection from low temperature stress [J].FrontiersinPlantScience,2016,7:1011.
[14]LI F,HAN Y Y,FENG Y,etal.Expression of wheat expansin driven by the RD29 promoter in tobacco confers water-stress tolerance without impacting growth and development [J].JournalofBiotechnology,2013,163(3):281.
[15] 霍秀文,米福贵,云锦凤,等.转录因子CBF4诱导型启动子的克隆及功能分析[J].分子植物育种,2005,3(3):363.
HUO X W,MI F G,YUN J F,etal.Cloning and functional analysis of an induced promoter of transcriptional factor CBF4 fromArabidopsis[J].MolecularPlantBreeding,2005,3(3):363.
[16]XU W,YU Y,DING J,etal.Characterization of a novel stilbene synthase promoter involved in pathogen- and stress-inducible expression from Chinese wildVitispseudoreticulata[J].Planta,2010,231(2):475.
[17] 杨 梅,熊立仲.水稻干旱诱导型启动子Oshox24P的分离与鉴定[J].华中农业大学学报,2011,30(5):525.
YANG M,XIONG L Z.Isolation and characterization of a drought-inducible promoter Oshox24P in rice [J].JournalofHuazhongAgriculturalUniversity,2011,30(5):525.
[18]LEE S C,KIM S H,KIM S R.Drought inducible OsDhn1 promoter is activated by OsDREB1A and OsDREB1D [J].JournalofPlantBiology,2013,56(2):115.
[19] 张新宇,赵兰杰,李艳军,等.盐胁迫对拟南芥 AtPUB18基因的诱导表达及其启动子分析[J].西北植物学报,2014,34(1):54.
ZHANG X Y,ZHAO L J,LI Y J,etal.Expression of AtPUB18 after salt stress treatment and analysis of its promoter fromArabidopsisthaliana[J].ActaBotanicaBoreali-OccidentaliaSinica,2014,34(1):54.
[20]TAO Y,WANG F T,JIA D M,etal.Cloning and functional analysis of the promoter of a stress-inducible gene( ZmRXO1) in maize [J].PlantMolecularBiologyReporter,2014,33(2):1.
[21]CLOUGH S I,STEVEN I,BENT A F.Floral dip:a simplified method forAgrobacterium-mediated transformation ofArabidopsisthaliana[J].PlantJournal,1998,16(6):735.
[22]VASEVA I I,GRIGOROVA B S,SIMOVA-STOILOVA L P,etal.Abscisic acid and late embryogenesis abundant protein profile changes in winter wheat under progressive drought stress [J].PlantBiology,2010,12:698.
[23] 梁雅珺,于正阳,强智全,等.小麦 TaDRLea3-2基因的克隆及胁迫诱导表达分析[J].西北植物学报,2016,36(6):1091.
LIANG Y J,YU Z Y,QIANG Z Q,etal.Cloning of a new gene TaDRLea3-2 from wheat and its expression under different stresses treatments [J].ActaBotanyBoreal-OccidentSinica,2016,36(6):1091.
[24] 李孟军,李亚青,张 楠,等.小麦 TaDHN2基因及其启动子的克隆与功能研究[J].麦类作物学报,2015,35(8):1031.
LI M J,LI Y Q,ZHANG N,etal.Cloning and functional analysis of coding and promoter regions of TaDHN2 gene in wheat [J].JournalofTriticeaeCrops,2015,35(8):1031.
Isolation and Functional Analysis of Coding and Promoter Regions ofLEAsmGene in Wheat
LI Mengjun,LI Yaqing,ZHANG Shichang,ZHANG Nan,SHI Zhanliang
(Shijiazhuang Academy of Agriculture and Forestry Sciences,Shijiazhuang,Hebei 050041,China)
Late embryogenesis abundant (LEA) proteins are a family of small highly hydrophilic proteins that accumulate markedly in plants under stress conditions and protect plants from the damage caused by stress. LEA proteins can be classified into seven groups,in which group 3 LEA proteins are characterized as a repeating motif of eleven amino acids. In order to analyze the mechanism of group 3 LEA proteins in response to stress conditions deeply,an osmotic-stress induced gene,TaLEAsm,was screened in DNA microarrays for wheat expression profiles. One BAC clone was isolated from Shimai 15 BAC library using primers designed based onTaLEAsmcDNA sequence.TaLEAsmand its promoter sequence were cloned by sequencing walking using BAC clone plasmid as template. Only an 105 bp intron was identified inTaLEAsm. The open reading frame ofTaLEAsmis 675 bp long,encoding 224 amino acids.TaLEAsm belonged to group 3 LEA protein containing ten repeating motifs of eleven amino acids.TaLEAsmwas strongly up-regulated induced by low temperature,high salt and osmotic stress,showing different expression patterns in root and leaf of wheat. Fourteen stress-responsecis-elements were predicted in 1 500 bp sequence upstream ofTaLEAsmstart codon.TaLEAsmpromoter could lead to the expression ofGUSgene,which was induced by osmotic stress inArabidopsis.The results above indicatedTaLEAsmmay be a dehydration-response gene and its promoter was an inducible promoter.
Triticumaestivum;TaLEAsm; Promoter; Cloning; Functional analysis
时间:2017-01-16
2016-09-27
2016-11-18
国家重点研发计划项目(2016YFD0101802);国家公益性行业(农业)科研专项(201203012);河北省科技支撑计划项目(16226320D)
E-mail:lmj199612@aliyun.com
史占良(E-mail:shizhanl@163.com)
S512.1;S330
A
1009-1041(2017)02-0145-08
网络出版地址:http://www.cnki.net/kcms/detail/61.1359.S.20170116.1833.002.html
